假单胞菌SJTD-1全蛋白质组学研究
孙文兵,侯敬丽
(1.上海交通大学生命科学技术学院,上海 200240;2.上海交通大学分析测试中心,上海 200240)
假单胞菌SJTD-1全蛋白质组学研究
孙文兵1,侯敬丽2
(1.上海交通大学生命科学技术学院,上海 200240;2.上海交通大学分析测试中心,上海 200240)
近年来,石油泄漏对海洋和土壤的污染事件频发,解决这一污染问题非常重要。本实验从原油污染的土壤中分离得到一株新的铜绿假单胞菌株SJTD-1,使用SDS-PAGE-1DLC-MS和offline-2DLC-MS两种方法研究其全蛋白质组。利用这两种技术平台分别鉴定获得1 453个和1 623个可信蛋白,共鉴定到1 912个可信蛋白。通过比较两种技术路线所鉴定到蛋白种类的差异,发现offline-2DLC-MS有利于SJTD-1菌株的膜蛋白、相对分子质量较大的蛋白和pI值不大于8的蛋白的鉴定。另外,对所有鉴定到的可信蛋白进行GO功能注释,亚细胞定位分析和代谢通路富集分析。通过对分离获得的SJTD-1菌株进行蛋白质组学分析,可为铜绿假单胞菌SJTD-1分解环烷烃以及SJTD-1在生物修复应用中的研究奠定基础。
铜绿假单胞菌;蛋白质组学;SDS-聚丙烯酰胺凝胶电泳;离线二维色谱
石油的开采、炼制、贮运和使用过程会对海洋和土壤造成极大的污染。一些石油烃类经过食物链进入动物体内后,对哺乳类动物及人类有致癌、致畸、致突变的作用,严重危害人类健康。目前,国际上通用的石油污染治理及回收方法可大致分为物理法、化学法和生物法。生物法依赖微生物对石油的降解作用[1-2],具有迅速、无残毒、成本低等优点,当当前研究的热点[3]。能降解石油烃的微生物很多[4],但对于大多数微生物来说,十六个碳以上的烷烃不易降解[5]。本课题组分离得到一个可以降解长链环烷烃的铜绿假单胞菌SJTD-1,已经测定了SJTD-1菌株的全基因组序列[6],并推测SJTD-1菌株包含5 647个CDS序列,其中95%的CDS都与铜绿假单胞菌PAO1基因组相同[7]。虽然SJTD-1全基因组草图已经发布,但是SJTD-1的蛋白质组还没有被研究。
本实验利用SDS-PAGE-1DLC-MS和offline-2DLC-MS串联质谱技术鉴定SJTD-1菌株的全蛋白质组,并分析两种技术路线鉴定的蛋白种类差异及其优缺点,研究SJTD-1菌株的全蛋白质组图谱,以期为铜绿假单胞菌SJTD-1分解环烷烃以及SJTD-1在生物修复中的研究奠定基础。
1 实验部分
1.1 主要仪器和数据分析软件
Dionex ultimate 3000纳升液相色谱仪,Thermo Surveyor高效液相色谱仪:美国Themo-Fisher公司产品;Maxis impact UHR四极杆飞行时间串联质谱仪:德国Bruker公司产品,配有纳升电喷雾离子源;Mascot软件:英国Matrix Science公司产品;数据质控软件Scaffold_4.0.5:美国Proteome Software公司产品。
1.2 主要材料与试剂
实验菌株铜绿假单胞菌SJTD-1:由本实验室分离得到,在环十八烷烃中生长良好;二硫苏糖醇(DTT)、碘乙酰胺(IAM)、碳酸氢铵、甲酸铵等:美国Sigma公司产品;测序用胰酶:美国Promega公司产品;10 K超滤管:德国Sartorius公司产品;乙腈、乙醇、异丙醇(色谱纯):德国Merk公司产品;其他常用试剂:上海生工生物工程(上海)股份有限公司产品。
1.3 实验方法
1.3.1 细菌培养 将铜绿假单胞菌SJTD-1菌株接种于20 mL LB液体培养基中,于37 ℃培养,转速180 r/min,生长至OD600约1.2后离心收集。
1.3.2 全蛋白的提取 离心收集细胞,加入3 mL细胞裂解液(40 mmol/L Tris-HCl pH 7.4,8 mol/L尿素,4% CHAPS,65 mmol/L DTT,1 mmol/L PMSF,1 mmol/L EDTA,0.5 mmol/L EGTA),混合振荡1 min,悬浮于冰面上,超声破碎10 min(超声功率300 W,超声3 s,停3 s)。将细胞裂解液于4 ℃,以20 000 g/min离心15 min去除细胞碎片。提取的上清液用5倍体积混合液(乙醇∶丙酮∶乙酸=50∶50∶0.1,V/V/V)过夜沉淀,以25 000 g/min于4 ℃离心1 h,收集沉淀。沉淀干燥后用还原溶液(6 mol/L盐酸胍,50 mmol/L碳酸氢铵)重溶,Bradford法测定蛋白浓度,剩余样品于-20 ℃保存,备用。
1.3.3 胶内酶解 取60~80 μg蛋白做10% SDS-PAGE电泳,考马斯亮蓝进行胶染色,脱色后,用干净刀片将胶条等分切割成10份,向每份中加入DTT至终浓度为20 mmol/L,60 ℃水浴还原1 h;再加入IAM至终浓度为25 mmol/L,避光反应30 min后,依次用50 mmol/L碳酸氢铵,100%乙腈洗涤,最后加入10 mg/L胰酶过夜酶解;次日用100%乙腈萃取;沉淀干燥后,用上样缓冲液(水∶乙腈∶甲酸=97.9∶2∶0.1,V/V/V)溶解,待测。
1.3.4 FASP酶解[8]取一定量蛋白加入DTT至终浓度为20 mmol/L,于56 ℃ 温育1 h还原蛋白;再加入IAM至终浓度为90 mmol/L,进行烷基化反应,将蛋白溶液转入10 K超滤管中,离心弃掉收集管底部溶液;加入50 mmol/L碳酸氢铵离心20 min,弃掉收集管底部溶液,重复操作3次;按质量比1∶50加入胰蛋白酶,过夜酶解;次日,离心收集酶解消化后的肽段溶液,并干燥保存。
1.3.5 高效液相色谱仪(HPLC)分级收集 酶解后的多肽混合物利用HPLC对多肽进行分级收集。多肽分离色谱柱为Durashell-C18分析柱(4.6 mm×250 mm×5 μm)。流动相A为20 mmol/L甲酸铵(pH 10),流动相B为20 mmol/L甲酸铵和80%乙腈混合液(pH 10)。酶解后的多肽混合物用30 μL 20 mmol/L甲酸铵(pH 10)溶解。HPLC分级收集条件:流速0.8 mL/min;分离梯度0~5 min、5%B,16~30 min、5%~15% B,31~45 min、15%~38%B,46~54.5 min、90%B,55~65 min、5%B。从第10 min开始,依次收集洗脱液,干燥后根据HPLC紫外监测情况,将得到的组分用上样缓冲液(水∶乙腈∶甲酸=97.9∶2∶0.1,V/V/V)溶解,并合并成10个组分。
1.4 实验条件
1.4.1 色谱条件 液相分离分析柱为C18反相分析柱(75 μm×15 cm×3 μm);多肽富集柱为C18反相柱(100 μm×2 cm×5 μm);流动相A为甲酸水溶液(0.1∶99.9,V/V),流动相B为甲酸-乙腈溶液(0.1∶99.9,V/V);流速为400 nL/min;多肽洗脱梯度:0~87 min、2~30%B,88~98 min、30%~50%B,99~109 min、50~80%B,110~120 min、80%B。
1.4.2 质谱条件 ESI+模式,质谱电喷雾毛细管电压1 900 V,气体流速2.0 L/min,离子源温度120 ℃,选择母离子m/z350~1 500进行二级碎裂。
1.5 数据分析
1.5.1 Mascot搜库 使用Data Analysis 4.1软件提取离子峰生成mgf文件。Mascot 4.0搜索引擎搜索Nr_Pseudomonas aeruginosa PA01_201312蛋白数据库(37 636条目);母离子质量容差2×10-5,二级谱图质量容差0.050 u,固定修饰为C+57,可变修饰为M+16,胰酶最大漏切位点数为1。利用数据质控软件Scaffold_4.0.5做搜库文件数据分析,多肽置信度设置为1%FDR,一个蛋白至少检出2个唯一肽段。
1.5.2 生物信息学分析 利用生物信息学网站(http:∥www.pantherdb.org/)对搜索到的蛋白进行GO分子功能注释分析;利用生物信息学网站(http:∥db.psort.org/)对蛋白进行亚细胞定位分析;利用生物信息学网站(http:∥david.abcc.ncifcrf.gov/home.jsp)对蛋白进行代谢通路的富集;另外利用在线蛋白分析网站(http:∥www.expasy.com)对两组蛋白质等电点和相对分子质量进行分析。
2 结果与讨论
2.1 SJTD-1的生长曲线
每2 h测定一次OD600,其生长曲线示于图1。结果显示,接种8 h后,细菌生长进入对数期,并且能够延续30 h。因此,选择培养15 h后的细胞进行收集能满足实验需求。

图1 SJTD-1在LB培养基中的生长曲线Fig.1 Growth curve of SJTD-1 cultured in LB medium
2.2 两种技术路线分析SJTD-1蛋白质组
两种技术路线示意图示于图2。
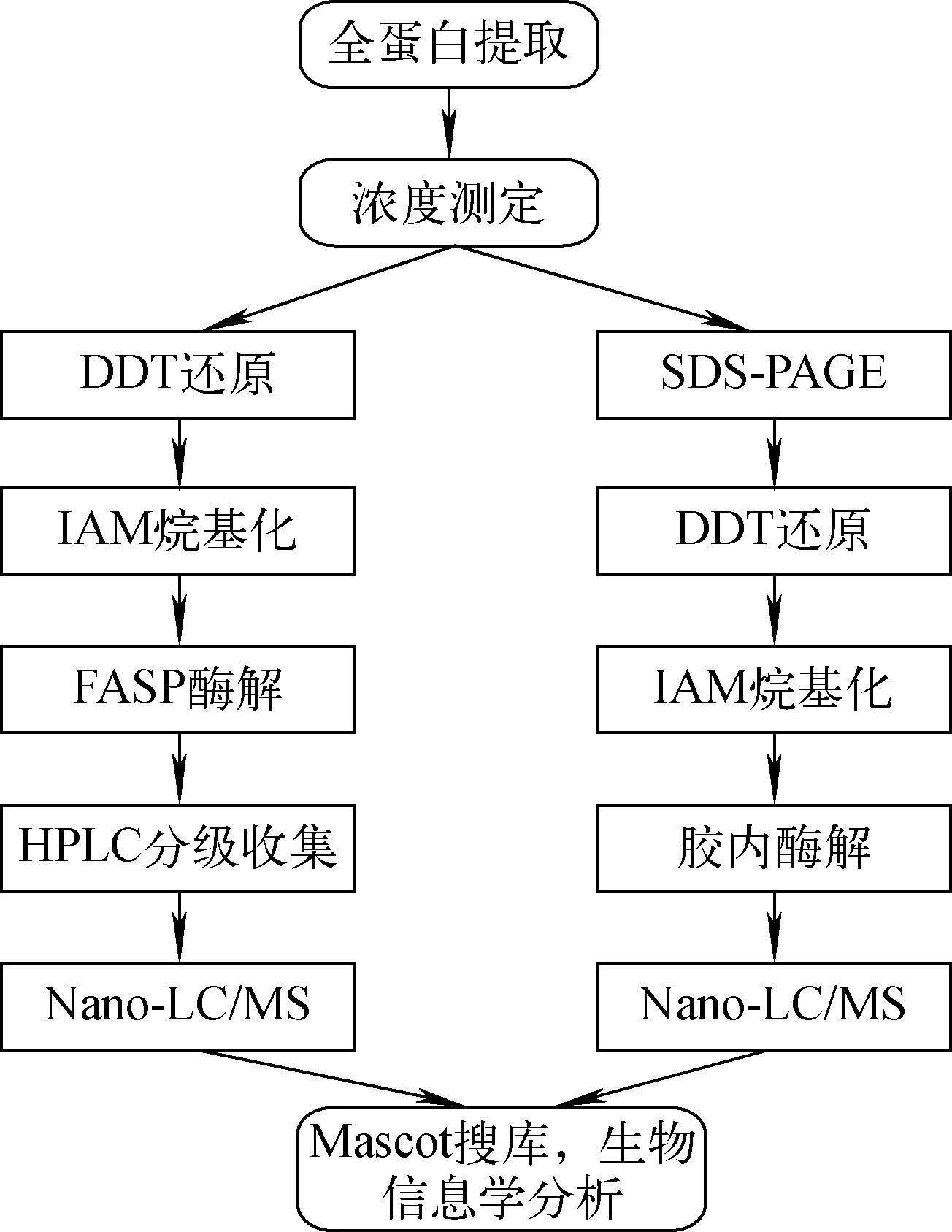
图2 两种方法技术路线示意图Fig.2 Schematic diagram of the two different techniques
利用SDS-PAGE-1DLC-MS和offline-2DLC-MS技术方法分别对SJTD-1菌株进行了蛋白质组分析,并进行了2次生物学重复实验,分别鉴定到1 453个和1 623个可信蛋白,共鉴定到1 912个可信蛋白,示于图3。

图3 SDS-PAGE-1DLC-MS和offline-2DLC-MS方法鉴定蛋白数目差异Fig.3 The difference between SDS-PAGE-1DLC-MS and offline-2DLC-MS for proteome identification
在PSORTb3.00数据库中分析显示,SJTD-1质膜蛋白和外膜蛋白分别占总蛋白的22.9%和3.1%。对SDS-PAGE-1DLC-MS和offline-2DLC-MS技术方法鉴定到的蛋白进行膜定位分析,比较结果示于图4。由图4可见,SDS-PAGE-1DLC-MS方法鉴定到的膜蛋白数目明显少于offline-2DLC-MS方法。与高丰度的可溶性蛋白相比,膜蛋白的相对分子质量大,丰度较低,而且在二维凝胶电泳过程中容易聚集,整合膜蛋白很难酶解[9]。因此,近年来研究者广泛采用SDS-PAGE-1DLC-MS和多维色谱蛋白质组学技术共同研究膜蛋白[10-11]。offline-2DLC-MS技术采用FASP 方法酶解提取全蛋白,FASP方法能够有效地酶解膜蛋白,从而提高膜蛋白的鉴定效率[12]。前人研究表明,近26%的人类基因编码为膜蛋白[13],因此在人类膜蛋白的鉴定研究中,offline-2DLC-MS技术可提供借鉴。
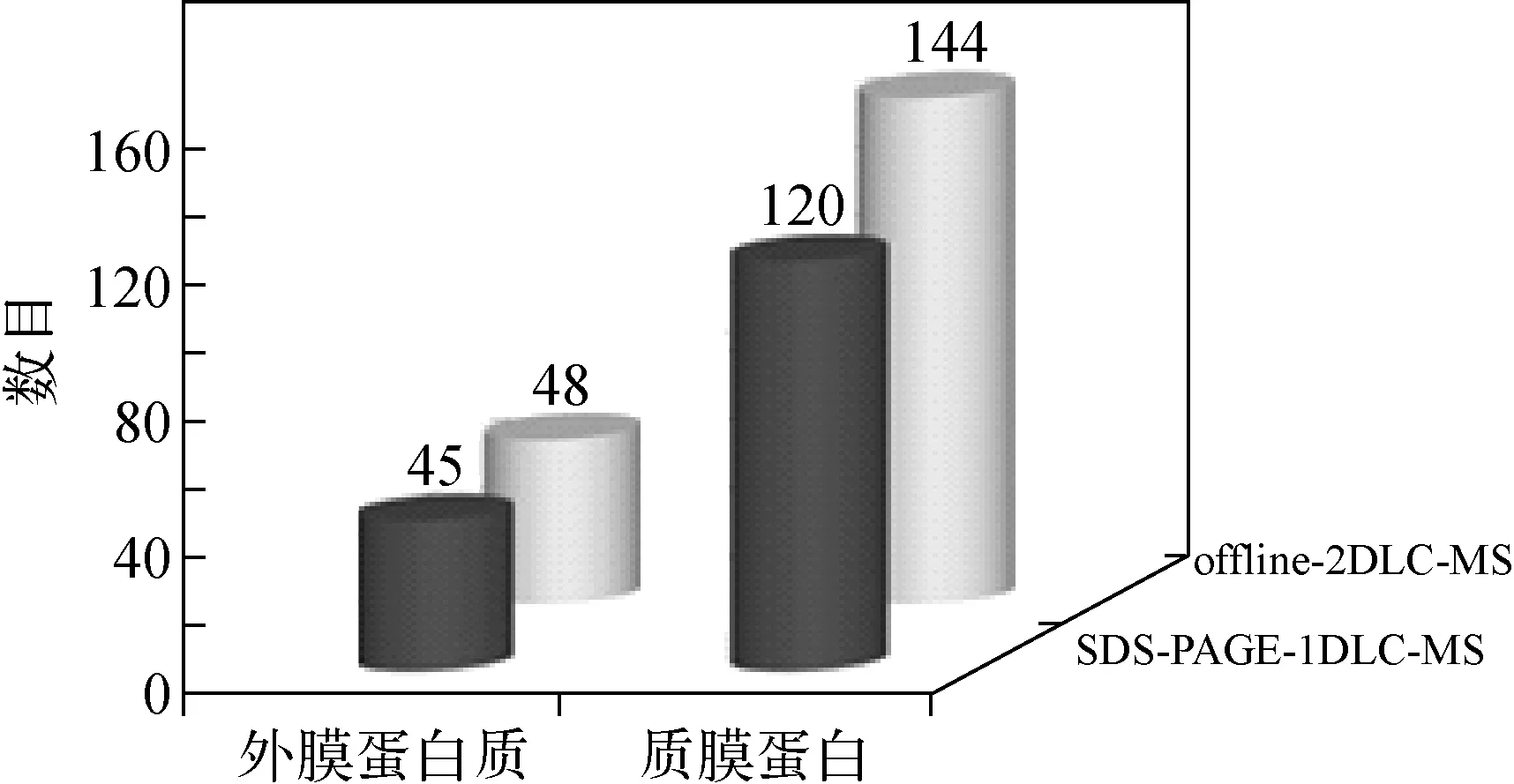
图4 两种技术方法对SJTD-1膜蛋白鉴定数目比较Fig.4 Comparison of membrane proteins of SJTD-1 identified by two techniques

图5 SJTD-1总蛋白相对分子质量分布Fig.5 Relative molecular weight distribution of identified proteins of SJTD-1
对鉴定蛋白的相对分子质量分布进行分析,结果示于图5。由图5可见,offline-2DLC-MS方法鉴定到的大于30 ku的蛋白数量明显高于SDS-PAGE-1DLC-MS方法,可见offline-2DLC-MS方法在检测相对分子质量高于30 ku的蛋白中有明显优势,更有利于相对分子质量较大蛋白的检测。

图6 SJTD-1总蛋白等电点分布Fig.6 Distribution of the identified proteins of SJTD-1 depending on pI value proteomeproteome proteome proteome
将分离得到的蛋白进行等电点pI值分析,结果示于图6。从图6可以看出:大部分蛋白等电点处于5~6;而offline-2DLC-MS鉴定到的pI≤8蛋白数目高于SDS-PAGE-1DLC-MS方法;鉴定到的pI≥9蛋白数目低于SDS-PAGE-1DLC-MS方法。可见offline-2DLC-MS方法能够实现pI≤8蛋白的高效鉴定,这与前人的研究结果一致[14-15]。这可能是由于在不同pH值环境下,多肽的反相保留行为不同,用一维高pH值反相色谱,二维低pH值反相色谱组成的分离多肽混合物,能显著提高2DLC分离效率,改善峰形,提高峰容量[16-17]。虽然一维高pH值反相色谱和二维低pH值反相色谱组成的2DLC能够在整体上大幅地提高多肽的鉴定效率,但是offline-2DLC-MS对pI≥9蛋白的鉴定能力不如SDS-PAGE-1DLC-MS,可能是由于随着多肽碱性提高,色谱柱效下降严重,无法达到中性化合物所需的柱效[18]。另外,铜绿假单胞菌蛋白质组本身在等电点分布方面与其他物种的蛋白质组分布相似[14,19-20]。因此,2DLC-MS对不同等电点蛋白的鉴定效率不同,这与铜绿假单胞菌蛋白质组本身的组成无关,而与2DLC分离能力有关。
综上所述,对于相对分子质量较大和pI≤8的细菌蛋白质鉴定,offline-2DLC-MS无疑是更好的鉴定方法,在后续的研究中,可以通过优化offline-2DLC-MS方法的一维洗脱条件,或者提高SDS-PAGE-1DLC-MS方法中切胶组分的数目来提高检测灵敏度和分辨率,进而鉴定到更多数目的可信蛋白[21-22]。
2.3 数据分析
利用SDS-PAGE-1DLC-MS和offline-2DLC-MS技术方法对共同鉴定到的蛋白进行GO蛋白生物学过程、GO蛋白生物学功能和蛋白亚细胞定位、代谢通路富集等分析。
通过GO注释对蛋白功能进行聚类分析,结果示于图7a。鉴定的蛋白中47%主要集中在代谢过程,这与铜绿假单胞菌是一种代谢过程多样的菌株有关。分子功能主要集中在起催化功能的相关蛋白,示于图7b;蛋白亚细胞定位示于图7c,蛋白主要分布在细胞质和细胞质膜上。利用David 网站相关软件进行KEGG代谢通路富集[23],结果列于表1。表1中排名前10的代谢通路均为合成与分解代谢,其中包括核酸、脂肪酸代谢以及一些氨基酸代谢,这些通路与烷烃降解转化为脂肪酸,最后经过脂肪酸代谢与TCA循环过程相关。后续的实验将以烷烃为唯一碳源培养SJTD-1菌株,研究以上相关代谢途径是否加强,以及相关蛋白表达是否提高,以找到烷烃分解代谢的相关蛋白,目前这部分工作正在进行。

注:a.蛋白生物学过程;b.蛋白生物学功能;c.蛋白亚细胞定位图7 GO功能分析总蛋白Fig.7 Protiens were classified by GO annotations
3 结论
利用SDS-PAGE-1DLC-MS和offline-2DLC-MS两种不同的技术路线,鉴定了铜绿假单胞菌SJTD-1的全蛋白质。通过比较两种方法,分析在细菌蛋白鉴定上的优劣,以提高蛋白质的鉴定水平;另外,本实验结果可为生物蛋白质组的鉴定提供借鉴,尤其是在以后铜绿假单胞菌膜蛋白和特定相对分子质量蛋白鉴定时,可选择不同的技术路线来满足不同的实验需求。为进一步分析铜绿假单胞SJTD-1与石油分解相关的关键代谢通路和相关蛋白提供了重要信息。

表1 全蛋白的KEGG代谢通路富集
注:富集分值≥1.3表示有意义;P值:Fisher's exact test值,P值≤0.1表示有意义; 数目:在该条目下聚类到的蛋白数
[1] JONES D M, HEAD I M, GRAYN D, et al. Crude-oil biodegradation via methanogenesis in subsurface petroleum reservoirs[J]. Nature,2008, 451(10):176-180.
[2] ATLASR M.Petroleum biodegradation and oil spill bioremediation[J].Marine Pollution Bulletin,1995,31 (4):178-182.
[3] KUYUKINA M S, IVSHINA I B,RITCHKOVA M I, et al.Bioremediation of crude oil-contaminated soil using slurry-phase biological treatment and land farming techniques[J]. Soil and Sediment Contamination, 2003,12(1): 85-99.
[4] RHAMAN K, THAHIRA-RAHMAN J, LAKSH-MANAPERUMALSAMY P, et al.Towards efficient crude oil degradation by a mixed bacterial consortium[J]. Bioresource Technology, 2002, 85(3): 257-261.
[5] ROJOF. Degradation of alkanes by bacteria[J]. Environ Microbiol, 2009, 11(10): 2 477-2 490.
[6] LIU H, LIANG R, TAO F, et al.Genome sequence of Pseudomonas aeruginosa strain SJTD-1, a bacterium capable of degrading long-chain alkanes and crude oil[J].Journal of Bacteriology, 2012, 194(17): 4 783-4 784.
[7] STOVER C, PHAM X, ERWIN A, et al.Complete genome sequence of Pseudomonas aeruginosa PAO1, an opportunistic pathogen[J]. Nature, 2000, 406(6 799): 959-964.
[8] WISNIEWSKI J R, ZOUGMAN A, NAGARAJ N,et al.Universal sample preparation method for proteome analysis[J]. Nat Methods, 2009, 6(5): 359-362.
[9] WASHBURN M P, WOLTERS D, YATES J R. Large-scale analysis of the yeast proteome by multidimensional protein identification technology[J]. Nat Biotechnol, 2001, 19(3): 242-247.
[10]PARK Y M, KIM J Y, KWON K H, et a1. Profiling human brain proteome by multi-dimensional separations coupled with MS[J]. Proteomics, 2006, 6(18): 4 978-4 986.
[11]MONNEUSE J M, SUGANO M, BECU T, et al. Towards the profiling of the Arabidopsis thaliana plasma membrane transportome by targeted proteomics[J]. Proteomics, 2011, 11(9): 1 789-1 797.
[12]WIS′NIEWSKI JR, ZOUGMAN A, NAGARAJ N, et al. Universal sample preparation method for proteome analysis[J]. Nat Methods, 2009, 6(5): 359-62.
[13]FAGERBERG L, JONASSONONASSON K, HEIJNEEIJNE G VON, et al. Prediction of the human membrane proteome[J]. Proteomics, 2010, 10(6): 1 141-1 149.
[14]DELMOTTE N, LASAOSA M, THOLEY A, et al. Two-dimensional reversed-phase x ion-pair reversed-phase HPLC: An alternative approach to high-resolution peptide separation for shotgun proteome analysis[J]. J Proteome Res, 2007, 6(11): 4 363-4 373.
[15]FERREIRA R, ROCHA H, ALMEIDA V. Mitochondria proteome profiling: a comparative analysis between gel- and gel-free approaches[J]. Talanta, 2013,15: 277-83
[16]ZHOU F, CARDOZA J D, FICARRO S B, et al. Online nanoflow RP-RP-MS reveals dynamics of multicomponent Ku complex in response to DNA damage[J]. J Proteome Res, 2010, 9(12): 6 242-6 255.
[17]GILAR M, OLIVOVA P, DALY A E, et al.Two-dimensionalseparation of peptides using RP-HPLC system with different pH in first and second separation dimensions[J]. J Sep Sci, 2005, 28(14):1 694-1 703.
[18]TRÜDINGER U, MÜLLER G, UNGER K K. Porous zirconia and titania as packing materials for high-performance liquid chromatography[J]. Unger Journal of Chromatography,1990, 535(28):111-125.
[19]LECOUTERE E, VERLEYEN P, HAENEN S, et al. A theoretical and experimental proteome map of Pseudomonas aeruginosa PAO1[J]. Microbiologyopen, 2012, 1(2): 169-181.
[20]SCHUBERT M, PETERSSON UA, HAAS B J, et al. Proteome map of the chloroplast lumen of Arabidopsis thaliana[J]. J Biol Chem, 2002, 277(10): 8 354-8 365.
[21]VOLLMER M, HORTHRTH P, NAGELE E. Optimization of two-dimensional off-line LC/MS separations to improve resolution of complex proteomic samples[J]. Analytical Chemistry, 2004, 76(17): 5 180-5 185.
[22]WANG N, XIE C, YOUNG J B, et al. Off-line two-dimensional liquid chromatography with maximized sample loading to reversed-phase liquid chromatography-electrospray ionization tandem mass spectrometry for shotgun proteome analysis[J]. Analytical Chemistry, 2009, 81(3): 1 049-1 060.
[23]HUANG D A W, SHERMAN B T, LEMPICKI R A. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources[J]. Nat Protoc, 2008, 4(1): 44-57.
Proteome Analysis of Pseudomonas Aeruginosa SJTD-1
SUN Wen-bing1, HOU Jing-li2
(1.CollegeofLifeandBiotechnologyofShanghaiJiaotongUniversity,Shanghai200240,China;2.InstrumentalAnalysisCenterofShanghaiJiaotongUniversity,Shanghai200240,China)
In recent years, oil spill and its impact on marine environment occurs frequently. To solve this problem, study of oil degrading was put on the agenda. A kind of pseudomonas aeruginosa named SJTD-1 was isolated from soil contaminated by crude oil. SDS-PAGE-1DLC-MS and offline-2DLC-MS methods were used to analysis proteome of SJTD-1 respectively. 1 453 proteins with SDS-PAGE-1DLC-MS and 1 623 protenis with offline-2DLC-MS were identified, respectively, and the total identified proteins were 1 912 by the two methods. Comparing the two different techniques, offline-2DLC-MS method is more benefit to identify membrane proteins, proteins with molecular weight larger than 30 ku and proteins with pI value less than or equal with eight. GO functional annotation, cellular component, functional enrichment and clustering were used to analysis the total proteins. The proteome analysis of pseudomonas aeruginosa SJTD-1 would promote the application on marine environment bioremediation by SJTD-1.
pseudomonas aeruginosa;proteome; SDS-PAGE; offline-2DLC
2014-05-09;
2014-07-15
酵母RNaseH2磷酸化调控机制及其生理功能的研究项目(31200062)资助
孙文兵(1988—),男(汉族),安徽人,硕士研究生,生物化学与分子生物学专业。E-mail: swb1008@sjtu.edu.cn
侯敬丽(1977—),女(汉族),山东人,副研究员,从事生物质谱与蛋白质组学研究。E-mail: hopie00@sjtu.edu.cn
时间:2014-12-02;
http:∥www.cnki.net/kcms/doi/10.7538/zpxb.youxian.2014.0069.html
O657.63
A
1004-2997(2015)03-0193-06
10.7538/zpxb.youxian.2014.0069

