蔬菜核心种质研究进展
陈学军,雷 刚,2,周坤华,方 荣
(1.江西省农业科学院 蔬菜花卉研究所,江西 南昌 330200;2.江西农业大学 农学院,江西 南昌 330045)
种质资源是作物遗传改良的基础。目前,全世界已有各类植物遗传资源610多万份(含重复),我国共收集保存各类作物种质资源38万份[1],其中,中国国家蔬菜种质资源中期库收集保存的有性繁殖蔬菜种质资源132种(变种)33 280份[2]。如此巨大数量的资源给种质资源的评价、研究和利用带来了诸多困难。针对这一问题,Frankel等[3]提出了基于遗传多样性构建核心种质的解决方案,之后,核心种质的研究逐步成为国际遗传资源研究的热点。迄今国内外已构建水稻[4-5]、小麦[6-7]、大麦[8]、大豆[9]、玉米[10]、油菜[11]等大田作物和苹果[12-13]、桃[14]、柚[15]、山葡萄[16]、橄榄[17]等果树以及大白菜[18]、马铃薯[19]、胡萝卜[20]、蚕豆[21]等蔬菜作物共80余种农作物的初级核心种质或核心种质,为农作物种质资源深入研究与有效利用奠定了基础。本文概述了国内外蔬菜核心种质研究进展,并对相关研究存在的问题和发展方向进行了探讨,以期为蔬菜核心种质的深入研究与有效利用提供理论参考。
1 核心种质的概念与构建方法
1.1 核心种质的概念
Frankel[3]提出核心种质(core collection)概念,即用科学的方法从某种植物所有种质资源中筛选出能够最大程度代表其遗传多样性、且数量尽可能少的种质材料作为核心材料,以便于保存,进一步的评价和利用,剩余的种质称为保留种质(reserve collection)。Brown[22]对概念进一步补充完善。Hodgkin[23]提出了构建核心种质的基本步骤。此后,Casler[24]、Basigalup 等[25]及 Spagnoletti等[26],对核心种质的基本特征进行了总结,并对多种构建方法进行了比较。随着研究不断的深入,核心种质的概念也进一步发展,出现了合成核心种质、分级核心种质和微核心种质等概念[27]。
1.2 核心种质的构建方法
1.2.1 数据收集 核心种质构建数据包括基本数据、特征数据和评价鉴定数据。基本数据即地理分布,起源及分类等,基本数据主要用于初步分组。在构建我国花生核心种质时,姜慧芳等[28]先按照花生植物学类型把材料分成5层,即多粒型、珍珠豆型、龙生型、普通型和中间型。特征数据包括形态、同工酶、蛋白及分子标记等数据,是构建核心种质的主要数据。其中,形态数据较易获得而使用最多,大多数蔬菜核心种质都是基于形态数据构建的。同工酶、分子标记数据更加准确,更加能反映出资源的遗传多样性,但数据获取相对较难。黎毛毛等[29]采用SSR标记法对3 187份江西地方稻种资源材料进行多样性和聚类分析,建立了占资源总量9.28%、包含296份材料的江西省地方稻种资源核心种质库。评价鉴定数据主要是一些农艺性状,如抗性、产量和品质等,用于检验核心样品的有效性。
目前,蔬菜作物基本数据尚不完整,在今后的工作中尽快完善所收集资源的地理分布、数量、分类及育种体系等信息,同时应对形态数据的评价、记录标准予以统一,确保研究结果的可比较性。分子数据能够较好的反应材料的遗传信息和群体的遗传关系且可以在短时间获取,因此,在研究过程中应加大分子数据的应用。各类数据在构建核心种质时,特征数据和鉴定评价数据没有明显的用途归类,且各类型数据综合利用可以相互补充,使构建的核心种质更具代表性。
1.2.2 数据分组 数据的分组有随机取样分组和分层取样分组,前者由于获得的材料代表性较差[24],目前多采用后者。分层分组就是依据材料遗传相似程度把要进行研究的实验材料归类,各组之间互不重叠,比如按来源地、栽培区、生态型、品种类群等分组。相同的来源地、栽培区等的材料由于长期的自然及人为选择,遗传结构较相近。Erskine等[30]对比分析了来自3个国家的扁豆种质材料的形态及同工酶数据后,认为同一来源地的种质平均等位基因、多态性比例及多样性指数无显著差异。因此,分层分组是一种简单可靠的分组方法。姜慧芳等[28]在构建我国栽培花生核心种质时,先按植物学类型把材料分为5层,然后在每层内按地理来源分组,国内按生态区分组,包括西北东北花生区、华北淮河花生区、长江流域花生区、南方花生区,国外材料按北美和南美洲、亚洲、欧洲、ICRISAT、非洲和大洋洲,把6 390份资源分成32组。对原始材料进行合理的分组,可以提高取样的效率和所构建核心种质的代表性。利用多种数据对材料进行更精细的分组,如董玉琛等[31]在构建普通小麦核心种质时,依据小麦和其他物种、地方品种和选育品种及不同的生态栽培区将材料逐层分组。
1.2.3 取样策略 取样策略即采用一定的方法从原始群体中选择抽取核心种质,是核心种质构建关键步骤之一,方法分为随机取样和系统取样。随机取样对所有样品以相同的概率抽取,但是由于种质资源生长环境的不同、遗传多样性分布不均匀,不同的等位基因在全部遗传资源中的重复次数也不尽相同,因此,随机取样法使用较少。在构建中国绿豆时,刘长友等[31]采用了聚类选择取样和随机取样构建了13个初选核心样本,通过性状符合度、遗传多样性指数、数量性状变异系数及品种间平均相似性系数的比较发现,聚类选择取样优于随机取样。系统取样又包括恒量法、对数法、平方根法、遗传多样性法、比例法。邱丽娟等[9]采用了20种取样方法构建中国大豆初级核心种质,对比分析发现,比例法和平方根法在确定组内取样量时优于遗传多样性法。Tohme等[32]构建菜豆核心种质时采用了比例法,先根据地理来源分组,再在组内以材料量占整个资源材料量的比例确定取样比例。在构建安第斯栽培种马铃薯的核心种质时,Huaman等[19]采用了平方根法,根据地理来源分组,在组内依据各组内材料数目平方根确定组内取样比例。恒量法需要各组材料数量及遗传多样性结构接近时才能得到可靠的结果,使用较少。其余的几种方法各有优缺点,在具体应用过程中,要依据材料的数量、分组情况及遗传多样性等信息来选择合适的方法,也可通过对比选取最优方法。
1.2.4 取样规模 Brown[34]认为核心种质一般占整个遗传资源的5%~10%,或总量不超过3 000份,但在后续诸多研究中发现5%~10%并不是既定的,Yonazawa等[35]认为对于遗传冗余度Dr值在0.2~0.9的群体,其最佳取样比例为20%~30%。目前报道的核心种质取样比例多在5%~40%[36],但所有研究都没有提供一个统一的总体取样比例,这主要是不同植物资源在收集程度、遗传多样性状况和内部遗传结构上存在较大差异,对整体取样比例的确定不能格式化、简单化,应视研究作物遗传结构及遗传多样性的不同而定。例如,在构建中国栽培大豆核心种质时,王丽侠等[37]根据不同取样策略和不同总体取样量水平的核心种质遗传多样性指数比较发现,3%的取样水平不仅能够保证对初选核心种质的代表性,且核心种质具有较大的遗传多样性。对于材料较少的资源,可以适当的增加取样规模,而对材料数目较大的材料,应依据所选核心材料的代表性和冗余度确定合适的取样规模。
1.2.5 核心种质的检验 核心种质的检验是运用一些指标对所筛选核心样品的代表性、所用构建方法的合理性及其在生产中的实用性[35]进行评价验证的过程,包括遗传多样性的符合性检验和实用性检验两方面。遗传多样性的符合性检验依据不同类型数据分别计算原始群体和筛选出的核心材料的各个指标来判断其代表性。Diwan等[38]认为,若核心种质和整个样品在平均数及变异幅度上存在显著差异的性状少于30%,且核心种质各性状变幅占整个样品变幅平均比率高于70%,就可以认为该核心种质基本代表了原始群体的遗传多样性。对核心种质遗传多样性的符合性检验分为离散型和连续型指标多样性:离散型指评价数据如同工酶、分子标记等离散型数据,常用Nei’s多样性指数(H)、Shannon and Weaver’s信息指数(I)、Brillouln’s指数等评价指标;连续型数据如农艺性状,生理生化等,多用方差、标准差、极差、均值、变异幅度等指标。例如,杨美等[39]对所构建的88份花莲核心种质评价时,应用多态性位点百分率保留率、等位基因数、有效等位基因数、Nei’s遗传多样性指数及Shannon’s信息指数等指标对核心种质的代表性进行了评价。核心种质的实用性检验指检验所构建的核心种质是否包含已知的农艺性状,该方法目前研究利用较少,评价指标还有待发展和补充,一般作为补充检验手段。
2 蔬菜核心种质构建与利用研究现状
2.1 蔬菜核心种质的构建
2.1.1 茄果类蔬菜 Zewdie等[40]基于21个形态性状和分层分组代表性取样法,以美国国家种质库1 202份辣椒种质为试料,以约10%总体取样规模建立了包含137份材料的辣椒核心种质。Mao等[41]以1 968份栽培茄子为试材,在构建核心种质过程中,采用了两种遗传距离(马氏和欧式距离)、4种聚类方法(UGPA、CL、SL及W)、3种取样策略(R、P、D策略)以及4种取样规模(10%、15%、20%及30%),通过比较,最终确定了欧式距离、UGPA、R或P策略、15%~20%取样规模为构建茄子核心种质合适的方法。Corrado等[42]利用152个SNP标记对100份番茄材料进行了检测,并探讨了番茄核心种质构建的策略和核心种质有效性检验方法,认为15%~25%的取样比例可以保证原始群体的遗传多样性(表1)。
2.1.2 豆类蔬菜 刘玉皎等[21]采用AFLP技术对来自青海和国外的149份蚕豆种质材料进行了遗传分析,建立了进化树,以进化树为基础,对亲缘关系较近的资源按比例取舍,初步构建了40份核心种质,经检验其等位变异数、有效等位变异数、多态性条带数、遗传相似系数、遗传距离及基因多样性指数等,发现遗传信息的符合率均在80%以上。Zong等[43]利用SSR分子标记数据对我国1 243份豌豆材料进行聚类分析,构建了包含146份豌豆资源的核心种质,约占取样群体的12%。Reddy等[44]在构建树豆(木豆)核心种质时,以收集的12 153份树豆为试材,基于14个形态数据,按地理起源把收集的12 153份树豆材料分成14组,组内采取随机取样,以10%的取样规模构建了1 290份树豆核心种质。
2.1.3 叶菜类蔬菜 李国强等[18]在构建大白菜核心种质时,比较了4种组内取样比例法、6种取样规模和2种取样方法后,以国家蔬菜种质资源中期库保存的1 651份大白菜为材料,基于43个形态数据,按大白菜分类系统把材料分成6组,组内按多样性比例聚类取样法,以15%为取样规模构建了248份大白菜核心种质。Jansen等[45]利用AFLP分子标记数据,以1 283份莴苣种质材料为研究对象,通过遗传距离抽样法,构建了128份材料的莴苣核心种质,占全部材料的9.98%。张明等[46]先利用15个农艺性状数据,通过非加权配对算术平均聚类法(UPGMA)对174份韭菜种质材料进行聚类,然后采用简单比例和人工选择结合的取样方法,以40%的取样比例构建了69份韭菜核心种质。
2.1.4 瓜类蔬菜 吕婧[47]应用23对SSR引物对3 318份黄瓜初级种质基因组进行DNA分析,采用M策略,通过逐渐增大的取样规模资源聚类得到预选核心种质,最后确定以3%为取样规模,构建了黄瓜核心种质。
2.1.5 根菜类蔬菜 李建春[48]在构建萝卜核心种质时,先依据11个性状对1 281份萝卜种质材料聚类分析,所有材料被分为8个类群,在每一类群中优先找出各性状最大值和最小值的样品作为核心材料预先保留。其余的材料按不同的抽样比例用多次聚类法抽取,比较后确定15%为最佳比例,以此构建了包含192份材料的萝卜核心种质。庄飞云等[20]以我国340份地方胡萝卜品种为试材,通过对13个农艺性状的评价进行聚类分组,设定适宜的阈值,以约17%的比例构建了胡萝卜初级核心种质。Elsayed等[49]通过25个形态学和农艺性状数据,以30%的取样比例,从210份秘鲁胡萝卜种质材料中选取了63份,构建了秘鲁胡萝卜核心种质。
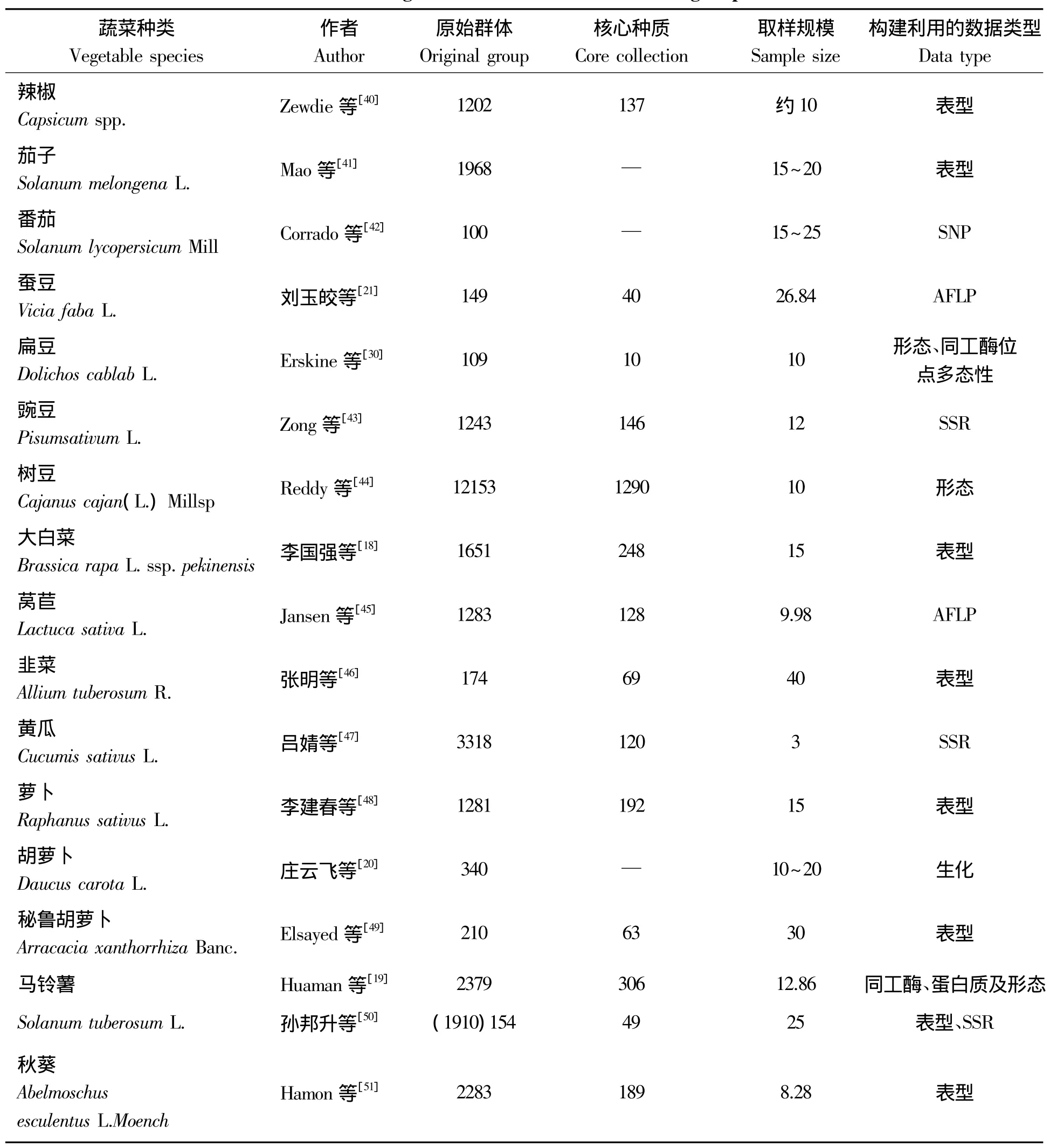
表1 部分蔬菜核心种质研究情况Tab.1 Some vegetables research situation of core germplasm
2.1.6 薯蓣类蔬菜 Huaman等[19]在构建安第斯栽培马铃薯核心种质时,通过比较形态及总蛋白量和酯酶电泳带型的相似性将10 722份材料压缩到2 379份,在利用25个形态数据、地理及评价数据对2 397份材料聚类,并以抗虫、抗病性及干物质含量对2 397份材料进行评价从中筛选出306份材料作为核心种质。孙邦升[50]在构建高淀粉马铃薯核心种质时,先以淀粉含量高于17%为标准,从1 910份马铃薯种质资源中筛选出154份高淀粉马铃薯种质,然后再以淀粉含量1%的差距,将高淀粉马铃薯种质资源分为4组,最后以25%的取样比例构建了包含40份种质的高淀粉马铃薯核心种质。其他蔬菜核心种质的构建情况详见表1。
2.2 蔬菜核心种质的初步利用
核心种质以最少的种质数囊括了原始资源群体的所有或大部分的变异,较好的解决了资源数量巨大和保存、管理、评价、鉴定及利用之间的矛盾,为资源的更深层次的研究提供了广阔的平台。柴敏等[52]以番茄属近缘野生种(L.pennellii)的16份核心种质为试材,开展了对红蜘蛛和蚜虫抗性的鉴定与评估。结果表明,16份核心种质中除1份高感红蜘蛛外,其余15份均对红蜘蛛表现出明显的抗性。Ellis等[53]在对芸苔属甘蓝的401份核心种质进行抗蚜性评价后,获得了中抗材料12份,部分抗性的材料43份。
3 蔬菜核心种质研究存在问题与发展方向
3.1 存在问题
虽然蔬菜核心种质构建研究发展迅速,并取得了一定的成果,但相对其它作物来说,蔬菜核心种质的构建研究总体滞后。我国蔬菜种质资源中期库保存的蔬菜达132种(含变种),目前国内外已构建核心种质(包括初级核心种质)的蔬菜种类还不超过20种,尚未达到蔬菜种类的1/6;且已构建的蔬菜核心种质大多是基于单一的表型数据或分子数据,仅马铃薯、扁豆等少数蔬菜作物是结合了多种类型的数据信息构建的(表1)。此外,大多数蔬菜种质资源包含的材料类型较为复杂,既有原始种、变种,也有品种、品系及性状较优的株系等,因此,在构建过程中应该区别对待,分组时应考虑按遗传关系和育种体系进行分组。总之,蔬菜作物的核心种质构建研究与大田作物如水稻、玉米和大豆等比较还有较大的差距,今后还有很多工作要做。
3.2 蔬菜核心种质研究发展方向
3.2.1 基于多种类型数据构建核心种质 到目前为止,蔬菜核心种质构建大多是基于形态数据,很少综合多类型数据进行研究。形态数据方便获取,且类型丰富,但受环境影响较大,不同环境及管理水平下获取的数据差异明显,导致构建核心种质过程中易产生重复材料或重要材料的遗漏。生理生化、分子数据可靠,但获取难度较大,尤其原始群体太大的情况下,更是耗时费力。因此,在构建时先通过形态数据将原始群体初步压缩,得到初级核心种质,在此基础上通过生理生化或分子数据把初级核心种质再次压缩,筛选出核心种质。不同类型数据可以相互补充,能较全面揭示资源间的关系,只有通过多种类型数据进行研究、构建的核心种质才能最大限度的代表该物种的遗传多样性。
3.2.2 多种方法结合构建核心种质并加强对核心种质评价检验研究 取样策略、取样比例仍是目前核心种质研究的重点和热点,关系到所构建核心种质的可靠性。核心种质构建过程就是去同留异的过程,面对杂乱的原始种质,采用不同的数据处理方法获得的核心种质的遗传结构、多样性差异都不尽相同。加之蔬菜种类繁多,不同蔬菜地理分布、栽培历史、起源地及进化情况都不相同,因此,在构建时应多种方法对比,筛选最适合的构建方法。此外,对核心种质的研究大多集中在取样策略和取样规模上,对已构建的核心种质的评价检验研究甚少,且大多集中在表型性状遗传多样性的验证方面,以后需加强蔬菜核心种质评价方法、分子水平遗传多样性检验等方面的研究。
3.2.3 重视核心种质库的动态性 核心种质库并不是一成不变的。首先,由于构建初期收集材料并未穷尽该作物所有材料,在后期还需继续补充新发现材料;其次,在构建过程中由于数据、构建方法等的原因导致某些材料的遗漏或重复,在后期的研究中要将此类遗漏材料及时收入,重复材料则置于保留种质中保存;此外,核心种质、保留种质及初级核心种质之间应是相互补充,相互完善的。
3.2.4 进一步加强科研合作 核心种质的构建过程涉及到多个学科,包括生理学、遗传学、生物化学、生态学、统计学、分子生物学等,因此,核心种质构建需要多领域研究人员的参与。鉴于蔬菜种类繁多,各研究单位构建核心种质时掌握的原始材料有限且材料的纯度、数据记录及处理标准、分析方法等都不尽相同,最终构建出的核心种质也不会一样,因此,需要多科研单位协作。在核心种质构建初期,可以资源共享,以获得更为全面的原始材料;对于已构建的核心种质的,可以将各单位构建的核心种质进行整合,以形成一个代表性更广的统一的核心种质。
[1]贾继增,张启发.为第二次“绿色革命”发掘基因资源-国家重点基础研究发展规划项目"农作物核心种质构建、重要新基因发掘与有效利用研究"总体设计及研究进展[J].中国基础科学,2001,7:4-8.
[2]李锡香,沈嫡,王海平,等.我国蔬菜种质资源研究进展与发展策略[J].中国蔬菜,2006,(Suppl):3-9.
[3]Frankel O H.Genetic perspectives of germplasm conservation[M]//Arber W,Llimensee K,Peacock W J,et al,(eds).Genetic manipulation:impact on man and society.Cambridge,UK:Cambridge University Press,1984:161-170.
[4]李自超.中国地方稻种资源核心种质取样策略和表型及同工酶遗传多样性研究[D].北京:中国农业大学,2001.
[5]魏兴华,汤圣祥,余勇汉,等.浙江粳稻地方品种核心样品的构建方法[J].作物学报,2001,27(3):324-328.
[6]Zeuli P L S,Qualset C O.Evaluation of five strategies for obtaining a core subset from a large genetic resource collection of durum wheat[J].Theor Appl Genet,1993,87(3):295-304.
[7]郝晨阳,董玉琛,王兰芬,等.我国普通小麦核心种质的构建及遗传多样性分析[J].科学通报,2008,53(8):908-915.
[8]Hintum van T H L.Comparison of marker systems and construction of a core collection in a pedigree of European spring barley[J].Theor Appl Genet,1994,89(7/8):991-997.
[9]邱丽娟,曹永生,常汝镇,等.中国大豆(Glycine max)核心种质构建Ⅰ:取样方法研究[J].中国农业科学,2003,36(12):1442-1449.
[10]Li Y,Shi Y S,Cao Y S,et al.Establishment of a core collection for maize germplasm preserved in Chinese national gene bank using geographic distribution and characterization data[J].Genetic Resource and Crop Evolution,2004,51(8):845-852.
[11]何余堂,涂金星,傅廷栋,等.陕西白菜型油菜核心种质的初步构建[J].中国油料作物学报,2002,24(1):6-9.
[12]Hokanson S C,Lamboy W F,Szewc-McFadden A K,et al.Microsatellite(SSR)variation in a collection of Malus(apple)species and hybrids[J].Euphytica,2001,118(3):281-294.
[13]张雨春.新疆野苹果(Malus sieversii)群体遗传结构与核心种质构建方法[D].泰安:山东农业大学,2008.
[14]李银霞,安丽君,姜全,等.桃品种核心种质的构建与评价[J].中国农业大学学报,2007,12(5):22-28.
[15]刘勇,孙中海,刘德春,等.利用分子标记技术选择柚类核心种质资源[J].果树学报.2006,23(3):339-345.
[16]刘闯萍,王军,沈育杰,等.山葡萄资源核心种质的初步构建[J].植物遗传资源学报,2008,9(3):372-374.
[17]Belaj A,Dominguez-García M,Atienza S G,et al.Developing a core collection of olive(Olea europaea L.)based on molecular markers(DArTs,SSRs,SNPs)and agronomic traits[J].Tree Genetics & Genomes,2012,8(2):365-378.
[18]李国强,李锡香,沈镝,等.基于形态数据的大白菜核心种质构建方法的研究[J].园艺学报,2008,35(2):1759-1766.
[19]Huaman Z,Ortiz R,Gomez R.Selecting a Solanum tuberosum subsp andigena core collection using morphological,geographical disease and pest descriptors[J].American Journal of Potato Research,2000,77(3):183-190.
[20]庄飞云,赵志伟,李锡香,等.中国地方胡萝卜品种资源的核心样品构建[J].园艺学报,2006,33(1):46-51.
[21]刘玉皎,侯万伟.青海蚕豆种质资源AFLP多样性分析和核心资源构建[J].甘肃农业大学学报,2011,46(4):62-68.
[22]Brown A H D.Core Collections:a practical approach to genetic resources management[J].Genome,1989,31(2):818-824.
[23]Hodgkin T.Core collection and conservation of genetic resources[M]//Arora R K,Riley K W,eds.Sesame Bio-diversity in Asia,Conservation,Evaluation and Improvement.New Delhi,India,1993.
[24]Casler M D.Patterns of variation in a collection of perennial ryegrass accessions[J].Crops Science,1995,35(4):1169-1177.
[25]Basigalup D H,Barnes D K,Stucker R E.Development of core collection for perennial Medicago plant introductions[J].Crops Science,1995,35(4):1163-1168.
[26]Spagnoletti Z P L,Qualset C O.Evaluation of five strategies for obtaining a core subset from a large genetic resource collection of durum wheat[J].Theor Appl Genet,1993,87(3):295-304.
[27]王建成,胡晋,黄歆贤,等.植物遗传资源核心种质新概念与应用发展[J].种子,2008,27(5):47-50.
[28]姜慧芳,任小平,廖伯涛,等.中国花生核心种质的建立[J].武汉植物学研究,2007,25(3):289-293.
[29]黎毛毛,黄永兰,余丽琴,等.利用SSR标记构建江西稻种资源核心种质库的研究[J].植物遗传资源学报,2012,136:952-957.
[30]Erskine W,Muehlbauer F J.Allozyme and morphological variability,outcrossing rate and core collection formation in lentil germplasm[J].Theor Appl Genet,1991,83(1):119-125.
[31]董玉琛,曹永生,张学勇,等.中国普通小麦初选核心种质的产生[J].植物遗传资源学报,2003,4(1):1-8.
[32]刘长友,王素华,王丽侠,等.中国绿豆种质资源初选核心种质构建[J].作物学报,2008,34(4):700-705.
[33]Tohme J,Jones P,Beebe S,et al.The combined use of agroecological and characterisation data to establish the CIAT phaseolus vulgaris core collection[M]//Hodgkin T,Brown A H D,Hintum van T H L,et al.(eds).core collection of plant genetic resources.International plant genetic resources institute(IPGRI):A Wiley-Sayce Publication,1995:95-107.
[34]Brown A H D.The case for core collections[M]//Brown A H D,Frankel O H,Marashall R D,et al.(eds).The use of plant genetic resources.Cambridge,England:Cambridge University Press,1989:136-156.
[35]Yonezawa K,Nomura T,Mrishima H.Sampling strategies for use in stratified germplasm collections[M]//Hodgkin T,Brown A H D,Hintum van T H L,et al.(eds).Core collections of plant genetic resources.International plant genetic resources institute(IPGRI):A Wiley-Sayce Publication,1995:35-54.
[36]李锡香,方智远.从核心种质的研究入手开展农作物优异基因的挖掘利用[J].中国蔬菜,2005(suppl):1-7
[37]王丽侠,关荣霞,常汝镇,等.中国栽培大豆核心种质的取样策略[C]//中国作物学会.中国粮食安全战略:第九十次中国科协青年科学家论坛文集,2004:119-129.
[38]Diwan N,Mclntosh M S,Bauchan G R.Methods of developing a core collection of annual Medicago species[J].Theor Appl Genet,1995,90(6):755-761.
[39]杨美,付杰,向巧彦,等.利用 AFLP分子标记技术构建花莲核心种质资源[J].中国农业科学,2011,44(15):3193-3205.
[40]Zewdie Y,Tong N,Bosland P.Establishing a core collection of Capsicum using a cluster analysis with enlightened selection of accessions[J].Genetic Resources and Crop Evolution,2004,51(2):147-151.
[41]Mao W H,Yi J X,Sihachakr D.Development of core subset for the collection of Chinese cultivated eggplants using morphological-based passport data[J].Plant Genetic Resources:Characterization and Utilization,2008,6(1):33-40.
[42]Corrado G,Caramante M,Piffanelli P,et al.Genetic diversity in Italian tomato landraces:Implications for the development of a core collection[J].Science Direct,2014,168:138-144.
[43]Zong X,Redden R J,Liu Q C,et al.Analysis of a diverse global Pisum sp.collection and comparison to a Chinese local P.sativum collection with microsatellite markers[J].Theor Appl Genet,2008,118(2):193-204.
[44]Reddy L J,Upadhyaya H D,Gowda C L L,et al.Development of core collection in Pigeonpea [Cajanus cajan(L.)Millspaugh]using geographic and qualitative morphological descriptors[J].Genetic Resources and Crop Evolution,2005,52(8):1049-1056.
[45]Jansen J,van Hintum T.Genetic distance sampling:a novel sampling method for obtaining core collections using genetic distances with an application to cultivated lettuce[J].Theor Appl Genet,2007,114(3):421-428.
[46]张明,王贞,李延龙.韭菜种质资源初级核心库的构建与分析[C]//河南省植物生理学会.河南省植物生理学会三十周年庆典暨学术研讨会论文集,2010:7.
[47]吕婧.黄瓜种质资源群体结构分析与核心种质集筛选[D].北京:中国农业科学院,2011.
[48]李建春.萝卜种质资源遗传多样性的SSR分析和初级核心种质构建[D].重庆:西南农业大学,2006.
[49]Elsayed A Y A M,Granate M J,da Silva D J H,et al.Developing a core collection of Brazilian Arracacha(Arracacia xanthorrhiza Banc.)based on morphological and agronomic descriptors character of Brazil[J].Gene Conserve,2010,9:1-10.
[50]孙邦升.高淀粉马铃薯核心种质的构建与验证[D].北京:中国农业科学院,2012.
[51]Hamon S,van Sloten D H.Characterization and evaluation of Okra[M]//Brown A H D,Frankel O H,Marshall D R,et al.(eds).The use of plant genetic resources.Cambridge,UK:Cambridge University Press,1989:173-196.
[52]柴敏,于拴仓,姜立纲,等.番茄野生种 L.pennellii核心种质抗虫性初步评价[J].华北农学报,2006,21(5):87-90.
[53]Ellis P R,Pink D A,Phelps K,et al.Evaluation of a core collection of Brassica oleracea accessions for resistance to Brevicoryne brassica,the cabbage aphid[J].Euphytica,1998,103(2):149-160.

