真核生物FANCJ-like蛋白的结构与进化
吉克伍合,武泽峰,范三红,2,奚绪光
真核生物FANCJ-like蛋白的结构与进化
吉克伍合1,武泽峰1,范三红1,2,奚绪光1
1. 西北农林科技大学生命科学学院,杨凌 712100;2. 旱区作物逆境生物学国家重点实验室,杨凌 712100
FANCJ-like蛋白是一类ATP依赖的5′-3′DNA解旋酶,参与DNA损伤修复、同源重组及G4-DNA拆解,在基因组稳定性维持过程中发挥重要功能。文章系统分析了47种真核生物的FANCJ-like蛋白,对其序列结构特征及起源进化进行了深入探讨。真核生物FANCJ-like蛋白包含4类成员——XPD、CHL1、RTEL1和FANCJ,但在真菌的一些世系及昆虫中存在严重缺失现象,如接合菌门(Zygomycota)缺失了RTEL1,担子菌门(Basidiomycota)和子囊菌门(Ascomycota)缺失了RTEL1和FANCJ,双翅目昆虫缺失了FANCJ。FANCJ-like蛋白不仅包含经典解旋酶共有HD1和HD2结构域,而且在HD1结构域中插入了自身特有的Fe-S、Arch和Extra-D结构域。Fe-S和Arch结构域在4类成员中较保守,Extra-D结构域在XPD中不存在,在其他3类成员中也各不相同。在FANCJ-like蛋白的Fe-S、Arch和Extra-D结构域中分别发现了7个、10个和2个特有模体;除了已报道的保守模体外,HD1和HD2中分别发现了5个和12个特有模体。从这些特有模体的组成和排布来看,RTEL1和FANCJ最为相近,它们在HD2区包含两个独有模体Vb2和Vc,可能与其G4-DNA解旋活性相关。进化方面的证据表明,FANCJ-like蛋白起源于一种HD1区插入了Fe-S和Arch结构域的DNA解旋酶,在多细胞真核生物出现之前,该蛋白通过3次复制事件和随后的特异化过程,依次形成了目前真核生物所包含的4类FANCJ-like蛋白。
FANCJ-like蛋白;解旋酶;系统发育;保守模体;空间结构
FANCJ(Fanconi anemia complementation group J)是一种ATP依赖的5′-3′DNA解旋酶,其突变体最初是从范可尼贫血症[1~3]和早发性乳腺癌患者[4]中鉴定出来,暗示FANCJ蛋白可能作为肿瘤抑制因子维持基因组的稳定性。FANCJ-like蛋白属于解旋酶超家族2(Superfamily 2,SF2)中的Rad3/XPD亚家族,该家族成员具有解旋酶共有的7个保守模体I、Ia、II、III、IV、V和VI,构成了解旋酶核心的ATP酶/解旋酶结构域。有别于其他亚家族成员,FANCJ-like蛋白在模体Ia和II之间插入一个Fe-S结构域,在模体II和III之间插入一个Arch结构域。已有研究显示,该亚家族至少包含FANCJ、XPD、CHL1和RTEL1 4类成员。
人类()基因位于17号染色体长臂的17q23.2,该基因编码一种长为1249个氨基酸残基的5′→3′DNA解旋酶。其中N-端(1-888)包含了核心的ATP酶/解旋酶结构域,其C-端(888- 1063)为肿瘤抑制因子BRCA1(Breast cancer type 1 susceptibility protein)结合结构域[4]。Cantor等最先证实FANCJ是一种解旋酶,优先结合并解旋叉状和双链底物,并需要一段5′单链DNA[5,6]。研究表明,FANCJ蛋白在DNA链间交联(Interstrand cross-link,ICL)修复、复制胁迫响应及G4-DNA结构拆解过程中发挥重要功能[3,7,8]。鸡()突变体对DNA交联剂高度敏感,其表型可被人类所互补[9]。线虫()直系同源基因突变不仅导致个体对DNA交联剂敏感,同时导致基因组中富G/C簇及其旁侧序列的缺失[10]。
XPD(Xeroderma pigmentosum D)是通用转录因子TFIIH组分之一,参与核苷酸切除修复、转录调控和细胞凋亡调节[11,12],突变会导致人类着色性干皮病的发生[13]。近年来古细菌XPD晶体结构研究取得了重大进展,为FANCJ-like蛋白结构和功能关系的研究提供了重要信息[14~16]。CHL1是另一种FANCJ-like蛋白,酵母()基因对于姐妹染色单体粘附和正确分离是必需的[17],其突变会导致染色体丢失或不正确分离。人类CHL1同源蛋白hChlR1和hChlR2在增殖细胞系中表达,与染色单体的正确粘着有关,可与粘着复合体紧密结合。此外,hChlR1也参与人类端粒长度的决定[18]。小鼠()中编码ChlR1蛋白的基因突变导致姐妹染色体不能正确粘着和分配,产生非整倍体细胞,从而引起胚胎致死[19]。RTEL1 (Regulator of telomere length)是 FANCJ-like蛋白的另一个重要成员。最初研究发现敲除该蛋白对应基因后,小鼠胚胎干细胞表现出端粒缺失和染色体断裂与融合现象[20]。Wu[21]等研究表明,RTEL1以端粒富含G的3′尾巴为靶标,能拆解可能形成的G4-DNA结构,从而保证端粒的正确复制或修复。Barber[22]等用遗传学和生物化学方法证明线虫RTEL1是酵母RTEL1同源蛋白Srs2功能类似物,突变后表现出基因重组率升高、DNA损伤敏感,在突变背景下导致组成型致死。人类RTEL1可以拆解具有D环结构的重组中间物,但是不能拆解Rad51-ssDNA形成的纤丝,暗示RTEL1作为抗重组酶以一种独特的方式确保基因组稳定性[22]。
迄今为止,国内外学者已对FANCJ-like蛋白家族成员进行了大量的研究,主要集中在其生物学功能及作用机制的阐明。本研究系统分析了47个真核生物的FANCJ-like蛋白,对其序列多样性、起源进化、模体组织模式及空间结构差异进行了深入探讨,以阐明各种FANCJ-like蛋白的起源关系及序列和结构特征,为FANCJ-like解旋酶功能的研究奠定基础,同时为相关疾病的治疗提供参考。
1 材料和方法
1.1 FANCJ-like蛋白鉴定
FANCJ-like蛋白包含两个特征结构域:DEAD_2和Helicase_C_2,分别对应于Pfam(v27.0)数据库中的两个蛋白家族PF06733和PF13307[23]。利用Pfam数据库提供的隐马尔科夫模型,使用HMMER 3.0搜索NCBI非冗余蛋白数据库,将两结构域搜索结果E-value均小于e-10的序列认定为候选序列。异水霉罗兹壶菌()、孢霉菌()、大雌异水霉()和德氏根霉()的蛋白序列下载自Broad institute的Fungal Genome Initiative及JGI的Fungal Genomes Project项目网站。挑选出4种古细菌和47种真核生物的候选FANCJ-like蛋白,去除冗余和不完整序列,最后获得非冗余序列160条。依据物种进化关系及基因组测序的完整度,选取37个代表性物种的100条完整序列用于真核生物FANCJ-like蛋白系统发育树的构建及保守模体分析。在分析真菌FANCJ-like蛋白缺失现象过程中使用了11种真菌的32条序列,分析昆虫FANCJ-like蛋白缺失现象时使用了13种昆虫的46条序列(附表1)。
1.2 多序列比对与系统发育树构建
利用MAFFT[24](v7.023b)软件的L-INS-i算法对37个物种的候选FANCJ-like蛋白进行多序列比对分析。以多序列比对结果为依据,用PHYML 3.0[25]构建跨物种FANCJ-like蛋白系统发育树。进化树生成采用极大似然法(ML method),使用ProtTest 3.2分析获得最优氨基酸替代模型LG+I+G+F[26],并使用1000次的 Bootstrap对节点的可靠性进行评估。同时使用Mega 5.0构建基于邻接法的系统发育树,综合两种方法获得最终的系统树。根据每种蛋白所在枝的不同对其进行基于类别的重命名(附表1)。
1.3 FANCJ-like蛋白保守模体和三维结构分析
利用MEME[27](v4.9.1)对37个物种的100条FANCJ-like蛋白中的保守模体进行识别,并对各亚类中共有和特有的模体开展进一步分析。保守模体识别时,最长模体含有的氨基酸数、最短模体含有的氨基酸数及不同模体数分别设置为50、 3和 50,其他参数均采用默认值。蛋白质三维结构预测采用EXPaSy提供的在线分析软件Swiss-Model[28],分子模型使用Accelrys公司的Discovery Studio 2.5进行可视化分析。
1.4 ARCH结构域聚类分析
提取100条FANCJ-like蛋白完整的ARCH区,用CLANS[29]软件的Convex方法进行聚类(e-value:1e-40),用Jacknife方法对分类结果的可靠度进行评价。
2 结果与分析
2.1 真核生物的FACNJ-like蛋白
利用HMMER3.0软件搜索NCBI非冗余蛋白数据库和真菌基因组计划数据库,从搜索到的51个物种(4种古细菌、8种原生生物、16种真菌、4种植物和19种动物)中共鉴定出候选FANCJ-like蛋白序列232条。人工逐条对序列进行分析,去除冗余和不完整序列并重新命名,最后获得非冗余完整FANCJ-like蛋白序列160条。除嗜酸热硫化叶菌()外,其他3种古细菌均只包含1个FANCJ-like蛋白,绝大多数真菌包含2个FANCJ-like蛋白,原生生物和无脊椎动物通常包含3~4个FANCJ-like蛋白,植物和脊椎动物则通常包含4~5个FANCJ-like蛋白。

图1 真核生物FANCJ-like蛋白系统发育树
2.2 真核生物FANCJ-like蛋白的系统发育
以4种古细菌嗜酸热硫化叶菌()、极端嗜热古菌()、嗜酸热原体()和热原体属古菌()为外群,通过构建系统发育树可知,不同来源的真核FANCJ-like蛋白主要聚为4个进化枝(图1),根据已有的功能研究表明,这4个分枝分别对应于已知功能的4种FANCJ-like蛋白XPD、CHL1、RTEL1和FANCJ。同时,也可以利用该进化树对那些功能尚不清楚FANCJ-like蛋白的功能进行初步界定。此外,我们发现大部分真菌只包含XPD和CHL1两类FANCJ-like蛋白,而缺少RTEL1和FANCJ的对应物。通常情况下,每一个物种中每一类FANCJ-like蛋白只存在一个拷贝,也有少数物种中同一类FANCJ-like蛋白存在两个拷贝,如人类基因组编码两个CHL1同源蛋白,拟南芥中存在两个FANCJ同源蛋白,大雌异水霉()包含两个CHL1和两个FANCJ同源蛋白。在单细胞真核生物牛焦虫()、胶球藻()和蓝隐藻()中均存在4类FANCJ-like蛋白,而且4类蛋白在动物、植物、原生生物以及真菌中均出现,因而推测这4类FANCJ-like蛋白产生于真核生物形成早期,其出现先于多细胞生物的形成。
2.3 真菌FANCJ-like蛋白进化历程
在鉴定真核生物FANCJ-like蛋白时,最引人关注的现象是大部分真菌缺失了FANCJ和RTEL1。为了阐明这一现象,本文参考已有的真菌系统发育关系[30],对真菌中FANCJ-like蛋白的进化历程进行了仔细梳理。图2展现了真菌主要类别的分歧时间,以及各个主要类别代表物种中FANCJ-like蛋白的数目和种类。壶菌门(chytridomycota,大约出现在5.5亿年前)是目前公认起源最早的真菌,有全基因组序列支持的两种壶菌纲真菌大雌异水霉和蛙壶菌()中均出现了4类FANCJ-like蛋白。同时被认为是真菌过度祖先的隐真菌门()生物异水霉罗兹壶菌()也存在4类FANCJ-like蛋白成员,因而可以确定真菌的祖先中存在4类FANCJ-like蛋白。接合菌门(Zygomycot,大约出现在4.6亿年以前)是另一类出现较早的真菌,有全基因序列支持的两个物种德氏根霉()和被孢霉黑藻()中均缺失了RTEL1的直系同源蛋白,而3.9亿年前出现的担子菌门(Basidiomycota)和3.3亿年前出现的子囊菌门(Ascomycota)真菌同时缺失了RTEL1和FANCJ蛋白的对应物。因而在真菌进化过程中出现过两次FANCJ-like蛋白丢失现象,一次发生在接合菌门形成时期,一次发生担子菌门形成时期。为验证上述推论的正确性,本文进一步分析了140种真菌的FANCJ-like蛋白的分布(附表2),分析结果完全支持上述结论。

图2 FANCJ-like蛋白在真菌中的进化历程
合成单源树表示真菌主要群组之间的系统发育关系。左边的时间尺度和细虚线表示不同组群的大概分化时间(百万年),右边标注的是真菌的5个门和相对应的物种名。“*”表示序列不完整。
2.4 昆虫FANCJ-like蛋白的进化
昆虫纲生物与高等动物的FANCJ-like蛋白存在差异,如果蝇()和蚊子()均缺失了FANCJ蛋白的直系同源蛋白。图3呈现了昆虫主要类别的进化关系以及各类别昆虫的代表物种中FANCJ-like蛋白的数目[31]。以甲壳纲生物水蚤()为外群,昆虫主要类别的进化早晚依次为虱目(Phthiraptera)—膜翅目(Hymenoptera)—鞘翅目(Coleoptera)—鳞翅目(Lepi->doptera)和双翅目(Diptera)。除双翅目昆虫外,其它昆虫中并没有明显的证据显示缺失FANCJ蛋白。因此,昆虫的祖先中存在4类FANCJ-like蛋白,但双翅目昆虫在进化过程中丢失了其中的FANCJ蛋白。
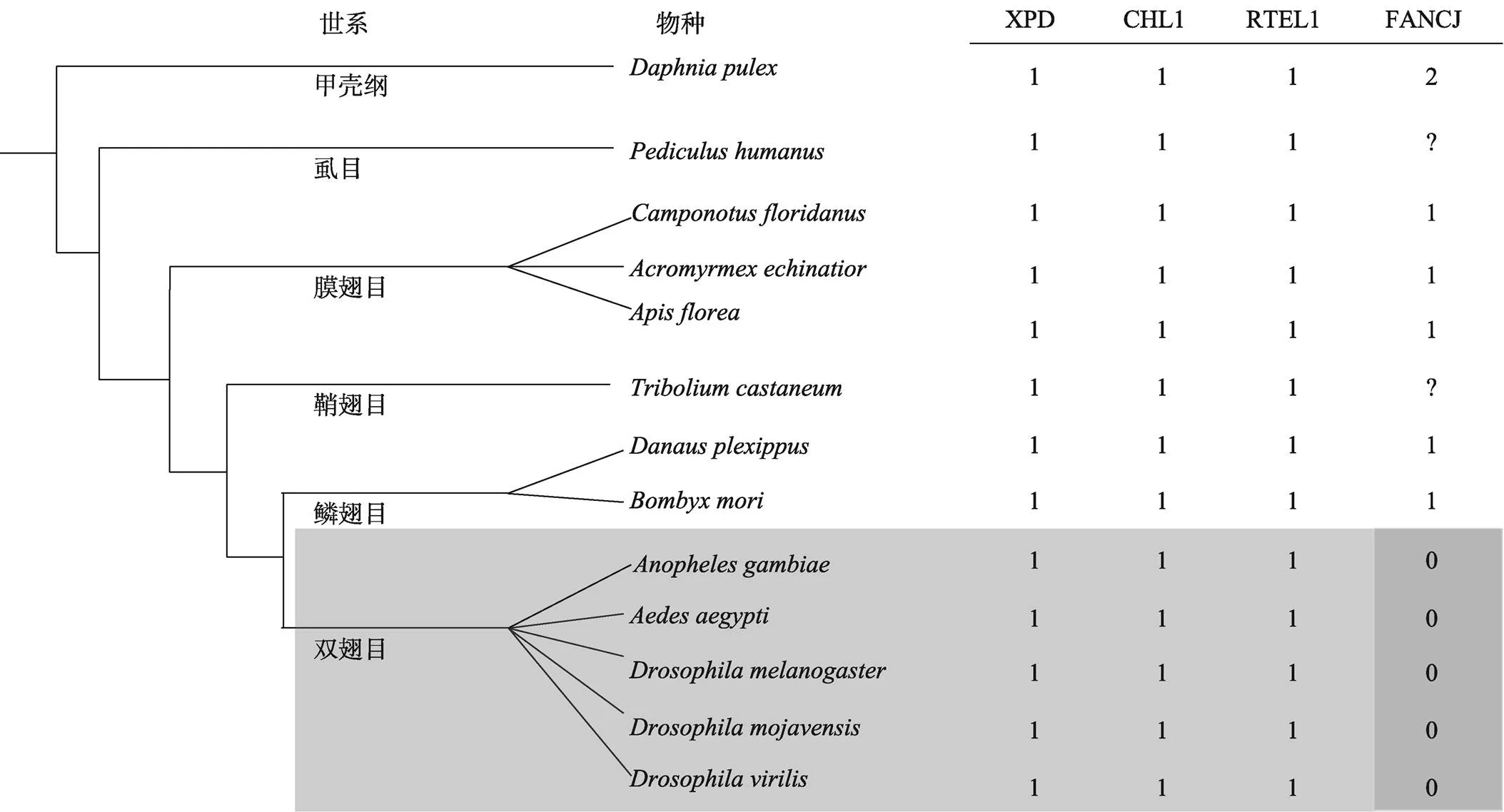
图3 节肢动物门中的FANCJ-like蛋白的分布
左侧合成单源树代表了昆虫纲中主要群组之间的系统发育关系,右侧数据统计了每个子类各个物种中所含有的FANCJ-like蛋白数。“?”表示该类FANCJ-like蛋白在该物种中未发现,但该物种是该类群中唯一全基因组测序完成的物种。
2.5 不同类别FANCJ-like蛋白保守模体的差异
FANCJ-like蛋白包含经典解旋酶所共有的两个保守结构域HD1和HD2,但其HD1结构域中插入了另外3个结构域:Fe-S、Arch和Extra-D,其中Fe-S和Arch区相对保守,Extra-D区在不同类的FANCJ-like蛋白间差异极大(图4A)。在HD1和HD2结构域对应区,4类FANCJ蛋白均包含先前报道的保守模体Q、I、Ia、Ib、Ic、II、III、IV、IVa、V、Va、VI和VII[15,32],但XPD的Q、Ib和V模体(Q1、Ib1和V1)与其他3类蛋白明显不同(Q2、Ib2和V2)。此外,本研究还在HD1和HD2区新发现了一些FANCJ-like蛋白特有保守模体:IIIa1、IIIb1 VIIa1为XPD特有,IIIa2、IIIb2、Ivb和VIIa2为CHL1、RTEL1和FANCJ1共有,Vb2和Vc为RTEL1和FANCJ共有。Extra-D区位于模体I和Ia之间,其长度和保守性在4类FANCJ-like蛋白两两间差异显著(图4B)。XPD中Extra-D区长约9~11个氨基酸残基,长度最短且变化幅度最小;CHL1中Extra-D区长约123~150,包含保守模体Ea和Eb;RTEL1中该区段长约42~52,变化幅度相对较小;而FANCJ中该区段长约30~123,变化幅度最大。不同物种FANCJ蛋白在Extra-D区段的低相似度暗示:该区段在不同物种中可能发挥着不同的作用。如脊椎动物FANCJ蛋白的Extra-D区段包含MLH1蛋白互作模体,线虫和植物FANCJ蛋白则不包含该模体。
在Fe-S区共发现了7种保守模体Fa~Fe,其中Fa和Fe为4类成员共有,它们分别包含一个半胱氨酸残基,可能参与铁硫簇的形成,而铁硫簇参与了双链和单链DNA结合点的识别并且直接参与双链的解旋。除XPD外,其他3类蛋白的Fe-S区均包含模体Fd2,其核心为DxE(D/E),包含3个连续的酸性氨基酸残基,其功能还有待研究。
在FANCJ-like蛋白的Arch区共发现了10种保守模体Aa~Ag,其中保守模体Ag为4类蛋白共有。XPD中该区包含的保守模体最多,CHL1次之,这种保守性反映出XPD和CHL1这两种蛋白的Arch区对其功能的重要性。图4C是利用CLANS 软件对4类蛋白Arch结构域序列进行聚类分析的结果,4类FANCJ-like蛋白的Arch区分别聚类在一起,其中XPD的Arch区最为收敛,其次是CHL1,而FANCJ和RTEL1较为分散。已有研究表明,XPD的Arch结构域能够募集活化激酶,作为分子开关,调控TFIIH的DNA修复和转录活性,其序列的保守性则说明该机制在进化中极其重要。
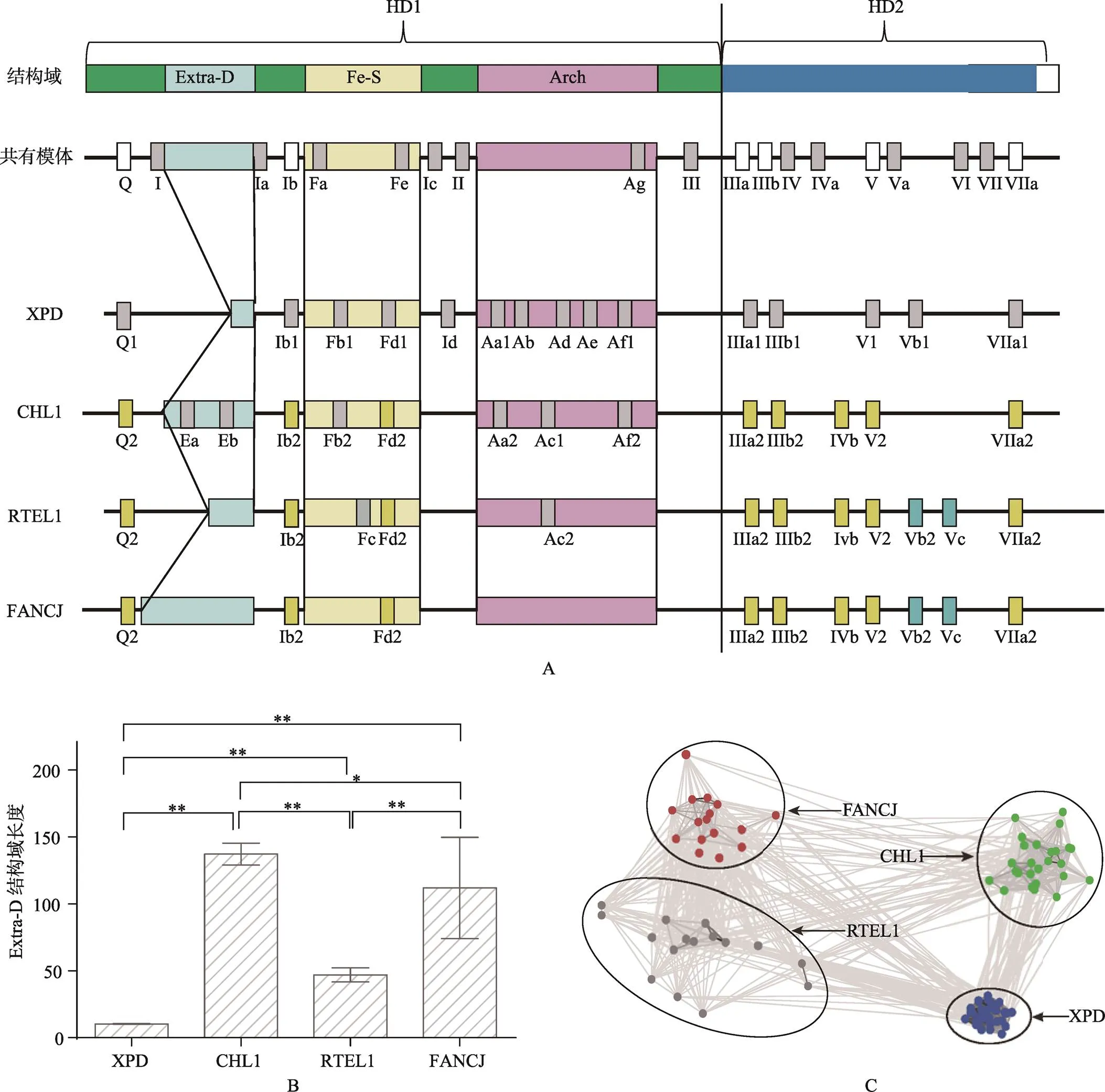
图4 FANCJ-like蛋白保守模体排布及Extra-D和Arch结构域变异度分析
A:FANCJ-like蛋白保守模体排布,灰色框代表了在一种蛋白中独有的模体,蓝绿色代表两类FANCJ-like蛋白共有的模体,黄色代表三类FANCJ-like蛋白共有的模体(详细信息见附表3和附表4);B:FANCJ-like蛋白Extra-D结构域长度统计,“*”表示两组数据间差异显著,“**”表示两组数据间差异极显著;C:FANCJ-like蛋白Arch 结构域聚类。
2.6 不同类别FANCJ-like蛋白空间结构比较
分别选取人类XPD、FANCJ和小鼠的CHL1、RTEL1作为代表,以古细菌嗜酸热菌体()XPD空间结构4a15为模板,利用在线分析软件Swiss-Model对4类FANCJ-like蛋白进行同源建模,结果如图5所示。从结构域组成和堆叠方式来看,4类FANCJ-like蛋白的三维结构非常类似,均包含经典解旋酶所必须的HD1和HD2结构域,同时包含独有Fe-S和Arch结构域。这4个结构域在4种蛋白中虽然有细微差异,但整体的排布方式完全一致,即两个马达结构域HD1和HD2被拱状Arch结构域所连接,Fe-S结构域从HD1结构域一侧伸出并与Arch结构域形成一个封闭通道。4类蛋白最大的差异在于Extra-D结构域,Extra-D在XPD中并不存在,而在其他3类蛋白质中也各不相同。从保守模体在三维结构中的位置来看,XPD包含的独有模体最多,其中一半以上都位于Arch区,这表明Arch结构域在XPD进化中比较保守,功能比较重要。Fd2模体仅出现在XPD以外的3种FANCJ-like蛋白中,该模体形成了两段短α-螺旋,中间被一段β-折叠间隔,位于Fe-S和HD1结构之间,其序列中保守的3个酸性氨基酸残基可能参与了两个结构域间的互作。Vb2和Vc是FANCJ和RTEL共有的两个模体,Vb2、Vc核心序列分别为Dx2VxLKx4D和Gx3WY,形成两段相连的α-螺旋,在HD2表面形成一个凸起。由于FANCJ和RTEL1具有G4-DNA解旋活性,而XPD和CHL1不具备该功能,因而Vb2和Vc的模体可能与G4-DNA解旋相关。

图5 FANCJ-like蛋白三维结构
3 讨 论
本研究以基因组序列为参考,对真核生物FANCJ-like蛋白的系统发育和结构差异进行了全面分析。以古细菌XPD为外群,真核FANCJ-like蛋白主要集中于4个进化枝,分别对应于XPD、CHL1、RTEL1和FANCJ蛋白。结合系统发育树、保守模体、空间结构及前人研究结果[33],本文提出了如图6所示的FANCJ-like蛋白进化历程。在原始的原核生物中首先进化出一种在HD1结构域中同时插入Fe-S和Arch结构域的DNA解旋酶,这种解旋酶随细菌和古细菌的分化而产生差异,并且一个物种中仅包含一个这样的蛋白。原始真核细胞继承了类古细菌XPD蛋白,并在多细胞真核生物分化之前发生了3次复制事件,依次演化形成目前真核生物拥有的XPD、CHL1、RTEL1和FANCJ蛋白。之所以说真核的4类FANCJ-like蛋白在多细胞真核生物形成之前就已形成,是因为牛焦虫()、胶球藻()和蓝隐藻()等单细胞真核生物基因组中已存在编码4种FANCJ- like蛋白的对应基因。细菌包含两类FANCJ-like蛋白,一种称为DinG一种称为DinG-Exo,后者比前者在N端多出了一个外切酶结构域。
在鉴定不同真核生物FANCJ-like蛋白时发现,大部分真菌缺失了FANCJ和RTEL1,而双翅目昆虫缺失了FANCJ。通过分析FANCJ-like蛋白在不同昆虫和真菌中的分布,我们证明两者祖先中均存在4类FANCJ-like蛋白,只是在进化过程中某些世系丢失了一类或两类成员。从另外一个角度而言,相较于XPD和CHL1,FANCJ和RTEL和功能并不是必不可少的,或者说它们的功能和其他蛋白的功能是重叠的,或者在缺失它们的世系中出现了其他蛋白可以互补它们的功能。FANCJ和RTEL1具有G4-DNA解旋活性,本研究对缺失FANCJ和RTEL1的节肢动物和真菌基因组中的G4-DNA进行分析(附表5),结果发现,G4-DNA含量和是否缺失FANCJ和RTEL1没有相关性。这暗示FANCJ和RTEL1的G4-DNA解旋、复制胁迫响应等功能可能被其他功能同源蛋白所行使。

图6 FANCJ-like蛋白起源与进化模式图
红色的短斜线代表了复制事件。
4类真核生物FANCJ-like蛋白具有相似的空间结构,均包含解旋酶超家族II成员共有的HD1和HD2结构域,但其HD1结构域中插入了两个特异结构域Fe-S和Arch。同时CHL1、RTEL1和FANCJ中HD1结构域的模体I和Ia之间插入了一个Extra-D结构域,该结构在XPD蛋白中不存在,在其他3类蛋白中也各不相同,甚至在同一类蛋白中也有显著差异。4类蛋白的HD1、HD2、Fe-S和Arch结构域虽然在排布和折叠方式上非常相似,但每类各有自己保守的模体,这些保守模体应该是各类蛋白功能差异的基础。XPD除了包含FANCJ-like蛋白共有的13个模体外,还包含15个特有模体,这说明XPD的结构、功能、互作蛋白及参与的生物学过程在进化中非常保守。这些特有模体突变会导致遗传疾病的发生。如人类XPD特有模体IIIa1中的R487突变为G,或VIIa1中的R722突变为W均会导致人类毛发低硫营养不良症(Trichothiodystrophy,TTD)的发生[14]。在CHL1、RTEL1和FANCJ中存在多种共有模体, 如Q2、Ib2、IIIa2、IIIb2、Ivb、V2和VIIa2,这反映出三者亲缘较近,也暗示三者存在一些相同的功能位点和调控模式。如XPD的Ib模体(Ib1)的核心保守序列为EL(K/R)(K/R)L,而CHL1、RTEL1和FANCJ中对应的Ib2模体核心保守序列为EL(K/R)(K/R)T,新出现的苏氨酸残基(T)为潜在的磷酸化位点,暗示后三者的活性可能通过该位点可逆磷酸化调节。具有G4-DNA解旋活性的RTEL1和FANCJ蛋白中发现了特有保守模体Vb2和Vc,它们的存在可能与G4解旋活性直接相关。本研究系统呈现了FANCJ-like蛋白进化历程及4类成员的序列和结构差异,为该家族蛋白功能的阐明及相关遗传疾病的诊断与治疗提供了重要参考。
www.Chinagene.cn
[1] Levitus M, Waisfisz Q, Godthelp BC, de Vries Y, Hussain S, Wiegant WW, Elghalbzouri-Maghrani E, Steltenpool J, Rooimans MA, Pals G, Arwert F, Mathew CG, Zdzienicka MZ, Hiom K, De Winter JP, Joenje H. The DNA helicase BRIP1 is defective in Fanconi anemia complementation group J, 2005, 37(9): 934–935.
[2] Levran O, Attwooll C, Henry RT, Milton KL, Neveling K, Rio P, Batish SD, Kalb R, Velleuer E, Barral S, Ott J, Petrini J, Schindler D, Hanenberg H, Auerbach AD. The BRCA1-interacting helicase BRIP1 is deficient in Fanconi anemia., 2005, 37(9): 931–933.
[3] Litman R, Peng M, Jin Z, Zhang F, Zhang J, Powell S, Andreassen PR, Cantor SB. BACH1 is critical for homologous recombination and appears to be the Fanconi anemia gene product FANCJ., 2005, 8(3): 255–265.
[4] Cantor SB, Bell DW, Ganesan S, Kass EM, Drapkin R, Grossman S, Wahrer DC, Sgroi DC, Lane WS, Haber DA, Livingston DM. BACH1, a novel helicase-like protein, interacts directly with BRCA1 and contributes to its DNA repair function., 2001, 105(1): 149–160.
[5] Cantor SB, Drapkin R, Zhang F, Lin YF, Han J, Pamidi S, Livingston DM. The BRCA1-associated protein BACH1 is a DNA helicase targeted by clinically relevant inactivating mutations., 2004, 101(8): 2357–2362.
[6] Gupta R, Sharma S, Sommers JA, Jin Z, Cantor SB, Brosh RM Jr. Analysis of the DNA substrate specificity of the human BACH1 helicase associated with breast cancer., 2005, 280(27): 25450–25460.
[7] Kumaraswamy E, Shiekhattar R. Activation of BRCA1/ BRCA2-associated helicase BACH1 is required for timely progression through S phase., 2007, 27(19): 6733–6741.
[8] Wu YL, Shin-ya K, Brosh R M Jr. FANCJ helicase defective in Fanconia anemia and breast cancer unwinds G-quadruplex DNA to defend genomic stability., 2008, 28(12): 4116–4128.
[9] Bridge WL, Vandenberg CJ, Franklin RJ, Hiom K. The BRIP1 helicase functions independently of BRCA1 in the Fanconi anemia pathway for DNA crosslink repair., 2005, 37(9): 953–957.
[10] Cheung I, Schertzer M, Rose A, Lansdorp PM. Disruption of dog-1 in Caenorhabditis elegans triggers deletions upstream of guanine-rich DNA., 2002, 31(4): 405–409.
[11] Friedberg EC, Bardwell AJ, Bardwell L, Feaver WJ, Kornberg RD, Svejstrup JQ, Tomkinson AE, Wang ZG. Nucleotide excision repair in the yeast Saccharomyces cerevisiae: its relationship to specialized mitotic recombination and RNA polymerase II basal transcription., 1995, 347(1319): 63–68.
[12] Hoeijmakers JH, Egly JM, Vermeulen W. TFIIH: a key component in multiple DNA transactions., 1996, 6(1): 26–33.
[13] Lehmann AR. The xeroderma pigmentosum group D (XPD) gene: one gene, two functions, three diseases., 2001, 15(1): 15–23.
[14] Fan L, Fuss JO, Cheng QJ, Arvai AS, Hammel M, Roberts VA, Cooper PK, Tainer JA. XPD helicase structures and activities: insights into the cancer and aging phenotypes from XPD mutations., 2008, 133(5): 789–800.
[15] Liu HT, Rudolf J, Johnson KA, McMahon SA, Oke M, Carter L, McRobbie AM, Brown SE, Naismith JH, White MF. Structure of the DNA repair helicase XPD., 2008, 133(5): 801–812.
[16] Wolski SC, Kuper J, Hänzelmann P, Truglio JJ, Croteau DL, Van Houten B, Kisker C. Crystal structure of the FeS cluster–containing nucleotide excision repair helicase XPD., 2008, 6(6): e149.
[17] Skibbens RV. Chl1p, a DNA helicase-like protein in budding yeast, functions in sister-chromatid cohesion., 2004, 166(1): 33–42.
[18] Vasa-Nicotera M, Brouilette S, Mangino M, Thompson JR, Braund P, Clemitson JR, Mason A, Bodycote CL, Raleigh SM, Louis E, Samani NJ. Mapping of a major locus that determines telomere length in humans., 2005, 76(1): 147–151.
[19] Inoue A, Li T, Roby SK, Valentine MB, Inoue M, Boyd K, Kidd VJ, Lahti JM. Loss of ChlR1 helicase in mouse causes lethality due to the accumulation of aneuploid cells generated by cohesion defects and placental malformation., 2007, 6(13): 1646–1654.
[20] Ding H, Schertzer M, Wu XL, Gertsenstein M, Selig S, Kammori M, Pourvali R, Poon S, Vulto I, Chavez E, Tam PPL, Nagy A, Lansdorp PM. Regulation of murine telomere length by: an essential gene encoding a helicase-like protein., 2004, 117(7): 873–886.
[21] Wu XL, Sandhu S, Ding H. Establishment of conditional knockout alleles for the gene encoding the regulator of telomere length (RTEL)., 2007, 45(12): 788–792.
[22] Barber LJ, Youds JL, Ward JD, McIlwraith MJ, O’Neil NJ, Petalcorin MIR, Martin JS, Collis SJ, Cantor SB, Auclair M, Tissenbaum H, West SC, Rose AM, Boulton SJ. RTEL1 maintains genomic stability by suppressing homologous recombination., 2008, 135(2): 261–271.
[23] Punta M, Coggill PC, Eberhardt RY, Mistry J, Tate J, Boursnell C, Pang N, Forslund K, Ceric G, Clements J, Heger A, Holm L, Sonnhammer ELL, Eddy SR, Bateman A, Finn RD. The Pfam protein families database., 2012, 40(Database issue): D290–D301.
[24] Katoh K, Standley DM. MAFFT multiple sequence alignment software version 7: improvements in performance and usability., 2013, 30(4): 772–780.
[25] Guindon S, Dufayard JF, Lefort V, Anisimova M, Hordijk W, Gascuel O. New algorithms and methods to estimate maximum-likelihood phylogenies: assessing the performance of PhyML 3.0., 2010, 59(3): 307–321.
[26] Darriba D, Taboada GL, Doallo R, Posada D. ProtTest 3: fast selection of best-fit models of protein evolution., 2011, 27(8): 1164–1165.
[27] Bailey TL, Williams N, Misleh C and Li W W. MEME: discovering and analyzing DNA and protein sequence motifs., 2006, 34(Web Server issue): W369- W373.
[28] Arnold K, Bordoli L, Kopp J, Schwede T. The SWISS- MODEL workspace: a web-based environment for protein structure homology modelling., 2006, 22(2): 195–201.
[29] Frickey T, Lupas A. CLANS: a Java application for visualizing protein families based on pairwise similarity., 2004, 20(18): 3702–3704.
[30] Berbee ML, Taylor JW. Dating the evolutionary radiations of the true fungi., 1993, 71(8): 1114–1127.
[31] Trautwein MD, Wiegmann BM, Beutel R, Kjer KM, Yeates DK. Advances in insect phylogeny at the dawn of the postgenomic era., 2012, 57: 449–468.
[32] Fairman-Williams ME, Guenther UP, Jankowsky E. SF1 and SF2 helicases: family matters., 2010, 20(3): 313–324.
[33] White M F. Structure, function and evolution of the XPD family of iron-sulfur-containing 5′→3′ DNA helicases., 2009, 37(Pt 3): 547–551.
(责任编委: 黄原)
Structure and evolution of the eukaryotic FANCJ-like proteins
Wuhe Jike1, Zefeng Wu1, Sanhong Fan1,2, Xuguang Xi1
The FANCJ-like protein family is a class of ATP-dependent helicases that can catalytically unwind duplex DNA along the 5′-3′ direction. It is involved in the processes of DNA damage repair, homologous recombination and G-quadruplex DNA unwinding, and plays a critical role in maintaining genome integrity. In this study, we systemically analyzed FNACJ-like proteins from 47 eukaryotic species and discussed their sequences diversity, origin and evolution, motif organization patterns and spatial structure differences. Four members of FNACJ-like proteins, including XPD, CHL1, RTEL1 and FANCJ, were found in eukaryotes, but some of them were seriously deficient in most fungi and some insects. For example, the Zygomycota fungi lost RTEL1, Basidiomycota and Ascomycota fungi lost RTEL1 and FANCJ, and Diptera insect lost FANCJ. FANCJ-like proteins contain canonical motor domains HD1 and HD2, and the HD1 domain further integrates with three unique domains Fe-S, Arch and Extra-D. Fe-S and Arch domains are relatively conservative in all members of the family, but the Extra-D domain is lost in XPD and differs from one another in rest members. There are 7, 10 and 2 specific motifs found from the three unique domains respectively, while 5 and 12 specific motifs are found from HD1 and HD2 domains except the conserved motifs reported previously. By analyzing the arrangement pattern of these specific motifs, we found that RTEL1 and FANCJ are more closer and share two specific motifs Vb2 and Vc in HD2 domain, which are likely related with their G-quadruplex DNA unwinding activity. The evidence of evolution showed that FACNJ-like proteins were originated from a helicase, which has a HD1 domain inserted by extra Fe-S domain and Arch domain. By three continuous gene duplication events and followed specialization, eukaryotes finally possessed the current four members of FANCJ-like proteins.
FANCJ-like proteins; helicase; phylogeny; conserved motifs; spatial structure
2014-04-05;
2014-05-09
国家自然科学基金项目(编号:31370798, 11304252)资助
吉克伍合,硕士研究生,专业方向:分子进化与系统发育分析。E-mail:wuhe@nwsuaf.edu.cn
范三红,博士,副教授,研究方向:生化与分子生物学,生物信息学。E-mail:shfan@nwsuaf.edu.cn奚绪光,博士,教授,研究方向:生物大分子的结构与功能。E-mail:xxi01@ens-cachan.fr
10.16288/j.yczz.14-115
网络出版时间: 2014-11-19 16:46:08
URL: http://www.cnki.net/kcms/detail/11.1913.R.20141119.1646.001.html

