海岛棉Sus活性变化特征及时空表达模式与纤维比强度的关系
贾春平,耿洪伟,倪志勇,朱亚夫,黄启秀,曲延英,陈全家
(新疆农业大学 农学院/农业技术重点实验室,乌鲁木齐 830052)
海岛棉Sus活性变化特征及时空表达模式与纤维比强度的关系
贾春平,耿洪伟,倪志勇,朱亚夫,黄启秀,曲延英,陈全家*
(新疆农业大学 农学院/农业技术重点实验室,乌鲁木齐 830052)
摘要:以海岛棉(Gossypium barbadense L.)品种‘C6015’和‘新海29’(高比强度组)以及‘巴1248’和‘比马1’(低比强度组)为实验材料,利用果糖和UDPG比色法以及qRT-PCR方法,对2个实验组不同纤维发育时期蔗糖合成酶(EC 2.4.1.13,Sus)活性变化特征及其基因家族时空表达模式进行测定,并分析与纤维比强度的关系,探讨海岛棉纤维比强度差异形成的主要生理与分子机理。结果显示:(1)品种‘C6015’和‘新海29’的平均纤维比强度分别为47.5和44.7 cN·tex-1,‘巴1248’和‘比马1’分别为31.2和32.6 cN·tex-1,两实验组平均纤维比强度差异极显著。(2)纤维发育过程中4个海岛棉品种的Sus活性变化特征均呈单峰曲线,且低比强度组的峰值出现较早,但高比强度组的峰值以及后期活性极显著高于低比强度组。(3)海岛棉纤维发育过程中高表达的Sus基因有Sus1A、Sus1D、Sus3A、Sus3D、Sus6A、Sus6D、Sus8D,但各基因成员在纤维发育过程中具有表达特异性;其中两实验组的Sus3A基因都是在纤维次生壁加厚初期(花后20 d)开始大量表达且达最大值后下降,说明Sus3A基因在纤维次生壁加厚初期起作用;Sus1A、Sus1D基因在高比强度实验组的纤维次生壁加厚后期和末期(花后30 d)相对表达量较高并有明显上升现象,而同期在低比强度实验组中相对表达量很低且无上升现象,说明Sus1A、Sus1D基因作用于纤维次生壁加厚后期和末期。(4)两实验组的Sus活性水平及其基因家族各成员相对表达量高低与纤维次生壁加厚后期维持高活性时间的长短存在明显差异,表现为高比强度组>低比强度组;且两组Sus活性高低差异与Sus3A、Sus1A、Sus1D基因的表达差异同步。研究表明,海岛棉Sus3A、Sus1A、Sus1D基因的表达差异与纤维比强度的形成有关,可能是影响纤维比强度的关键基因。
关键词:海岛棉;蔗糖合成酶活性;纤维比强度;表达模式
Sus催化Sucrose+UDP↔fructose+UDPG的可逆反应,但更偏向蔗糖裂解进程[1],参与库器官中蔗糖强度和细胞生长分化的相关调控[2],是蔗糖代谢调节中的关键酶之一[3]。在淀粉及纤维素的合成、种子发育、逆境胁迫、生物固氮及调节植物细胞碳水化合物的分配及贮藏过程中发挥重要作用[4-10]。另外Sus基因在进化过程中功能上发生分化,表现出组织和植株发育阶段特异性[11-14]。Sus是一个多基因家族[15],四倍体棉花以系统进化命名法可能有15个Sus基因(Sus1A-Sus8D),各成员与纤维生长发育的关系尚不明确[16]。
纤维比强度的形成及高低与纤维素的合成及平缓累积密切相关。纤维素合成的直接底物是尿苷二磷酸-D-葡萄糖(UDPG),该反应受多个酶和基因的调控[17-18],Sus可催化生成UDPG,为该反应提供前体物质。故其活性及基因表达水平直接影响纤维素合成速度和沉积质量,进而影响纤维比强度[19]。
纤维比强度是原棉的重要品质指标之一[20-21]。海岛棉以纤维较细、结构致密、比强度高著称。因此,本研究选择比强度差异明显的2组海岛棉品种为实验对象,利用果糖和UDPG比色法及qRT-PCR技术,探究纤维生育时期Sus活性变化特征及其基因家族各成员表达特异性与作用,分析对纤维比强度的影响。旨在探索海岛棉纤维比强度差异形成的主要生理与分子机理,深入研究纤维比强度的形成机制,为在分子水平上提升棉花产量及改良纤维品质的辅助育种方法提供理论依据。
1材料和方法
1.1实验材料
海岛棉品种‘C6015’、‘巴1248’、‘新海29’和‘比马1’选自新疆农业大学农学院作物遗传育种海岛棉资源群体库。并于2015年4月12日种植于阿克苏地区阿拉尔市16团科研基地。2015年7月8日盛花期时开始,每天早晨9:00选择中部果枝一致已盛开但未授粉的花朵挂牌并记录开花日期,分别摘取4个品种开花后0、5、10、15、20、25、30和35 d的棉铃,将纤维和种子(胚珠)剥离后用锡箔纸包好并标记样品,液氮速冻后分2份置于-20 ℃和-80 ℃冰箱中保存备用。
1.2Sus活性及纤维品质的测定
利用测定果糖和UDPG比色法测定不同比强度品种Sus活性[22]。包括缓冲液配制、粗酶液制备和酶活性测定3个步骤。酶活性的单位为mg·g-1·h-1,即为1 g样品经酶促反应1 h后产生的蔗糖量(以mg计)。采用BioTek公司的全功能微孔板检测仪测定蔗糖在480 nm处吸光值,纤维样品的的Sus活性计算公式为:
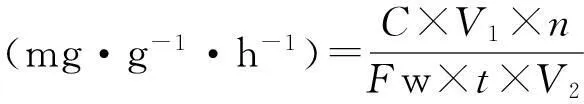
式中,C为依据样品吸光值根据标准方程计算的蔗糖量;V1为提取的粗酶液的体积;V2为在反应时加的粗酶液体积;Fw为样品质量;n为反应时稀释倍数;t为酶促反应时间。

表1 本研究用到的引物信息
注:引物设计模板来自黄圣等的Sus序列[28]。
Note:The primer template for qRT-PCR comes from HUANG Setal[28].
棉花采收轧花后,不同比强度品种各送15 g左右混合纤维样品至农业部棉花质量监督检验测试中心(乌鲁木齐)检测纤维品质。
1.3实时荧光定量(qRT-PCR)检测
采用北京全式金公司的改良CTAB法(TransZolPlant RNA)试剂盒提取海岛棉纤维中的总RNA。提取纤维总RNA所用的全部实验耗材均确保RNase-free。每个纤维发育时期样品独立提取3份总RNA,置于-80 ℃冰箱保存备用。利用Titertek-Berthold公司的超微量分光光度计(Colibri)检测总RNA浓度和质量,通过北京全式金公司的DNase Ⅰ(RNase-free)处理总RNA样品中的DNA杂质。
每个采用Thermo公司逆转录试剂盒(RevertAid First strand cDNA Synthesis kit)合成的cDNA样品,都用ddH2O稀释20倍后作为qRT-PCR模板。使用北京全式金公司的qRT-PCR试剂盒(TransStart Green qPCR SuperMix),通过ABI Prism 7500 Fast System检测15个Sus基因在纤维不同生育时期的表达水平。扩增体系为25 μL,包括cDNA 1 μL约2 ng、2×Trans Start Green qPCR SuperMix 10 μL、上下游引物各0.4 μL,Passive Reference Dye Ⅱ(50×)0.4 μL,不足体系部分用ddH2O补足。采用两步法增加特异性,扩增条件为94 ℃预变性30 s;94 ℃变性5 s,60 ℃退火34 s,共40个循环。根据定量扩增曲线达到平台期时的Ct值,以ubiquitin为内参基因计算相对表达量。相对表达量计算公式为2-△Ct。实时荧光定量PCR引物序列(表1),使用SPSS 17.0中独立样本t检验进行方差分析。
2结果与分析
2.1海岛棉纤维品质性状分析
4个海岛棉品种的3年共8项纤维品质性状数据列于表2。通过SPSS 17.0中独立样本t检验对‘C6015’和‘巴1248’(第1组)及‘新海29’和‘比马1’(第2组)进行方差分析发现,这2组在断裂比强度上均达到极显著差异(P<0.01);在上半部平均长度上,第2组呈极显著差异;在整齐度比率上,第2组呈极显著差异;在纺纱均匀性指数上,第1组呈显著差异(P<0.05),第2组呈极显著差异(P<0.01);其余各项纤维品质性状数据均无显著差异。
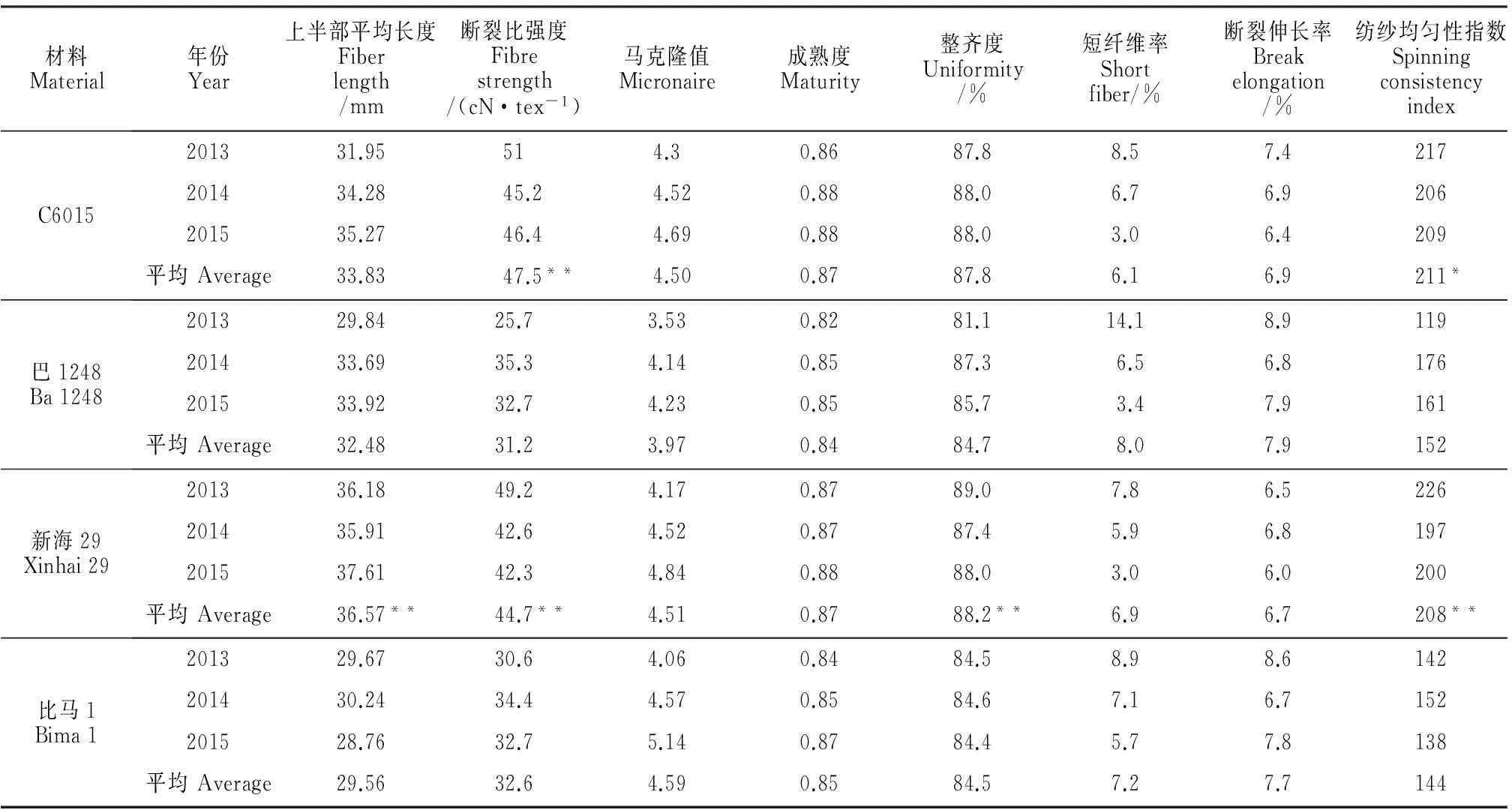
表2 海岛棉4个材料的纤维品质性状数据
注:海岛棉纤维品质数据由农业部棉花质量监督检验测试中心(乌鲁木齐)提供;*和**分别表示0.05和0.01的差异显著水平。下同。
Note:Fiber quality traits ofGossypiumbarbadensewere provided by ministry of agriculture cotton quality supervising and testing center (Urumqi);* and ** mean significant difference at the levels of 0.05 and 0.01,respectively.The same as below.
2.2海岛棉纤维发育过程中Sus活性变化特征
图1显示,纤维发育过程中4个海岛棉品种的Sus活性变化特征均呈单峰曲线。其中‘C6015’和‘新海29’Sus活性峰值出现在花后30 d,‘巴1248’和‘比马1’峰值出现在花后25 d。另外,前二者维持高Sus活性时间长于后二者,突出表现在花后25~35 d。前二者花后25~30 dSus活性继续呈快速上升现象,而后二者Sus活性开始出现下降现象;花后30~35 d前二者Sus活性小幅下降但依然维持在较高水平,而后二者Sus活性已呈快速下降趋势并降至较低水平。通过SPSS 17.0中独立样本t检验发现‘C6015’和‘巴1248’(第1组)与‘新海29’和‘比马1’(第2组)纤维发育过程中Sus活性差异显著(图1)。
2.3不同棉纤维发育时期Sus基因表达模式分析
为了对上述Sus活性变化特征结果进行更为深入的阐释,利用15对特异性引物,通过qRT-PCR对4个海岛棉品种不同纤维发育时期Sus基因家族各成员表达模式进行了检测。通过SPSS 17.0中独立样本t检验分别对‘C6015’和‘巴1248’(第1组)及‘新海29’和‘比马1’(第2组)不同纤维发育时期Sus基因家族各成员相对表达量进行显著性分析。结果显示,纤维中高表达的Sus基因有Sus1A、

图1 纤维发育过程中Sus活性的动态变化
Sus1D、Sus3A、Sus3D、Sus6A、Sus6D、Sus8D。Sus1A、Sus1D分别在纤维发育的起始期和伸长期(花后0~15 d)和起始期(花后0~5 d)大量表达,且这2个基因在次生壁加厚后期(花后30~35 d)表达量又重新回升(图2,A、B);Sus3D、Sus8,Sus8A和Sus7A、Sus7D分别在起始期(花后0~5 d)和纤维突起和快速伸长期(花后0~10 d)大量表达(图2,E、H);Sus4A、Sus5D、Sus8D都是在纤维伸长期(花后15 d)达到峰值(图2,F、I);Sus3A在次生壁加厚初期(花后20 d)开始大量表达为最大值随后快速下降(图2,D);海岛棉纤维生长发育全时期Sus6D、Sus6A的表达量远高于其他13个Sus基因,它们可能是纤维中优势表达的基因(图2,G)。而Sus2A、Sus4D全时期表达量极低或不表达,可能与纤维发育关系不大(图2,C)。
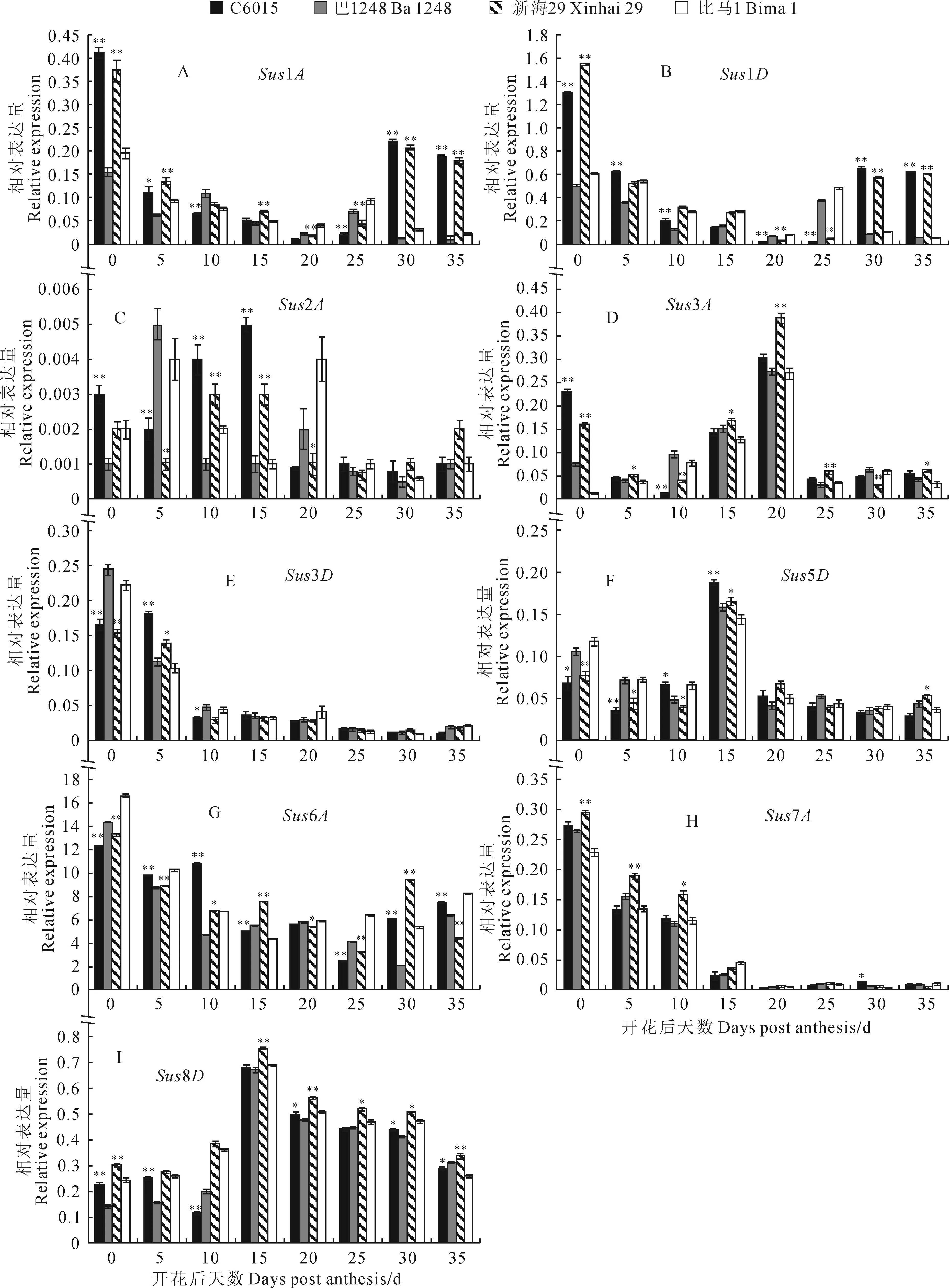
图2 纤维发育过程中蔗糖合成酶基因表达模式分析

图3 纤维发育过程中Sus基因相对高表达情况及分布模式
2.4纤维发育过程中Sus基因相对高表达情况及分布模式
利用相对表达量公式:{‘C6015’(Sus1A,花后0 d)+‘新海29’(Sus1A,花后0 d)}/2-{‘巴1248’(Sus1A,花后0 d)+‘比马1’(Sus1A,花后0 d)}/2取正值舍负后得到Sus1A基因在花后0 d的相对高表达情况。利用公式依次可计算出其它14个Sus基因在花后0 d的相对高表达情况。同理,可计算Sus1A~Sus8D基因在花后5~35 d的相对高表达情况。通过直观的观察比较Sus基因各成员相对高表达情况及分布模式可发现(图3),纤维发育全时期Sus6D、Sus6A的表达量远高于其他13个Sus基因,尤其在花后5~15 d。花后20 d相对高表达的Sus基因有Sus3A、Sus5D、Sus6D、Sus8D;花后25 d为Sus3A、Sus8D;花后30 d为Sus1A、Sus1D、Sus6D、Sus8D;花后35 d为Sus1A、Sus1D、Sus3A、Sus6D。排除可能是纤维中优势表达Sus基因(Sus6D),可能是组成型表达Sus基因(Sus8D)和其它相对高表达不明显Sus基因(Sus5D)的影响后,我们认为Sus3A、Sus1A、Sus1D为纤维次生壁加厚期(花后20~35 d)相对高表达的Sus基因。
3讨论
目前,异源四倍体陆地棉(GossypiumhirsutumL.)全基因组图谱(AADD基因组)已于2015年完成解析工作,结束了棉花没有参考基因组图谱的历史[23]。在注释过程中发现陆地棉有15个Sus基因[16],它们在结构上具有保守性,但功能上又发生分化。Brill等[24]对陆地棉纤维发育不同阶段SusA、SusB、SusC、SusD基因研究发现,这4者在不同的纤维发育阶段发挥不同的调控作用。Salnikov等[25]发现纤维次生壁加厚期Sus活性和纤维素沉积量与比强度关系密切且呈显著正相关。张文静等[26]和束红梅等[27]研究也表明高比强度品种Sus活性动态变化,基因表达量与纤维素快速累计增长期的协调性好,纤维素累积平缓,纤维比强度增幅大;低比强度品种反之。
本研究选用比张文静等[26]和束红梅等[27]所用研究材料平均纤维比强度更高,纤维比强度差异更明显的海岛棉材料作为研究对象,在研究Sus基因对纤维比强度的影响方面具有一定的说服力。同时,设立的研究时期比Zou等[16]和黄圣等[28]的更多,可更全面分析多个Sus基因参与棉纤维生长发育阶段,且不同的Sus基因在不同阶段作用不同的情况。结合花后0~35 d的Sus活性变化特征测定结果,可对Sus基因在纤维发育全时期相关作用机制进行深入细致的阐释。结果表明,2个实验组Sus3A基因在花后20 d开始大量表达为最大值随后下降趋势明显,表达模式同于黄圣等[28]对陆地棉GhSus3A的研究结果,不同于Chen等[29]对二倍体亚洲棉(GossypiumarboretumL.)GaSus3在花后15 d达到最大值的研究结果,说明四倍体棉花中Sus3A主要作用于纤维次生壁加厚初期而不是纤维快速伸长期。2个实验组Sus1A基因在花后0~15 d的表达模式与陆地棉GhSus1A基因相似,但本研究进一步发现,高比强度实验组Sus1A基因在花后30~35 d表达量又重新回升。低比强度实验组Sus1A基因除在花后20 d表达量略高于前者外,同期相对表达量并没有上升且远低于前者。海岛棉Sus1D、Sus3D、Sus8,Sus8A基因在2个实验组花后0~5 d大量表达,其中Sus3D、Sus8、Sus8A基因和低比强度实验组Sus1D基因与黄圣等[28]对陆地棉GhSus1D基因和Chen等[29]对二倍体亚洲棉GaSus1基因的研究结果相同,呈现出纤维起始期明显特异高表达,纤维伸长期及次生壁加厚初期下降的表达模式。本研究还发现,高比强度实验组Sus1D基因在花后30~35 d相对表达量又重新开始上升,低比强度实验组Sus1D基因除在花后25 d表达量高于前者外,同期Sus1D基因表达量并没有上升且远低于前者,以上说明Sus1A、Sus1D基因作用于纤维次生壁加厚后期和末期。2个实验组Sus7A、Sus7D基因在花后0~10 d相对表达量较高,其余时期相对于其他13个Sus基因表达量很低或基本不表达。该现象与黄圣等[28]对陆地棉GhSus7D基因的研究结果基本相一致。2个实验组Sus4A、Sus5D、Sus8D基因都是在花后15 d达到峰值,Sus4A、Sus5D基因的表达模式与Zou等[16]和黄圣等[28]对陆地棉GhSus4A、GhSus5D基因的研究结果相一致,即在纤维伸长期依赖Sus4A、Sus5D基因合成其所必须的大量纤维素。本研究还发现海岛棉Sus8D基因在达到峰值后并没有像Sus4A、Sus5D基因一样快速回落到较低的表达水平而是全程持续较高表达,且Sus8D基因在纤维发育各时期表达量都高于Sus4A、Sus5D基因。这与陆地棉GhSus8D基因转录丰度低、表达水平差的现象不同。因除了花后10 d和花后35 d外都有Sus8D基因高表达情况发生,故推测可能原因是其在海岛棉纤维发育过程中属组成型表达基因。通过实时荧光定量结果分析,我们认为海岛棉Sus3A、Sus1A、Sus1D基因与纤维比强度的形成与差异有关,可能是影响纤维比强度的关键基因。
参考文献:
[1]GEIGENBERGER P,STITT M.Sucrose synthase catalyses a readily reversible reactioninvivoin developing potato tubers and other plant tissues[J].Planta,1993,189(3):329-339.
[2]KLECZKOWSKI LA,KUNZ S,WILCZYNSKA M.Mechanisms of UDP-Glucose synthesis in plants[J].PlantSciences,2010,29(4):191-203.
[3]KOUCH K E,NOLTE K D,DUKE E R,etal.Sugar levels modulate differential expression of maize sucrose synthase genes[J].PlantCell,1992,4(1):59-69.
[4]FU H,PARK W D.Sink and vascular-associated sucrose synthase functions are encroded by different gene classes in potato[J].ThePlantCellOnline,1995,7(9):1 369-1 385.
[5]FARRAR J,POLLOCK C,GALLAGHER J.Sucrose and the integration of metabolism in vascular plants[J].PlantSciences,2000,154(1):1-11.
[6]HEIM U,WEBER H,BAUMLEIN H,etal.A sucrose synthase gene ofViciafabaL.:expression pattern in developing seeds in relation to starch synthesis and metabolic regulation[J].Planta,1993,191(3):394-401.
[7]BAI W Q,XIAO Y H,ZHAO J,etal.Gibberellin overproduction promotes sucrose synthase expression and secondary cell wall deposition in cotton fibers[J].PLoSOne,2014,9(5):e96537.
[8]XIAO X H,TANG C R,Fang Y J,etal.Structure and expression profile of the sucrose synthase gene family in the rubber tree:indicative of roles in stress response and sucrose utilization in the laticifers[J].FEBSJournal,2014,281(1):291-305.
[9]BAIER M C,KECK M,GOBBE V,etal.Knockdown of the symbiotic sucrose synthaseMtSucS1 affects arbuscule maturation and maintenance in mycorrhizal roots ofMedicagotruncatula[J].PlantPhysiology,2010,152(2):1 000-1 014.
[10]COLEMAN H D,YAN J,MANSFIELD S D.Sucrose synthase affects carbon partitioning to increase cellulose production and altered cell wall ultrastructure[J].ProceedingsoftheNationalAcademyofSciences,2009,106(31):13 118-13 123.
[11]HIROSEA T,SCOFIELDB G N,TERAOA T.An expression analysis profile for the entire sucrose synthase gene family in rice[J].PlantScience,2008,174(5):534-543.
[12]ISLAM M Z,HU X M,JIN L F,etal.Genome-wide identification and expression profile analysis of citrus sucrose synthase genes:investigation of possible roles in the regulation of sugar accumulation[J].PLoSOne,2014,9(11):e113623.
[13]ZHANG C H,YU M L,MA R J,etal.Structure,expression profile,and evolution of the sucrose synthase gene family in peach (Prunuspersica)[J].ActaPhysiologiaePlantarum,2015,37(4):1-15.
[14]ZHANG D Q,XU B H,YANG X H,etal.The sucrose synthase gene family inPopulus:structure,expression,and evolution[J].TreeGenetics&Genomes,2011,7(3):443-456.
[15]GOREN S,HUBER S C,GRANOT D.Comparison of a novel tomato sucrose synthase,SlSUS4,with previously described SlSUS isoforms reveals distinct sequence features and differential expression patterns in association with stem maturation[J].Planta,2011,233(5):1 011-1 023.
[16]ZOU C S,LU C R,SHANG H H,etal.Genome-wide analysis of theSusgene family in cotton[J].JournalofIntegrativePlantBiology,2013,55(7):643-653.
[17]DELMER D P,HAIGLER C H.The regulation of metabolic flux to cellulose,a major sink for carbon in plants[J].MetabolicEngineering,2002,4(1):22-28.
[18]HAIGLER C H,MILKA I D,HOGAN P S,etal.Carbon Partitioning to Cellulose Synthesis[M].PlantCellWalls,2001,47:29-51.
[19]BROWN R M,SAXENA I M,KUDLICKA K.Cellulose biosynthesis in higher plants[J].TrendsinPlantScience,1996,1(5):149-155.
[20]MEREDITH W R.Minimum number of genes controlling cotton fiber strength in a backcross population[J].CropScience,2005,45(3):1 114-1 119.
[21]倪志勇,吕萌,李波,等.棉花GhCCR1基因结构及表达分析[J].西北植物学报,2011,31(1):14-18.
NI Z Y,LÜ M,LI B,etal.Structure and expression ofGhCCR1 fromGossypiumhirsutumL.[J].ActaBot.Boreal.-Occident.Sin.,2011,31(1):14-18.
[22]汤章程.现代植物生理学实验指南[M].北京:科学出版社,1999:126-128.
[23]LI F G,FAN G Y,LU C R,etal.Genome sequence of cultivated upland cotton (GossypiumhirsutumTM-1) provides insights into genome evolution[J].NatureBiotechnology,2015,33(10):524-530.
[24]BRILL E,THOURNOUT M V,WHITE R G,etal.A novel isoform of sucrose synthase is targeted to the cell wall during secondary cell wall synthesis in cotton fiber[J].PlantPhysiology,2011,157(1):40-54.
[25]SALNIKOV V V,GRIMSON M J,SEAGULL R W,etal.Localization of sucrose synthase and callose in freeze-substituted secondary-wall-stage cotton fibers[J].Protoplasma,2003,221(3):175-184.
[26]张文静,胡宏标,王友华,等.棉纤维发育相关酶活性的基因型差异与纤维比强度的关系[J].中国农业科学,2007,40(10):2 177-2 184.
ZHANG W J,HU H B,WANG Y H,etal.Fiber strength and enzyme activities of different cotton genotypes during fiber development[J].ScientiaAgriculturaSinica,2007,40(10):2 177-2 184.
[27]束红梅,王友华,张文静,等.2个棉花品种纤维发育关键酶活性变化特征及其与纤维比强度的关系[J].作物学报,2008,34(3):437-446.
SHU H M,WANG Y H,ZHANG W J,etal.Activity changes for enzymes associated with fiber development and relation to fiber strength in two cotton cultivars[J].ActaAgronomicaSinica,2008,34(3):437-446.
[28]黄圣,何鹏,田莉莉,等.棉花蔗糖合成酶基因家族在纤维发育期时空表达模式分析[J].棉花学报,2015,27(4):317-328.
HUANG S,HE P,TIAN L L,etal.Expression profiles of the cottonSusgene family during fiber development[J].CottonScience,2015,27(4):317-328.
[29]CHEN A Q,HE S E,LI F F,etal.Analyses of the sucrose synthase gene family in cotton:structure,phylogeny and expression patterns[J].BMCPlantBiology,2012,12(3):1-17.
(编辑:宋亚珍)
Activity Changes and Expression Profiles ofSuswith Relation to Fiber Strength in Island Cotton (GossypiumbarbadenseL.)
JIA Chunping,GENG Hongwei,NI Zhiyong,ZHU Yafu,HUANG Qixiu,QU Yanying,CHEN Quanjia*
(College of Agronomy,Key Laboratory of Agricultural Biological Technology,Xinjiang Agricultural University,Urumqi 830052,China)
Abstract:In this study,we used island cotton (Gossypium barbadense L.) variety as experimental materials,which are ‘C6015’ and ‘Xinhai 29’,(high fiber strength experimental group);‘Ba 1248’ and ‘Bima 1’,(low fiber strength experimental group).Activity changes and expression profiles of sucrose synthase (EC 2.4.1.13,Sus) during different development periods were measured by fructose and UDPG colorimetric with qRT-PCR methods in two experimental groups,which differences of fiber strength are obvious.Analysis the relationship between activity changes,expression profiles of Sus and fiber strength.In addition,we discussed the main physiological and molecular mechanisms,which lead to form differences of fiber strength in island cotton.The results showed that:(1)the average strength of ‘C6015’ and ‘Xinhai 29’:47.5 cN·tex-1and 44.7 cN·tex-1,respectively;the average strength of ‘Ba 1248’ and ‘Bima 1’:31.2 cN·tex-1and 32.6 cN·tex-1,respectively.The difference was extremely significant about the average strength in two experimental groups.(2)Activity change features of Sus were all single peak curve appear in 4 island cotton varieties,as well as peak appear more early in low fiber strength experimental group.However,peak and later activity of Sus in high fiber strength experimental group were higher and reached to very significant as compared with the low fiber strength experimental group.(3)High expression of Sus genes were Sus1A,Sus1D,Sus3A,Sus3D,Sus6A,Sus6D and Sus8D,but each member of Sus genes had specific expression during fiber development in island cotton.Expression of Sus3A gene start increased to maximum then droped at initial stage of secondary wall thickening (20 days post anthesis) in two experimental groups.Showed that it emphasized on the role of this period;expression of Sus1A and Sus1D genes happened increased phenomenon at late and final period of secondary wall thickening (30-35 days post anthesis) in high fiber strength experimental group.However,expression of Sus1A and Sus1D genes were very low and did not existence this phenomenon at those periods in low fiber strength experimental group.The results showed that both of them work in these periods.(4)The obvious differences about Sus activity level,relative expression of Sus gene each member,as well as Sus activity maintain time during late stage of secondary wall thickening in two experimental groups showed as high fiber strength experimental group>low fiber strength experimental group.Furthermore,the differences of Sus activity about high or low was synchronize with the differences of expression in Sus3A,Sus1A and Sus1D genes.Research showed that:Sus3A,Sus1A,Sus1D genes associated with formation and differences of fiber strength.They may key genes,which affect the fiber strength.
Key words:Gossypium barbadense L.;sucrose synthase activity;fiber strength;expression profiles
中图分类号:Q789
文献标志码:A
作者简介:贾春平(1992-),在读硕士研究生,主要从事棉花纤维品质改良研究。E-mail:sevenjcp@163.com*通信作者:陈全家,博士,教授,硕士生导师,主要从事棉花遗传育种研究。E-mail:chqjia@126.com
基金项目:新疆维吾尔自治区高技术研究发展计划(201411103)
收稿日期:2015-11-25;修改稿收到日期:2016-01-18
文章编号:1000-4025(2016)02-0249-08
doi:10.7606/j.issn.1000-4025.2016.02.0249

