昆明地区妊娠糖尿病孕妇网膜下脂肪的全基因组甲基化差异研究
钱源 刘君 李晓红 马忠蕊 王艳梅 肖雪 郭知 张兰 屈在卿
(昆明医科大学第一附属医院 妇产科产前诊断室,云南 昆明 650032)
昆明地区妊娠糖尿病孕妇网膜下脂肪的全基因组甲基化差异研究
钱源刘君李晓红马忠蕊王艳梅肖雪郭知张兰屈在卿
(昆明医科大学第一附属医院 妇产科产前诊断室,云南 昆明650032)
【摘要】目的本研究应用甲基化芯片技术研究妊娠糖尿病网膜下脂肪与正常对照组的全基因组甲基化差异,提供妊娠期糖尿病网膜下脂肪全基因组范围内的甲基化差异数据背景,为寻找妊娠糖尿病网膜脂肪基因表达差异原因提供线索。方法收集3例通过OGTT实验确诊但未经过治疗的妊娠期糖尿病患者和同期3例年龄,孕次产次,孕前BMI与之无差异的健康对照者网膜下脂肪组织,提取总RNA后,采用Illumina Methylation BeadChipchip芯片进行检测,并进行基因甲基化结果进行比较,寻找具有甲基化差异的基因。结果结果发现两研究组中网膜下脂肪的全基因组DNA甲基化存在差异。妊娠糖尿病组中总共有1298个基因发生了低甲基化,1570个基因发生了高甲基化。这些基因参与了细胞骨架构建,细胞凋亡调控,细胞核内信号转导,糖和脂代谢,炎症反应等。进一步数据分析发现,两组样本在miRNA启动区上位点甲基化变化水平不一致的基因有3个,包括:PSORS1C1,PCDHB13和DKFZp686A1627。在同一CpG岛区域位点甲基化变化水平不一致的基因有7个,在miRNA区域具有甲基化差异的基因有13个。结论正常对照组与GDM组中基因的甲基化位点和水平存在显著差异,这可能与该基因在组织中表达的蛋白水平在两组中的差异表达密切相关。是否甲基化的差异是导致相应基因表达水平差异的原因还需要深入的研究。未来需要进一步弄清DNA甲基化在网膜下脂肪中对基因表达目标蛋白的调控作用,为妊娠糖尿病的治疗和预防提供线索。
【关键词】妊娠期糖尿病;甲基化谱;全基因组;网膜下脂肪组织
妊娠糖尿病(GDM)的病因涉及遗传、环境、疾病等诸多方面,其发病机制也相当复杂,到目前为止,还没有完全揭示清楚。GDM是由基因和环境相互作用引起的代谢紊乱,主要特点是胰岛素抵抗。胰岛素抵抗的本质就是单位胰岛素的生物效应的降低,即胰岛素刺激葡萄糖利用能力的降低。最初,可以通过代偿性增加胰岛素分泌,产生高胰岛素血症,维持血糖水平正常。当这一过程发展到超过机体代偿极限,则表现为糖尿病。
与皮下组织不同,网膜下组织在解剖学上拥有更丰富的血流和神经分布,它表达更高的受体和具有更活跃的细胞代谢活性,使其对人体交感神经系统敏感,分泌更多的脂肪因子和输出更多的游离的脂肪酸,导致其与代谢相关疾病的关联性更高[1-9]。
大量经典遗传学手段已经证明,不同的个体在GDM易感性上存在差异,但是单纯从基因变异的角度是无法完整解释妊娠糖尿病的发生和发展。
研究发现,DNA甲基化在脂肪组织生长发育过程中发挥重要作用,DNA甲基化,特别是启动子区域的甲基化可以调控脂肪细胞分化转录因子,转录辅助因子以及很多脂肪组织特异性基因的表达[10-12]。糖尿病患者通过长期饮食控制和运动会使得胰岛素抵抗的情况得到改善。这些证据都支持DNA甲基化的异常非常可能是导致妊娠糖尿病发生的重要原因,同时,也可能成为改善或者治疗妊娠糖尿病的突破口。
因此,本研究应用甲基化芯片技术研究妊娠糖尿病网膜下脂肪与正常对照组网膜下脂肪的全基因组甲基化水平,比对和分析正常对照组与GDM组中基因的甲基化位点和水平的差异,提供了妊娠期糖尿病网膜下脂肪全基因组范围内的甲基化差异数据背景,为寻找妊娠糖尿病网膜脂肪基因表达差异原因提供线索。
1资料与方法
1.1一般资料本次研究对象为2012年1月至2014年5月在昆明医科大学第一附属医院妇产科门诊定期产前检查并入院分娩的孕妇。在知情同意的前提下,选取3位妊娠期糖尿病孕妇为病例组(GDM组),选取同期的3位正常孕妇为对照组(NGT组)。
1.1.1GDM的纳入标准既往无高血压、糖尿病以及心、肝、肾疾病以及自身免疫性疾病,并符合75g OGTT的妊娠糖尿病诊断标准。
1.1.2NGT的纳入标准既往以及妊娠期无高血压,糖尿病以及心,肝,肾疾病和自身免疫性疾病。75gOGTT试验正常。
入选本研究的孕妇均无重大疾病家族史,且在年龄,身高,体质量和孕周等方面无明显差异。病例均为单病种病例,除本文提及的疾病外未发现其他疾病。
1.2方法
1.2.1样本采集在剖宫产术中迅速剥离大网膜脂肪,大小约为0.5cm×0.5cm×0.5cm,立即存储于RNALater中,冻存于-80℃超低温冰箱中。
1.2.2采用 Illumina Methylation BeadChip芯片进行检测甲基化检测将基因组DNA亚硫酸盐处理,变性和扩增后,用随机内切酶酶切扩增产物。再将这些DNA片段与Methylation BeadChip芯片进行杂交,通过芯片上的特异性捕获探针与相互补的酶切基因片段结合,杂交过夜,清洗后进行单碱基延伸和染色。根据只有与gDNA发生互补结合的探针才能得到延伸,染色后扫描。
1.2.3数据分析将扫描得到的原始数据,通过GenomeStudio 软件,根据Methylation Analysis Algorithms 生成每个样本每个位点的甲基化水平,经过偏差校正和位点过滤,得到甲基化水平结果。两组样本进行配对的甲基化分析,筛选差异甲基化位点的标准为:adjustP<0.05,Beta 差异值≥0.2。
2结果
对妊娠糖尿病组和正常对照组的网膜下脂肪进行全基因组甲基化水平检测,比对并分析两组样本组中网膜下脂肪的全基因组DNA甲基化差异。结果发现总共有1298个基因发生了低甲基化,1570个基因发生了高甲基化。
进一步数据分析发现,两组样本在miRNA启动区上位点甲基化变化水平不一致的基因有3个,包括PSORS1C1、PCDHB13和DKFZp686A1627。在同一CpG岛区域位点甲基化变化水平不一致的基因有7个,在miRNA区域具有甲基化差异的基因有13个。具体基因列表见表1~3。
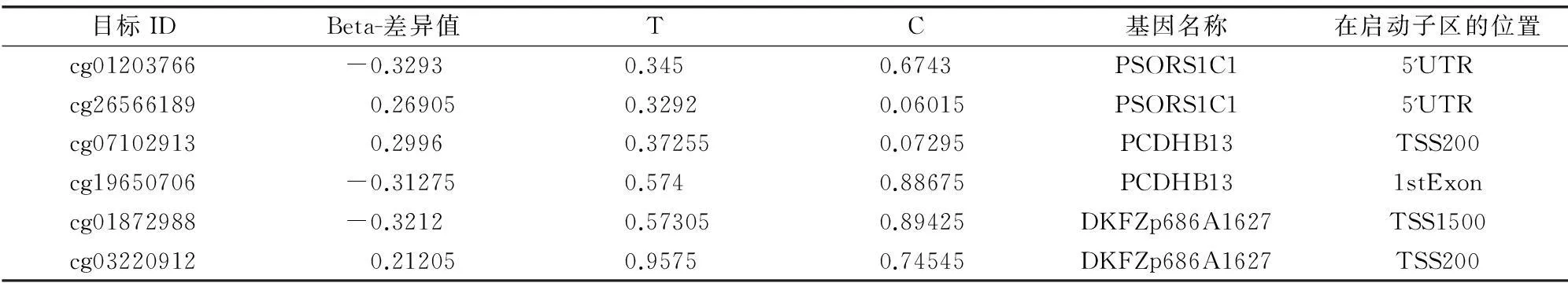
表1 在miRNA启动区上位点甲基化变化水平不一致的结果列表

表2 在同一CpG岛区域位点甲基化变化水平不一致结果表
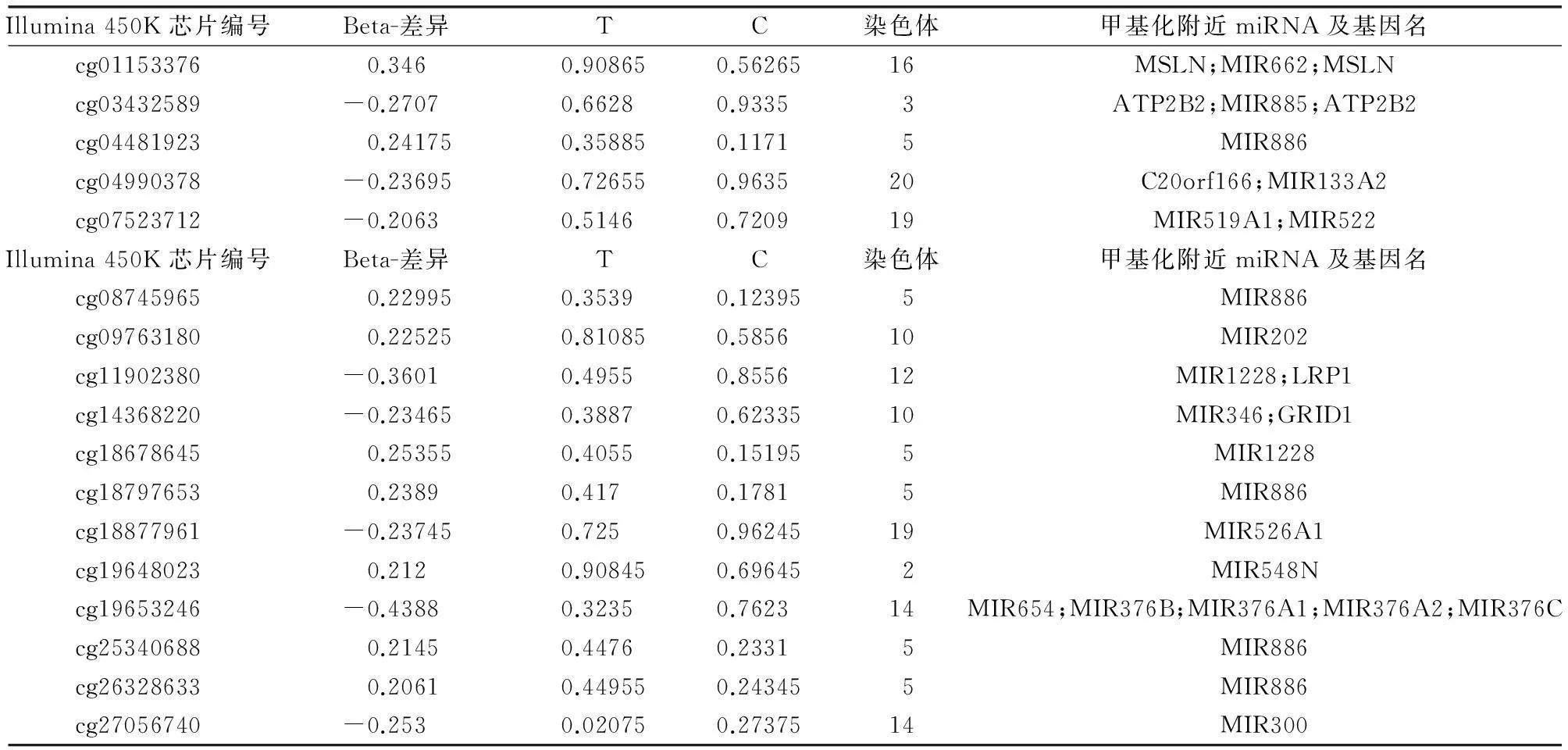
表3 在miRNA区域具有甲基化差异的位点
3讨论
本研究比对并分析两研究组中网膜下脂肪的全基因组DNA甲基化差异,结果发现总共有1298个基因发生了低甲基化,1570个基因发生了高甲基化。这些基因参与了细胞骨架构建、细胞凋亡调控、细胞核内信号转导、糖和脂代谢、炎症反应等。
在表1中,两组样本组中miRNA启动区上位点甲基化变化水平具有差异的基因包括:PSORS1C1、PCDHB13和DKFZp686A1627。PSORS1C1又称为SEEK1,位于染色体6p21.3上HLA-C基因座的终端,两个基因呈紧密连锁。Cheung YH等[13]报道PSORS1C1可能和TCF19、POU5F1、CCHCR1等是1型糖尿病潜在的关联致病基因。PCDHB13编码原钙黏附蛋白β13,这个基因是原钙黏附β基因簇的一员,定位在5号染色体上。该蛋白由一个外显子编码,在特异的细胞对细胞的神经连接中起到重要的作用。DKFZp686A1627又名为PHF2P1,是一个假基因。
表2中,在同一CpG岛区域位点甲基化变化水平不一致的基因包括:PFKP、GSTT1、ZFYVE28、PCDHB、RNF39、HLA-C、HLA-DRB。其中,PFKP基因又名为PFKF、PFK-C、PFK-P或者 ATP-PFK,定位在10号染色体上。该基因编码PFKP蛋白酶,主要在糖代谢中不可逆的催化6磷酸果糖成为1,6双磷酸果糖,在糖酵解中发挥重要作用[14,15]。Morgan AR等[16]报道过PFKP的基因变异与SGA的发生密切相关。GSTs(谷胱甘肽-S-转移酶)是人体内一类具有重要解毒作用的多基因酶族。GSTs催化各种亲电子致癌物与谷胱甘肽反应。其中GST-θ同工酶由GSTT1基因编码,分别对多环芳烃类和卤代烷烃类具有很强的解毒作用[17,18]。RNF39又称为HZF,HZFW, LIRF。该基因存在于6号染色体MHC I基因区域内中间,HLA-J的前面。该基因编码的蛋白在小鼠功能实验中表明在神经突触可塑性中起到非常重要的作用。人类白细胞抗原(HLA)基因位于第6对染色体短臂上,为一组密切连锁的基因群。Ⅰ类基因区域包括HLA-A、HLA-B、HLA-C和其他一些功能未明的基因及假基因,其编码的抗原分子存在于全部有核细胞的表面,负责递呈外来抗原给CD8+的T淋巴细胞;Ⅱ类基因区域主要包括HLA-DR、HLA-DQ和HLA-DP3个亚区,分别编码DR、DQ和DP抗原,存在于成熟B淋巴细胞及抗原递呈细胞表面,负责递呈抗原给CD4+细胞。HLA通过主要组织相溶性复合体(MHC)限制,参与T淋巴细胞识别抗原和其他免疫细胞的相互作用,以及自身耐受的形成和维持,在识别自身和异己、诱导和调节免疫反应等多个方面均具有重要作用。研究支持HLA与糖尿病的发生有强烈的相关性[19]。
表3中,在miRNA区域具有甲基化差异位点的基因包括:MSLN、ATP2B2、MIR886、MIR202、MIR1228、MIR346、MIR886、MIR526A1、MIR548N、MIR654、MIR886和MIR300。其中,MSLN称为间皮素,是一种分子量为40KDa的细胞表面糖蛋白,高表达于多种肿瘤组织中,一些研究表明,特殊信号途径的激活在肿瘤间皮素表达增加中起重要作用[20]。ATP2B2称为钙离子转运ATP酶B2。ATP酶能将三磷酸腺苷(ATP)催化水解为二磷酸腺苷(ADP)和磷酸根离子的酶,是一个释放能量的反应。小鼠动物模型中的研究支持ATP2B2的变异与听力丢失和运动平衡密切相关[21]。Hu CC等[22]报道C20orf166-AS1在前列腺癌组织中具有显著性差异表达,推测其可能是前列腺癌的特异lncRNAs。研究支持MiR-886-3p 在人骨髓细胞中下调SDF1的表达[23]。Mir346作用于RIP140蛋白的5'非转录区,上调RIP140蛋白的表达。受体相互作用蛋白140(receptor-interacting protein 140, RIP140)是一种转录辅抑制因子,其与核受体结合后能够负向调节多种代谢组织中靶基因的转录,包括脂肪组织、肌肉组织以及肝脏等。基因沉默RIP140后,多种代谢途径相关基因表达上调,主要涉及糖酵解、甘油三酯合成、三羧酸循环、脂肪酸β氧化、线粒体电子传递以及氧化磷酸化等能量代谢过程[24]。RIP140有望成为治疗代谢综合征的候选靶点。He J等[25]认为MiR300可能在调节细胞应对辐射时参与了调节细胞凋亡,细胞周期以及DNA损伤和修复的信号通路。
综上,本研究支持正常对照组与GDM组中基因的甲基化位点和水平存在显著差异,这可能与该基因在组织中表达的蛋白水平在两组中的差异表达密切相关。是否甲基化的差异是导致相应基因表达水平差异的原因还需要深入的研究。未来需要进一步弄清DNA甲基化在网膜下脂肪中对基因表达目标蛋白的调控作用,为妊娠糖尿病的治疗和预防提供线索。
参 考 文 献
[1]骆天红,赵萸,李果,等.肥胖及2型糖尿病患者内脏脂肪基因差异表达研究[J].中国应用生理学杂志,2007,23(2):229-234.
[2]李晓南,陈荣华,Tommy Olsson,等.人类皮下和网膜脂肪组织脂肪细胞因子的表达[J].江苏医药,2005,31(12):914-916.
[3]张磊,石琳琳,卢丹,等.单纯性肥胖及肥胖伴2型糖尿病患者血清中瘦素水平的变化及其与胰岛素抵抗的关系[J].吉林大学学报(医学版),2005,31(1):130-132.
[4]叶小龙,王长江,林刚,等.Visfatin在2型糖尿病患者腹部皮下和网膜脂肪组织中的表达及其与胰岛素抵抗的关系[J].安徽医科大学学报,2007,42(2):205-207.
[5]李晓南,陈荣华,潘亚,等.人类皮下和网膜脂肪组织中脂联素基因的表达和调控的研究[J].南京医科大学学报(自然科学版),2006,26(1):6-9.
[6]吴海娅,贾伟平,魏丽,等.肥胖及2型糖尿病患者血清视黄醇结合蛋白4水平的变化及其临床意义[J].中华内分泌代谢杂志,2006,22(3):290-293.
[7]计晨琳,孙斌,韩素萍,等.不同年龄女性脂肪组织11βHSD1 mRNA表达水平及临床意义[J].中国老年学杂志,2012,32(18):3878-3880.
[8]陈卓,马润玫,胡大春,等.妊娠期糖尿病脂肪组织Omentin mRNA表达差异与糖代谢的相关性[J].中国妇幼保健,2011,26(24):3791-3794.
[9]李冰,杨菊红,王慧杰,等.Chemerin基因在自发性2型糖尿病大鼠脂肪组织中表达的研究[J].天津医科大学学报,2010,16(3):447-450.
[10]张思佳,乔虹.DNA甲基化与人类糖尿病的研究进展[J].中国老年学杂志,2014,(10):2896-2898.
[11]杨梅,孙家忠,孙妍蕾,等.Leptin基因甲基化与糖尿病的相关性研究[J].中华医学遗传学杂志,2012,29(4):474-477.
[12]易斌,蔡旭,张浩,等.基因组DNA和结缔组织生长因子基因启动子甲基化状态与2型糖尿病肾病的关系[J].中华肾脏病杂志,2012,28(3):189-193.
[13]Cheung YH, Watkinson J, Anastassiou D. Conditional meta-analysis stratifying on detailed HLA genotypes identifies a novel type 1 diabetes locus around TCF19 in the MHC[J]. Hum Genet,2011, 129(2): 161-176.
[14]Andreasen CH, Mogensen MS, Borch-Johnsen K, et al. Non-replication of genome-wide based associations between common variants in INSIG2 and PFKP and obesity in studies of 18,014 Danes[J]. PLoS One,2008, 3(8): e2872.
[15]Sharifi S, Daghighi S, Motazacker MM, et al. Superparamagnetic iron oxide nanoparticles alter expression of obesity and T2D-associated risk genes in human adipocytes[J]. Sci Rep,2013,3: 2173.
[16]Morgan AR, Thompson JM, Murphy R, et al. Obesity and diabetes genes are associated with being born small for gestational age: results from the Auckland Birthweight Collaborative study[J]. BMC Med Genet,2010,11: 125.
[17]Olsson AH, Volkov P, Bacos K, et al. Genome-wide associations between genetic and epigenetic variation influence mRNA expression and insulin secretion in human pancreatic islets[J]. PLoS Genet,2014,10(11): e1004735.
[18]Zhou SF, Liu JP, Chowbay B. Polymorphism of human cytochrome P450 enzymes and its clinical impact[J]. Drug Metab Rev,2009,41(2): 289-295.
[19]Smigoc Schweiger D, Mendez A, Kunilo Jamnik S, et al. Genetic risk for co-occurrence of type 1 diabetes and celiac disease is modified by HLA-C and killer immunoglobulin-like receptors[J]. Tissue Antigens,2014,84(5): 471-478.
[20]Kendrick ZW1, Firpo MA, Repko RC, et al. Serum IGFBP2 and MSLN as diagnostic and prognostic biomarkers for pancreatic cancer[J]. HPB (Oxford) ,2014,16(7):670-676.
[21]Carpinelli MR, Manning MG, Kile BT, et al. Two ENU-induced alleles of Atp2b2 cause deafness in mice[J]. PLoS One, 2013, 8(6):e67479.
[22]Hu CC, Gan P, Zhang RY,et al. Identification of Prostate Cancer LncRNAs by RNA-Seq[J].Asian Pac J Cancer Prev,2014,15(21):9439-9444.
[23]Pillai MM, Yang X, Balakrishnan I, et al.MiR-886-3p down regulates CXCL12 (SDF1) expression in human marrow stromal cells[J].PLoS One,2010,5(12):e14304.
[24]Tsai NP, Lin YL, Wei LN. MicroRNA mir-346 targets the 5'-untranslated region of receptor-interacting protein 140 (RIP140) mRNA and up-regulates its protein expression[J]. Biochem J,2009,424(3):411-418.
[25]He J, Hua J, Ding N, et al. Modulation of microRNAs by ionizing radiation in human gastric cancer[J]. Oncol Rep, 2014,32(2):787-793.
编辑:宋文颖
·视频导读·
【Abstract】ObjectiveIn order to investigate the pathogenesis of gestational diabetes mellitus (GDM), the differences of genes methylation patterns in omenta adipose tissues between pregnant women with GDM and the normal pregnancies were compared.These data provided the background of methylation patterns in whole genome, which could provide the clues for explaining the discrepancy of gene expression between the two study groups. Method3 cases of omenta adipose tissues in insulin untreated GDM pregnancies diagnosed by oral glucose tolerance test (OGTT) were collected. Meanwhile, the same cases of omenta adipose tissues in normal pregnancies were collected. There was no discrepancy between the two groups , including gestation age, parity and pre-pregnancy BMI. The RNAs of these tissues were extracted, and the gene methylation of these tissues were detected by Illumina Methylation BeadChipchip. The differences of gene expression profiles between GDM and normal controls were analyzed in order to find the possible disease genes. ResultsThere was discrepancy of mathylation pattern in whole genome between the two groups. 1298 genes were hypomethylated and 1570 genes were hypermethylated in GDM group. These genes were involved in many physiological functions such as: cytoskeleton conformation, cell apoptosis, ignal transduction,fat and sugar metabolism and inflammation. Futher data supported that there were different methylation patterns in three genes, PSORS1C1,PCDHB13and DKFZp686A1627. There were seven genes with different methylation patterns in the same CpG island regions, thirteen genes with diggerent methylation patterns in miRNA regions.ConclusionsThere are notable CpG methylation differences in omental adipose tissues between GDM and the normal pregnancie, which would be closely relatively with the expression difference of target genes. Whether the discrepancy of methylation pattern was the lead course of the exprssion were needed for further research. The mechanisms of gene methylation on target protein should be elucidated, which could provide the clues for prevention and treatment of GDM.
【Key words】GDM; methylation profile; genome; OVAT
DOI:10.13470/j.cnki.cjpd.2016.01.001
基金项目:云南省科技厅-昆明医学院应用基础研究联合专项资金项目(2012FB039);国家自然科学基金(81360103)
*通讯作者:钱源,E-mail:yuanqian2x@hotmail.com
【中图分类号】R714.256
【文献标识码】A
(收稿日期:2015-07-01)

