水稻bHLH转录因子Os11g39000的功能研究
刘选明 彭玉冲 杨远柱 李懿星++夏妙林 王文文++林建中

摘要:水稻Os11g39000 为非典型的bHLH家族成员,其功能尚不清楚.酵母双杂交实验表明,转录因子Os11g39000可以形成同源蛋白聚合体.随机结合位点筛选实验表明,转录因子Os11g39000可以与DNA结合,并且其结合位点初步确定为TT/CG/CACC/GT/C.Os11g39000基因的组织表达模式分析发现,Os11g39000基因主要在叶片和根中表达,推测该基因可能在叶和根的发育调控中发挥作用.水培实验发现,该基因功能缺陷型转基因株系的根长显著短于野生型,表现出明显的根部发育缺陷,表明Os11g39000在水稻根的发育中起调控作用.QPCR分析进一步证明,功能缺陷型转基因植株体内生长素合成和信号转导相关基因表达量显著上升,表明转录因子Os11g39000参与了水稻生长素的合成和信号转导的调控.
关键词:水稻;bHLH转录因子;Os11g39000;生长素
中图分类号:S601 文献标识码:A
转录因子(tarnscription factor, TF)是能够与目的基因上游特定DNA序列结合的蛋白质,又被称为反式作用因子,通过与顺式作用元件相互作用,从而保证目的基因以特定强度在特定时间与空间表达的蛋白质分子[1].bHLH(basic HelixLoopHelix, 碱性螺旋环螺旋)转录因子是真核生物蛋白质中的一个大家族,其成员在动植物界行使着多种重要的功能.例如,一个bHLH蛋白BPEp可以通过与生长素应答因子ARF8相互作用调控拟南芥花蕊的早期发育[2].研究发现bHLH在光敏色素的信号转导,细胞分化,应答油菜素内酯的基因表达等过程中都起着重要作用[3-5].
典型的bHLH结构域包含大约60个氨基酸,由位于bHLH结构域N端能够结合DNA的basic区域和HLH区域组成[6].HLH区域包含两个α螺旋,通过它们bHLH蛋白可以相互作用,形成同源或者异源蛋白聚合体[1, 7-8].一类以人类Id蛋白为代表缺少basic区域的HLH蛋白虽然不能够与DNA直接相互作用,但是可以通过与其它bHLH蛋白的HLH区域形成特异的异源蛋白聚合体来抑制相应bHLH转录因子的活性[9-10].通过抑制像E这样的bHLH蛋白,从而在细胞分化和发育过程中起到关键作用[11].在拟南芥中,发现一个HLH蛋白KIDARI,通过与一个在光调控过程中起作用的bHLH转录因子相互作用来抑制该转录因子活性,从而调控植物生长发育[12].
随着水稻全基因组测序的完成,利用生物信息学分析发现,水稻中包含有167个bHLH转录因子,绝大多数成员功能未知.有研究表明,bHLH家族在水稻生长发育过程中起着关键作用,如Zhang[13]等发现一对参与BR下游的bHLH基因ILI1和PRE1可以通过形成异源二聚体的形式参与调控与BR相关的基因的表达.ili1的水稻突变体表现出与使用BR处理时相同的叶片倾角的表型,过表达和RNA干扰的ILI1转基因水稻株系都分别表现出了叶片倾角增加和减小的相反表型.ILI1和PRE1的相互作用导致了水稻叶片的直立.因此该家族蛋白功能的揭示将有助于丰富和完善水稻基因功能调控网络.
本研究选取了一个水稻非典型的bHLH基因家族成员Os11g39000 基因进行了初步的功能研究,发现转录因子Os11g39000可以以同源蛋白聚合体的形式结合DNA,从而实现生长素(IAA)信号转导,调节下游基因的表达,最终实现对水稻根部生长发育的调控.
1材料与方法
1.1生物信息学分析
利用TIGR(http://tigrblast.tigr.org/tgi, Rice Genome Annotation Project)和NCBI(http://www.ncbi.nlm.nih.gov, National Center for Biotechnology information)数据库获得Os11g39000全长CDS序列、氨基酸序列及启动子序列.通过基于隐马科夫模型的蛋白质结构域分析数据库SMART(http://smart.emblheidelberg.de, Simple Modular Architecture Research Tool)对Os11g39000氨基酸序列比对分析,获得Os11g39000蛋白的结构域类型.同时,利用PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)数据库对Os11g39000起始密码子ATG上游1 500 bp的启动子序列进行分析,预测该基因启动子区域所存在的调控元件,根据调控元件推测其可能的调控途径.
1.2载体构建与功能缺陷型转基因株系的获得
以粳稻品种日本晴的cDNA为模板,利用Gateway方法,设计含有attB接头的引物(F:5'CAAAAAAGCAGGCTTCATGTCGTCGAGCCGGCG3'; R: 5'CAAGAAAGCTGGGTCTTACATGAGTAGGCTACGGATGAGG3'),PCR克隆目的片段.PCR反应程序为:98 ℃, 5 min;98 ℃,10 s;58 ℃,15 s;72 ℃,20 s;共35个循环.利用BP ClonaseII (Invitrogen)经BP重组反应将PCR产物克隆到入门载体pDONR/Zeo,测序正确后,利用LR ClonaseII (Invitrogen)将Os11g39000克隆到玉米Ubiquitin启动子驱动表达的pCAMBIA1301GWEAR表达载体中,得到N端添加转录抑制基序EAR的合成型转录因子[14],并抑制目标转录因子的转录活性.
根据林建中等的水稻转化方法[15],将重组的pCAMBIA1301GWEAROs11g39000质粒经过电击法转入农杆菌,通过农杆菌侵染水稻粳稻品种Kitaake的愈伤组织,获得功能缺陷型转基因株系Ubi::EAROs11g39000.
1.3酵母双杂交
pGADT7Os11g39000重组质粒能够编码融合Os11g39000和GAL4激活域的蛋白,pGBKT7Os11g39000重组质粒能够编码融合Os11g39000和GAL4结合域的蛋白,将两个重组质粒按照酵母手册进行酵母转化,涂布于SD/Leu/Try平板上培养.28 ℃培养2~3 d,挑选4~5个单克隆用无菌水混匀后涂布于SD/Leu/Try/His平板上,分析结果.为了进一步检测相互作用的真实性,同时参照COLONTECH说明书进行滤纸显色实验.
1.4蛋白表达和随机结合位点筛选
根据Liu等[14]的蛋白诱导纯化方法,分别将pGEX4T1Os11g39000和pGEX4T1质粒转入大肠杆菌BL21(DE3),诱导表达.离心收集菌体,超声破碎,离心,谷胱甘肽琼脂糖珠(Ferments)亲合纯化蛋白,SDSPAGE检测.将已纯化的重组蛋白保存,用于后续随机结合片段筛选实验.
根据Blackwell的方法[16],使用引物RBSS4和CPDF4进行PCR扩增,得到双链随机DNA片段并与结合目的蛋白的谷胱甘肽琼脂糖珠室温孵育10 min.使用DNA结合缓冲液洗涤离心,重复5次,最后加入30 μL蛋白洗脱缓冲液,洗脱后离心取上清为模板,PCR扩增目的片段,正向引物为RBSS3,反向引物为CPDF4,琼脂糖凝胶电泳观察结果.重复6~8次,直到扩增出目的条带,然后将扩增片段克隆至pMD18T载体(TaKaRa),测序.测序结果使用在线分析工具分析(http://weblogo.berkeley.edu/).引物见表1.
1.5植物材料种植、激素处理及取材设置
参照Lin等[17]水稻种子催芽及种植方法,用3%次氯酸钠溶液对水稻种子表面消毒,37 ℃下种子吸胀处理48 h,28 ℃恒温培养箱催芽48 h.待种子露白后,用1∶1 000浓度的潮霉素(Hyg)溶液对种子进行初筛,然后将阳性植株转入1/2 MS培养液置于恒温培养箱中(28 ℃,光照16 h/黑暗8 h).
参照Song等[18]检测基因时空表达的取材方法,分别对营养生长期和生殖生长期的水稻材料进行取材.营养生长期的水稻材料取自温室生长20 d的野生型水稻日本晴,分别取根、茎和叶3部分;生殖生长期的水稻材料取自大田内灌浆时期的野生型水稻日本晴,分别取根、茎、叶、节间、叶鞘和幼穗等组织材料.液氮速冻,-80 ℃低温保存.
参照Li等[19]植物生长素诱导基因表达检测的方法, 将正常条件生长10 d的日本晴幼苗,转入含有10 μmol/L生长素的水培溶液中,分别处理0,1,3,6和12 h并取材,-80 ℃保存.设置生长素浓度梯度为0,10-8,10-7,10-6和10-5 mol/L分别处理野生型材料Kitaake和功能缺陷型转基因材料,处理7 d后观察表型并统计分析.
1.6实时荧光定量PCR分析
按照RNAeasy Mini Kit (TaKaRa)试剂盒说明提取总RNA,然后利用Maxima First Strand cDNA Synthesis Kit (TaKaRa)合成cDNA,用于实时定量PCR分析.利用Ferments 定量试剂盒(Ferments),Mx3000P仪器 (Stratagene)进行PCR分析.实时荧光定量PCR反应程序为:95 ℃,2 min;95 ℃,10 min;95 ℃,30 s;55 ℃,30 s;72 ℃,30 s;45个循环.实验重复3次,以持家基因OsACTIN1为内参,Mx3000P软件分析检测基因的相对表达水平.定量PCR引物见表2.
2结果
2.1生物信息学分析与基因克隆
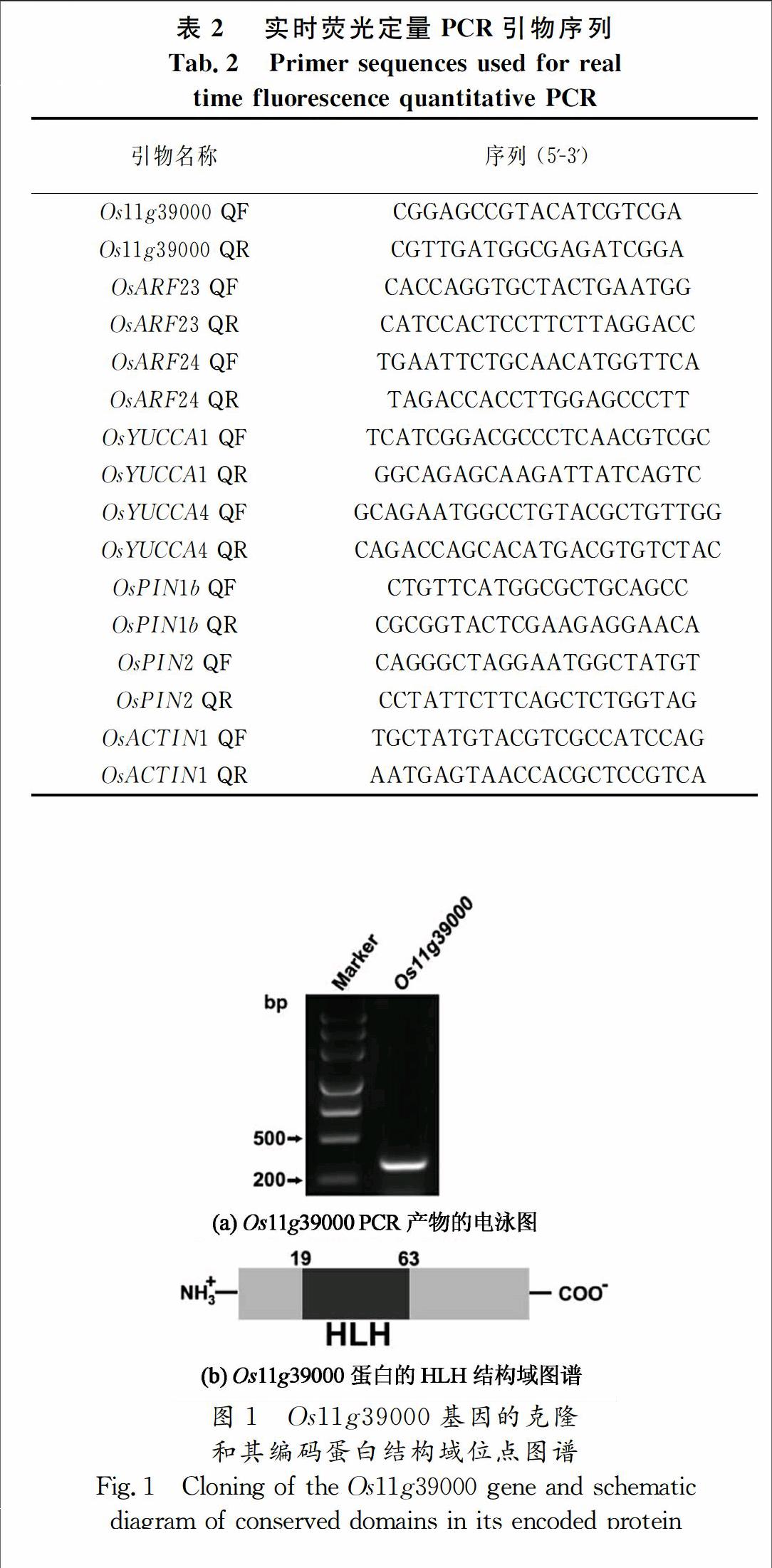
根据TIGR和NCBI数据库的序列信息,设计引物,以水稻cDNA为模板克隆出和预测分子量大小相似的DNA片段(图1(a)).测序发现该片段的序列和数据库中预测相同,命名为Os11g39000.该基因的编码序列含有306个核苷酸,编码含102个氨基酸序列的蛋白质.利用蛋白质结构域分析数据库SMART对Os11g39000蛋白结构进行分析,发现Os11g39000蛋白在第19~63个氨基酸区域包含一个HLH保守结构域(图1(b)),但是并没有结合DNA的basic区域,因此Os11g39000是一个非典型的bHLH蛋白,单个的Os11g39000蛋白不能够直接结合DNA片段.有研究表明bHLH蛋白可以通过HLH结构域形成同源或异源聚合体行使功能[7-8].因此推测该蛋白可能是以蛋白聚合体的形式行使功能.
2.2Os11g39000蛋白与自身的相互作用
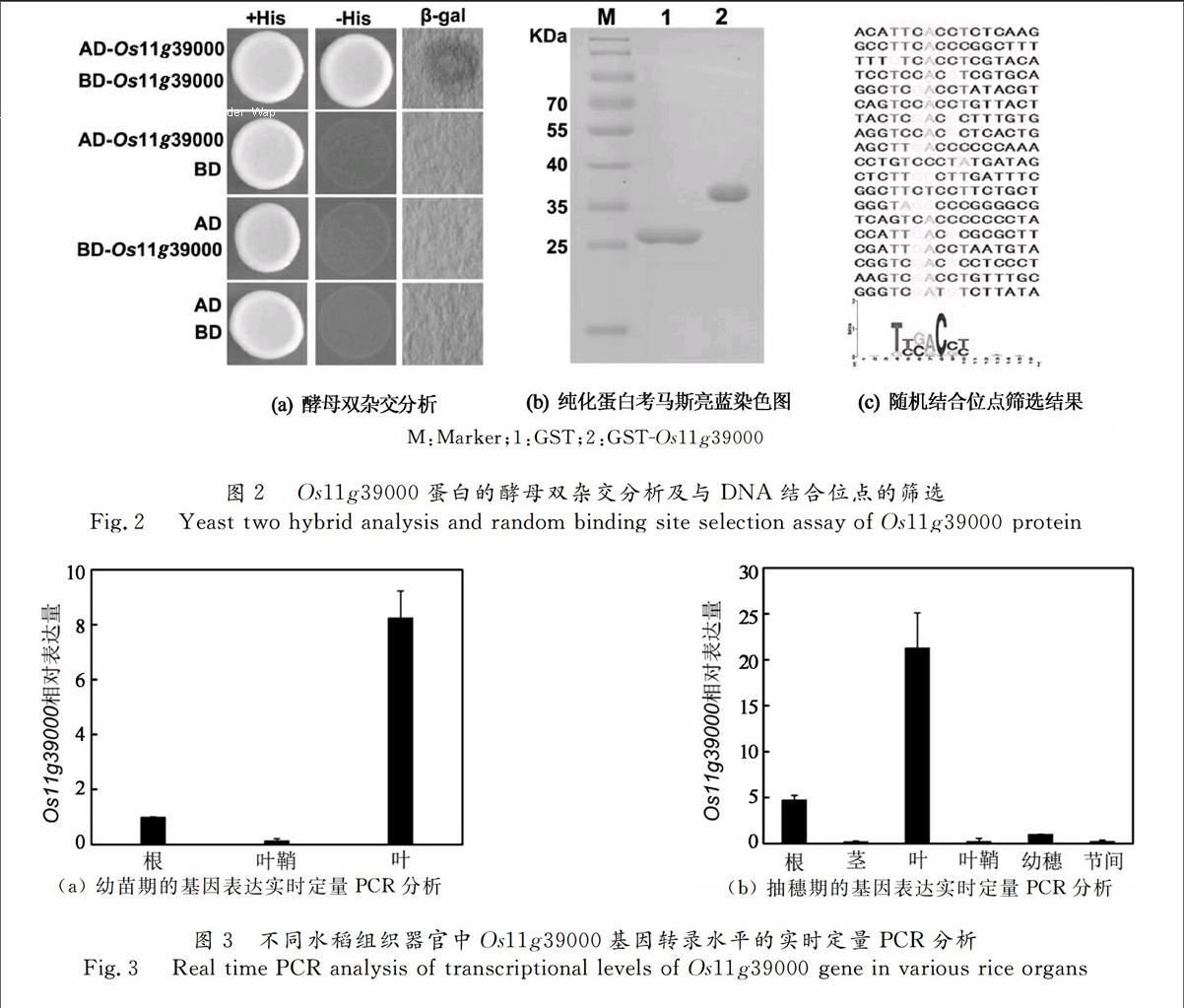
为了探究该编码蛋白能够形成同源蛋白聚合体,进行了酵母双杂交实验.首先构建了pGADT7Os11g39000重组质粒和pGBKT7Os11g39000重组质粒,然后将重组质粒共转入AH109酵母中,同时用共转pGADT7Os11g39000/pGBKT7,pGADT7/ pGBKT7Os11g39000和pGADT7/pGBKT7载体的酵母作为阴性对照,涂布于含有10 mmol/L 3AT的SD/Leu/Try固体平板,以及于含有10 mM 3AT的SD/Leu/Try/His固体平板.结果显示只有共转入pGADT7Os11g39000/ pGBKT7Os11g39000的酵母在含有10 mmol/L 3AT的SD/Leu/Try/His固体平板上继续生长.滤纸显色实验也发现只有共转入pGADT7Os11g39000和pGBKT7Os11g39000重组质粒的酵母可以显色(图2(a)),说明Os11g39000蛋白可以形成同源蛋白聚合体.
2.3Os11g39000蛋白的DNA随机结合位点筛选
酵母双杂交实验证明Os11g39000蛋白可以形成同源蛋白聚合体,为了进一步验证Os11g39000蛋白形成的同源蛋白聚合体是否可以与DNA直接相互作用,构建了pGEX4T1Os11g39000重组质粒.将重组质粒通过热激法转化入大肠杆菌BL21(DE3)并成功诱导纯化了GSTOs11g39000重组蛋白(图2(b)).DNA随机结合位点筛选实验.结果显示,该蛋白可以通过形成同源蛋白聚合体与DNA片段直接相互作用,且直接结合的DNA片段为TT/CG/CACC/GT/C(图2(c)).表明该蛋白可以通过HLH区域相互作用形成同源蛋白聚合体结合特定DNA片段,从而行使调控下游基因表达的功能.
2.4Os11g39000在水稻不同组织器官中的表达
基因的时空表达模式可以为预测和研究其生物学功能提供参考依据.实时定量PCR分析Os11g39000基因在水稻不同组织器官中的相对表达量,表明该基因在苗期和抽穗期都主要在叶和根表达,其它的组织器官表达量极少(图3(a),(b)),推测该基因可能参与植物根和叶的生长发育调控.
2.5转基因植物鉴定与表型分析
利用嵌合抑制因子基因沉默技术[14,20](chimeric repressor genesilencing technology CREST)及Gateway技术获得了pCAMBIA1301GWEAROs11g39000重组质粒(图4(a)).通过农杆菌侵染水稻Kitaake愈伤组织的方法,得到Os11g39000转录抑制型的转基因株系,即该转录因子的功能缺陷型转基因株系.通过DNA和RNA水平上的分子鉴定(图4(b),(c)),筛选出2个阳性功能缺陷型转基因株系Ubi::EAROs11g3900010和Ubi::EAROs11g3900011.水培实验表明,转基因株系Ubi::EAROs11g3900010和Ubi::EAROs11g3900011的根长比野生型的短,表现为明显的根部发育缺陷(图4(c),(d)).该结果表明,当抑制Os11g39000的转录活性时严重干扰了水稻根部的正常生长发育,说明Os11g39000基因参与了水稻根部发育的调控.
2.6生长素对Os11g39000的表达调控
研究表明,生长素对植物根部发育具有显著影响[21].利用Plant CARE数据库对Os11g39000基因起始密码子ATG上游1 500 bp的启动子序列进行分析,发现该基因启动子-792 bp处含有一个生长素应答元件(图5(a)),推测该基因功能可能与生长素有关.实时荧光定量PCR分析表明,随着生长素处理时间的延长,该基因表达量持续升高(图5(b)),表明该基因表达受到生长素诱导.同时,不同浓度生长素对野生型Kitaake和转基因株系Ubi::EAROs11g3900010和Ubi::EAROs11g3900011处理7 d发现,随着生长素浓度的升高,野生型根长受到明显抑制,而转基因株系根长没有明显变化(图5(c),(d)),表现为对生长素不敏感,即抑制转录因子Os11g39000的转录活性干扰了植物对生长素的响应,暗示该基因参与了生长素的相关途径.
为了进一步探究该基因对生长素生物合成、信号转导和极性运输途径的影响,选取这些途径中的相关基因,检测其表达量.结果表明,在功能缺陷型转基因株系中,生长素信号转导相关基因OsARF23,OsARF24和生长素合成相关基因OsYUCCA1,OsYUCCA4的表达量显著提高(图6(a),(b));生长素极性运输相关基因OsPIN1b和OsPIN2表达量没有明显变化(图6(c)).该结果说明,Os11g39000基因的功能缺陷主要影响了生长素的信号传导和生物合成途径,而对于生长素的极性运输并没有显著影响,推测Os11g39000参与了生长素合成与显著信号转导的调控.
3讨论
bHLH蛋白通常通过HLH区域相互作用形成同源或异源蛋白聚合体行使对下游基因的调控[8].本研究发现,Os11g39000蛋白不能直接结合DNA的basic区域(图1(b)),但Os11g39000蛋白可以通过形成同源蛋白聚合体来直接结合DNA片段,从而调控下游基因表达(图2(a)~(c)).同时通过DNA随机结合位点筛选实验分析了该基因的直接DNA结合位点TT/CG/CACC/GT/C(图2(c)),为深入研究Os11g39000蛋白的生理功能提供了重要线索.
分析基因在植物不同组织器官中的表达可以对该基因行使的功能进行预测[22].通过QPCR分析发现Os11g39000基因主要在水稻叶和根中表达(图3(a),(b)),暗示该基因可能参与植物叶和根的发育.水培实验发现,功能缺陷型转基因株系的根相对于野生型明显变短(图4(d),(e)),证实了该基因在根发育过程中起作用.但叶片中我们没有发现明显差异,对于该结果还有待进一步研究.
bHLH基因家族在动植物界广泛存在且行使多种多样的功能,已知 PIF3,PIF4,HFR1在光敏色素信号传导中起作用,SPT和ALC与雌蕊发育有关,AMS在花粉粒发育中起作用,AtMYC2与脱落酸诱导的基因表达有关[23].植物激素是调控植物生长发育的重要因子.生长素影响植物生长发育的各个方面,包括细胞分裂与生长,细胞分化,顶端优势,根的向地性等[24].本研究通过Plant CARE对Os11g39000基因的启动子顺式元件进行分析,结果显示该基因启动子区域存在生长素响应元件(图5(a)).Os11g39000基因的表达受到生长素诱导并且随着生长素处理时间的延长,该基因的表达量显著提高(图5(b)).不同浓度的生长素对转基因株系处理发现其根长对生长素不敏感(图5(c),(d)),说明该基因的表达受到生长素的调控.为了进一步研究该基因对生长素途径的影响,对功能缺陷型转基因株系中生长素的相关基因进行定量分析,发现与生长素信号转导和合成相关的基因表达量显著提高(图6(a),(b)),表明抑制转录因子Os11g39000的转录活性主要影响了生长素的信号转导和合成途径.这些结果表明,Os11g39000是生长素对植物调节途径中的相关基因,为进一步阐明Os11g39000的作用机理奠定了良好的基础.
参考文献
[1]MURRE C, MCCAW P S, BALTIMORE D. A new DNA binding and dimerization motif in immunoglobulin enhancer binding, daughterless, MyoD, and myc proteins[J]. Cell, 1989, 56(5): 777-783.
[2]VARAUD E, BRIOUDES F, SZECSI J, et al. AUXIN RESPONSE FACTOR8 regulates Arabidopsis petal growth by interacting with the bHLH transcription factor BIGPETALp[J]. Plant Cell, 2011, 23(3): 973-983.
[3]BERNHARDT C, ZHAO M, GONZALEZ A, et al. The bHLH genes GL3 and EGL3 participate in an intercellular regulatory circuit that controls cell patterning in the Arabidopsis root epidermis[J]. Development, 2005, 132(2): 291-298.
[4]DUEK P D, FANKHAUSER C. bHLH class transcription factors take centre stage in phytochrome signaling[J]. Trends Plant Sci, 2005, 10(2): 51-54.
[5]SERNA L. bHLH proteins know when to make a stoma[J]. Trends Plant Sci, 2007, 12(11): 483-485.
[6]ATCHLEY W R, TERHALLE W, DRESS A. Positional dependence, cliques, and predictive motifs in the bHLH protein domain[J]. J Mol Evol, 1999, 48(5): 501-516.
[7]ELLENBERGER T, FASS D, ARNAUD M, et al. Crystal structure of transcription factor E47: Ebox recognition by a basic region helixloophelix dimmer[J]. Genes Dev, 1994, 8(8): 970-980.
[8]NESI N, DEBEAUJON I, JOND C, et al. The TT8 gene encodes a basic helixloophelix domain protein required for expression of DFR and BAN genes in Arabidopsis siliques[J]. Plant Cell, 2000, 12(10): 1863-1878.
[9]SUN X H, COPELAND N G, JENKINS N A, et al. Id proteins Id1 and Id2 selectively inhibit DNA binding by one class of helixloophelix proteins[J]. Mol Cell Biol, 1991, 11(11): 5603-5611.
[10]RUZINOVA M B, BENEZRA R. Id proteins in development, cell cycle and cancer[J]. Trends Cell Biol, 2003, 13(8): 410-418.
[11]KEE B L. E and ID proteins branch out[J]. Nat Rev Immunol, 2009, 9(3): 175-184.
[12]HYUN Y, LEE I. KIDARI, encoding a nonDNA binding bHLH protein, represses light signal transduction in Arabidopsis thaliana[J]. Plant Mol Biol, 2006, 61(1/2): 283-296.
[13]ZHANG L Y, BAI M Y, WU J, et al. Antagonistic HLH/bHLH transcription factors mediate brassinosteroid regulation of cell elongation and plant development in rice and Arabidopsis [J]. Plant Cell, 2009, 21(12):3767-3780.
[14]LIU H, YU X, LI K, et al. Photoexcited CRY2 interacts with CIB1 to regulate transcription and floral initiation in Arabidopsis[J]. Science, 2008, 322(5907): 1535-1539.
[15]林建中,杨远柱,周波,等.一种简单高效的水稻体细胞诱变育种新方法[J]. 湖南大学学报:自然科学版,2013,40(9):79-85.
LIN Jianzhong, YANG Yuanzhu, ZHOU Bo, et al. A new and effective somatic mutagenesis rice breeding method[J]. Journal of Hunan University: Natural Sciences, 2013,40(9):79-85 .(In Chinese)
[16]BLACKWELL T K, WEINTRAUB H. Differences and similarities in DNAbinding preferences of MyoD and E2A protein complexes revealed by binding site selection[J]. Science, 1990, 250(4984): 1104-1110.
[17]LIN J, ZHOU B, YANG Y, et al. Piercing and vacuum infiltration of the mature embryo: a simplified method for agrobacteriummediated transformation of Indica rice[J]. Plant Cell Rep, 2009, 28(7): 1065-1074.
[18]SONG X Q, LIU LF, JIANG Y J, et al. Disruption of secondary wall cellulose biosynthesis alters cadmium translocation and tolerance in rice plants[J]. Mol Plant, 2013, 6(3): 768-780.
[19]LI Gang, LIANG Wanqi, ZHANG Xiaoqing, et al. Rice actinbinding protein RMD is a key link in the auxinactin regulatory loop that controls cell growth[J]. Pro Natl Acad Sci USA, 2014, 111(28): 10377-10382.
[20]HIRATSU K, MATSUI K, KOYAMA T, et al. Dominant repression of target genes by chimeric repressors that include the EAR motif, a repression domain in Arabidopsis[J]. Plant J, 2003, 34(5): 733-739.
[21]TANAKA H, DHONUKSHE P, BREWER P B, et al. Spatiotemporal asymmetric auxin distribution: a means to coordinate plant development[J]. Cell Mol Life Sci, 2006, 63(23): 2738-2754.
[22]QIN Y, LI X, GUO M, et al. Regulation of salt and ABA responses by CIPK14, a calcium sensor interacting protein kinase in Arabidopsis[J]. Sci China C Life Sci, 2008, 51(5): 391-401.
[23]TOLEDOORTIZ G, HUQ E, QUAIL P H. The Arabidopsis basic/helix loophelix transcription factor family[J]. Plant Cell, 2003, 15(8): 1749-1770.
[24]VANNESTE S, FRIML J. Auxin: a trigger for change in plant development[J]. Cell, 2009, 136(6): 1005-1016.

