结核分枝杆菌 Rv2986c T、B细胞表位分析
孙 妍,王心倩,占卫红,雷 航,陈高瞻,余晓丽
(武汉轻工大学 生物与制药工程学院,湖北 武汉 430023)
结核分枝杆菌 Rv2986c T、B细胞表位分析
孙妍,王心倩,占卫红,雷航,陈高瞻,余晓丽
(武汉轻工大学 生物与制药工程学院,湖北 武汉 430023)
预测并分析结核分枝杆菌Rv2986c的 CD4+T 、CD8+T 和B细胞表位分布情况。方法在NCBI数据库上查询并获得Rv2986c的氨基酸序列,用 BLAST分析其与结核分枝杆菌复合群及人类蛋白的同源性。利用NetMHC数据库预测并分析目的蛋白的CD4+T细胞表位的分布情况;利用SYFPEITHI、NetMHC、BIMAS和IEDB数据库预测并分析目的蛋白的CD8+T 细胞表位的分布情况。然后,综合CD4+T与CD8+T细胞表位预测情况,筛选出优势T细胞表位肽段。利用IEDB和ABCpred数据库分析目的蛋白的B细胞抗原表位,筛选出优势B细胞表位肽段。通过预测与分析,初步筛选出CD4+T细胞的强结合候选表位10个,弱结合候选表位245个,CD8+T 细胞候选表位3个;综合CD4+T与CD8+T细胞表位预测结果,最终筛选出1个优势T细胞表位肽段。综合IEDB和ABCpred数据库分析得出8条优势B细胞抗原表位。预测出来的T、B细胞候选表位将为结核病的特异性诊断及新的抗结核多表位疫苗的研究奠定基础。
结核分枝杆菌;Rv2986c;T细胞表位;B细胞表位
1 引言
结核病是一种严重危害人类身体健康的慢性传染病,由结核分枝杆菌(Mycobacteriumtuberculosis,MTB)感染所致[1]。据2015年WHO报[2],全球有960万新发结核病病例,其中包括48万耐药结核病(multiple drug resistant tuberculosis,MDR-TB)病例。我国2014年的新发肺结核人数为93万,仅次于印度(220万)和印度尼西亚(100万)而位居全球第三位。但是有研究发现,现在唯一用来预防结核病的疫苗——卡介苗(bacilluscalmette-guerinvaccine,BCG)只能预防婴儿和儿童肺外血源播散性结核[3],对成年人的保护作用效力不确定(0%—80%)[4],这说明有些人在接种卡介苗后仍然有患结核病的风险。除此之外,对多重耐药结核病而言其治疗失败率比一般敏感性结核病高出80倍[5],这表明多重耐药结核病在今后的治疗中存在无药可治的风险。面对如此严峻的结核病形势我们迫切的需要寻找一种能代替BCG来预防结核病感染的新型疫苗。
对传统疫苗来说,我们往往是将病原微生物或者完整的外源物质作为抗原来激发机体的免疫应答。但实际上仅仅只是外源物的一小段区域(抗原表位)在发挥免疫应答作用。由此可见在表位水平筛选特定的优势B细胞表位与T细胞表位,对设计开发新型表位疫苗和抗体,和对疾病的预防和诊断有重要意义。目前尚未有关于Rv2986c的研究报道,旨在分析Rv2986c作为免疫标志物对结核病的诊断检测方面作用的可行性。
2 材料与方法
2.1材料
通过NCBI数据库获得MTB的Rv2986c蛋白序列(Gene ID: 888166;214aa)。
2.2同源性分析
通过NCBI数据库中的BLAST(http://blast.ncbi.nlm.nih.gov/Blast.cgi)程序,将Rv2986c蛋白序列与结核分枝杆菌复合群蛋白序列和人类蛋白序列分别进行同源比对。
2.3CD4+T细胞表位预测
用在线软件NetMHCⅡpan3.1 Server(http://www.cbs.dtu.dk/services /NetMHC -IIpan/ )对目的蛋白的CD4+T细胞表位进行预测,设定表位多肽长度为15个氨基酸残基,选取经典人白细胞抗原II类基因(HLA-DP、HLA-DQ、HLA-DR,包括A、B两个基因座)中的HLA-DRB1-0101、0301、0401、0701、0802、0901、1101、1302、1501亚型为限定条件[6],表位多肽与等位基因的结合性能评价值设定为50 nm,预测结果以结合值由强到弱次序排列输出(强结合亲和值<50 nm ,50 nm<弱结合亲和值<500 nm )。
2.4CD8+T细胞表位预测
2.4.1SYFPEITHI对目的蛋白CD8+T细胞表位预测
用在线软件SYFPEITHI(http://www.syfpeithi.de/bin/MHCServer.dll/Epitope Predic-tion.htm)对目的蛋白的CD8+T细胞表位进行预测,选取经典人白细胞抗原I类基因(HLA-A、HLA-B、HLA-C)HLA- A* 02: 01和HLA- A* 03: 01等位基因作为限定条件,设定表位多肽长度为9个氨基酸残基,选取前2%的氨基酸表位进行分析[7]。
2.4.2NetMHC对目的蛋白CD8+T细胞表位预测
用在线软件NetMHC ( http://www.cbs.dtu.dk/service/NetMHC/)对目的蛋白的CD8+T细胞表位进行预测。选取人类可能性最大的HLA-A 02 :01(A2 )和HLA-A0301(A3 )等位基因作为预测表位肽段的限定条件[8],设定表位多肽长度为9个氨基酸残基,结果以结合值由强到弱次序排列输出(强结合亲和值<50 nm ,50 nm<弱结合亲和值<500 nm )。
2.4.3BIMAS对目的蛋白CD8+T细胞表位预测
用在线软件BIMAS ( http://www- bimas.cit.nih.gov/molbio /hla_bind / )对目的蛋白的蛋白的CD8+T细胞表位进行预测。选取HLA-A_0201及A3作为限定条件,设定表位多肽长度为9个氨基酸残基。
2.4.4IEBD对目的蛋白CD8+T细胞表位预测
用在线软件IEBD(http://tools.iedb.org/mhci/)对目的蛋白的CD8+T细胞表位进行预测。选取HLA-0201、HLA-0301亚型作为限定条件,设定表位多肽长度为9个氨基酸残基。选定亲和值为0—500 nm的序列进行分析。
2.5B细胞表位预测
2.5.1IEDB对目的蛋白B细胞表位预测
用在线软件IEBD(http://tools.immuneepitope.org/tools/bcell/iedb_input)对目的蛋白进行B细胞抗原表位预测。分别用IEBD提供的五种参数方案(亲水性方案、表面可及性方案、柔韧性方案、抗原性方案、二级结构预测方案)进行抗原表位预测。
2.5.2ABCpred对目的蛋白B细胞表位预测
在线软件ABCpred(http://www.imtech.res.in/raghava/abcpred/ABC _submis -sio n.html)是通过递归神经网络训练得到的分数对目的蛋白的B细胞表位进行预测。提交目的蛋白序列,设定临界值为0.51,选定表位肽段长度为16个氨基酸序列。
3 结果
3.1同源性分析结果
通过同源比对发现目的蛋白与MTB复合群(人型MTB、牛型MTB、非洲分枝杆菌和田鼠分枝杆菌和减毒牛分枝杆菌菌株,BCG)的同源性均为100% ,与非MTB的同源性有所降低(80%—60%,平均70% ),与其他细菌的同源性更低(最高80%,平均65%),抗原序列与人类蛋白无同源性,该蛋白具有特异性抗原的潜质,并不会出现与人类自身蛋白交叉感染的情况,具有较高的安全性[9]。
3.2CD4+T细胞表位预测结果
利用在线数据库NetMHCⅡpan Server对目的蛋白表位进行预测,预测结果见表1。从预测结果发现,不同等位基因所结合的短肽数目相差比较显著。其中目的蛋白仅在DRB1-0101和DRB1-1101等位基因处有强结合位点,分别为7个和3个。预测结果中,9个HLA-DRB1等位基因处均有弱结合位点,其中DRB1-1101结合位点最多,DRB1-1302结合位点最少。与CD8+T 细胞表位预测方法相比,CD4+T细胞表位预测更为复杂一些,NetMHCⅡpan Server是目前预测效果较好的在线工具。将NetMHCⅡpan Server 预测出来的15个氨基酸的表位肽与CD8+T细胞的表位肽综合起来分析才能找到更可靠的表位肽段,然后再合成验证。
3.3CD8+T细胞表位预测结果
综合4个不同的在线预测工具( SYFPEITHI、NetMHC、BIMAS和IEBD)对目的蛋白的CD8+T细胞表位进行预测,结果见表2。综合4种工具预测出来结合能力都较强的九肽共3条,其表位氨基酸序列起始位点为94—102、105—113和113—121。
表1Rv2986c抗原CD4+T 细胞表位分布情况
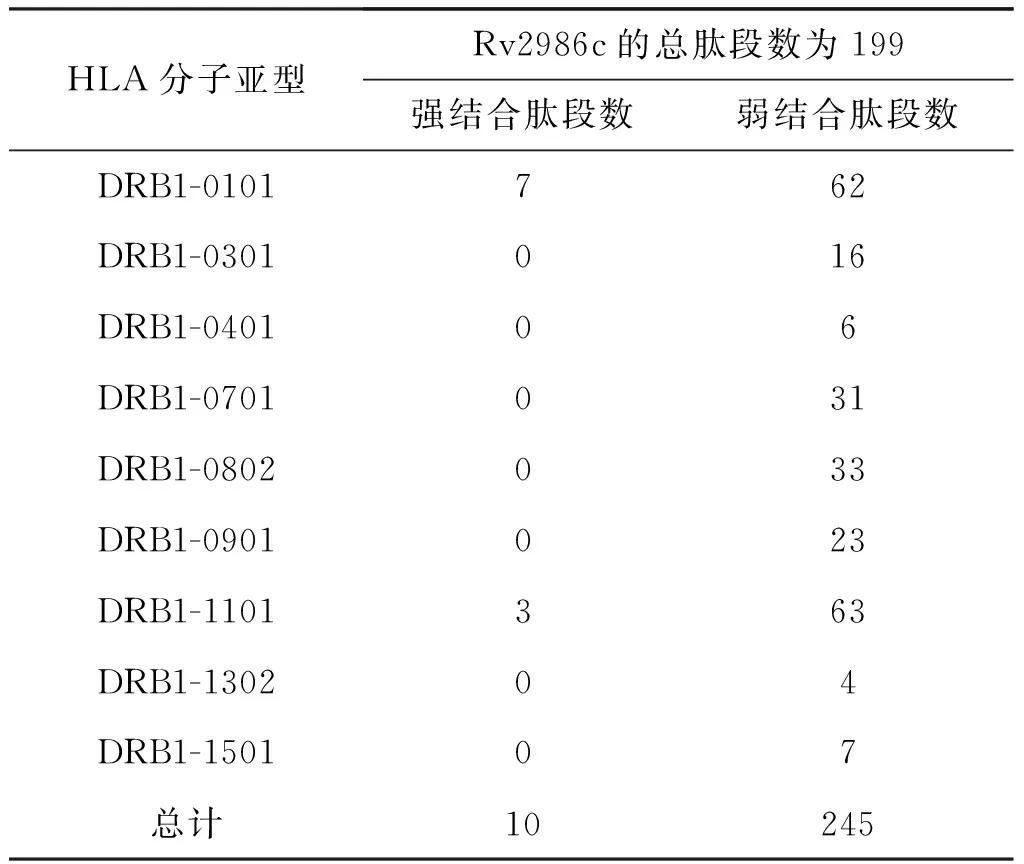
HLA分子亚型Rv2986c的总肽段数为199强结合肽段数弱结合肽段数DRB1-0101762DRB1-0301016DRB1-040106DRB1-0701031DRB1-0802033DRB1-0901023DRB1-1101363DRB1-130204DRB1-150107总计10245
表2Rv2986c抗原CD8+T 细胞表位的综合分析结果
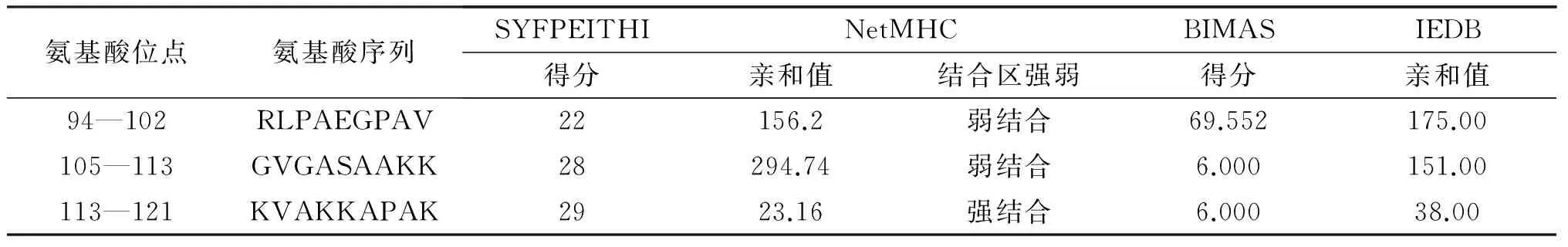
氨基酸位点氨基酸序列SYFPEITHINetMHCBIMASIEDB得分亲和值结合区强弱得分亲和值94—102RLPAEGPAV22156.2弱结合69.552175.00105—113GVGASAAKK28294.74弱结合6.000151.00113—121KVAKKAPAK2923.16强结合6.00038.00
注:亲和值越低表明结合能力越强;得分越高表示结合能力越强。
3.4CD4+T和CD8+T细胞表位综合分析结果
将在线软件预测得到的CD4+T和CD8+T细胞表位进行整理分析。将预测得到的8条CD4+T细胞抗原和3条CD8+T细胞抗原综合分析根据CD4+T细胞表位15肽覆盖CD8+T细胞表位的9肽筛选出共同的优势肽段(表3)。通过综合分析共得到1条优势抗原肽段,其起始位点为105—113,其序列为GVGASAAKK。
表3Rv2986c抗原CD4+T和CD8+T表位综合分析结果
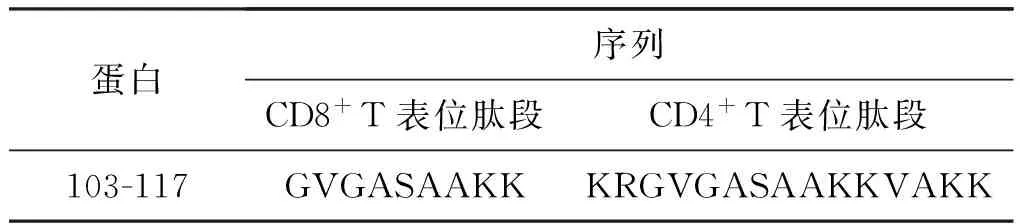
蛋白序列CD8+T表位肽段CD4+T表位肽段103-117GVGASAAKKKRGVGASAAKKVAKK
注:表中划线部分即为优势抗原表位。
3.5B细胞表位预测结果
综合两种在线软件分析共筛选出8条优势B细胞抗原表位,见表4。由于B细胞处理抗原时不需要抗原提呈细胞(APC)处理,其抗原表位为构象型表位具有一定的空间构象,B抗原表位是由那些空间上邻近但实际上不连续的氨基酸组成,因此其表位肽段长度不一。
表4B细胞优势表位
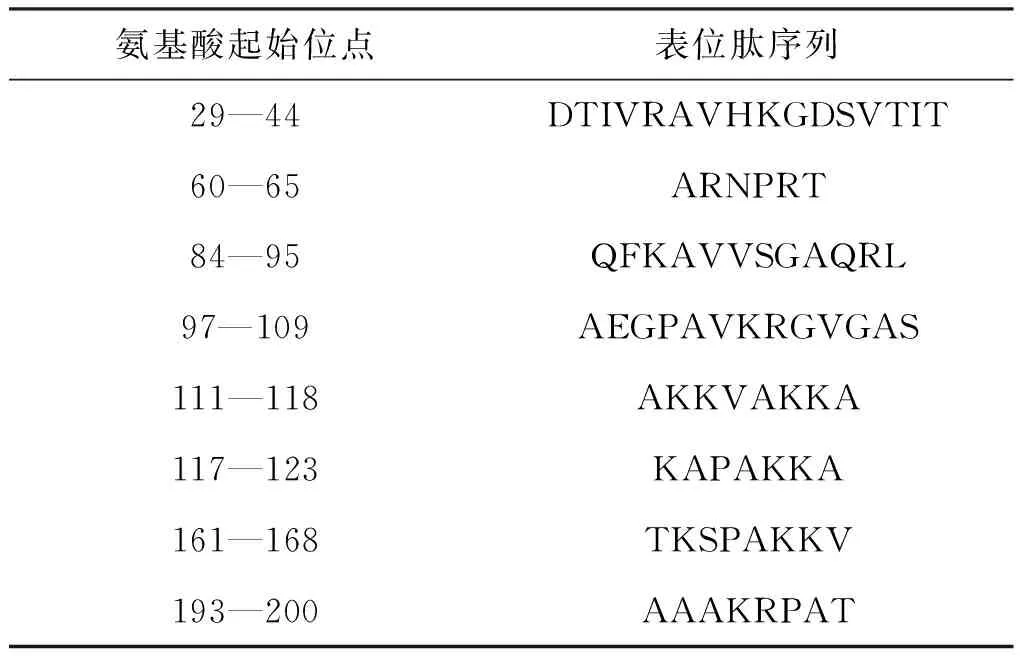
氨基酸起始位点表位肽序列29—44DTIVRAVHKGDSVTIT60—65ARNPRT84—95QFKAVVSGAQRL97—109AEGPAVKRGVGAS111—118AKKVAKKA117—123KAPAKKA161—168TKSPAKKV193—200AAAKRPAT
4 讨论
研究表明人体在对抗结核分枝杆菌的过程中主要依赖于特异性免疫和非特异性免疫反应[10-12]。但是结核分枝杆菌与其他革兰氏阳性菌不同,存在宿主逃逸现象[13]。这给我们利用结核分枝杆菌自身抗原来作为免疫标志物诱导其产生特异性免疫反应以及新型疫苗的开发带来挑战。在机体特异性免疫应答过程中主要是T、B两类淋巴细胞参与免疫应答,因此对抗原T、B细胞表位的分析筛选显得尤为重要。
具体来说,CD4+T细胞结合MHC-I类分子提呈的抗原表位,有辅助型和调节型两类,主要产生IFN-γ,IL-2,IL-12,TNF-β/α这几类细胞因子[14],研究发现CD4+T细胞产生的IFN-γ不仅在控制结核分枝杆菌感染也在诱导强大CD8+T细胞方面发挥作用[15]。CD8+T细胞结合MHC-II类分子提呈的抗原表位,主要产生IFN-γ和TNF-α等细胞因子[16]。有研究报道,由于基因缺失无CD8+T细胞功能的小鼠更容易感染结核分枝杆菌[17]。且有研究发现少量的记忆CD8+T细胞也能发挥强大的免疫作用,分泌IFN-γ的CD8+T细胞在对抗结核分枝杆菌的过程中发挥重要作用[18]。B细胞表面的抗原表位能识别和辅助呈递抗原,且其在免疫过程中能产生抗体杀灭细菌。
本研究利用生物信息学方法,通过表位分析数据库对MTB的Rv2986c蛋白抗原的CD4+T、CD8+T细胞以及B细胞进行表位预测分析,找出与等位基因结合比较强的优势肽段。通过对比4种在线预测工具预测的结果,得出了抗原指数和表位较强的优势抗原集中区103—117。通过B细胞抗原表位分析筛选出了8条优势抗原表位。通过预测结果说明用Rv2986c蛋白作为抗体可以刺激机体产生免疫应答,可作为免疫标志物用于结核病的诊断,也可以与现有已知的诊断抗原组合使用,从而提高对结核病患者的检测敏感性。
[1]张贺秋,赵雁林.现代结核病诊断技术[M].北京:人民卫生出版社. 2013:5-6.
[2]MarioRaviglione. Global tuberculosis report 2015[J].Global Tuberculosis Report,2015.
[3]Trunz BB,Fine P,Dye C.Effect of BCG vaccination on childhood tuberculous meningitis and miliary tuberculosis worldwide:a meta-analysis and assessment of cost- effectiveness[J].Lancet, 2006,367( 9517):1173-1180.
[4]Black GF,Weir RE,Floyd S,et al.BCG- induced increase in interferon- gamma response to mycobacterial antigens and efficacy of BCG vaccination in Malawi and the UK:tworandomisedcontrolledstudies[J].Lancet, 2002,359( 9315):1393-1401.
[5]沈洪波,王洪海. 治疗性结核病疫苗研究进展[J]. 微生物与感染,2010(02):111-116.
[6]叶娟,张舒林,刘文第. 结核分枝杆菌RD12区T细胞表位分布情况预测及分析[J]. 上海交通大学学报(医学版),2014,01:7-12.
[7]吴祖建,高芳銮,沈建国.生物信息学分析实践[M].北京:科学出版社, 2010:147-148.
[8]Gordon SV,BroschR,Billault A,et al.Identification of variable regions in the genomes of tubercle bacilli using bacterial artificial chromosome arrays[J].MolMicrobiol, 1999, 32( 3) : 643-655.
[9]Mommaas B, Kamp J, Drijfhout JW, et al. Identification of a novelHLA-B60-res-ricted T cell epitope of the minor histocompatibilityantigen HA-1 locus[J]. J Immunol, 2002, 169(6):3131-3136.
[10]Kashion S S,Vallerskog T, Martens G, et al. Initiation of acquired immunity in the lungs of mice lacking lymph nodes after infection with cersolizedMycobacterium tuberculosis[J].Am J Pathol,2010,176(1):198-204.
[11]Van Crevel R,Ottenhoff T H,Van der Meer J W, Innate immunity to Mycobacterium tuberculosis[J].ClinMicrobiol Rev,2002,15(2):294-309.
[12]LeuilletC,Martinon F, Perez C, et al.Mycobacteriumtuberculosissubverts innate immunity to evade specific effectors[J].J Immunol,2006,177(9):6245-6255.
[13]Cambier C J, Stanley Falkow, LatitaRamakrishnam. Host evasion and exploitation schemes of Mycobacterium tuberculosis[J].Cell.2014,159:1497-1509.
[14]卢佳丽,杨晓明. 结核分枝杆菌的免疫学研究进展[J]. 微生物学免疫学进展,2010(01):74-78.
[15]Flynn JL. Lessons from experimental Mycobacterium tuberculosis infections[J]. Microbes Infect, 2006, 8( 4):1179-1188.
[16]董毅,吴利先. T细胞免疫在抗结核杆菌感染中的研究进展[J]. 现代生物医学进展,2014,18:3593-3595.
[17]OGarra A, Redford P S, McNab F W, et al. The Immune Response in Tuberculosis [J].Annu Rev Immunol, 2013,31:475-527.
[18]Sud D, Bigbee C, Flynn JL, et al.Contribution of CD8+ T cells tocontrol of myc -obacterium tuberculosis infection [J].The Journal ofImmunology, 2006,176(7):4296- 4314.
Prediction and analysis of T,B cell epitopes’s distribution of theMycobacteriumtuberculosisRv2986c
SUNYan,WANGXin-qian,ZHANWei-hong,LEIHang,ChENGao-zhan,YUXiao-li
(School of Biology and Pharmaceutical Engineering, Wuhan Polytechnic University, Wuhan 430023, China)
To predict and analyze the distribution of CD4+T cell CD8+T-cell and B-cell epitopes encoded by the Rv2986c of Mycobacterium tuberculosis. Obtaining the amino acid sequences by online NCBI database and analyzing the homology between Mycobacterium tuberculosis complex and human proteins by BLAST.NetMHC databases was used to predict CD4+Tcell epitopes.Pre- dicting the distribution of CD8+T cell epitopes by SYFPEITH- I, NetMHC,BIMASand IEDB dat-abases.IEDB and ABCpreddatabases was used to predict Bcell epitopes. After the prediction,we can select the superior epitopes . For the results,there are 10 strong and 245 weak candidates CD4+T cell epitopes candidates3 CD8+T cell epitopes candidates. In the end,1 superiorT-cell epitopecan be selected. 8 superior B-cell epitopes can be selected .Prediction of T and B cell epitopes will be the basis diagnosis of tuberculosis and the study of new tuberculosis vaccine.
Mycobacteriumtuberculosis; Rv2986c;T-cell epitope; B-cell epitope
2016-03-20.
孙妍(1992-),女,硕士研究生,E-mail:1928125710@qq.com.
余晓丽(1963-),女,教授,E-mail:yxll268@126.com.
武汉轻工大学研究生创新基金项目(2014cx019).
2095-7386(2016)03-0045-04
10.3969/j.issn.2095-7386.2016.03.008
Q 93
A

