食源性金黄色葡萄球菌粘附素基因的检测及蛋白酶K和DNaseⅠ对菌株被膜形成的影响
马伊萨兰,余博旸,陈 娟,刘 骥,唐俊妮,*
(1.西南民族大学生命科学与技术学院,四川成都 610041;2.成都七中,四川成都 610041)
食源性金黄色葡萄球菌粘附素基因的检测及蛋白酶K和DNaseⅠ对菌株被膜形成的影响
马伊萨兰1,余博旸2,陈娟1,刘骥1,唐俊妮1,*
(1.西南民族大学生命科学与技术学院,四川成都 610041;2.成都七中,四川成都 610041)
本研究以实验室保存的17株金黄色葡萄球菌食品分离菌株为研究对象,通过提取细菌DNA,采用PCR检测14种粘附素基因的分布情况,随后采用试管法和微孔板法对细菌成膜能力进行检测,进一步研究蛋白酶K和DNase Ⅰ对生物被膜形成能力的影响。结果表明:17株菌株均携带clfB基因,检出率为100%,均不携带bap,bbp和clfA基因,有13株菌扩增出icaBC基因(76.5%),12株均扩增出cna基因(70.6%),11株菌扩增出eno基因(64.7%),10株菌分别扩增出fib和icaAD基因(58.8%),9株菌扩增出fnbA基因(52.9%),4株菌分别扩增出sasC,sasG和ebpS基因(23.5%),2株菌扩增出fnbB基因(11.8%)。试管法和微孔板法观察到17株菌株均有生物被膜形成,但不同菌株的被膜形成能力有差异,10号和30号菌株被膜形成能力显著强于其它15株菌(p<0.05)。在细菌生长过程中,蛋白酶K、DNase Ⅰ以及两种酶的联合处理发现蛋白酶K和DNase Ⅰ处理组细菌生物被膜形成能力明显低于对照未处理组(p<0.05),并且蛋白酶K对金黄色葡萄球菌生物被膜形成的抑制效果比DNase Ⅰ好,但是两种酶联合处理效果与对照组差异不显著(p>0.05)。本研究旨在为食源性金黄色葡萄球菌生物被膜的形成与控制策略提供基础数据。
食源性金黄色葡萄球菌,生物被膜形成,粘附素相关基因,蛋白酶K,DNaseⅠ
细菌生物被膜是细菌细胞大量附着在固体和活的生物体表面形成的三维结构聚合物[1]。经常引起医院感染和食物中毒的金黄色葡萄球菌具有形成生物被膜的能力,研究表明金黄色葡萄球菌生物被膜结构复杂,主要是由β-1,6-聚-N-乙酰葡萄糖胺(PNAG)或黏附多糖(PIA)[2]、胞外DNA[3-4]、胞外蛋白质、细胞壁磷壁酸[5]、脂质[6]和其它环境中的物质等一系列复杂生物大分子包裹细菌细胞聚集而成。作为病原菌,金黄色葡萄球菌通常通过能够粘附到生物材料表面(如细胞外基质组成成分或者宿主组织),或者非生物材料表面(如医疗器械装置等),一旦粘附上去细菌就开始增殖扩散,并且通过形成生物被膜的方式定植在生物材料或非生物材料表面[7]。生物被膜的形成有助于增加细菌抵御抗生素和逃脱寄主免疫攻击的能力造成持续性感染和疾病的难以治疗,也增加了食品加工过程的污染频率[8]。目前的研究认为,在金黄色葡萄球菌生物被膜的形成过程中,一些菌株依赖于细胞分泌的胞外粘附多糖(PIA/PNAG)形成生物被膜,这种类型主要是由icaADBC操纵基因编码产物进行调控[9]。还有一些菌株不依赖于PIA/PNAG进行粘附,这类金黄色葡萄球菌生物被膜的形成主要是通过粘附基质分子(粘附基质分子是一类具有保守结构和功能特征相似,在生物膜形成和发展中起着重要作用的细胞表面锚定蛋白以及胞外分泌蛋白的总称[10])的共价结合而引起细胞相互聚集。金黄色葡萄球菌全基因组中大约含有几十个编码表面锚定粘附基质分子的相关基因[11-15]。如由fnbPA,fnbPB,bap,sasC和sasG基因编码的表面粘附因子FnBPA和FnBPB[11],Bap[12],SasC和SasG[13-14]等在金黄色葡萄球菌生物被膜形成作用已被证实。另外,Atshan等[15]还发现另一些金黄色葡萄球菌菌株是由其自身分泌的基质粘附分子与宿主细胞的配体分子相互作用而进行粘附,这些基质粘附分子包括弹力蛋白结合蛋白EbpS,层粘连蛋白结合蛋白Eno,胶原结合蛋白Cna,纤维蛋白原结合蛋白Fib,纤维蛋白原结合蛋白ClfA和ClfB,骨唾液酸蛋白结合蛋白Bbp等,它们分别由ebpS,eno,cna,fib,clfA,clfB和bbp基因进行编码。这些研究反映了金黄色葡萄球菌粘附基质分子在介导粘附和生物被膜形成发展中的重要性。
针对细菌生物被膜形成带来的危害,学者们也在不断探索抑制细菌生物被膜形成的方法,目前这方面的研究主要集中在消杀剂及酶对生物被膜形成的影响[16-18]。为了进一步了解食源性金黄色葡萄球菌生物被膜形成以及相关粘附素基因的分布,本文以实验室保存的17株食品分离菌株为研究对象,探索不同粘附素相关基因bap,bbp,cna,clfA,clfB,ebpS,eno,fib,fnbPA,fnbPB,sasC,sasG,以及icaAD和icaBC基因在食源性菌株中的携带情况,同时,研究蛋白酶K和DNAaseⅠ对食源性金黄色葡萄球菌生物被膜形成的影响,旨在为食源性金黄色葡萄球菌生物被膜的形成与控制策略提供基础数据。
1 材料与方法
1.1材料与仪器
菌株来源:实验室保存的17株经分离鉴定的食品源金黄色葡萄球菌(17株菌株中均未检测出传统肠毒素基因型sea-see,其中3,14,23,26,30号菌株检测出sek基因,47,48,49,57,67号菌株检测出sei基因)。胰蛋白胨大豆肉汤培养基(TSB)、Baird-Parker(BP)琼脂基础青岛海博生物技术有限公司;酵母提取物、胰化蛋白胨英国OXOID公司;蛋白酶K(10 mg/mL)、DNase1(1 mg/mL)大连宝生生物工程有限公司;分析纯的草酸铵、甲醇、冰乙酸成都市科龙化工试剂厂;结晶紫Amresco公司。
Eppendorf 5804R型冷冻离心机Eppendorf中国有限公司;Bio-Rad PTC-200 PCR仪购自Bio-Rad公司;Elx808酶标仪日本BIO-TEK公司;DYY-6C型电泳仪北京六一仪器厂;DL-720E智能超声波振清洗器上海之信仪器有限公司;GHP-9270隔水式恒温培养箱上海齐欣科学仪器有限公司。
1.2实验方法
1.2.1金黄色葡萄球菌DNA提取将17株实验室保存的金黄色葡萄球菌食品分离菌株按照1%的比例接种于TSB培养基中,37 ℃,转速150 r/min培养18 h。取1 mL菌悬液4 ℃条件下12000 r/min离心2 min后,收集菌体,提取细菌DNA方法参考我们之前发表的文献[19]进行,提取好的样本DNA于-20 ℃保存,用于后续基因检测。
1.2.2金黄色葡萄球菌生物被膜形成相关基因检测生物被膜形成相关基因的引物参考文献[20],由生工生物工程(上海)有限公司合成。PCR的反应体系:10×PCR buffer(Mg2+free)2 μL,25 mmol/L MgC121.6 μL,dNTPs(1.0 mmol/L)1.2 μL,Taq DNA聚合酶(5 unit/μL)0.3 μL,20 μmol/L的上下游引物各0.5 μL,模板1 μL,无菌超纯水补足20 μL体系。PCR扩增程序:95 ℃预变性5 min,再按95 ℃变性40 s,退火50 s(各基因退火温度见参考文献[20]),72 ℃延伸40 s,进行35个循环,最后72 ℃延伸10 min,4 ℃保存。PCR产物琼脂糖电泳检测:配置1%琼脂糖凝胶,取5 μL PCR扩增产物上样,80 V电泳45 min,凝胶成像系统观察与储存电泳图片。每一种基因的PCR阳性产物送至上海生工生物工程技术服务公司进行测序和比对,以保证每对引物扩增产物的真实性。
1.2.3试管法和微孔板法检测菌株生物被膜的形成将实验室保存的17株金黄色葡萄球菌食品分离菌株在BP平板上纯化,挑取单菌落接种于TSB液体培养基中,37 ℃,150 r/min过夜培养,作为原始接种菌液。试管法和微孔板法均采用我们之前发表的文献描述的具体步骤进行操作[21]。其中,96孔板法中阈值(ODc)的定义为阴性控制吸光值的2倍。判定标准是根据三次重复所测得的吸光值,将金黄色葡萄球菌生物被膜形成能力分为不形成生物被膜菌株(OD≤ODc),生物被膜弱形成菌株(ODc≤OD≤2×ODc),以及生物被膜强形成菌株(2×ODc≤OD)三类。
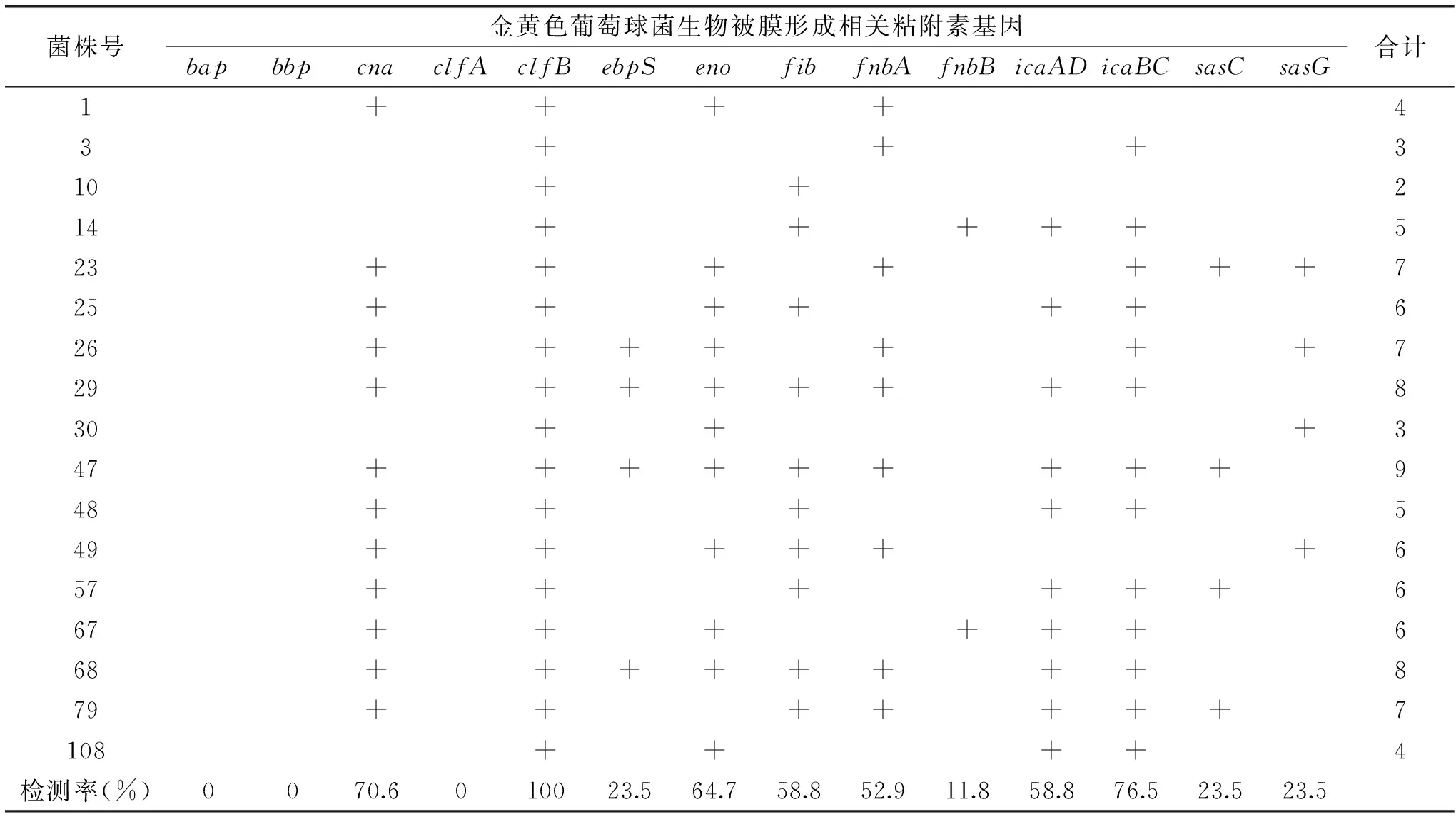
表1 食源性金黄色葡萄球菌粘附素基因携带情况
1.2.4添加蛋白酶K和DNase Ⅰ对金黄色葡萄球菌生物被膜形成影响根据试管法和微孔板法的生物被膜实验结果筛选出2株强生物被膜形成菌株,用于探索蛋白酶K和DNase Ⅰ单独使用以及两种酶联合使用分别对金黄色葡萄球菌生物被膜形成能力的影响。具体操作如下:将培养过夜的2株菌分别接种于无菌的聚苯乙烯96 孔板中,每孔中加入100 μL的TSB培养基,10 μL(约106CFU)过夜培养的新鲜菌液,1 μL的酶(蛋白酶K或DNase Ⅰ或两种酶联合使用),37 ℃培养,观察细菌生长的不同时间点(4、8、12、16、20、24、28、32 h)两种酶对被膜形成影响以及随时间变化趋势,每一组别都要做三次平行,同时设置对照组。
1.2.5数据处理和分析96孔板测得的三个平行数据,均采用Excel软件初步处理,进一步采用SPSS 18.0数据分析软件处理,组内比较采用Duncan方法,以p<0.05为差异具有统计意义的标准。
2 结果与分析
2.1食源性金黄色葡萄球菌粘附素基因的分布情况
以提取的17株金黄色葡萄球菌食品分离菌株DNA作为模版,对14种粘附素基因(bap,bbp,cna,clfA,clfB,ebpS,eno,fib,fnbA,fnbB,icaAD,icaBC,sasC,sasG)进行PCR检测,检测结果详见表1。从表1可以看出,17株菌株均携带clfB基因,检出率为100%,均不携带bap,bbp和clfA基因。有13株菌扩增出icaBC基因(76.5%),12株均扩增出cna基因(70.6%),11株菌扩增出eno基因(64.7%),10株菌分别扩增出fib和icaAD基因(58.8%),9株菌扩增出fnbA基因(52.9%),4株菌分别扩增出sasC,sasG和ebps基因(23.5%),2株菌扩增出fnbB基因(11.8%)。从各菌株自身携带的基因来看,47号菌株携带9种检测的粘附素基因,29和68号携带8种,23、26和79携带7种,25、49、57和67号携带6种,14和48号携带5种,1和108号菌株携带4种,3和30号携带3种,10号携带2种。
2.2试管法和微孔板检测生物被膜形成结果
根据试管法的结果,肉眼可以观察到17株菌株在玻璃试管壁上均有生物被膜形成(详见图1),但不同菌株的被膜形成能力有差异,其中,10、26、30、48、49、57、67和79号菌株能够在试管上形成明显的被膜,结晶紫染色特别清晰可见,见图1。根据96孔板的判定标准,从图1中可以看出1、23和25号菌株为弱生物被膜形成菌株,3、10、14、26、29、30、47、48、49、57、67、68、79和108号菌株均为强生物被膜形成菌株。但是通过Duncan分析17株菌株在微孔板上生物被膜形成能力的数据发现,菌株10和30被膜形成能力显著强于其它15株菌(p<0.05),这两株菌株不论是在试管上,还是微孔板上都表现出很强的生物被膜形成能力。因此,选取10和30号菌株进行后续的酶处理。
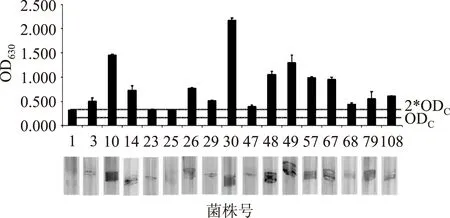
图1 微孔板和试管法分析17株金黄色葡萄球菌分离菌株的生物被膜形成情况Fig.1 The results of biofilm formation ability for 17 S. aureus isolates by tube test and microtiter-plate test
2.3蛋白酶K和DNase Ⅰ处理对细菌生物被膜形成能力的影响
结合试管法与96孔板法的结果,挑选10号与30号两株菌株进行蛋白酶K、DNase Ⅰ以及两种酶的联合处理。两种酶对两株菌生物被膜形成影响以及随时间的变化趋势见图2。其中,图2A为10号菌株酶处理与对照组的分析结果,2B为30号菌株酶处理与对照组的分析结果。由图2中的数据和变化趋势,可以明确得出蛋白酶K和DNase I处理组的生物被膜形成能力明显低于对照未处理组(p<0.05),并且蛋白酶K对金黄色葡萄球菌生物被膜形成抑制效果比DNase Ⅰ好。但是两种酶联合处理效果反而不好,与对照组比较,差异不显著(p>0.05),详见图2。
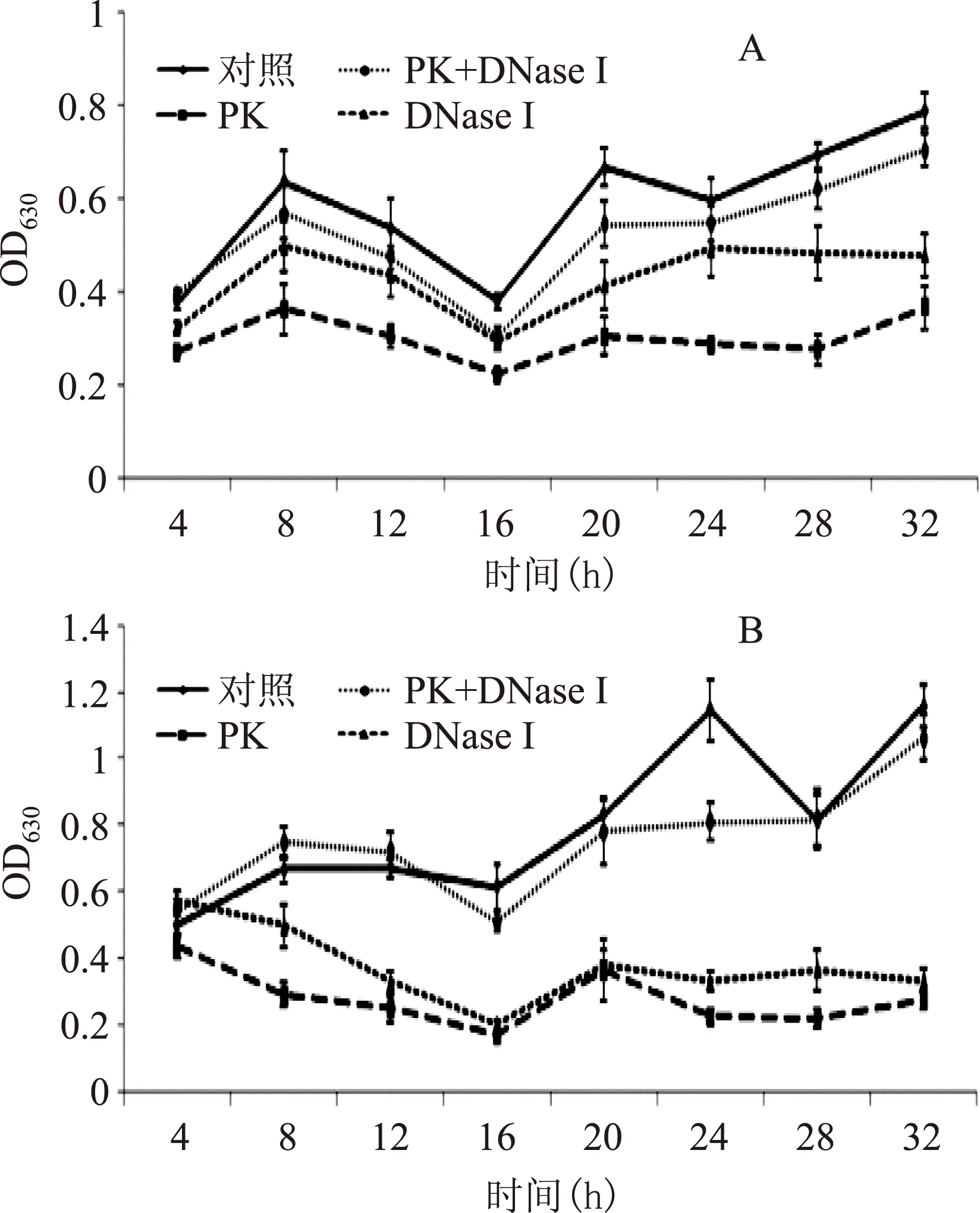
图2 蛋白酶K和DNase Ⅰ对金黄色葡萄球菌食品分离菌株的生物被膜形成影响Fig.2 The effect of Proteinase K(PK)and DNase I on the biofilm formation of S. aureus food isolates注:A为10号菌株;B为30号菌株。
3 结论与讨论
从九十年代末,学者们就开始关注到食源性病原细菌可粘附到食品和食品接触器具表面引发严重的卫生问题和经济损失,导致食物的污染和腐败变质,其原因就是细菌生物被膜的出现[22]。而对于金黄色葡萄球菌生物被膜形成的研究始于医院病人体内的慢性感染[8]。从医院感染病人体内最频繁分离到的生物被膜形成菌株是葡萄球菌属,其中,金黄色葡萄球菌可占到22%~39%,凝固酶阴性葡萄球菌占到15%~37.5%[23]。在食品行业,金黄色葡萄球菌肠毒素引起的食源性疾病一直以来备受关注,但是随着研究的深入,这些食品分离菌株生物被膜形成以及它们对食品安全造成的隐患也引起了重视。由于食品介质表面的类型不同,各种不同食品分离菌株在生物被膜形成能力和粘附素基因的分布上可能存在差异。因此,本研究以实验室保存的17株食品分离菌株为研究对象,这些菌株全部来源于菜市场的凉拌熟食和肉类,对这些菌株生物被膜形成相关基因的检测以及生物被膜形成影响的研究旨在为细菌生物被膜的控制策略提供部分基础数据。
学者们从葡萄球菌属医院分离株和动物源分离株中识别出不同的粘附基质分子,发现不同来源的菌株中粘附基质分子的基因携带情况差异较大[24-27]。本研究通过对食品金黄色葡萄球菌分离菌株进行14种生物被膜形成相关基因的检测,结果发现17株食品分离菌株中clfB基因检出率最高,为100%,其次是icaBC基因76.5%,cna基因70.6%,eno基因64.7%,fib和icaAD基因分别为58.8%,fnbA基因52.9%,sasC,sasG和ebps基因分别为23.5%,fnbB基因11.8%,所有菌株均未检测出bap,bbp和clfA基因。Xu等[28]的研究发现28株牛奶源金黄色葡萄球菌分离菌株中eno基因的携带率最高,为100%,其次是fib基因为92.9%,clfA基因为89.3%和clfB基因为85.7%,由于他们检测的粘附素基因数量种类少,研究结果间存在一定的差异,不一致的原因可能是菌株来源不同。本次检测菌株均来源于食品,这些菌株肠毒素基因携带率不高,根据我们以前的研究结果,我们推测携带传统肠毒素基因的金黄色葡萄球菌产被膜形成能力差,原因可能是金黄色葡萄球菌传统肠毒素的产生与耐热核酸酶之间的相关性强,而耐热核酸酶能够抑制细菌被膜形成[21]。本研究筛选的17株菌株均能在试管壁上形成肉眼可见的生物被膜,全部不携带传统肠毒素基因,因此,从另一个侧面支持了我们的猜测:大多强生物被膜形成的金黄色葡萄球菌可能不携带传统肠毒素基因。当然,这种猜测还需要在以后的研究中进一步验证。另外,学者认为不同粘附素基因和群集感应系统调控因子的协调表达是细菌生物被膜形成起始过程的关键[26-27]。从本研究的检测结果来看,不同分离菌株携带粘附素基因差异性较大,结合试管法以及微孔板法生物被膜形成能力检测结果,我们发现并非携带粘附素基因种类越多的菌株其生物被膜形成能力就越强。事实上,10号和30号菌株的生物被膜形成能力强,但携带的粘附素基因种类却很少,相反,29,47和68号菌株携带的基因种类最多,反而这几株菌株的生物被膜形成能力弱。基于细菌生物被膜的形成受多种因素影响,猜测也许某些关键基因的组合表达才是生物被膜形成的关键。例如,有学者认为FnBPA和FnBPB粘附蛋白分子可促使医院感染分离的耐甲氧西林金黄色葡萄球菌(MRSA)的生物被膜聚集,并且认为同时拥有fnbA和fnbB这两个基因的菌株生物被膜形成能力明显高于只拥有单一基因的菌株[29]。本研究中检测的17株食源性菌株未发现fnbA和fnbB两个基因同时共存的菌株。由于金黄色葡萄球菌生物被膜形成除了受环境的影响,更多依赖于不同分离菌株个体特性和不同粘附素基因特异性组成情况等,如研究者们对引起牛乳腺炎的金黄色葡萄球菌分离菌株V329进行研究,证实生物被膜相关蛋白Bap在金黄色葡萄球菌生物被膜形成的起始粘附和胞间聚集过程是必须的,并且V329的ica位点突变对生物膜形成表型没有任何影响[12,25]。但bap基因只是从牛乳腺炎分离株中发现,到目前为止在任何人源的金黄色葡萄球菌分离菌株中还没有发现[30]。在本研究中17株食源性分离菌株也均未检测出bap基因。在我们先前针对四川地区不同动物源(鸡和羊)部分菌株的研究也未检测出bap基因[20],暗示这些菌株的生物被膜形成与bap基因之间关联性不强。另外,研究者认为金黄色葡萄球菌胞外粘附多糖(PIA/PNAG)在金黄色葡萄球菌生物被膜形成中占有重要的作用,PIA/PNAG是由icaADBC操纵子编码的基因产物,因此,有人认为ica操纵子编码的胞外粘附多糖(PIA/PNAG)是理解生物被膜形成机制的桥梁[9]。本文中,ica基因的检出率相对较高,说明食源性分离菌株大多可能是依赖于PIA/PNAG介导的生物被膜形成类型,但同时也存在不依赖于PIA/PNAG介导的生物被膜形成类型。另外,我们的研究中发现sasC和sasG的携带率约在23%左右,这与Schroeder等[30]的研究结果较一致,他们主要针对医院源金黄色葡萄球菌的分离菌株进行监测,sasG基因的检出率为33%,并且从部分菌株中还筛选到了新的SasC分子,同样,他们在这些菌株中也未检测出bap基因。值得一提的是,所有菌株中均能检测出clfB基因,我们推测这个基因也许在细菌生物被膜形成过程中起着较为重要的作用,我们未来的研究可以针对该基因进行敲出验证,或许能够得到新的发现。
进一步在针对10号和30号菌株的酶处理实验表明蛋白酶K和DNase Ⅰ的单独作用对食源性金黄色葡萄球菌生物被膜形成抑制效果明显,并且蛋白酶K效果优于DNase Ⅰ,但联合作用时效果反而变差,我们分析的原因是蛋白酶K和DNase Ⅰ之间有相互作用,导致酶的活性降低而引起抑制效果变弱。针对DNaseⅠ对生物被膜形成抑制的研究有多次报道,例如,我们以前的研究发现葡萄球菌耐热核酸酶和DNaseⅠ对金黄色葡萄球菌生物被膜的形成可以起到抑制作用[21]。Mann[31]等也发现葡萄球菌核酸酶能够降解胞外DNA,促进细胞的分散,从而能够抑制自身生物被膜的形成。相比较采用蛋白酶K处理生物被膜的报道较少,从本文的结果来看,蛋白酶K的处理效果很好,这也从另一个侧面反映蛋白质粘附素分子在分离菌株生物被膜形成中的贡献。
关于食源性细菌生物被膜的研究目前已成为食品安全关注的一个热点问题[32],还有许多新的因子和新的机制等待我们去探索。特别是针对金黄色葡萄球菌不同分离菌株开展粘附素基因的相关研究能够让我们从中发现一些新的规律和新的特点,所有这些为进一步探索食源性金黄色葡萄球菌生物被膜形成起主要作用的粘附素基因的调控机制奠定基础,也为食源性金黄色葡萄球菌生物被膜形成菌株的检测、消除和控制提供新的目标靶点。
[1]Costerton JW,Stewart PS,Greenberg EP. Bacterial biofilms:a common cause of persistent infections[J]. Science,1999,284(5418):1318-1322.
[2]Sadovskaya I,Vinogradov E,Flahaut S,et al. Extracellular carbohydrate-containing polymers of a model biofilm-producing strain,Staphylococcus epidermidis RP62A[J]. Infection and Immunity,2005,73(5):3007-3017.
[3]Das T,Sharma PK,Busscher HJ,et al. Role of extracellular DNA in initial bacterial adhesion and surface aggregation[J]. Applied and Environmental Microbiology,2010,76(10):3405-3408.
[4]Kiedrowski MR,Kavanaugh JS,Malone CL,et al. Nuclease modulates biofilm formation in community-associated methicillin-resistant Staphylococcus aureus[J]. PloS one,2011,6(11):e26714.
[5]Kogan G,Sadovskaya I,Chaignon P,et al. Biofilms of clinical strains of Staphylococcus that do not contain polysaccharide intercellular adhesin[J]. FEMS Microbiology Letter,2006,255(1):11-16.
[6]Flemming HC,Wingender J. The biofilm matrix[J]. Nature reviews Microbiology,2010,8(9):623-633.
[7]Van Houdt R,Michiels CW. Biofilm formation and the food industry,a focus on the bacterial outer surface[J]. Journal of applied microbiology,2010,109(4):1117-1131.
[8]Garzoni C,Kelley WL. Staphylococcus aureus:new evidence for intracellular persistence[J]. Trends in microbiology,2009;17:59-65.
[9]Gad GF,El-Feky MA,El-Rehewy MS,et al. Detection of icaA,icaD genes and biofilm production by Staphylococcus aureus and Staphylococcus epidermidis isolated from urinary tract catheterized patients[J]. Journal of Infection in Developing Countries,2009,3(5):342-51.
[10]Seo YS,Lee DY,Rayamahji N,et al. Biofilm-forming associated genotypic and phenotypic characteristics of Staphylococcus spp. isolated from animals and air[J]. Research in Veterinary Science,2008,85(3):433-438.
[11]O’Neill E,Pozzi C,Houston P,et al. A novel Staphylococcus aureus biofilm phenotype mediated by the fibronectin-binding proteins,FnBPA and FnBPB[J]. Journal of Bacteriology,2008,190(11):3835-3850.
[12]Cucarella C,Solano C,Valle J,et al. Bap,a Staphylococcus aureus surface protein involved in biofilm formation[J]. Journal ofBacteriology,2001,183(9):2888-2896.
[13]Schroeder K,Jularic M,Horsburgh SM,et al. Molecular characterization of a novel Staphylococcus aureus surface protein(SasC)involved in cell aggregation and biofilm accumulation[J]. PLoS One. 2009;4(10):e7567.
[14]Corrigan RM,Rigby D,Handley P,et al. The role of Staphylococcus aureus surface protein SasG in adherence and biofilm formation[J]. Microbiology,2007,153:2435-2446.
[15]Atshan SS,Nor Shamsudin M,Sekawi Z,et al. Prevalence of adhesion and regulation of biofilm-related genes in different clones of Staphylococcus aureus[J]. Journal of Biomedicine & Biotechnology,2012,2012:976972.
[16]陈秋云,韩北忠,李春雷. 金黄色葡萄球菌生物被膜在不锈钢表面的形成及其对二氧化氯的敏感性[J]. 中国农业大学学报,2004,9(4):10-13.
[17]张娜,韩北忠,李敏,等. 食品接触表面对金黄色葡萄球菌生物被膜形成的影响[J]. 食品研究与开发,2006,7:10-14.
[18]Furukawa S,Akiyoshi Y,O’Toole GA,et al. Sugar fatty acid esters inhibit biofilm formation by food-borne pathogenic bacteria[J]. International Journal of Food Microbiology. 2010,138(1-2):176-80.
[19]王琼,唐俊妮,汤承,等. 一种采用微波炉加热快速提取细菌DNA用于PCR扩增的方法[J]. 西南民族大学学报:自然科学版,2015,41(2):150-155.
[20]Tang J,Chen J,Li H,et al. Characterization of adhesin genes,staphylococcal nuclease,hemolysis,and biofilm formation among Staphylococcus aureus strains isolated from different sources[J]. Foodborne Pathogens and Disease,2013,10(9):757-763.
[21]唐俊妮,康名松,陈焕春,等. 葡萄球菌核酸酶对金黄色葡萄球菌和其他细菌生物被膜形成的抑制作用[J]. 中国科学:生命科学,2011,41(7):586-592.
[22]Kumar CG,Anand SK. Significance of microbial biofilms in food industry:a review[J]. International Journal of Food Microbiology,1998,42(1-2):9-27.
[23]Lentino J R. Prosthetic joint infections:bane of orthopedists,challenge for infectious disease specialists[J]. Clinical Infectious Diseases,2003,36:1157-1161.
[24]Vu B,Chen M,Crawford RJ,et al. Bacterial extracellular polysaccharides involved in biofilm formation[J]. Molecules,2009,13;14(7):2535-54.
[25]Cucarella C,Tormo MA,Ubeda C,et al. Role of biofilmassociated protein Bap in the pathogenesis of bovine Staphylococcus aureus[J]. Infection and Immunity,2004,72(4):2177-2185.
[26]Martí M,Trotonda MP,Tormo-Más MA,et al. Extracellular proteases inhibit protein-dependent biofilm formation in Staphylococcus aureus[J]. Microbes and Infection. 2010;12(1):55-64.
[27]Conrady DG,Brescia CC,Horii K,et al. A zinc-dependent adhesion module is responsible for intercellular adhesion in staphylococcal biofilms[J]. Proceedings of the National Academy of Sciences of the United States of America,2008,105(49):19456-19461.
[28]Xu J,Tan X,Zhang X,et al. The diversities of staphylococcal species,virulence and antibiotic resistance genes in the subclinical mastitis milk from a single Chinese cow herd[J]. Microbial Pathogenesis,2015,88:29-38.
[29]O’Neill E,Humphreys H,O’Gara JP. Carriage of both the fnbA and fnbB genes and growth at 37 degrees C promote FnBP-mediated biofilm development in meticillin-resistant Staphylococcus aureus clinical isolates[J]. Journal of Medical Microbiology,2009,58(Pt 4):399-402.
[30]Zuniga E,Melville PA,Saidenberg AB,et al. Occurrence of genes coding for MSCRAMM and biofilm-associated protein Bap in Staphylococcus spp. isolated from bovine subclinical mastitis and relationship with somatic cell counts[J]. Microbial Pathogenesis,2015,89:1-6.
[31]Mann EE,Rice KC,Boles BR,et al. Modulation of eDNA release and degradation affects Staphylococcus aureus biofilm maturation[J]. PloS one,2009,4(6):e5822.
[32]Shi XM,Zhu XN. Biofilm formation and food safety in food industries[J],Trends in Food Science & Technology,2009,(20):407-413.
Detection of adhesin genes ofStaphylococcusaureusfood isolates and the effect of Proteinase K and DNaseⅠon biofilm formation
MA Yisalan1,YU Bo-yang2,CHEN Juan1,LIU Ji1,TANG Jun-ni1,*
(1.College of Life Science and Technology,Southwest University for Nationalities,Chengdu 610041,China; 2.Chengdu No. 7 High School,Chengdu 610041,China)
To investigate 14 adhesin genes distribution inStaphylococcusaureusfood isolates,the DNA of 17S.aureusisolates was extracted and used for PCR amplification. Then,the biofilm formation ability of 17S.aureusisolates was detected by tube test and microtiter-plate test methods. Further,the effect of Proteinase K and DNaseⅠon biofilm formation was explored and discussed. The results showed that all the 17S.aureusisolates carriedclfBgene and the detection rate was 100%;then followed theicaBCgene(13/17,76.5%);cnagene(12/17,70.6%);enogene(11/17,64.7%);fibandicaADgene(10/17,58.8%);fnbAgene(9/17,52.9%);sasC,sasGandebpSgene(4/17,23.5%);and thefnbBgene(2/17,11.8%). Thebap,bbpandclfAgenes were not exist in all 17 strains. The tube test and microtiter-plate test methods showed all the 17 strains could form biofilm;however,the ability of biofilm formation among different strains had obvious difference. Especially,the strains of No. 10 and No. 30 had stronger biofilm formation ability than others(p<0.05). Proteinase K,DNaseⅠand the combination of these two enzymes were used to treat the biofilm formation during the bacteria growth process. The results showed the biofilm formation ability treated by Proteinase K and DNaseⅠwas weaker than control,and the effect of Proteinase K was better than that of DNaseⅠ.While the effect of two enzymes combination did not present significant difference in comparison with the control(p>0.05). The results of this study would provide basic data for food sourcesS.aureusbiofilm formation and control strategy.
Staphylococcusaureusfood isolates;Biofilm formation;Adhesin genes;Proteinase K;DNaseⅠ
2016-04-08
马伊萨兰(1991-),女,硕士,研究方向:畜产品加工与安全,E-mail:403884179@qq.com。
唐俊妮(1971-),女,博士,教授,研究方向:食品安全与食品微生物,E-mail:junneytang@aliyun.com。
国家自然科学基金(31071515, 31371781);四川省应用基础项目(2014JY0253);中央高校基本科研业务费专项资金项目(2015NZYQN59)。
TS201.3
A
1002-0306(2016)17-0185-06
10.13386/j.issn1002-0306.2016.17.028

