向日葵E3泛素连接酶基因克隆及表达分析
张艳芳,孙瑞芬*,郭树春,于海峰,石慧芹
(1 内蒙古农牧业科学院,呼和浩特 010031;2 内蒙古园艺研究院,呼和浩特 010010)
向日葵E3泛素连接酶基因克隆及表达分析
张艳芳1,孙瑞芬1*,郭树春1,于海峰1,石慧芹2
(1 内蒙古农牧业科学院,呼和浩特 010031;2 内蒙古园艺研究院,呼和浩特 010010)
该研究从向日葵中克隆了E3泛素连接酶基因HERC2,并进行了生物信息学分析和不同胁迫条件的表达分析。序列分析表明,HERC2(登录号为KT832066)序列的CDS为1 608 bp,编码535个氨基酸,预测其分子量131 kD,等电点为5.03。HERC2编码的蛋白质为疏水性蛋白质,且为细胞质蛋白;亚细胞定位预测分析表明,向日葵HERC2可能定位在高尔基体中;该蛋白质有5个RCC1保守结构域。向日葵HERC2与已报道的其他植物同源蛋白有相似的保守区域,与醉蝶花亲缘关系最近,而与大豆和野生大豆的亲缘关系最远。与HERC2 cDNA对应的gDNA(登录号为KT832067)的ORF长度为3 409 bp,与cDNA编码序列比对结果表明,该gDNA由5个外显子和4个内含子组成。实时荧光定量PCR分析表明,向日葵HERC2基因表达受非生物胁迫调节,在不同器官及不同非生物胁迫下存在特异性表达差异。研究认为,HERC2基因应答逆境胁迫具有其特定的表达模式,研究结果为加强对HERC2的利用奠定了基础。
向日葵;E3泛素连接酶;基因克隆;序列分析;基因表达
蛋白质是生命的物质基础,在生物细胞生命周期中不断合成与降解,参与生物体内几乎全部的生理活动[1]。蛋白质降解是蛋白质代谢中不可或缺的部分,生物通过降解蛋白来实现生命活动的动态平衡,完成整个生理代谢和生长发育,以适应新的环境[2]。真核细胞内蛋白质的降解途径主要有溶酶体途径、泛素化途径和胱天蛋白酶途径三种。其中,泛素介导的蛋白酶体途径(ubiquitin-proteasome pathway,UPP)是真核生物体内具有高度特异性、最为重要的蛋白质降解途径,参与细胞周期调整、信号传导、转录、离子通道、生长与凋亡、生物与非生物抗性等重要的生理过程[3-5]。泛素蛋白酶体途径主要由泛素(ubiquitin,Ub)、泛素激活酶(ubiquitin-activating enzyme,E1)、泛素结合酶(ubiquitin-conjugating enzyme,E2)、泛素连接酶(ubiquitin-ligating enzyme,E3)和26S蛋白酶体(proteasome)等组成,其对靶蛋白的降解是一种级联反应过程[6]。在这个途径中,泛素用来标记需要降解的蛋白质,被标记的靶蛋白进而被26S蛋白酶体识别并降解,细胞内80%~90%的蛋白通过泛素途径降解[7]。蛋白质的泛素化修饰涉及一系列的酶参与反应,包括泛素激活酶E1、结合酶E2以及连接酶E3。E3是泛素蛋白酶体途径中种类最多,在底物的特异性选择降解过程中作用最重要的成员,对靶蛋白的特异性识别起关键作用。E3分子量较大,约100~300 kD。根据其亚基成分和作用机制,可分为单亚基类型(如HECT、RING/Ubox)和多亚基类型(如SCF复合物、后期促进复合物APC、CUL3-BTB、CUL4-DDB等)[8]。泛素连接酶所有的E3都具有连接靶蛋白和特定E2的能力,它通过调节蛋白的泛素化过程参与细胞内的生理过程,主要功能是识别应该被泛素化的靶蛋白,然后使活化的泛素靠近特异靶蛋白的赖氨酸,从而将泛素转移到底物蛋白上去[9-10]。蛋白质特定的翻译后的修饰经常作为其相应的E3泛素连接酶识别的标志。宋素胜等[11]认为E3泛素连接酶在植物响应逆境胁迫过程中起关键作用。宁约瑟等[12]认为E3泛素连接酶介导植物干旱胁迫响应。杨玖霞等[13]认为E3泛素连接酶参与了各种抗病信号反应途径的调控,在植物免疫反应从最初病原物的识别到下游信号途径等各个过程中发挥正调控或负调控作用。张雅芬[14]研究认为E3泛素连接酶在番茄对灰霉病抗性反应中起作用。阮松林等[15]研究发现,E3泛素连接酶在拟南芥响应水分、盐胁迫的信号转导中起作用。吴建民等[16]研究表明拟南芥E3泛素连接酶基因能够被脱水、NaCl和ABA处理强烈诱导。
本研究以‘内葵杂4号’为试验材料,克隆了向日葵E3泛素连接酶基因HERC2的cDNA和gDNA的ORF,并分别对其进行了生物信息学分析和基因结构分析,同时进行了不同胁迫条件下HERC2的表达模式分析。研究结果对探索向日葵E3泛素连接酶基因的功能及利用具有重要意义,为进一步解析其对非生物胁迫的响应机制提供新线索。
1 材料和方法
1.1 试验材料
‘内葵杂4号’种子由内蒙古农牧业科学院作物研究所向日葵课题组提供。试验在内蒙古农牧业科学院生物技术研究中心植物培养室中进行。选取饱满、无病虫害的种子,常温下浸泡24 h后,播种于装有等量蛭石的塑料盆(上口直径16.5 cm、下口直径12 cm、高12 cm)中,每盆播种30粒种子。待下胚轴伸长3 cm左右将长势一致的幼苗从蛭石中取出,用清水洗净根部的蛭石。将塑料盆中装满等量的1/2MS营养液,盖上已打孔的塑料盆盖(每个盖子均匀打15个孔),每3株小苗为一组,用海绵轻轻裹住下胚轴,放到1个孔中,根部全部浸入到营养液中进行培养以适应液体环境,培养期间每天更换1次新鲜的培养液。第1对真叶完全展开时,将塑料盆中的1/2MS营养液换为含有等量的0、120、150和180 mmol·L-1NaCl,0、5、10和50 μmol·L-1ABA,0、5%、10%和20%PEG6000的1/2MS溶液中进行胁迫处理,每种胁迫处理各1盆,每盆45株。以0、6、24和48 h为时间点取样,每个处理每个时间点取3个孔(9株)中的幼苗混合,分别取根、下胚轴、叶,立即于液氮中速冻,-80℃冰箱保存。
1.2 核酸提取及cDNA合成
1.2.1 总RNA的提取及cDNA合成 前期通过高通量测序完成的向日葵转录组[17]和数字化基因表达谱测序结果中,得到120 mmol·L-1NaCl胁迫处理2 d向日葵叶片中候选基因HERC2(CL5734.Contig2-All)的表达量最高。因此,本研究以120 mmol·L-1NaCl胁迫2 d向日葵幼苗叶片为材料,利用TaKaRa公司的RNAiso Plus提取总RNA,并用1%琼脂糖凝胶电泳检测其完整性和纯度。用TaKaRa公司的PrimeScript 1st Strand cDNA Synthesis Kit合成第一链cDNA。具体操作步骤依据说明书进行。
1.2.2 基因组DNA的提取 利用天根生化试剂(北京)有限公司的植物基因组DNA提取试剂盒提取相同材料的DNA,并用1%琼脂糖凝胶电泳检测其完整性和纯度。具体操作步骤依据说明书进行。
1.3 HERC2基因cDNA和gDNA开放阅读框(ORF)的克隆
根据NaCl胁迫下向日葵根的mRNA转录组测序结果中获得的HERC2序列,在其ORF(Open Reading Frame)两端设计特异性引物。以向日葵叶片的cDNA为模板,用TaKaRa公司LATaq,经PCR扩增cDNA片段。同时以基因组DNA为模板,用LATaq,PCR扩增其对应的gDNA(genomic DNA)片段。cDNA和gDNA反应体系为50 μL:LATaq0.5 μL,10×LA PCR Buffer II(Mg2+Free) 5 μL,dNTP Mixture 8 μL,MgCl25 μL,E3F1 1 μL,E3R1 1 μL,cDNA/DNA 1 μL,ddH2O补足到50 μL。反应程序为94 ℃ 1 min;98 ℃ 10 s,60 ℃ 30 s,72 ℃ 3 min,35个循环;72 ℃ 10 min。PCR扩增产物用0.8%琼脂糖电泳分析后,用Roche公司的High Pure PCR Product Purification Kit回收。分别连接到pGM-T载体上,进行TA克隆并测序。pGM-T克隆试剂盒、TOP10感受态细胞购自天根生化试剂(北京)有限公司。HERC2相关的引物合成在TaKaRa公司完成,序列测定在南京金斯瑞生物工程技术服务有限公司完成,引物序列见表1。
1.4 HERC2基因的生物信息学分析
运用NCBI数据库中的(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)查找和翻译HERC2 cDNA序列的CDS(Coding Sequence);运用在线分析软件ProtParam(http://www.expasy.org/tools/protparam.html)对该基因编码蛋白进行理化性质分析;运用在线分析软件ProtScale(http://www.expasy.org/tools/protscale.html)进行疏水性/亲水性预测;运用在线分析软件TMHMM Server 2.0(http://www.cbs.dtu.dk/services/TMHMM/)分析目的蛋白的跨膜结构域;运用PSORT(http//psort.hgc.jp/form.html)分析蛋白质的亚细胞定位;运用在线分析软件Blast(http://blast.ncbi.nlm.nih.gov/Blast.cgi)分析蛋白质结构域;运用NCBI数据库中的BLAST对氨基酸序列进行同源性搜索和比对分析;运用DNAMAN软件将目的序列与已报道其他物种的E3泛素连接酶基因同源序列进行多重比对,用MEGA5.0软件构建系统进化树;运用DNAMAN软件将获得的gDNA核苷酸序列与cDNA核苷酸序列比对分析基因结构。
1.5 HERC2基因在不同胁迫下的表达分析
根据HERC2 cDNA的CDS设计实时荧光定量PCR引物E3RTF1和E3RTR1,以向日葵18S rRNA(HM638219)为内参基因(表1)。用RNAiso Plus提取不同胁迫处理的向日葵根、下胚轴、叶的总RNA,并稀释到相同浓度,用TaKaRa公司的PrimeSript RT reagent Kit with gDNA Eraser试剂盒反转录成单链cDNA,具体方法按照说明书进行。采用Real-time PCR分析各处理向日葵叶中HERC2的相对表达量,并对每种胁迫处理相对表达量最高的时间点上进行HERC2在不同器官中的相对表达量分析。Real-time PCR用TaKaRa公司的SYBRPremixExTaqII(Tli RNaseH Plus)试剂盒进行,向日葵18S rRNA作为内参。实时荧光定量PCR反应体系为E3RTF1 0.5 μL、E3RTR1 0.5 μL、cDNA 1 μL、SYBR Premix Ex TaqⅡ 10 μL、ddH2O补足到20 μL。反应程序为95 ℃ 30 s;95 ℃ 5 s,60 ℃ 30 s,40个循环,每个反应设3次重复。采用2-ΔΔCt法进行基因相对表达量分析。采用方差分析检测向日葵HERC2基因在不同非生物胁迫下不同时间表达水平的显著性。

表1 HERC2相关的引物序列
2 结果与分析
2.1 HERC2基因cDNA和gDNA的ORF克隆
根据高通量测序结果得到HERC2基因的保守序列设计特异性扩增引物,以120 mmol·L-1NaCl胁迫处理2 d向日葵叶片的总RNA为模板,经PCR扩增,获得1条约1 700 bp的目的片段(图1,A)。回收克隆测序结果表明该片段的长度为1 690 bp,且与高通量测序结果一致。以DNA为模板,经PCR扩增,获得1条约3 500 bp的目的片段(图1,B),回收克隆测序结果表明该片段的长度为3 491 bp。最终获得HERC2基因的cDNA长度为1 994 bp(基因登录号为KT832066),gDNA长度为3 795 bp(基因登录号为KT832067)。
2.2 HERC2基因cDNA和gDNA序列分析
对HERC2基因cDNA全长序列分析表明,该基因具有完整的开放阅读框,自起始密码子ATG至终止密码子TAG长度为1 608 bp,编码535个氨基酸(图2)。运用ProtParam软件推测其编码蛋白的理论分子量为131 kD,等电点为5.03。对HERC2编码的氨基酸组成进行分析,其中含量最高的是丙氨酸(Ala),占28%,其后依次是苏氨酸(Thr)占27.4%、甘氨酸(Gly)占26.4%,含量最少的为半胱氨酸(Cys),占18.2%。分子式为C4841H8076N1608O2050S293,总原子数11 970,亲水性平均系数0.662。运用ProtScale软件的Kyte & Doolittle算法对HERC2编码的氨基酸进行了疏水性分析,氨基酸在疏水性区域所占比重远大于亲水性区域,预测HERC2编码的蛋白质为疏水性蛋白。TMHMM结果表明HERC2整条肽链都位于细胞膜外,不存在跨膜域,可能为细胞质蛋白。运用PSORT软件在线预测该蛋白的亚细胞定位情况,发现存在于高尔基体中的可能性比较大。通过Blast发现,有5个RCC1(Regulator of chromosome condensation 染色体浓缩调节因子)保守结构域,分别为第33~88、91~153、224~272、287~333和337~395氨基酸。通过Blast比对发现HERC2与已报道的芝麻(XP_011081781.1)、黄色猴脸花(XP_012857820.1)、蓖麻(XP_015573157.1)、木豆(KYP65138.1)、马铃薯(XP_015170023.1)、香瓜(XP_008445010.1)、大豆(XP_003545609.1)、蒺藜状苜蓿(XP_003603615.2)、野生大豆(KHN12959.1)、绿豆(原变种)(XP_014504264.1)和醉蝶花(XP_010541254.1)的E3泛素连接酶氨基酸序列相似性分别为58%、58%、57%、57%、57%、57%、56%、55%、54%、54%和53%。氨基酸序列多重比对发现HERC2与以上11个物种基因推导氨基酸序列有相似的高度保守区域(图3)。利用MEGA5.0软件将向日葵与芝麻等11种植物的E3泛素连接酶氨基酸序列进行系统进化分析,表明向日葵与醉蝶花亲缘关系最近,而与大豆和野生大豆的亲缘关系最远(图4)。HERC2 cDNA对应的gDNA,经序列测定发现该基因编码区自起始密码子ATG至终止密码子TAA长度为3 409 bp,与cDNA编码序列比对结果表明该gDNA由5个外显子和4个内含子组成,5个外显子长度分别为94、174、36、113和1 191 bp,4个内含子长度分别为101、1 300、339和61 bp,内含子两端具有典型的GT/AG结构。
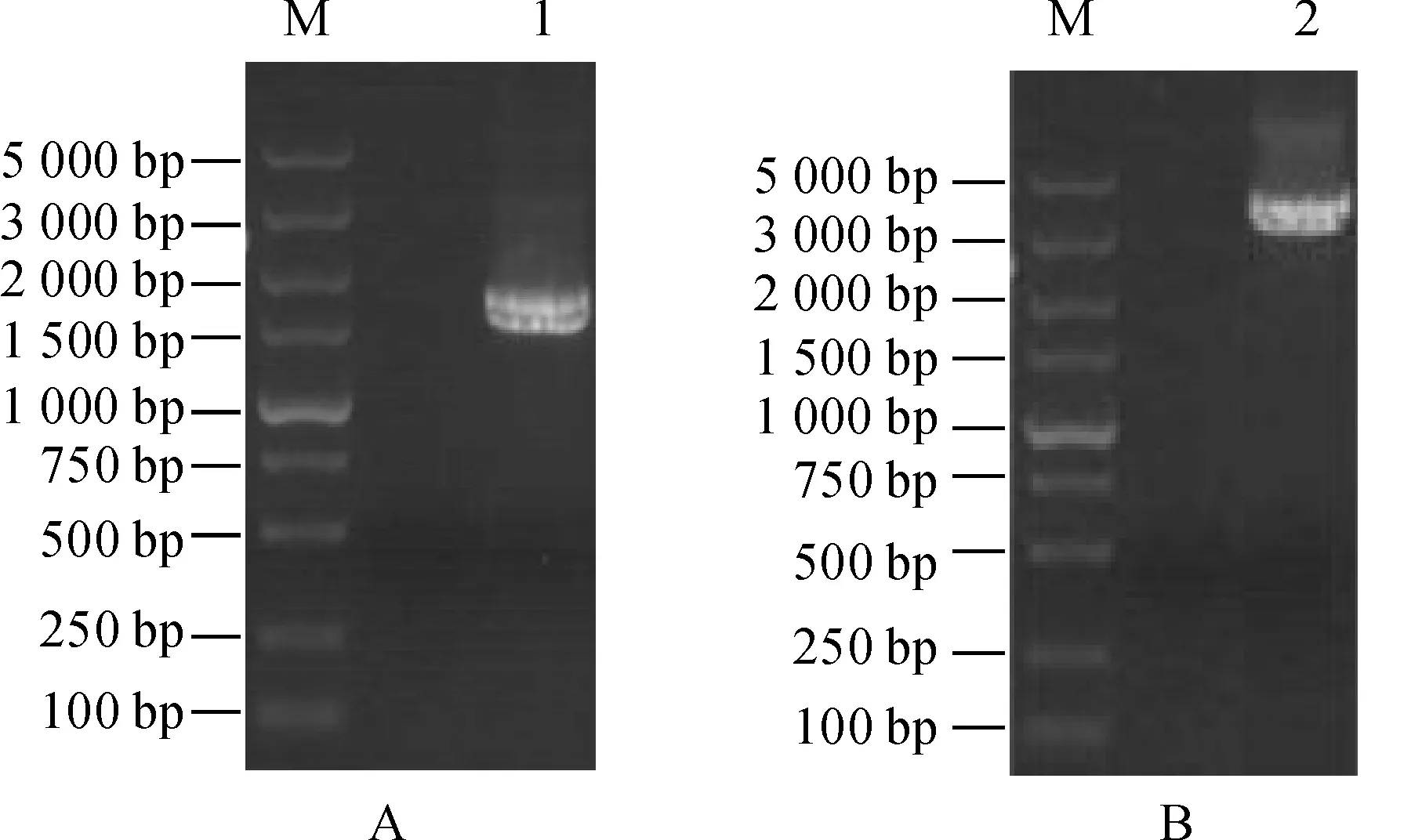
M.DL5000;1.cDNA;2.gDNA图1 HERC2 基因开放阅读框cDNA和gDNA PCR扩增结果Fig.1 PCR amplification of ORF cDNA and gDNA of HERC2 gene

图2 HERC2 cDNA的CDS序列及其推导的氨基酸序列Fig.2 CDS and its deduced amino acid sequence of HERC2 cDNA
2.3 HERC2基因的表达分析
实施荧光定量PCR分析表明,向日葵幼苗受到NaCl、ABA和PEG的胁迫后,叶片中的HERC2表达量较对照均有所增加,但增加的倍数有所不同。说明HERC2在不同胁迫条件下的表达模式不同。
不同浓度NaCl胁迫下,HERC2的表达量随胁迫浓度的增大而增高,120 mmol·L-1NaCl胁迫处理较对照增加幅度最小,当浓度达到180 mmol·L-1时的表达量增高幅度最大。随时间的延长, 每种浓度处理HERC2的表达量均呈现先增高后降低的趋势;其中NaCl胁迫24 h时基因的表达量最大,此时120、150和180 mmol·L-1处理分别为对照(0 h)的15.96、38.32和45.99倍(图5,A)。
KT832066. 向日葵;XP_011081781.1. 芝麻;XP_012857820.1. 黄色猴脸花;XP_015573157.1. 蓖麻;KYP65138.1. 木豆;XP_015170023.1. 马铃薯;XP_008445010.1. 香瓜;XP_003545609.1. 大豆;XP_003603615.2. 蒺藜状苜蓿;KHN12959.1. 野生大豆;XP_014504264.1. 绿豆(原变种);XP_010541254.1. 醉蝶花图3 HERC2与其他物种E3泛素连接酶基因的氨基酸序列比对KT832066. Helianthus annuus; XP_011081781.1. Sesamum indicum; XP_012857820.1. Erythranthe guttata; XP_015573157.1. Ricinus communis; KYP65138.1. Cajanus cajan; XP_015170023.1. Solanum tuberosum; XP_008445010.1. Cucumis melo; XP_003545609.1. Glycine max; XP_003603615.2. Medicago truncatula; KHN12959.1. Glycine soja; XP_014504264.1. Vigna radiata var. radiata; XP_010541254.1. Tarenaya hasslerianaFig.3 Comparison of the amino acid sequences of HERC2 and other E3 ubiquitin ligase genes

分支上的数值表示Bootstrap验证中基于1 000次重复节点的可信度;标尺表示遗传距离图4 HERC2的氨基酸序列与其他物种同源序列的进化分析The numbers on the branches represent the reliability percent of Bootstrap values based on 1 000 replication; The scale bar represents genetic distanceFig.4 Phylogenetic analysis of amino acid sequences compare with other species of HERC2
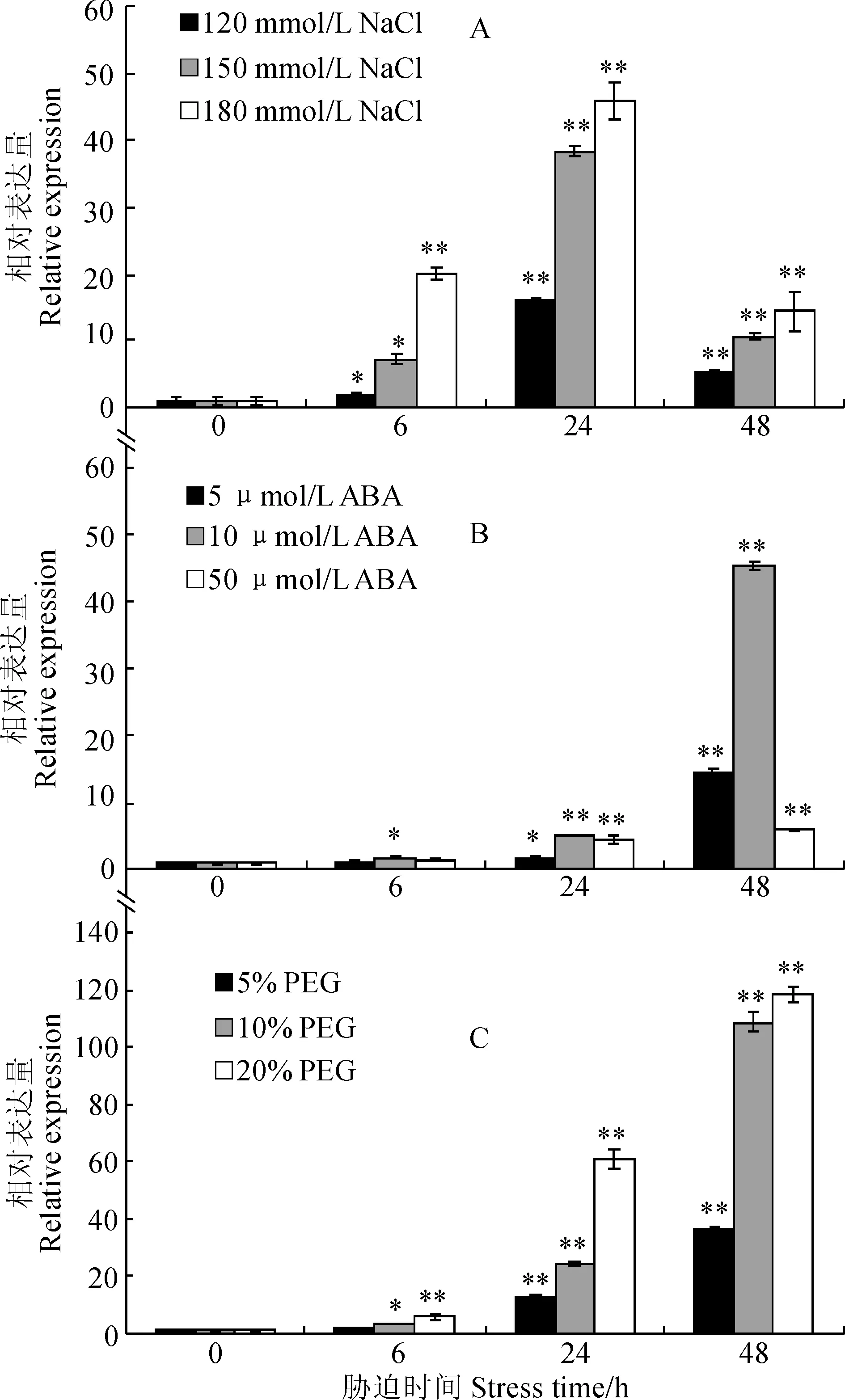
*和**分别表示各浓度胁迫处理与对照之间在0.05和0.01水平存在显著性差异图5 不同浓度NaCl、ABA和PEG胁迫下HERC2在向日葵叶中的表达* and ** represent significant difference between every concentration stress and control at 0.05 and 0.01 levels, respectivelyFig.5 Expression analysis of HERC2 gene in sunflower leaf under different NaCl, ABA and PEG concentration stress
不同浓度ABA胁迫下,HERC2的表达量在50 μmol·L-1时增高幅度最小,在10 μmol·L-1时增高幅度最大。每种处理浓度HERC2的表达量均随时间的延长呈现增高的趋势;其中ABA胁迫处理48 h时基因的表达量最大,此时50、5和10 μmol·L-1处理分别为对照(0 h)的5.98、14.29和和45.25倍(图5,B)。
不同浓度PEG胁迫下,HERC2表达量随胁迫浓度的增大而增高,5%时增高幅度最小,20%时的增高幅度最大。每种处理浓度HERC2的表达量随处理时间的延长均呈现增高的趋势。其中PEG胁迫处理48 h时基因的表达量最大,5%、1%和2%处理HERC2表达量分别为对照(0 h)的36.09、108.63和118.06倍(图5,C)。

**表示根与下胚轴、叶之间在0.01水平存在显著性差异图6 HERC2在不同胁迫处理下的器官特异性表达** represent significant difference between root and hypocotyl, leaf at 0.01 levelFig.6 Expression of HERC2 gene in various organs of sunflower under different kinds of stress
向日葵幼苗受到NaCl、ABA和PEG的胁迫后,HERC2在根、下胚轴、叶中的相对表达量均呈现极显著性差异。其中,180 mmol·L-1NaCl胁迫处理24 h,HERC2在叶片中的表达量最高,是根中表达量的1.18倍;其次为根;在下胚轴中的表达量最低,是根的0.87倍。10 μmol·L-1ABA胁迫处理48 h,HERC2在根中的表达量最高;其次是叶,是根的0.53倍;在下胚轴中的最少,是根的0.36倍。20%PEG胁迫处理48 h,HERC2在根中的表达量最高;其次是下胚轴,是根的0.66倍;在叶中的最少,是根的0.64倍(图6)。说明HERC2在向日葵中存在器官特异性表达差异。
3 讨 论
在植物的发育过程中,E3泛素连接酶参与从胚胎发育到花器官产生几乎所有的发育过程,控制植物生长发育和逆境胁迫响应等过程中的关键步骤。E3泛素连接酶在植物生长发育过程中参与自交不亲和[18]、花发育[19-20]和植物衰老等反应[21-22]。E3泛素连接酶积极参与激素反应,有针对性地降解激素应答基因下游的特异性转录因子抑制生长素、赤霉素、脱落酸、乙烯和茉莉酸信号转导通路,限制激素生物合成[23-24]。Dong等[25]研究发现拟南芥通过泛素/蛋白酶体途径中的E3泛素连接酶HOS1降解ICE1蛋白减弱冷胁迫的伤害。对U-box蛋白CaPUB1、PUB22/23以及RING-finger蛋白DRIP1/DRIP2、Rma1H1的研究均表明,E3泛素连接酶可以通过泛素化修饰靶蛋白调控植物对干旱胁迫的响应过程[26-28]。
在许多E3泛素蛋白连接酶中发现,都有至少一个拷贝的与细胞周期调节RCC1同源的N末端。RCC1是目前唯一已知的Ran鸟嘌呤核苷酸交换因子,是位于间期细胞核内与染色质相结合与Ran相互作用的一种真核蛋白,它作为细胞周期调节因子在许多有机体的细胞中发挥作用[29]。RCC1和Ran的相互作用可能在基因表达调控中起主要作用。RCC1与Ran蛋白结合,促进Ran-GDP转变为RanGTP结合态,从而促进核质运输,在核内输、外运,有丝分裂期纺锤体的形成及核膜的重塑,防止S期DNA多重复制等过程中发挥作用,同时在染色体端粒沉默、细胞增殖、细胞周期调控、微管的组装和DNA复制等过程中发挥重要功能[30]。向日葵HERC2基因含有5个RCC1保守结构域,向日葵HERC2基因多重复RCC1保守结构域的特点可能使该基因在抵御盐胁迫的伤害上发挥作用。
通过对高通量测序获得的向日葵E3泛素连接酶基因序列的分析,得到该基因具有完整的CDS。因此,本研究以该序列为基础,克隆了cDNA和gDNA的ORF。又进一步对HERC2进行了盐胁迫、ABA激素胁迫和PEG模拟干旱胁迫3种非生物胁迫的表达分析,结果表明该基因可以被盐胁迫、激素胁迫、干旱等非生物胁迫诱导,来抵抗或削弱逆境对植物的伤害。对HERC2基因在向日葵受到胁迫后不同器官的表达特征进行研究,表明HERC2基因在向日葵中存在器官特异性表达差异。本研究认为HERC2基因参与了非生物胁迫响应的信号应答,是一个正调节因子,可以作为通过抗逆基因工程提高农作物抗逆性的候选基因,本研究为HERC2基因的功能和作用机制的研究奠定了基础。
[1] SMALLE J, VIERSTRA R. The ubiquitin 26S proteasome proteolytic pathway[J].AnnualReviewofPlantBiology, 2004, 55(1): 555-590.
[2] DREHER K, CALLIS J. Ubiquitin, hormones and biotic stress in plants[J].AnnalsofBotany, 2007, 99(5): 787-822.
[3] 郭启芳, 邹 琦, 王 玮. 植物泛素/26S蛋白酶体通路的生理功能和分子生物学[J]. 植物生理学通讯, 2004, 40(5): 533-539.
GUO Q F, ZOU Q, WANG W. Physiological function of plant ubiquitin/26S proteasome pathway and its molecular biology[J].PlantPhysiologyCommunications, 2004, 40(5): 533-539.
[4] GLICKMAN M H, CIECHANOVER A. The ubiquitin-proteasome proteolytic pathway: destruction for the sake of construction[J].PhysiologicalReviews, 2002, 82(2): 373-428.
[5] CRAIG A, EWAN R,MESMAR J,etal. E3 ubiquitin ligases and plant innate immunity[J].JournalofExperimentalBotany, 2009, 60(4): 1 123-1 132.
[6] HERSHKO A, CIECHANOVER A. The ubiquitin system[J].AnnualReviewofBiochemistry, 1998, 67: 425-479.
[7] 倪晓光, 赵 平.泛素-蛋白酶体途径的组成和功能[J]. 生理科学进展,2006, 37(3):255-258.
NI X G, ZHAO P. The composition and function of ubiquitin proteasome pathway[J].ProgressinPhysiologicalSciences, 2006, 37(3):255-258.
[8] VIERSTRA R D. The ubiquitin-26S proteasome system at the nexus of plant biology[J].NatureReviewsMolecularCellBiology, 2009, 10(6): 385-397.
[9] 黄海杰, 陈雄庭. 植物泛素/26S蛋白酶体途径研究进展[J].中国生物工程杂志, 2008, 28(7): 127-132.
HUANG H J, CHEN X T. The progress on the ubiquitin 26S proteasome pathway in plants[J].ChinaBiotechnology, 2008, 28(7): 127-132.
[10] HARTMANN-PETERSEN R, SEEGER M, GORDON C. Transferring substrates to the 26S proteasome[J].TrendsinBiochemicalSciences, 2003, 28(1): 26-31.
[11] 宋素胜, 谢道昕. 泛素蛋白酶体途径及其对植物生长发育的调控[J]. 植物学通报, 2006, 23(5): 564-577.
SONG S S, XIE D X. The ubiquitin-proteasome pathway and plant development[J].ChineseBulletinofBotany, 2006, 23(5): 564-577.
[12] 宁约瑟, 王国梁, 谢 旗. E3泛素连接酶介导的植物干旱胁迫反应[J]. 植物学报, 2011, 46(6): 606-616.
NING Y S, WANG G L, XIE Q. E3 ubiquitin ligase-mediated drought responses in plants[J].ChineseBulletinofBotany, 2011, 46(6): 606-616.
[13] 杨玖霞, 张 浩, 王志龙,等. E3泛素连接酶调控植物抗病分子机理研究进展[J]. 植物保护, 2015, 41(4):1-8.
YANG J X, ZHANG H, WANG Z L,etal. Recent progresses in the regulation mechanism of E3 ligases in plant disease resistance[J].PlantProtection, 2015, 41(4): 1-8.
[14] 张雅芬. 组蛋白单泛素化E3连接酶、mediator complex亚基和维生素B6合成途径在番茄对灰霉病抗性反应中的功能研究[D]. 杭州: 浙江大学, 2014.
[15] 阮松林, 武维华. 一个E3泛素连接酶在拟南芥响应水分胁迫信号转导中的作用[C]// 中国植物生理学会第九次全国会议论文摘要汇编, 2004.
RUAN S L, WU W H. A E3 ubiquitin ligase in response to water stress action in signal transduction inArabidopsis[C]//ProceedingsoftheNinthNationalConferenceoftheChineseSocietyforPlantPhysiology, 2004.
[16] 吴建民.拟南芥泛素连接酶SRAR1的功能研究[D]. 兰州: 兰州大学, 2009.
[17] 孙瑞芬, 张艳芳, 郭树春, 等. 基于RNA-Seq技术的盐胁迫向日葵转录组信息分析[J]. 分子植物育种, 2015, 13(12): 2 736-2 742.
SUN R F, ZHANG Y F, GUO S C,etal. Analysis on transcriptome of sunflower under salt stress based on RNA-Seq technology[J].MolecularPlantBreeding, 2015, 13(12): 2 736-2 742.
[18] 于晓敏,蓝兴国,李玉花,等. 泛素/26S蛋白酶体途径与显花植物自交不亲和反应[J]. 植物学通报, 2006, 23(2): 197-206.
YU X M, LAN X G, LI Y H,etal. The Ub/26S proteasome pathway and self-incompatible responses in flowering plants [J].ChineseBulletinofBotany, 2006, 23(2): 197-206.
[20] PARK B S, EO H J, JANG I C,etal. Ubiquitination of LHY by SINAT5 regulates flowering time and is inhibited by DET1[J].Biochemical&BiophysicalResearchCommunications, 2010, 398(2): 242-246.
[21] ZENG L R, QU S H, BORDEOS A,etal. Spotted leaf11, a negative regulator of plant cell death and defense, encodes a U-box/armadillo repeat protein endowed with E3 ubiquitin ligase activity[J].PlantCell, 2004, 16(10): 2 795-2 808.
[22] LIN S S, MARTIN R, MONGRAND S,etal. RING1 E3 ligase localizes to plasma membrane lipid rafts to trigger FB1-induced programmed cell death inArabidopsis[J].PlantJournal, 2008, 56(4): 550-561.
[23] DREHER K, CALLIS J. Ubiquitin, hormones and biotic stress in plants[J].AnnalsofBotany, 2007, 99(5): 787-822.
[24] SANTNER A, ESTELLE M. The ubiquitin-proteasome system regulates plant hormone signaling[J].PlantJournal, 2010, 61(6):1 029-1 040.
[25] DONG C H, AGARWAL M, ZHANG Y Y,etal. The negative regulator of plant cold responses,HOS1, is a RING E3 ligase that mediates the ubiquitination and degradation of ICE1[J].ProceedingsoftheNationalAcademyofSciences, 2006, 103(21): 8 281-8 286.
[26] CHO S K, CHUNG H S, RYU M Y,etal. Heterologous expression and molecular and cellular characterization of CaPUB1 encoding a hot pepper U-Box E3 ubiquitin ligase homolog[J].PlantPhysiology, 2006,142(4): 1 664-1 682.
[27] QIN F, SAKUMA Y, TRAN LS P,etal.ArabidopsisDREB2A-interacting proteins function as RING E3 ligases and negatively regulate plant drought stress-responsive gene expression[J].PlantCell, 2012, 20(6): 1 693-1 707.
[28] LEE H K, CHO S K, SON O,etal. Drought stress-induced Rma1H1, a RING membraneanchor E3 ubiquitin ligase homolog, regulates aquaporin levels via ubiquitination in transgenicArabidopsisplants[J].PlantCell, 2009, 21(2): 622-641.
[29] BISCHOFF F R, PONSTINGL H. Catalysis of guanine nucleotide exchange on Ran by the mitotic regulator RCC1[J].Nature, 1991, 354:80-82.
[30] 杨立琳. 小鼠卵母细胞成熟、受精和早期胚胎发育过程中RCC1的表达及定位[D]. 哈尔滨:东北农业大学, 2008.
(编辑:宋亚珍)
Cloning and Expression Analysis of E3 Ubiquitin Ligase Gene HERC2 of Sunflower (Helianthus annuus L.)
ZHANG Yanfang1, SUN Ruifen1*, GUO Shuchun1, YU Haifeng1, SHI Huiqin2
(1 Inner Mongolia Academy of Agricultural & Animal Husbandry Sciences, Huhhot 010031, China; 2 Inner Mongolia Horticulture Research Institute, Huhhot 010010, China)
In order to understand the functions ofHERC2 and effectively make use it, we cloned theHERC2 cDNA and gDNA sequence from sunflower and analyzed it by bioinformatics method. Expression pattern ofHERC2 of sunflower was revealed under abiotic stresses. The CDS ofHERC2 is 1 608 bp, encoding 535 amino acids. The predicted molecular weight is 131 kD, isoelectric point is 5.03. TheHERC2 encoded protein is hydrophobic proteins, and it is cytoplasmic protein. The protein was localized in the Golgi apparatus. The protein has 5 RCC1 conserved domains. Phylogenetic analysis of amino acid sequence of E3 ubiquitin ligase was close toTarenayahassleriana, was far withGlycinemaxandGlycinesoja. Gene accession number is KT832066. The ORF of gDNA ofHERC2 is 3 409 bp, which have 5 exons and 4 introns. Gene accession number is KT832067. RT-qPCR showed thatHERC2 has different expression characteristics under different abiotic stresses. The expression characteristics also exist in different organs.
sunflower; E3 ubiquitin ligase; gene cloning; sequence analysis; gene expression
1000-4025(2016)10-1933-08
10.7606/j.issn.1000-4025.2016.10.1933
2016-07-04;修改稿收到日期:2016-10-17
内蒙古农牧业创新基金(2013CXJJN14)
张艳芳(1982-),女,在读博士研究生,助理研究员,主要从事向日葵生物技术研究。E-mail:35812653@qq.com
*通信作者:孙瑞芬,研究员,主要从事向日葵生物技术研究。E-mail:sunruifen3231@sina.com
Q785;Q786
A

