猪流行性腹泻病毒变异毒株、经典毒株及弱毒疫苗株多重RT-PCR鉴别检测方法的建立及应用
秦毅斌,卢冰霞,段群棚,何 颖,李 斌,梁家幸,苏乾莲,周英宁,蒋冬福,卢敬专,赵 武
(广西兽医研究所,广西畜禽疫苗新技术重点实验室,广西 南宁 530001)
猪流行性腹泻病毒变异毒株、经典毒株及弱毒疫苗株多重RT-PCR鉴别检测方法的建立及应用
秦毅斌,卢冰霞,段群棚,何 颖,李 斌,梁家幸,苏乾莲,周英宁,蒋冬福,卢敬专,赵 武*
(广西兽医研究所,广西畜禽疫苗新技术重点实验室,广西 南宁 530001)
为建立猪流行性腹泻病毒(PEDV)变异毒株、经典毒株及弱毒疫苗株快速鉴别检测方法,根据GenBank中公布的PEDV变异毒株、经典毒株及弱毒疫苗株的S基因和ORF3基因,设计合成2对特异性扩增引物,用以扩增PEDV S基因和ORF3基因,通过目的片段的数量和片段大小来判断PEDV的毒株类型;通过对退火温度等反应条件的优化,建立了检测不同PEDV毒株类型的多重RT-PCR鉴别检测方法。结果显示,所建立的多重RT-PCR鉴别检测方法能特异性区分PEDV变异毒株、经典毒株和弱毒疫苗株;PEDV变异毒株扩增出2条目的条带,分别为234 bp的ORF3基因片段和826 bp的S基因片段;PEDV经典毒株扩增出1条目的条带,片段大小为234 bp的ORF3基因片段;而弱毒疫苗株扩增出1条目的条带,片段大小为185 bp的ORF3基因片段;与猪传染性胃肠炎病毒、A群猪轮状病毒、猪嵴病毒、猪繁殖与呼吸综合症病毒、猪伪狂犬病毒、猪瘟病毒、猪圆环病毒2型、猪乙型脑炎病毒、猪细环病毒及猪细小病毒均无交叉反应;敏感性试验显示,该方法能检测到的最低核酸浓度为2.3×10-4ng/μl;且该方法具有良好的重复性。利用该方法对采集自广西部分地区的91份临床腹泻样品进行检测,样品中PEDV阳性样品有64份,其中变异毒株占89.06 %(57/64),经典毒株占4.69 %(3/64),弱毒疫苗株占6.25 %(4/64)。结果表明,该多重RT-PCR鉴别检测方法特异性强、灵敏度高、操作简单,为猪流行性腹泻的流行病学及病原的鉴别诊断研究提供了可供借鉴的技术手段。
猪流行性腹泻病毒;变异毒株;经典毒株;弱毒疫苗株;RT-PCR;鉴别诊断
猪流行性腹泻(porcine epidemic diarrhea,PED)是由猪流行性腹泻病毒(porcine epidemic diarrhea virus,PEDV)引起的猪的一种高度接触性肠道传染病,该病的主要特征是急性水样腹泻、呕吐和脱水死亡;各种年龄阶段的猪只均易感,对1~2周龄哺乳仔猪危害最为严重,病死率可高达90 %~100 %,给养猪业造成严重的危害和经济损失[1-2]。20世纪70年代,英国和比利时各自报道了该病的发生[3-4],随后该病在世界上多个主要养猪国家相继暴发和流行[5],近年来亚洲一些国家如泰国、韩国、越南和中国多次报道了PED[6-8]。
PEDV是套式病毒目(Nidovirales)冠状病毒科(Coronaviridae)冠状病毒亚科(Coronavirinae)甲型冠状病毒属(Alphacoronavirus)的成员,为有囊膜的单股正链RNA病毒。PEDV基因组全长约28 Kb,包括5’端非编码区、3’端非编码区以及至少7个开放阅读框(ORF1a, ORF1b, ORF2~6),分别编码主要结构蛋白:纤突蛋白(S)、膜蛋白(M)、核衣壳蛋白(N)、小包膜蛋白(E)和非结构蛋白(复制酶1a、1b、ORF3蛋白)[9]。S蛋白为Ⅰ型跨膜蛋白,位于病毒粒子的表面,它在诱导感染宿主中和抗体的产生、与特异性受体的结合及细胞膜融合方面发挥着十分重要的作用[10-11]。此外,冠状病毒S基因的变异可以引起病毒组织嗜性的改变和病毒毒力的变化[12-13]。ORF3基因是PEDV的一个附属基因,编码一种离子通道蛋白,可调节病毒产量,可能与病毒的毒力相关[14]。PEDV弱毒疫苗株的ORF3基因在245~294 bp存在49或51个核苷酸的缺失,是弱毒株重要的分子标记[15]。
2010年底开始,PED在我国大范围暴发流行,临床上主要表现为高发病率和高死亡率的哺乳仔猪腹泻(病死率达80 %~100 %),造成了大量的哺乳仔猪死亡。当前,我国流行PEDV毒株主要存在2种类型,即PEDV经典毒株和PEDV变异毒株,并且以变异毒株为优势流行毒株[16-17]。PEDV基因组中变异最大的区域位于S基因,且大多数的变异都聚集在S基因的N端。与早期流行的经典毒株(PEDV CV777)相比,PEDV变异毒株的S基因在N端存在15个碱基的插入和6个碱基的缺失,从而导致流行毒株S蛋白的抗原位点、糖基化位点和跨膜螺旋均有明显变化[18]。此外,研究显示在采集的临床样品中还存在与弱毒疫苗株高度同源的毒株[16]。
当前我国广泛流行的PEDV有变异毒株为优势毒株、经典毒株并存的特点,再加上弱毒株的广泛使用,使得临床上的PEDV毒株错综复杂。常规检测方法如病毒分离、免疫组化、免疫荧光试验及常规RT-PCR等并不能区分PEDV毒株类型;基因测序是鉴定毒株类型的有效方式,但该方法操作复杂、消耗时间长、费用成本高。我国目前尚无鉴别诊断猪流行性腹泻病毒变异株、经典毒株和弱毒疫苗株的多重RT-PCR方法。因此,有必要建立一种快速简便有效的猪流行性腹泻病毒变异株、经典毒株和弱毒疫苗株的多重RT-PCR鉴别检测方法。
基于以上的研究和当前的需求,本研究根据PEDV弱毒疫苗株ORF3基因缺失49个核苷酸以及变异毒株S基因连续的核苷酸插入的特征,设计并合成了2 对检测猪流行性腹泻病毒的引物,采用多重RT-PCR方法扩增ORF3基因和S基因片段,通过扩增片段的数量和分子量大小,判断是PEDV变异毒株、经典毒株还是弱毒疫苗株,从而特异、敏感、快速简便、低成本、有效地鉴别诊断猪流行性腹泻病毒的毒株类型。
1 材料与方法
1.1 毒株
猪流行性腹泻病毒弱毒疫苗株(PEDV attenuated strain CV777)、猪流行性腹泻病毒经典毒株(PEDV classical strain)、猪流行性腹泻病毒变异毒株(PEDV variant strain)、猪传染性胃肠炎病毒(TGEV)、A群猪轮状病毒(PRoVA)、猪瘟病毒(CSFV)、猪繁殖与呼吸综合征病毒(PRRSV)、猪细小病毒(PPV)、伪狂犬病病毒(PRV)、猪嵴病毒(PKV)、猪乙型脑炎病毒(JEV)、猪圆环病毒2型(PCV-2)、猪细环病毒(TTV)均由本实验室保存提供。
1.2 临床样品
腹泻粪便及拉稀仔猪肠道样品采自广西壮族自治区南宁、柳州、桂林、贵港、玉林、崇左等地养猪场发生腹泻的仔猪。
1.3 主要试剂
病毒基因组DNA/RNA快速抽提试剂盒为Axygen生物科技有限公司产品;反转录试剂盒(PrimeScript RT reagent Kit)均为大连宝生物工程有限公司产品;DL 500 DNA Marker、2×TaqPCR Master Mix购自北京天根生化科技有限公司。
1.4 引物的设计与合成
根据GenBank上登录的猪流行性腹泻病毒弱毒疫苗株CV777、经典毒株及近些年的变异毒株的ORF3基因、S基因序列,利用Meg Align(DNA Star)和Primer Premier 7.0 软件在ORF3基因缺失片段的两端及S基因变异性最大的区域设计两对特异性引物,分别命名为ORF3-F、ORF3-R和S-P1、S-P2,引物序列详见表1。引物ORF3-F、ORF3-R扩增PEDV的ORF3基因片段,变异毒株和经典毒株预期目的片段大小为234 bp,弱毒疫苗株预期目的片段大小为185 bp;引物S-P1、S-P2仅能扩增变异毒株,产生预期目的片段大小为826 bp。上述引物由大连宝生物工程有限公司合成。

表1 用于扩增ORF3基因和S基因片段的引物序列
1.5 粪便样品的处理及病毒RNA的提取
将采集的粪便样品、肠道内容物用PBS(0.01 mol/L,pH 7.2)制成10 %的悬液,涡旋震荡后,4 ℃条件下10 000 r/min离心10 min后收集上清,进行RNA抽提或于-20 ℃储存备用。按照AxyPrep Body Fluid Viral DNA/RNA Miniprep Kit使用说明书对PEDV变异毒株、经典毒株、弱毒疫苗株细胞毒病毒液及粪便样品、肠道内容物上清进行RNA提取,所提取RNA直接进行RT-PCR或置于-20 ℃保存。
1.6 反转录-聚合酶链式反应(RT-PCR)
反转录合成cDNA:按照RT(反转录)试剂盒说明,采用20 μl体系:RNA 模板14 μl,5×PrimeScript TM缓冲液4 μl,RT Enzyme Mix 1.0 μl,反转录引物Oligo18T 1.0 μl,按以下程序进行反转录反应:37 ℃ 20 min,85 ℃ 30 s,12 ℃保存。PCR反应体系25 μl,灭菌水7.5 μl,2×TaqPCR Master Mix 12.5 μl,引物ORF3-F、ORF3-R和S-P1、S-P2各0.5 μl,模板3.0 μl。按照下列条件进行扩增:95 ℃预变性3 min;94 ℃变性40 s,52 ℃复性40 s,72 ℃延伸50 s,40个循环;最后72 ℃延伸8 min。取扩增产物于10 g/L琼脂糖凝胶中电泳,并观察结果。
1.7 特异性试验
分别取猪繁殖与呼吸综合症病毒(PRRSV)、猪嵴病毒(PKV)、猪传染性胃肠炎病毒(TGEV)、猪流行性腹泻病毒(PEDV)、A群猪轮状病毒(PRoVA)、猪瘟病毒(CSFV)、猪乙型脑炎病毒(JEV)的RNA反转录产物以及猪细小病毒(PPV)、猪伪狂犬病毒(PRV)、猪圆环病毒2型(PCV-2)、猪细环病毒(TTV)的DNA为模板,用所建立的方法进行扩增,以验证该方法的特异性。
1.8 敏感性试验
分别提取PEDV变异毒株、经典毒株、弱毒疫苗株细胞毒病毒液的RNA,用紫外分析仪测定其浓度并将不同毒株的RNA调整为相同浓度,然后用RNA-Free Water连续10倍倍比稀释成9个稀释度,取各个稀释度作为模板按上述体系及最适反应条件进行RT-PCR反应,以确定该方法的敏感性。
1.9 重复性及稳定性试验
用所建立的多重RT-PCR鉴别检测方法分别对PEDV变异毒株、经典毒株和弱毒疫苗株混合物,以及单纯的变异毒株、经典毒株和弱毒疫苗株,PEDV阳性病料和阴性病料的各5份重复检测3次,以验证本方法的重复性和稳定性。
1.10 临床样品的检测
利用本研究建立的多重RT-PCR鉴别检测方法对来自广西南宁、柳州、桂林、贵港、玉林、崇左等地养猪场发生腹泻的仔猪粪便样品(91份)进行PEDV检测,统计当前腹泻病例中PEDV变异毒株、经典毒株、弱毒疫苗株所占比例。
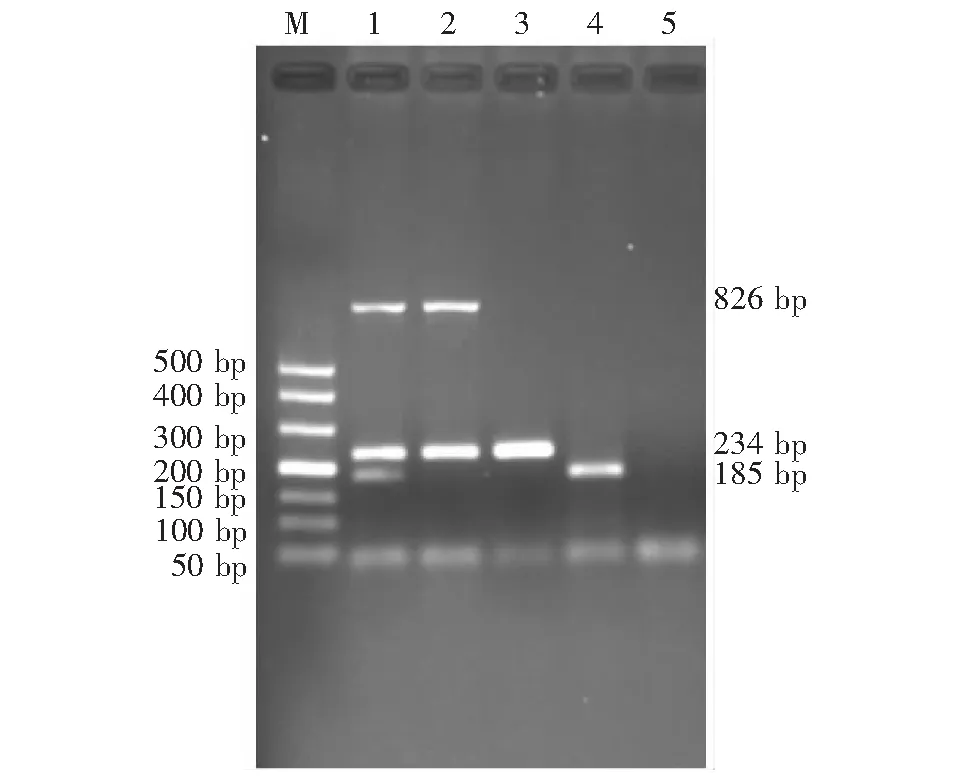
M:DNA分子质量标准;1:PEDV变异毒株、经典毒株、弱毒疫苗株混合物;2:PEDV变异毒株;3:PEDV经典毒株;4:PEDV弱毒疫苗株;5:阴性对照M:DL500 DNA Marker; 1:PEDV mixture of variant, classical and attenuated strains; 2:PEDV variant strain; 3:PEDV classical strain; 4:PEDV attenuated strain CV777; 5:Negative control图1 目的基因的RT-PCR扩增Fig.1 RT-PCR amplification of target gene
2 结果与分析
2.1 多重RT-PCR 结果
所建立的多重RT-PCR方法扩增PEDV变异毒株、经典毒株和弱毒疫苗株混合物获得3条目的片段,分别为826、234和185 bp;扩增PEDV变异毒株获得2条目的片段,分别为片段826和234 bp;扩增PEDV经典毒株获得1条目的片段,为234 bp;扩增PEDV弱毒疫苗株获得1条目的片段,为185 bp;均与预期的目的条带数量和片段大小相符,电泳结果详见图1。
2.2 多重RT-PCR 最佳退火温度
为获得该反应最佳扩增效果,对多重PCR扩增的条件进行了摸索和调整。梯度性地改变多重PCR反应的退火温度,以确定最佳的退火温度。结果显示,退火温度在49.0-52.5 ℃均可以扩增出预期的目的片段(图2),且以50.4 ℃为最佳。最终确定多重PCR的最佳扩增条件为95 ℃预变性,3 min;94 ℃变性40 s,50.4 ℃退火45 s,72 ℃延伸50 s,40个循环;最后72 ℃延伸8 min。
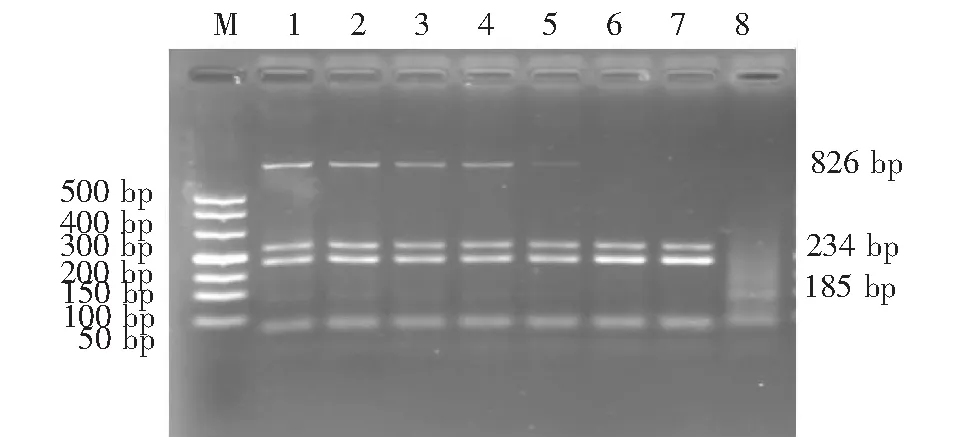
M:DNA分子质量标准;1:49.0 ℃;2:49.6 ℃;3:50.4 ℃;4:51.6 ℃;5:52.5 ℃;6:53.4 ℃;7:54.3 ℃;8:阴性对照M:DL500 DNA Marker;1:49.0 ℃;2:49.6 ℃;3:50.4 ℃;4:51.6 ℃;5:52.5 ℃;6:53.4 ℃;7:54.3 ℃;8:Negative control图2 多重RT-PCR的不同退火温度Fig.2 Multi-PCR at different annealing temperature

M:DNA分子质量标准;1:2.3 ng/μl;2:2.3×10-1 ng/μl;3:2.3×10-2 ng/μl;4:2.3×10-3 ng/μl;5:2.3×10-4 ng/μl;6:2.3×10-5 ng/μl;7:2.3×10-6 ng/μl;8:2.3×10-7 ng/μl;9:2.3×10-8 ng/μl;10:H2OM:DL500 DNA Marker; 1:2.3 ng/μl; 2:2.3×10-1 ng/μl; 3:2.3×10-2 ng/μl; 4:2.3×10-3 ng/μl; 5:2.3×10-4 ng/μl; 6:2.3×10-5 ng/μl; 7:2.3×10-6 ng/μl; 8:2.3×10-7 ng/μl; 9:2.3×10-8 ng/μl; 10:H2O图3 多重RT-PCR的敏感性试验Fig.3 The sensitivity test of multi-RT-PCR
2.3 敏感性试验
3种不同毒株的PEDV病毒液的RNA提取物,经浓度测定后调整为相同的浓度,再将相同的浓度3种RNA等量混合,每种毒株RNA的浓度均为2.3 ng/μl,然后取RNA-Free Water 10倍倍比稀释物为模板,用所建立多重RT-PCR方法进行扩增,并以ddH2O作阴性对照。结果表明,该方法可以同时检测出最低浓度为2.3×10-4ng/μl的3种毒株的猪流行性腹泻病毒的RNA(图3)。
2.4 特异性试验
试验结果(图4)显示,本研究建立的RT-PCR方法能特异地检测出PEDV变异毒株、经典毒株、弱毒疫苗株混合物及单纯的猪流行性腹泻病毒变异毒株、经典毒株、弱毒疫苗株,而对猪传染性胃肠炎病毒、A群猪轮状病毒、猪嵴病毒、猪伪狂犬病毒、猪瘟病毒、猪繁殖与呼吸综合症病毒、猪细小病毒、猪圆环病毒2型、猪乙型脑炎病毒、猪细环病毒的检测结果均无条带,表明所建立的方法具有较好的特异性。
2.5 重复性试验
用本研究建立的多重RT-PCR鉴别检测方法对PEDV猪流行性腹泻病毒变异毒株、经典毒株和弱毒疫苗株混合物,以及单纯的变异毒株、经典毒株和弱毒疫苗株,PEDV阳性病料和阴性病料各5份重复检测3次,结果所有样品3次检测结果均一致,表明该方法稳定可靠,重复性好。

M:DNA分子质量标准;1:PEDV变异毒株、经典毒株、弱毒疫苗株混合物;2:PEDV变异毒株;3:PEDV经典毒株;4:PEDV弱毒疫苗株;5:猪传染性胃肠炎病毒;6:A群猪轮状病毒;7:猪嵴病毒;8:猪伪狂犬病毒;9:猪瘟病毒;10:猪细小病毒;11:猪繁殖与呼吸综合症病毒;12:猪圆环病毒2型;13:猪乙型脑炎病毒;14:猪细环病毒;15:阴性对照M:DL500 DNA Marker; 1:PEDV mixture of variant, classical and attenuated strains; 2:PEDV variant strain; 3:PEDV classical strain; 4:PEDV attenuated strain CV777; 5:TGEV; 6:PRoVA; 7:PKV; 8:PRV; 9:CSFV; 10:PPV; 11:PRRSV; 12:PCV-2; 13:JEV; 14:TTV; 15:Negative control图4 多重RT-PCR的特异性试验Fig.4 The specificity test of multi-RT-PCR for PEDV
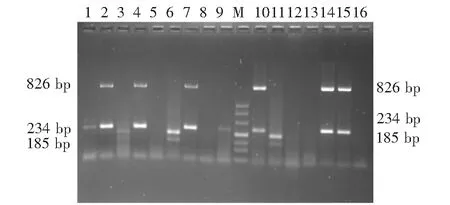
M:DNA分子质量标准;1~14:临床样品 ;15:阳性对照(PEDV变异毒株);16:阴性对照M:DL500 DNA Marker; 1-14:Clinical samples; 15:Positive control(PEDV variant strain); 16:Negative control图5 临床样品检测Fig.5 The test of RT-PCR for PEDV clinical samples
2.6 临床样品的检测
对采集的91份粪便样品,用本研究建立的鉴别诊断RT-PCR方法进行PEDV检测(图5)。结果显示,其中57份样品为PEDV变异毒株阳性,有3份样品为PEDV经典毒株阳性,此外有4份样品为PEDV弱毒疫苗株阳性;PEDV阳性样品共64份。PEDV变异毒株占总PEDV毒株的89.06 %(57/64),经典毒株占总PEDV毒株的4.69 %(3/64),弱毒疫苗株占总PEDV毒株的6.25 %(4/64),变异毒株是当前猪流行性腹泻的优势毒株。
3 讨 论
早在1973年我国就出现了PEDV的病例,随后研制了油佐剂灭活疫苗并在全国范围的养猪场中应用,在预防和控制PED中起到了积极的作用。2010年以前,我国猪群PEDV感染率相对较低,仅是散在发生。2010年底以来,PED在我国主要养猪地区持续暴发。当前,我国流行的PEDV主要有经典毒株和变异毒株两种类型,且以变异毒株为优势流行毒株,致病力较经典毒株更强[16-17,19]。基于S基因的遗传进化分析显示,经典毒株与我国早期的毒株亲缘关系密切;变异毒株与2009年前后韩国流行毒株的分子特征较为接近,据此推测我国PEDV变异毒株可能来源于韩国[16]。2013年5月以来美国猪群暴发PED疫情,猪群中也存在两种毒株类型,高毒力(highly virulent)PEDV毒株和致病力较低的PEDV变异(variant)毒株,并以高毒力毒株为优势毒株[20-21]。美国流行的高毒力PEDV毒株与中国PEDV流行的变异毒株(AH2012株)有密切的亲缘关系,它们很可能源自中国[22]。美国流行的PEDV变异毒株与该国最先出现的高毒力PEDV毒株相比,在S基因的N端有核苷酸插入和缺失且致病力较低,所以称为S基因插入缺失(S INDEL)毒株或者变异毒株[20];其S基因的分子特征与我国流行的经典毒株(CH/HBQX/10)相近,遗传关系较为接近[23]。PEDV通过何种途径从亚洲大陆传播到北美大陆仍然是个谜。最近的研究发现,实验猪只在急性感染PEDV后栏舍内空气颗粒物中可以检测到较高浓度的PEDV RNA,这提示了PEDV可以通过气溶胶传播[24]。
PED对亚洲国家的养猪业造成了一定的危害,韩国、日本及中国均培育开发了弱毒疫苗用于PED的防控。弱毒疫苗的广泛使用,可能引起PEDV疫苗毒在免疫猪场的循环存在。Li等人研究显示在我国发生PED的部分猪场PEDV毒株的S基因与DR13弱毒疫苗株高度同源[16]。Chen等人针对我国福建地区PEDV的分子流行病学研究中发现P55株与弱毒疫苗株在ORF3基因有一致的长片段核苷酸缺失,与弱毒疫苗株的亲缘关系最为接近[25]。这些与弱毒疫苗株高度亲近的类弱毒株,究竟是残留的弱毒疫苗毒还是重组演化的病毒需要全基因序列的测定及动物致病性试验来进行证实。
PEDV ORF3基因缺失49个核苷酸的是弱毒疫苗株区分于野毒株的重要分子标记,根据这一特征可以用RT-PCR方法对PEDV弱毒疫苗株和野毒株进行鉴别诊断[26]。S基因N端15个核苷酸的插入和6个核苷酸的缺失是区分PEDV变异毒株与经典毒株的主要分子特征,秦毅斌等基于S基因的差异建立了检测PEDV变异毒株与经典毒株的RT-PCR鉴别检测方法[27]。本研究在此前的基础上,建立了可以检测并鉴别3种PEDV毒株类型的多重RT-PCR方法,在一个反应管中同时扩增ORF3基因和S基因片段,通过电泳条带的数量和片段大小,即可判断所检测到的PEDV是变异毒株、经典毒株还是弱毒疫苗株。该方法有良好的特异性,可以扩增出PEDV不同毒株或者混合物的预期片段;而对本试验中其他的几种病毒无交叉反应。此外,该试验有高度的敏感性,可以检测到低至2.3×10-4ng/μl的PEDV RNA。
利用所建立的PEDV鉴别诊断RT-PCR方法对临床腹泻粪便样品检测结果显示,变异毒株占总PEDV毒株的89.06 %,是当前猪流行性腹泻的优势毒株。这一结果和国内外其他研究团队所获得的数据是相似的。南京农业大学Zhao等人根据S基因的差异建立了区别PEDV变异毒株和经典毒株的多重TaqMan实时荧光定量RT-PCR检测方法,并对42份腹泻仔猪的肠道内容物进行检测,从其中的36份样品当中检测到PEDV变异毒株,另外有3份样品PEDV经典毒株为阳性[19]。美国俄亥俄州农业部门动物疫病诊断实验室Wang等人也建立了针对美国PEDV高毒力毒株和变异毒株的二重实时荧光定量RT-PCR方法,对295份基于M基因的实时荧光RT-PCR检测为阳性的样品进行了进一步的鉴别诊断,其中250份为PEDV高毒力毒株,45份为变异毒株,并且对检测为变异毒株的样品还通过S基因N端约1100 bp的序列测定再一次的认证[21]。本试验在4份临床样本中检测到ORF3基因存在缺失的PEDV弱毒株,这些样本来自有猪流行性腹泻弱毒疫苗免疫史的2个猪场;使用疫苗前腹泻发病率在80 %以上,免疫之后哺乳仔猪腹泻发病率约为40 %,死亡率有所降低。使用PEDV弱毒疫苗后可能造成疫苗毒的残留或引起疫苗毒株与野毒株的重组,从而加剧病毒变异,应该引起关注。
综上所述,本研究建立了一种可以检测和区分PEDV变异毒株、经典毒株和弱毒疫苗株的多重RT-PCR方法,为PEDV的流行病学及鉴别诊断研究提供了一种特异的、敏感的、快速高效的检测手段。
[1]DEBOUCK P, PENSAERT M. Experimental infection of pigs with a new porcine enteric coronavirus, CV777[J]. AM J Vet Res, 1980, 41(2):219-223.
[2]DEBOUCK P, PENSAERT M, COUSSEMENT W. The pathogenesis of an enteric infection in pigs, experimentally induced by coronavirus-like agent, CV777[J]. Vet Microbiol, 1981, 6(2):157-165.
[3]PENSAERT M B, DEBOUCK P. A new coronavirus-like particle associated with diarrhea in swine[J]. Arch Virol, 1978, 58(3):243-247.
[4]CHASEY D, CARTWRIGHT S F. Virus-like particles associated with porcine epidemic diarrhea[J]. Res Vet Sci, 1978, 25(2):255-256.
[5]SUEYOSHI M, TSUDA T, YAMAZAKI K, et al. An immunohistochemical investigation of porcine epidemic diarrhea[J]. J Comp Pathol, 1995, 113(1):59-67.
[6]PRANAVEJA S, POOLPERM P, LERTWATCHARASARAKUL P, et al. Chinese-like strain of porcine epidemic diarrhea virus, Thailand[J]. Emerg Infect Dis, 2009, 5(7):1112-1115.
[7]PARK S J, KIM H K, SONG D S, et al. Molecular characterization and phylogenetic analysis of porcine epidemic diarrhea virus(PEDV) field isolates in Korea[J]. Arch Virol, 2011, 156(4):577-585.
[8]DUY D T, TOAN N T, PURANAVEJA S, et al. Genetic characterization of porcine epidemic diarrhea virus(PEDV) isolates from Southern Vietnam during 2009-2010 outbreaks[J]. Thai J Vet Med, 2011, 41(1):55-64.
[9]KOCHERHANS R, BRIDGEN A, ACKERMANN M, et al. Completion of the porcine epidemic diarrhea coronavirus(PEDV) genome sequence[J]. Virus Genes, 2001, 23(2):137-144.
[10]DUARTE M, LAUDE H. Sequence of the spike protein of the porcine epidemic diarrhea virus[J]. J Gen Virol, 1994, 75:1195-1200.
[11]CRUZ D J, KIM C J, SHIN H J. The GPRLQPY motif located at the carboxy-terminal of the spike protein induces antibodies that neutralize porcine epidemic diarrhea virus[J]. Virus Res, 2008, 132(1-2):192-196.
[12]BALLESTEROS M L, SáNCHEZ C M, ENJUANES L. Two amino acid changes at the N-terminus of transmissible gastroenteritis coronavirus spike protein result in the loss of enteric tropism[J]. Virology, 1997, 227(2):378-388.
[13]KWON H M, SAIF L J, JACKWOOD D J. Field isolates of transmissible gastroenteritis virus differ at the Molecular level from the Miller and Purdue virulent and attenuated strains and from porcine respiratory coronavirus[J]. J Vet Med Sci, 1998, 60(5):589-597.
[14]WANG K, LU W, CHEN J, et al. PEDV ORF3 encodes an ion channel protein and regulates virus production[J]. FEBS Letters, 2012, 586:384-391.
[15]PARK S J, MOON H J, LUO Y, et al. Cloning and further sequence analysis of the ORF3 gene of wild-and attenuated-type porcine epidemic diarrhea viruses[J]. Virus Genes, 2008, 36(1):95-104.
[16]LI W, LI H, LIU Y, et al. New variants of porcine epidemic diarrhea virus, China, 2011[J]. Emerg Infect Dis, 2012, 18(8): 1350-1353.
[17]CHEN J, LIU X, SHI D, et al. Detection and molecular diversity of spike gene of porcine epidemic diarrhea virus in China[J]. Viruses, 2013, 5(10):2601-2613.
[18]郑逢梅,霍金耀,赵军,等. 2010-2012年华中地区猪流行性腹泻病毒分子特征和遗传进化分析[J].病毒学报,2013,29(2):197-204.
[19]ZHAO P D, BAI J, JIANG P, et al. Development of a multiplex TaqMan probe-based real-time PCR for discrimination of variant and classical porcine epidemic diarrhea virus[J]. J Virol Methods, 2014, 206:150-155.
[20]LIN C M, ANNAMALAI T, LIU X, et al. Experimental infection of a US spike-insertion deletion porcine epidemic diarrhea virus in conventional nursing piglets and cross-protection to the original US PEDV infection[J]. Vet Res, 2015, 46:134.
[21]Wang L, Zhang Y, Byrum B. Development and evaluation of a duplex real-time RT-PCR for detection and differentiation of virulent and variant strains of porcine epidemic diarrhea viruses from the United States[J]. J Virol Methods, 2014, 207:154-157.
[22]HUANG Y W, DICKERMAN A W, PINEYRO P, et al. Origin, evolution, and genotyping of emergent porcine epidemic diarrhea virus strains in the United States[J]. MBio, 2013, 4(5):e00737-13.
[23]WANG L, BYRUM B, ZHANG Y. New variant of porcine epidemic diarrhea virus, United States, 2014[J]. Emerg Infect Dis, 2014, 20(5):917-919.
[24]ALONSO C, RAYNOR P C, DAVIES P R, et al. Concentration, Size Distribution, and Infectivity of Airborne Particles Carrying Swine Viruses[J]. PLoS One, 2015, 10(8):e0135675.
[25]CHEN X, ZENG L, YANG J, et al. Sequence Heterogeneity of the ORF3 Gene of Porcine Epidemic Diarrhea Viruses Field Samples in Fujian, China, 2010-2012[J]. Viruses, 2013(5):2375-2383.
[26]吴玉璐,程群,虞凌雪,等. 猪流行性腹泻病毒RT-PCR鉴别诊断方法的建立[J]. 中国农业科学,2013,46(20):4370-4377.
[27]秦毅斌,卢冰霞,赵 武,等. 猪流行性腹泻病毒变异毒株与经典毒株RT-PCR鉴别检测方法的建立及应用[J]. 中国兽医科学,2014,4(5):509-514.
(责任编辑 温国泉)
Development and Application of Multiplex RT-PCR Method for Differentiation of Variant, Classical and Attenuated Vaccine Strains of Porcine Epidemic Diarrhea Viruses
QIN Yi-bin, LU Bing-xia, DUAN Qun-peng, HE Ying, LI Bin, LIANG Jia-xing, SU Qian-lian,ZHOU Ying-ning, JIANG Dong-fu, LU Jing-zhuan, ZHAO Wu*
(Guangxi Veterinary Research Institute, Guangxi Key Laboratory of Animal Vaccines and New Technology, Guangxi Nanning 530001, China)
In order to establish a rapid multiplex RT-PCR assay for detection and differentiation of variant, classical and attenuated vaccine strains of porcine epidemic diarrhea virus (PEDV). Two pairs of specific primers were designed based on S gene sequences and ORF3 gene sequences of variant, classical and attenuated vaccine strains PEDV published in GenBank, to amplify the partial S gene fragment and ORF3 gene fragment of different PEDV strains, the type of PEDV strain could be differentiated according to the number of the fragment and the size of the fragment. Results showed that the multiplex RT-PCR could detect and differentiate variant, classical and attenuated vaccine strains PEDV. Variant PEDV strain generated two fragments, they were 826 bp of the S gene and 234 bp of the ORF3 gene, respectively. Classical PEDV strain only amplified 234 bp of the ORF3 gene, while attenuated vaccine strain produced 185 bp of the ORF3 gene. The multiplex RT-PCR did not cross-react with TGEV, PRoVA, PKV, PRRSV, PRV, CSFV, PCV-2, JEV, TTV and PPV used in the study. Testing of the sensitivity of RT-PCR indicated as low as 2.3×10-4ng/μl nuclear acids could be detected accurately and rapidly. Ninety-one stool specimens collected from different farms in Guangxi Province were detected by the established multiplex RT-PCR, 64 samples were positive for PEDV, of which 89.06 %(57/64) was variant PEDV strains, 4.69 %(3/64) was classical PEDV strain and 6.25 %(4/64) was attenuated vaccine PEDV strain. Therefore the multiplex RT-PCR could be used as an effective tool for differentiating diagnosis of PEDV in epidemiological investigations.
PEDV; Variant strain; Classical strain; Attenuated vaccine strain; RT-PCR; Differential diagnosis
1001-4829(2016)11-2746-06
10.16213/j.cnki.scjas.2016.11.043
2016-06-23
广西水产畜牧科技推广应用项目(桂渔牧科201633 044,201633041,201633034);南宁市西乡塘区科学研究与技术开发计划项目(2014304)
秦毅斌(1983-),男,广西临桂人,硕士,助理研究员,主要从事动物传染病病原与分子生物学研究,E-mail:qinyibin5188@163.com,*为通讯作者,E-mail:zhaowu168866@163.com。
S858.28
A

