基于CO1基因的渔山列岛厚壳贻贝遗传资源评估
袁文斌, 李长保, 焦海峰, 林志华, 包永波
(1. 浙江万里学院, 浙江 宁波 315100; 2. 宁波市海洋与渔业研究院, 浙江 宁波 315012)
厚壳贻贝(Mytiluscoruscus)属于双壳纲(Bivalvia),翼形亚纲(Pteriomorphia), 贻贝目(Mytioida)。也称淡菜, 海红, 中国古书上还称之为“东海夫人”[1], 主要分布于黄、渤海和东海沿岸[2], 野生厚壳贻贝多分布在外侧海岛低潮线下至 20 m之间的海底礁岩上[3],厚壳贻贝(Mytiluscoruscus)是渔山列岛重点保护的贝类资源之一, 也是当地重点采捕的种类之一, 由于其世代更替速度较慢, 大规模、高强度的采捕势必造成贻贝资源的减少。本文运用CO1基因分析对渔山列岛代表性潮间带生物厚壳贻贝进行遗传学研究,对五个地理群体(渔山、山东、大连、舟山、南麂)进行遗传多样性和单倍性分析等, 构建反映群间体关系的系统进化树, 观察不同群体的遗传多样性,探讨野生群体遗传多样性之间的差异, 从分子水平了解渔山列岛厚壳贻贝遗传背景。通过本课题的研究, 为渔山列岛其他海洋生物的恢复和保护提供了借鉴。
1 材料与方法
1.1 实验材料来源
贻贝: 5个地点的野生厚壳贻贝: 渔山列岛、大连獐子岛、南麂列岛、山东南隍城乡、舟山嵊泗。以上厚壳贻贝采集后活体运送至实验室, 取闭壳肌放于无水乙醇中, –20℃保存备用。
1.2 实验步骤
1.2.1基因组DNA的提取
用酚-氯仿法抽提各样品DNA。检测DNA完整性后, 放入–20℃保存备用。
1.2.2PCR扩增
采用常规PCR, 反应体系为25 μL, 其中12.5 μL taq master mix, 9 μL 双蒸水, 1.5 μL 模板, 上下引物各1 μL。PCR程序为: 94℃预变性10 min; 循环过程为: 94℃ 30 s, 55℃ 30 s, 72℃ 90 s, 35 个循环; 72℃延伸10 min, 引物序列为: CO1-F(5′-GGTCAACAAA TCATAAAGATATTGG-3′), CO1-R(5′-TAAACTTCA GGGTGACCAAAAAATCA-3′)。
1.2.3PCR产物鉴定
PCR产物在1%琼脂糖中电泳检测, 700 bp处出现单一亮条带则证明扩增成功。

表1 5个厚壳贻贝群体样品信息Tab. 1 Messages for sample collection
1.2.4扩增产物纯化及序列测定
本部分工作由上海桑尼公司完成。
1.2.5数据分析
使用Bioedit, Cexpress等软件进行序列比对, 并辅以人工校对, 并抹平单碱基差异。使用 DnaSP软件分析碱基组成、单倍型状况和遗传多样性指数, 使用MEGA7.0软件分析遗传距离, 使用MEGA7.0软件的 NJ方法构建分子系统树, 计算系统分支遗传差异程度。使用DnaSP转换格式后使用Network软件辅以人工绘制单倍型网络关系图, 绘出碱基差异数图。
2 结果与分析
测序结果通过 Cexpress与 MEGA软件处理后,获得用于分析的序列, 渔山列岛群体 30条, 大连獐子岛群体26条, 南麂列岛群体39条, 山东南隍城乡群体16条, 舟山嵊泗群体45条, 共156条序列, 统一截取576 bp的CO1序列。
在 mtDNACO1基因同源片段上共检测到了 39个多态位点, 其中 39个简约信息位点, 无单突变位点, 构成40个单倍型, 其中16个共享单倍型, 24个特有单倍型(表 2), Network绘制的碱基不配对分析图中可以看到每个单倍型间的核苷酸差异数(图1), 5个群体总的单倍型多样性为 0.888±0.014, 核苷酸多样性为 0.00558±0.00049, 两两对比的碱基差异度为3.216±1.669。基因片段中的A+T含量(60.5%)明显大于C+G的含量(39.5%)。
基于mtDNACO1基因同源性片段获得的遗传多样性结果显示, 40个单倍型中, 有4个单倍型为5个群体的共享单倍型, 特有单倍型居多, 单倍型共享率为 40.0%。就核苷酸多样性指数和两两对比的碱基差异度而言, 渔山群体(π=0.00890±0.00193, k=5.126±2.556)> 山 东 群 体 (π=0.00535±0.00062, k=3.083±1.691)>大连群体(π=0.00503±0.00051, k=2.895±1.571)>舟山群体(π=0.00442±0.00038, k=2.547±1.395)>南麂群体(π=0.00427±0.00028, k=2.459±1.359); 但是单倍型多样性却是山东群体(h=0.942±0.048)>渔山群体(h=0.929±0.023)>南麂群体(h=0.883±0.031)>大连群体(h=0.868±0.042)>舟山群体(h=0.855±0.033)。

表2 厚壳贻贝5个群体的单倍型频率分布Tab. 2 Distribution of haplotypes in five Mytiluscoruscus populations

图1 厚壳贻贝单倍型间碱基不配对分析图Fig. 1 Mismatch distributions analyses of the nucleotide of CO1 gene for Mytiluscoruscus haplotypes
基于mtDNACO1基因片段的群体间遗传距离构建了5个群体之间的NJ系统树(图2), 看到渔山列岛群体与其他 4个群体的遗传差异较大, 山东群体与大连群体间的关系较近, 渔山群体与南麂群体, 舟山群体的关系较近。表 4中可以看出群体内遗传距离为: 渔山(0.0089)>山东(0.0054)>大连(0.0050)>舟山(0.0044)>南麂(0.0043)(表 4)结果显示渔山与其他四个群体间的遗传距离较大。
基于 mtDNACO1基因同源性片段用 MEGA7.0软件构建了单倍型NJ系统树, 系统发育结果显示, 5个群体内存在三个显著分化的单倍型类群 A、B、C, 其中A类群含15个单倍型, B类群含23个单倍型, C类群含2个单倍型(均为渔山列岛的样品), 其中渔山列岛在上述三个类群中都有分布。使用Network软件绘制的单倍型中介连接网络关系图与 MEGA7.0构建的NJ树结果一致(图3-4)。类群间的遗传距离为0.0099~0.0268(A/B=0.0099, A/C=0.0268, B/C=0.0208)(图 3)。

表3 厚壳贻贝5个群体的遗传多样性指数Tab. 3 Molecular diversity indices of five Mytiluscoruscus populations
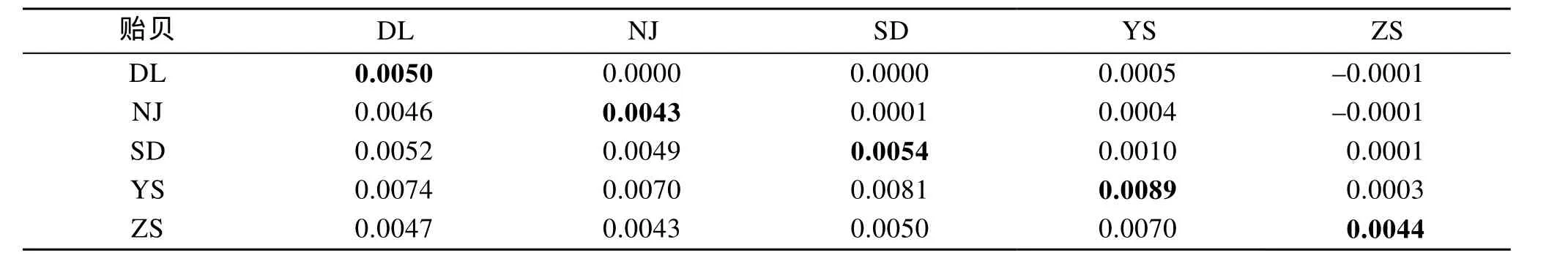
表4 基于CO1基因片段序列的5个厚壳贻贝群体间遗传距离(对角线下)、群体内遗传距离(粗体显示)和群体间平均净遗传距离(对角线上)Tab. 4 Genetic distances between populations (below diagonal), genetic distances within populations (in bold values), and genetic net distance between populations (above the diagonal) of the five Mytiluscoruscus populations based on CO1 gene

图2 基于5个群体间遗传距离的NJ系统树Fig. 2 Neighbor-joining tree of five Mytiluscoruscus populations based on genetic distance
基于厚壳贻贝 CO1基因的分歧速率为(0.7%~2.4%/MY)[4]。计算所有单倍型类群的分化时间: A/B的净遗传距离为0.0052, A/C的净遗传距离为0.0149,B/C的净遗传距离为0.0088; 类群A/B的分化事件发生于21~74万年前, 类群A/C的分化事件发生于62~212万年前, 类群 B/C的分化事件发生于 36.67~125.7万年前。上述分化事件的发生主要集中在更新世晚中期。
3 讨论

图3 基于CO1基因的各个单倍型NJ系统树Fig. 3 Neighbor-joining tree of haplotypes for Mytiluscoruscus from CO1 gene
线粒体DNA是一种共价闭合的, 环状的双链分子由于具有高拷贝数, 高置换率和母系遗传等特点,在近十年间已经成为研究动物遗传多样性的重要工具[5-9]。最近有关人类线粒体 DNA的研究文献也屡见不鲜[10]。目前对贻贝的研究方向主要在人工养殖或者遗传学分析[11]上, 也有少许分析形态[12], 研究药理病理[13-14]的方向。研究表明, 海洋贝类的遗传多样性水平的高低受多种因素的影响, 比如洋流, 温度等等。此外, 海洋贝类的自身生活史特征也会印象其遗传多样性。因此, 加大对海洋贝类种群遗传多样性的监测及遗传多样性分布格局的分析, 对于海洋贝类遗传多样性的保护和合理的资源利用有意义。
在多态位点的研究中, 渔山群体拥有较多的多态位点, 有较为丰富的遗传变异。单倍型多样性(h),核苷酸多样性(π), 两两比较的碱基差异度(k)是衡量种群遗传多样性水平的重要参数[15]。本研究中, 渔山群体的核苷酸多样性与两两比较的碱基差异度值要显著大于大连、山东、舟山和南麂群体。而单倍型多样性却是山东群体更高, 可能因为山东群体的个体数较少导致。大连獐子岛与山东南隍城乡地同处渤海黄海交界处附近, 地理位置相隔不远, 南麂列岛、渔山列岛、舟山嵊泗同处浙江沿海, 渔山列岛距大陆最远, 群体 NJ系统树的结果也发现: 渔山列岛群体与其他四个群体之间的遗传距离较远, 相对来说渔山列岛群体与舟山群体以及南麂群体的遗传距离较近; 山东群体与大连群体之间遗传距离较近。推测群体间遗传距离的远近可能与各群体间的相对地理位置的远近有关。综上所述, 虽然渔山列岛不仅与大陆的距离较远, 而且与其他群体之间的遗传距离也较远, 但是却具有较为丰富的遗传多样性水平,可能与渔山列岛厚壳资源丰富, 群体数量较大, 种子资源状况较好有关。
Zuckerkandl和 Pauling[16]提出分子钟假说认为对于任意给定的大分子(蛋白质或DNA分子)在其所有的进化谱系中核苷酸替代速率是近似恒定的。对于 5个群体的各个单倍型做系统发育分析后显示,厚壳贻贝被分为 3个较为明显的类群 A、B、C。4个类群间的净遗传距离为 0.0052~0.0149。采用0.7%~2.4%的分歧速率计算获得 4个类群的分化时间在大概一百万年之前。而那时候的海平面下降的非常剧烈, 海平面下降了120~140 m[17], 边缘海的面积和结构发生了巨大的变化, 从而导致了许多海洋生物的分布范围经历了收缩和扩张, 并在遗传信息上留下明显的印迹[18]。厚壳贻贝是群体附着生长的双壳类, 而且生长于近海, 受此次海洋的大变动的影响应该很大。从而导致了A、B群体和C群体的分化。C群体的两个单倍型是渔山群体的样本, 而其他渔山的单倍型在A、B两个类群里都有渗透, 推测可能是渔山列岛群体在一百万年前隔离后又与其他群体重新扩张混合。

图4 厚壳贻贝单倍型中介连接网络关系图Fig. 4 The median joining network of Mytiluscoruscus haplotypes
参考文献:
[1] 常抗美, 吴剑锋. 厚壳贻贝人工繁殖技术的研究[J].南方水产, 2007, 3: 26-30.Chang Kangmei, Wu Jianfeng. Study on artificial propagation of mussel Mytiluscoruscus[J]. South China Fisheries Science, 2007, 3: 26-30.
[2] 叶莹莹, 徐梅英, 郭宝英, 等. 厚壳贻贝(Mytilus coruscus)4个群体遗传多样性的 ISSR分析[J]. 海洋与湖沼, 2012, 1: 113-119.Ye Yingying, Xu Meiying, Guo Baoying, et al.Thick shell mussel (Mytilus coruscus) analysis of 4 population genetic diversity in ISSR[J]. Oceanologia et Limnologia Sinica, 2012, 1: 113-119.
[3] 杨顺良, 杨璐, 赵东波, 等. 福建台山列岛厚壳贻贝资源调查与评估[J]. 海洋科学, 2015, 39(11): 32-38.Yang Shunliang, Yang Lu, Zhao Dongbo, et al. The investigation and evaluation of Fujian Taishan islands’Mytilus coruscus resources[J]. Marine Sciences, 2015,39(11): 32-38.
[4] 毛阳丽. 贻贝属的系统发育及群体的形态学和遗传学研究[D]. 青岛: 中国海洋大学, 2011, 63-64.Mao Yangli. Phylogenetic groups, Morphological and genetic studies of the genus of Mytilus[D]. Qingdao:The master degree thesis of Ocean University of China,2011: 63-64.
[5] Moritz C, Dowling T E, Borwn W M. Evolution of animal mitochochondrial DNA: Relevance for population biology and systematics[J]. Annual Review of Ecology & Systematics, 1987, 18: 269-292.
[6] Englbrecht C C, Freyhof J, Nolte A, et al. Phylogeography of the bullhead Cottusgobio (Pisces: Teleostei:Cottidae) suggests a pre-Pleistocene origin of the major central European populations[J]. Molecular Ecology,2000, 9(6): 709-722.
[7] Whitehead A, Anderson S L, Kuivila K M, et al. Genetic variation among interconnected populations of Catostomus occidentalis: implications for distinguishing impacts of contaminants from biogeographical structuring[J]. Molecular Ecology, 2003, 12(10): 2817-2833.
[8] Zhou Benjiang, Yang Bingbing, Pham Ngoc Doanh, et al. Sequence analyses of ITS2 and CO1 genes of Paragonimus proliferus obtained in Yunnan province,China and their similarities with those of P. hokuoensis[J]. Parasitology Research, 2008, 102(6): 1379-1383.
[9] 杨振雄, 毛阳丽, 宋娜. 浙江和福建沿海厚壳贻贝Mytilus coruscus群体的CO1序列比较分析[J]. 海洋湖沼通报, 2014, 2: 82-88.Yang Zhenxiong, Mao Yangli, Song Na. Comparative analysis of CO1sequence in Zhejiang and the Fujian coast Mytilus coruscus group. Transactions of Oceanology and Limnology, 2014, 2: 82-88.
[10] Zhen Xiumei, Wu Bailin, Wang Jian.Increased Incidence of Mitochondrial Cytochrome C Oxidase 1 Gene Mutations in Patients with Primary Ovarian Insufficiency[J]. PLoS ONE, 2015, 10(7): 1-8.
[11] Shen Yubang, Li Jiale, Feng Bingbing. Genetic Analysis of cultured and wild populations of Mytilus coruscus based on mitochondrial DNA[J]. Zoological research,2009, 30(3): 240-246.
[12] Vekhova E E. Growth and Shell Morphology of Three Mytilidae (Bivalvia) Species from the Sea of Japan[J].Zoologicheskii Zhurnal, 2013, 92(4): 399-408.
[13] Xu Hongli, Guo Tingting, Guo Yifeng, et al. Characterization and protection on acute liver injury of a polysaccharide MP-I from Mytilus coruscus[J]. Glycobiology, 2008, 18(1): 97-103.
[14] Wu Jinchao, Cheng Jie, Shi Xiaolai. Preparation of ACE inhibitory peptides from Mytilus coruscus hydrolysate using uniform design[J]. Biomed Research International, 2013: 1-6.
[15] 肖永双, 程启群, 任桂静, 等. 中国近海条石鲷(Oplegnathus fasciatus)群体遗传多样性线粒体 DNA序列分析[J]. 海洋与湖沼, 2013, 44(2): 427-432.Xiao Yongshuang, Chen Qiqun, Ren Guijing, et al.China rock bream (Oplegnathus fasciatus) analysis of genetic diversity of mitochondrial DNA sequences[J].Oceanologia et Limnologia Sinica, 2013, 44(2): 427-432.
[16] Zuckerkandl E, Pauling L. Evolving Genes and Proteins[M]. New York: Academic press, 1965, 1965.
[17] Kurt Lambeck, Tezer M Esat, Emma-kate Potter. Links between climate and sea levels for the past three million years[J]. Nature, 2002, 419(6903): 199-206.
[18] Hewitt Godfrey. The genetic legacy of the Quaternary ice ages[J]. Nature, 2000, 405(6789): 907-913.

