长江上游特有鱼类红唇薄鳅线粒体控制区遗传多样性研究
申绍祎,田辉伍,汪登强,陈大庆,刘绍平
(1.西南大学生命科学学院,淡水鱼类资源与生殖发育教育部重点实验室,水产科学重庆市市级重点实验室,重庆 400715;2.中国水产科学院长江水产研究所,农业部长江中上游渔业资源环境科学观测实验站,武汉 430223)
长江上游特有鱼类红唇薄鳅线粒体控制区遗传多样性研究
申绍祎1,田辉伍2,汪登强2,陈大庆2,刘绍平2
(1.西南大学生命科学学院,淡水鱼类资源与生殖发育教育部重点实验室,水产科学重庆市市级重点实验室,重庆 400715;2.中国水产科学院长江水产研究所,农业部长江中上游渔业资源环境科学观测实验站,武汉 430223)
红唇薄鳅(Leptobotiarubrilabris)是长江上游特有鱼类,近年来资源量不断下降。本研究利用线粒体控制区序列片段(896 bp)分析了长江上游红唇薄鳅的遗传多样性及遗传结构,为其资源保护提供科学依据。共采集了119尾样本,来自长江上游江津、四川南溪、岷江下游蕨溪等。结果显示,共检测到58个单倍型,平均单倍型多样性为0.967,平均核苷酸多样性0.006 7,表明红唇薄鳅种群具有较高的遗传多样性。单倍型NETWORK网络关系图和分析系统树结果显示红唇薄鳅单倍型不按地理分布聚类,但是明显分成了2个谱系,表明红唇薄鳅群体发生了同域遗传分化。AMOVA分析结果显示,采样点和采样时间的群体间没有发生遗传分化。中性检验、错配分析及 BSP(Bayesian skyline plot)分析表明红唇薄鳅Lineage 1种群在距今0.007 5~0.055 Ma(百万年)期间发生过种群选择或扩张事件。
红唇薄鳅(Leptobotiarubrilabris);长江上游;线粒体控制区;遗传多样性
红唇薄鳅(Leptobotiarubrilabris)隶属鲤形目(Cypriniformes)鳅科(Cobitidae)沙鳅亚科(Botiinae)薄鳅属(Leptobotia),俗称红鱼、红鱼片儿等,体延长,较高,侧扁,头长,呈锥形,吻较长,唇厚,有许多皱褶。红唇薄鳅为长江上游特有鱼类[1],就个体大小而言,在沙鳅亚科中仅次于长薄鳅。因肉质细腻,味道鲜美,其深受人们欢迎而具有很高的经济价值。红唇薄鳅自然资源量不高,又因过度捕捞等原因,使其资源量明显下降,一些有历史记载的分布区,如嘉陵江、乌江、沱江等现今已难觅踪迹[2-3]。
另一方面,水电梯级开发等原因使其生境不断破碎、萎缩[4-5],进一步威胁红唇薄鳅的生存。红唇薄鳅产漂流性卵,其受精胚胎需要一定长度的流水完成发育过程。当前长江上游干流、岷江和金沙江等是红唇薄鳅主要栖息地和产卵场,但自2012年开始,位于金沙江下游的向家坝水电站正式蓄水,长江上游干支流其它多座水电站也在加紧前期准备,梯级电站的实施开发,将会对长江上游水生生物以及珍稀土著鱼类造成重大影响[6],尤其是产漂流性鱼卵鱼类。因此研究红唇薄鳅群体遗传多样性,对其资源保护及评估水电工程对其遗传结构的影响有重要的意义。目前对红唇薄鳅的研究主要在于资源的调查[7],遗传多样性研究上仅见微卫星标记开发[8]和微卫星遗传多样性分析[9]。
本研究采用线粒体(mt)DNA控制区序列分析采自长江上游红唇薄鳅的群体遗传多样性,研究其遗传结构,以期为红唇薄鳅种质保护和资源恢复提供基础数据,同时为评估和预测梯级电站开发对长江上游水生动物及生态环境产生的影响提供科学依据。
1 材料与方法
1.1 样本采集
样本采自长江上游干流重庆江津江段(JJ)和四川宜宾南溪江段(NX),以及岷江下游宜宾蕨溪镇江段(MJ),共采集红唇薄鳅119尾,其中93尾采于2010-2012年,26尾采于2015-2016年,具体采样信息见表1。所有样本经鉴定后剪取胸鳍条,清洗干净后置入5 mL离心管中以100%乙醇保存密封后带回实验室。
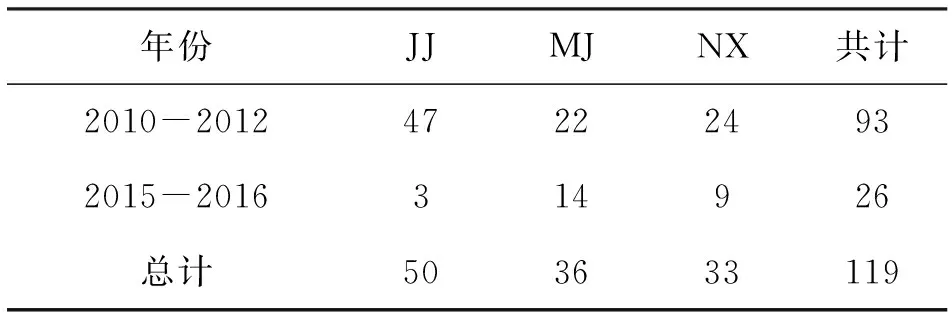
表1 红唇薄鳅采样点及采样数Tab.1 The sample sites information of L.rubrilaris
1.2 基因组 DNA 提取
用高盐法提取基因组DNA[10]。用琼脂糖凝胶电泳检测其基因组DNA的提取质量。
1.3 PCR扩增及测序
线粒体DNA控制区片段的引物序列如下: MitDI-F: 5′-CACCCYTRRCTCCCAAAGCYA-3′和MitDI-R: 5′-GGTGCGGRKACTTGCATGTRTAA-3′。PCR 的扩增体系为50 μL,其中Mix酶(北京天一辉远生物技术有限公司)25 μL、上下游引物各2 μL,DNA模板2 μL,灭菌双蒸馏水补至50 μL.PCR扩增条件:94 ℃ 预变性 3 min;94 ℃ 变性 30 s,55 ℃ 退火 30 s,72 ℃ 延伸 1 min,35 个循环;72 ℃ 再延伸 8 min。PCR产物检测及测序:取2.5 μL PCR反应产物在1.0%的琼脂糖凝胶进行电泳,在凝胶成像系统(Bio-rad)下观察拍照,检测扩增效果,扩增产物送至测序公司进行双向测序。
1.4 数据分析
用 Lasergene v6软件包对测序结果进行拼接校对及对位排列。单倍型数(HN)、变异位点数(S)、简约信息位点数、单倍型多样性指数(Hd)和核苷酸多样性指数(Pi)等参数用DnaSP v5.0[11]计算,同时进行Tajima’D、Fu’s Fs值检验、核苷酸错配分析。单倍型网络结构图在Network 4.6.1.0[12]软件构建,并对各个单倍型进行群体对应关系的分析。
序列的转换/颠换比率、统计其平均碱基组成用MEGA 6.0软件[13]分析,并构建K2-P单倍型系统发育树(NJ树),系统树中节点的自举置信水平应用自引导(Bootstrap) 估计,循环验证次数为1 000。分子变异方差(AMOVA)用Arlequin v3.1[14]软件进行分析,估算遗传变异在群体间和群体内的分布及遗传分化指数(FST),1 000次重复随机抽样重排后分析其统计学显著性。Beast 2.4.3.0进行Bayesian Skyline Plots(BSP)分析[15],采用鱼类线粒体控制区的平均突变率每百万年3.6%[16],MCMC长度为15 000 000,在TRACER ver.1.4构图,各项参数的ESS均大于200。
2 结果与分析
2.1 序列变异及多样性
序列比对后获得119条红唇薄鳅线粒体DNA控制区序列896 bp, A、T、C、G的平均含量分别为36.07%、31.05%、19.75%和13.14%,A+T 含量(67.12%) 远大于 G + C 含量(32.88%),呈现明显的碱基偏向。共检测到46个变异位点,其中10个为单一变异位点、36个为简约信息位点,转换颠换比例为11.23。3个采样点中,变异位点最多的是江津群体(37个),最少的是南溪群体(30个)。种群平均单倍型多样性(Hd)为0.967,平均核苷酸多样性(Pi)0.006 7。单倍型多样性和核苷酸多样性指数均是岷江群体最高(表2),119条序列共定义58个单倍型(Genebank登录号: MF072432-MF072489),其中频率最高的是Hap4,拥有14个个体(11.76%),其次是Hap10,拥有10个个体(8.40%),仅出现1次的单倍型有41个,拥有两个以上个体的单倍型有17个(表3)。江津群体单倍型数最多(29个),南溪群体单倍型数最少(20个)。
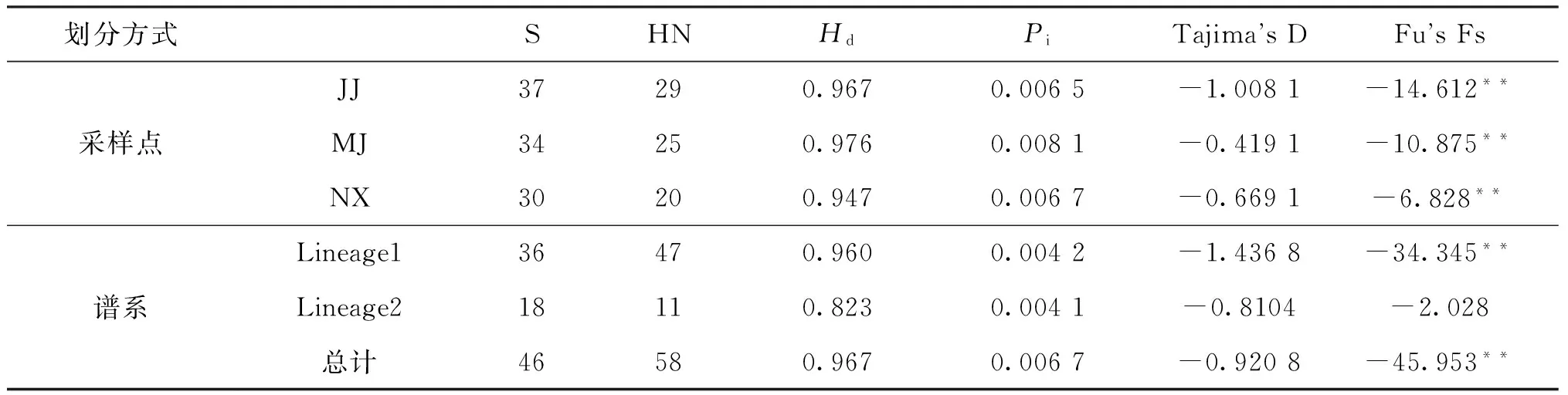
表2 红唇薄鳅种群遗传多样性、Tajima’s D和Fu’s FsTab.2 Genetic diversity parameters and Tajima’s D and Fu’s Fs of L.Rubrilaris population S.多态性位点数,HN.单倍型数,Hd.单倍型多样性,Pi.核苷酸多样性。
注:**表示差异极显著P<0.01。
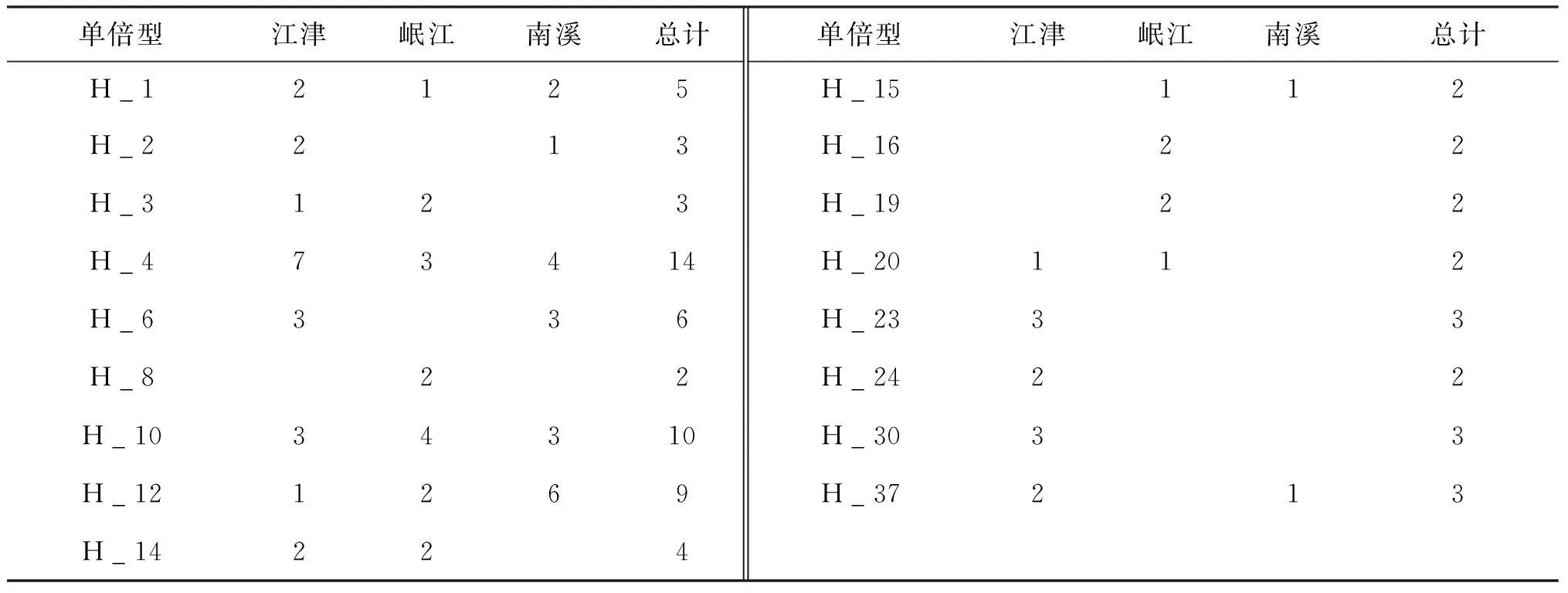
表3 红唇薄鳅单倍型地理分布(仅列频次为2或以上的单倍型)Tab.3 Distribution of haplotype in sampled sites of L.Rubrilaris(Only haplotypes with two or more samples were listed)
2.2 种群遗传分化
单倍型NETWORK网络连接见图1,单倍型关系比较复杂,形成许多环形连接,并且出现21个缺失单倍型(mv1~mv21)。根据连接单倍型之间的突变步长,可将单倍型划分为2个谱系(Lineage 1、Lineage 2),谱系内连接单倍型的突变步长为1~4,谱系间则通过两条步长为6的路径来连接。
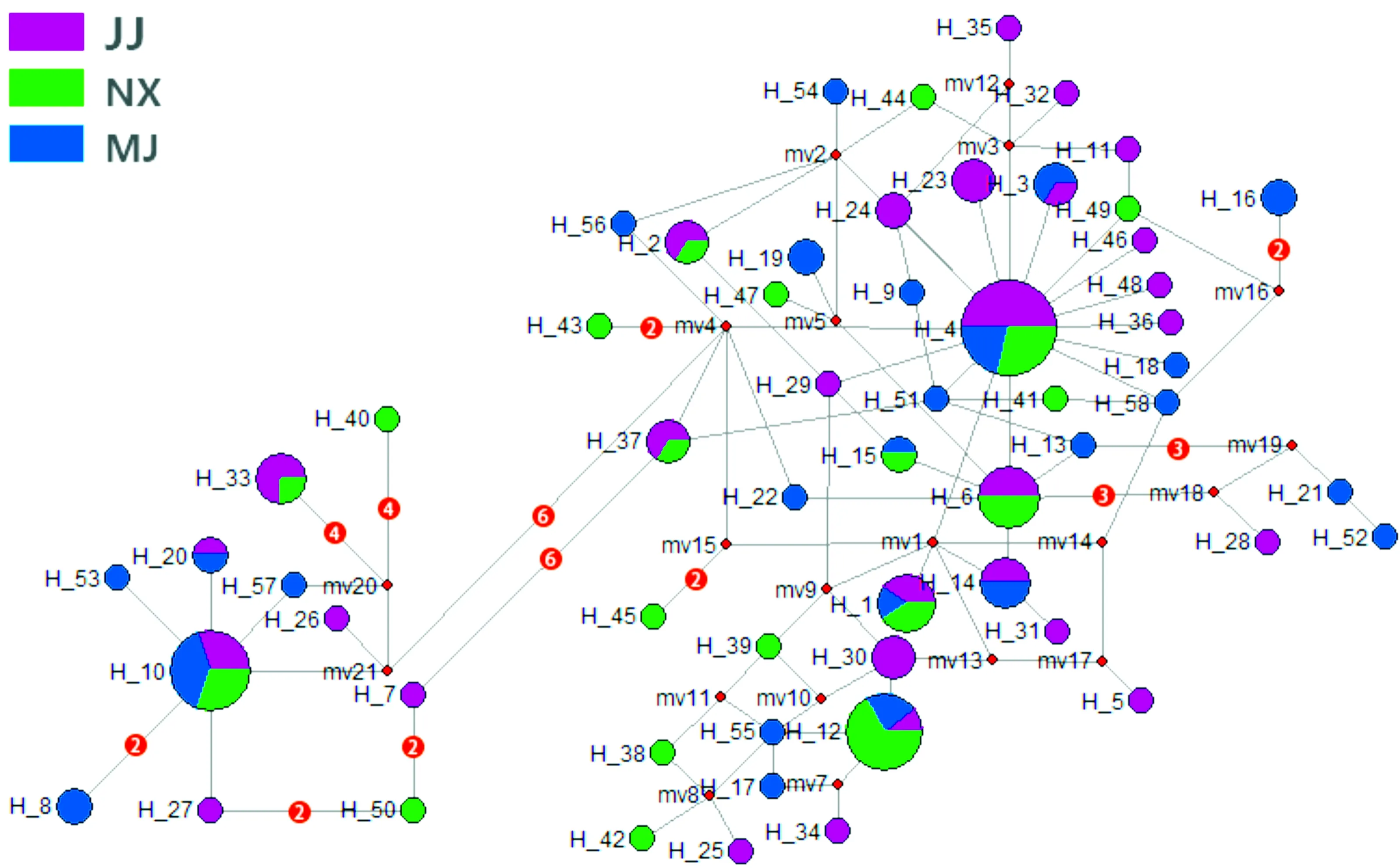
图1 红唇薄鳅线粒体控制区单倍型网络结构
Fig.1 Haplotype network ofL.rubrilabrismtDNA control region
圆圈大小表示单倍型的频率;mv代表缺失的单倍型,橙色数字代表突变步骤
以长薄鳅(L.elongata)为外类群(Genebank登录号: DQ105271.1;AY017144.1)进一步构建单倍型NJ系统发育树(图2)显示,红唇薄鳅群体线粒体控制区形成两个大的分支,支持率为100%。这两大分支所包含的单倍型分别与NETWORK网络结构中所划分两个谱系的单倍型完全一致。两个谱系的样本与采样点没有明显的对应关系,换言之,两个谱系均包含本研究中3个采样点的样本(表4)。
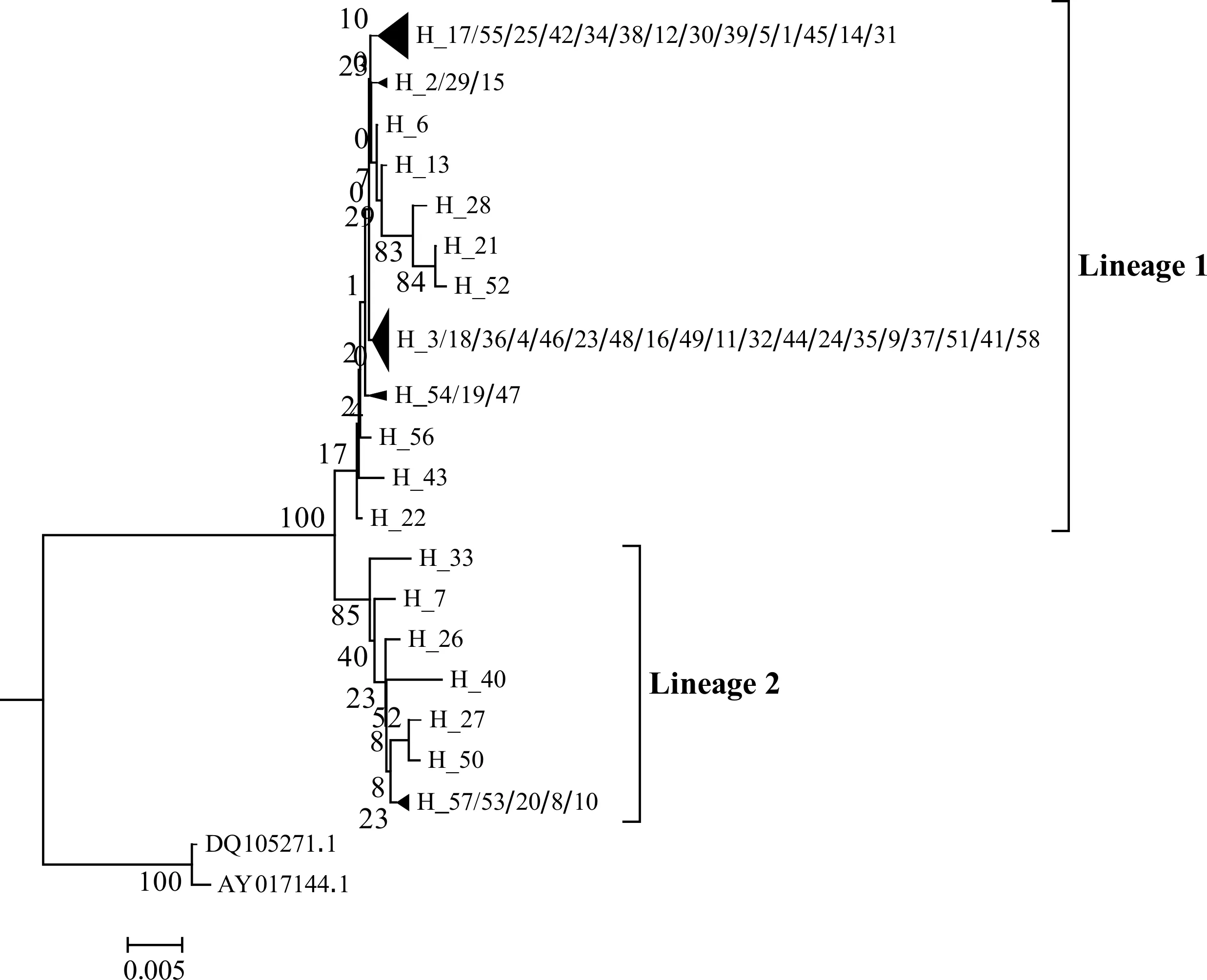
图2 红唇薄鳅线粒体控制区单倍型 NJ 树Fig.2 NJ phylogenetic tree of mt DNA control region in L.rubrilabris
对红唇薄鳅3个采样点群体的控制区序列作分子变异分析(AMOVA),结果显示,群体遗传分化指数FST=0.002 3(P>0.1),群体内的变异占99.77%,变异主要来自群体内(表5),表明红唇薄鳅3个采样点群体间未发生遗传分化。为了检验不同采样时间对遗传结构的影响,本研究对向家坝截流前后样本进行AMOVA分析,结果FST=-0.0068,即向家坝截流前后采集的样本没有发生遗传分化。进一步对基于NETWORK图和系统发育分析划分的2个谱系作AMOVA分析,结果FST=0.659 5(P<0.01)(表5),两谱系间具有显著遗传分化。
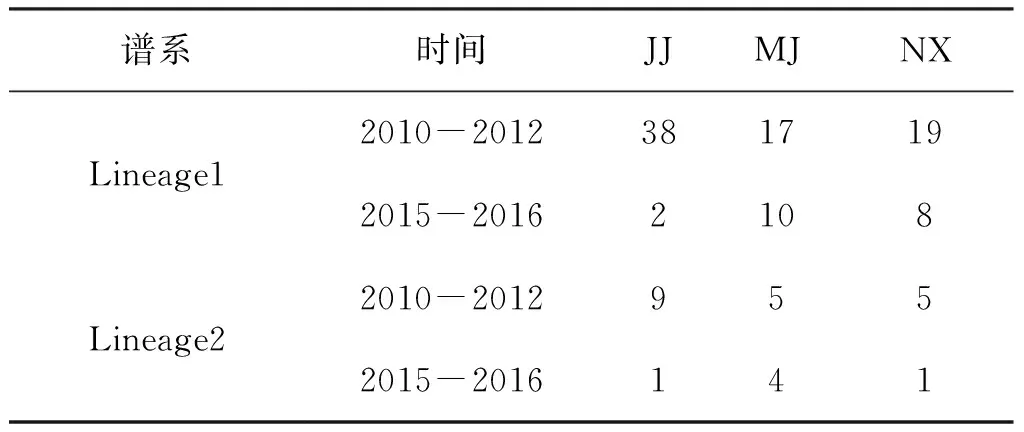
表4 两个谱系个体来源数目统计Tab.4 Statistics of the individuals according to the sourcesfor the 2 lineages
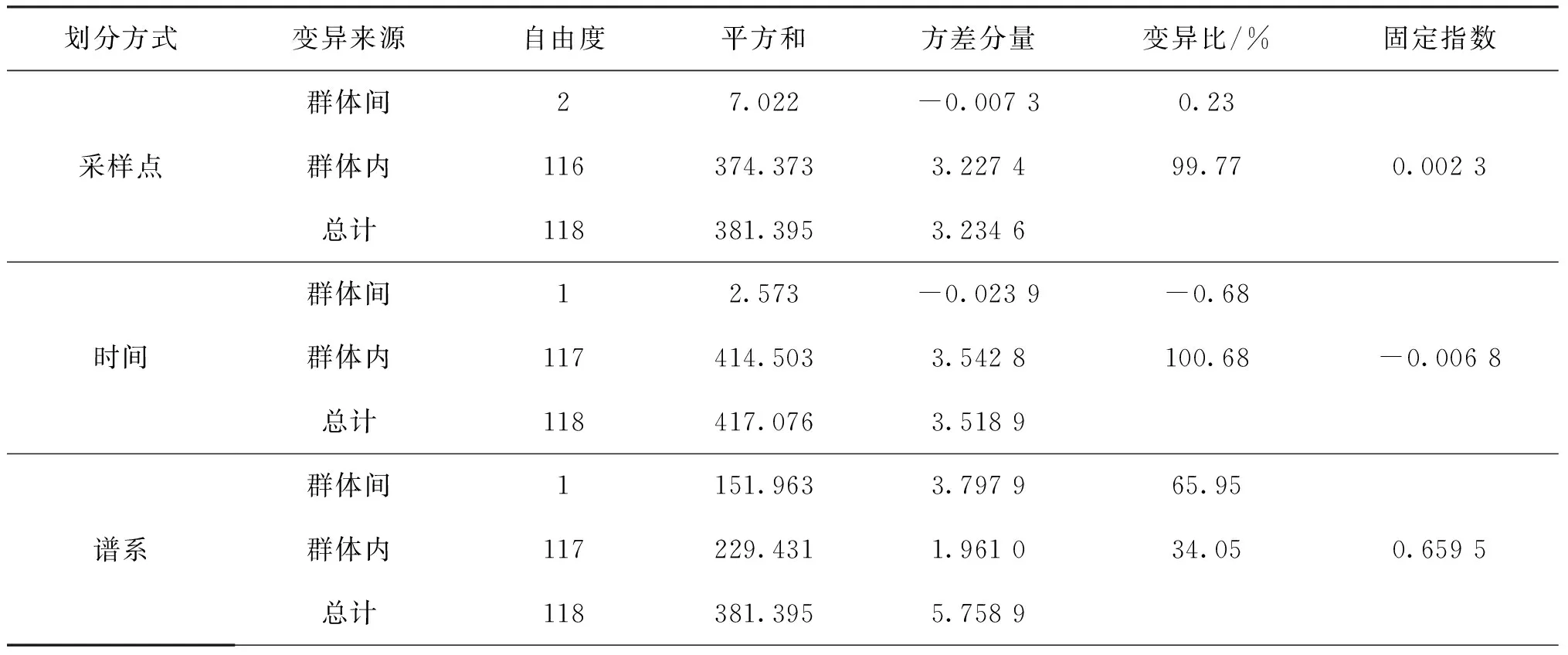
表5 红唇薄鳅群体分子方差分析(AMOVA)Tab.5 Analysis of molecular variance ( AMOVA) of L.rubrilaris
对2个谱系Lineage 1和Lineage 2遗传多样性分析见表2,2个谱系核苷酸多样性和单倍型多样性均低于总样本。计算两谱系的核苷酸变异系数显示,Lineage 1和Lineage 2谱系内的变异系数分别为3.795和3.407,谱系间的核苷酸差异系数为10.562,总样本的核苷酸差异系数为6.043。
2.3 种群历史
采用核苷酸错配分布[17]和中性检验Tajima’s D[18]、Fu’s Fs等方法分析种群历史,其中Tajima’s D检验更倾向于检测古老的突变和揭示古老种群发生扩张的历史,而Fu’s Fs检验则对近期种群扩张的检测更为敏感[19]。基于AMOVA的结果,红唇薄鳅采样点和采集时间对群体遗传分化无明显影响,因此首先将总样本作种群动态检验,再分别对基于NETWORK网络图和系统发育分析的2个谱系进行分析。结果显示,总样本和Lineage 2碱基错配分布呈多峰结构,Lineage 1为单峰(图3);各个群体的Tajima’s D值均为不显著负值,而Fu’s Fs除Lineage2为不显著负值外,其余群体的Fu’s Fs均为极显著负值(表2)。BSP结果进一步表明,长江上游红唇薄鳅总群体和Lineage1历史上经历了种群选择或扩张事件,时间在距今0.007 5 ~ 0.055 Ma(百万年)间。
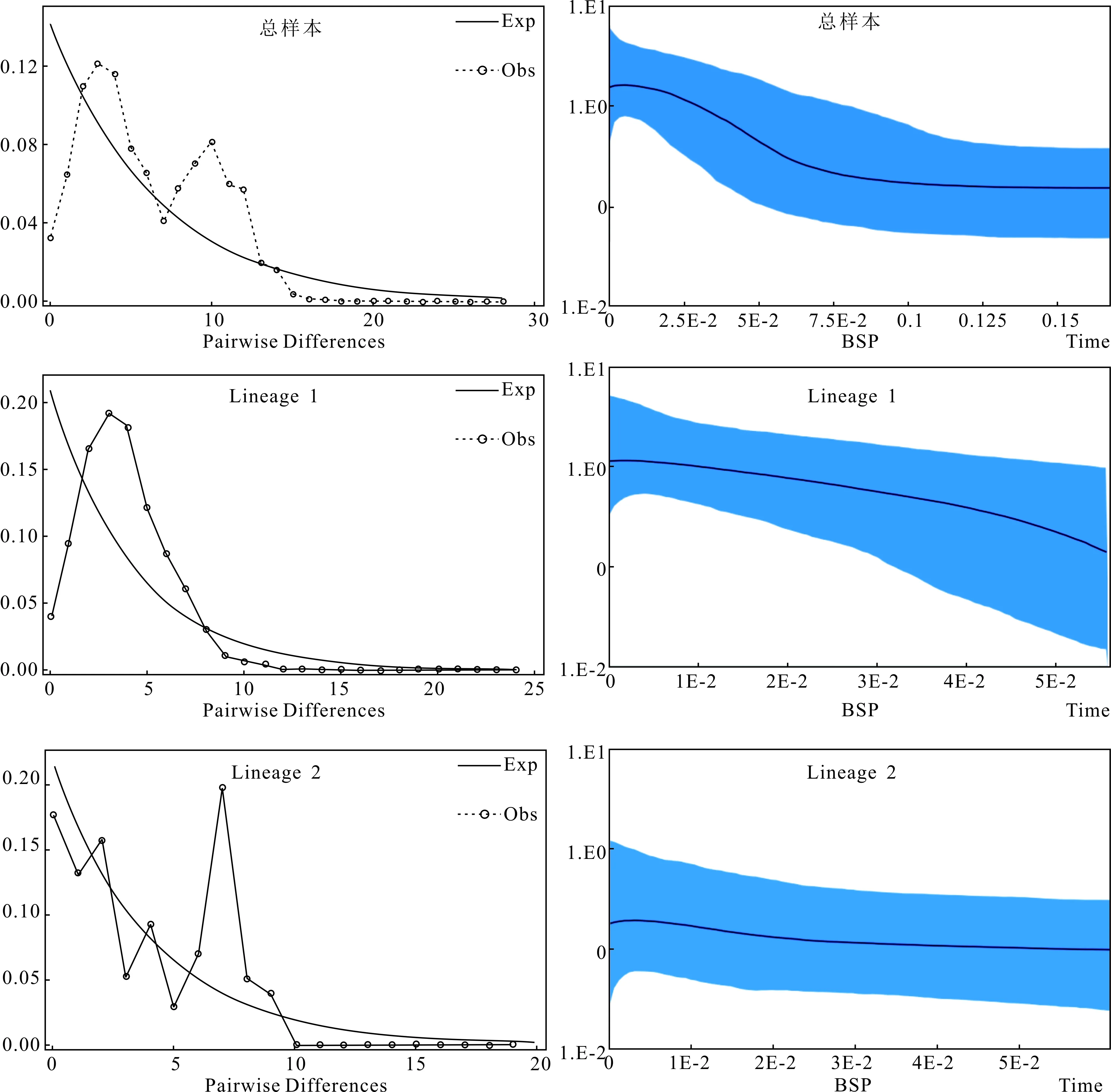
图3 红唇薄鳅的Mis-match分布和BSP分析Fig.3 Mis-match distribution and BSP analysis of L.rubrilabris
3 讨论
3.1 红唇薄鳅种群遗传多样性
遗传多样性是衡量物种进化潜力的重要指标,遗传多样性越高,表明物种对环境改变的适应能力更强,进化潜力越大。与长江上游其它鱼类线粒体控制区遗传多样性相比较,如长薄鳅(L.elongata)(Hd=0.916,Pi=0.004 5)[20]、圆筒吻鮈(Rhinogobiocylindricus)(Hd=0.840,Pi=0.011 4)[21]、铜鱼(Coreiusheterodon) (Hd=0.849,Pi=0.003 0)[23]和圆口铜鱼(C.guichenoti)(Hd=0.902,Pi=0.004 2)[22]等,除了圆筒吻鮈核苷酸多样性外,红唇薄鳅的遗传多样性均大于这几种鱼,说明红唇薄鳅遗传多样性暂处于较高水平,这与微卫星遗传多样性的结果是一致的[9]。
3.2 红唇薄鳅种群分化及种群历史
Wright[23]认为,若种群FST在 0 ~ 0.05之间,说明种群不存在分化;若0.05
但是网络连接图(图1)和NJ树的拓扑结构(图2)表明红唇薄鳅群体出现两个分化明显的谱系,谱系间的核苷酸差异系数明显大于谱系内,且谱系间FST>0.25(表5),表明两个谱系间的分化程度非常高[23]。这提示红唇薄鳅种群内部存在一定的遗传分化,这种分化是发生在同一区域内,即同域遗传分化(Sympatric Genetic Divergence)。同域遗传分化现象在其他鱼中也有发现,如进入美国阿拉斯加州伍德河流域繁殖的红大麻哈鱼(Oncorhynchusnerka)发现同域遗传分化,原因在于不同河段水温的差异,对红大麻哈鱼的繁殖产生了选择[24];又如生活在英国温德米尔(Windermere)湖的北极红点鲑(Salvelinusalpinus)在各个湖区的群体均发生了同域遗传分化,繁殖习性对此现象的发生具有重要作用[25-26]。本研究发现红唇薄鳅群体同域遗传分化的现象,是否也因存在某种繁殖习性上的差异,如产卵场、产卵时间等还有待进一步研究。
碱基错配分布和中性检验表明红唇薄鳅Lineage 1发生过群体扩张(图3和表2),采用BSP分析显示Lineage 1扩张发生在距今0.007 5 ~ 0.055 Ma,处于大理亚冰期(1万—11万年)[27]时期。已有研究表明长江上游一些鱼类也受冰期影响,种群历史上发生过瓶颈和扩张现象,如长薄鳅[28]、中华沙鳅[29]、蛇鮈[30]等。但是红唇薄鳅Lineage 2未检测到群体扩张发生,该谱系的种群大小低于Lineage 1,遗传多样性也相对较低(表2),暗示Lineage 1可能更适应长江生态环境的变迁。
3.3 保护建议
红唇薄鳅与同属鱼类的长薄鳅相比,分布范围更为狭窄,资源量更少,因此红唇薄鳅更容易受到人类活动的影响。加上红唇薄鳅生长潜力小且拐点年龄低,所以应该关注补充群体[3]。由于红唇薄鳅大小与中华沙鳅相近,容易受到中华沙鳅捕捞的协同作用影响,以及近年来由于渔业资源量在不断减少,渔民为了得到更充足的捕捞量而降低网具规格,致使捕捞规格不断下降,导致尚未性成熟的幼鱼个体被大量捕捞[31]。加上近几年的野外调查中,我们并未在这三个点以外的地域采到红唇薄鳅样本,侧面反映出渔业压力加大,资源前景不容乐观,因此持续对红唇薄鳅的资源动态进行关注是很有必要的。
[1] 丁瑞华.四川鱼类志[M].成都: 四川科学技术出版社,1994: 113-114.
[2] 高天珩,田辉伍,叶 超,等.长江上游珍稀特有鱼类国家级自然保护区干流段鱼类组成及其多样性[J].淡水渔业,2013,43(2):36-42.
[3] 田辉伍.长江上游保护区长薄鳅和红唇薄鳅种群生态及遗传结构比较研究[D].重庆: 西南大学,2013:97-126.
[4] 熊 飞,刘红艳,段辛斌,等.长江上游江津江段鱼类群落结构及资源利用[J].安徽大学学报(自科版),2014,(3):94-102.
[5] 徐 薇,乔 晔,龚昱田.长江上游鱼类资源变迁及其保护评析[J].人民长江,2012,43(1):67-71.
[6] Liu S,Xie G,Wang L,et al.Different roles of environmental variables and spatial factors in structuring stream benthic diatom and macroinvertebrate in Yangtze River Delta,China[J].Ecol Indic,2016,61:602-611.
[7] 段辛斌,田辉伍,高天珩,等.金沙江一期工程蓄水前长江上游产漂流性卵鱼类产卵场现状[J].长江流域资源与环境,2015,24(8):1358-1365.
[8] 刘红艳,熊 飞,宋丽香.长江上游特有鱼类红唇薄鳅微卫星DNA分离及序列特征分析[J].江西农业大学学报,2017,39(1): 145-152.
[9] 刘红艳,熊 飞,段辛斌,等.红唇薄鳅2个野生群体的遗传多样性研究[J].水产科学,2017,(2):192-196.
[10] Aljanabi S M,Martinez I.Universal and rapid salt-extraction of high quality genomic DNA for PCR-based techniques[J].Nucl Acids Res,1997,25(22):4692-4903.
[11] Rozas J,Rozas R.DnaSP version 3: an integrated program for molecular population genetics and molecular evolution analysis.[J].Bioinformatics,1999,15(2):174-175.
[12] Horng T,Barton G M,Medzhitov R.TIRAP: an adapter molecule in the Toll signaling pathway[J].Na Immunol,2001,2(9):835-841.
[13] Tamura K,Stecher G,Peterson D,et al.MEGA6: Molecular Evolutionary Genetics Analysis Version 6.0[J].Mol Biol Evol,2013,30(12):2725.
[14] Excoffier L,Lischer H E.Arlequin suite ver 3.5: a new series of programs to perform population genetics analyses under Linux and Windows[J].Mol Ecol Resour,2010,10(3):564-567.
[15]Drummond A J,Rambaut A.BEAST: Bayesian evolutionary analysis by sampling trees[J].BMC Evol Biol,2007,7(1):214.
[16]Donaldson K A,Jr W R.Amphi-panamic geminates of snook (Percoidei: Centropomidae) provide a calibration of the divergence rate in the mitochondrial DNA control region of fishes.[J].Mol Phylogen Evol,1999,13(1):208.
[17] Rogers A R,Harpending H.Population growth makes waves in the distribution of pairwise genetic differences.[J].Mol Biol Evol,1992,9(3):552-69.
[18] Tajima F.Statistical method for testing the neutral mutation hypothesis by DNA polymorphism.[J].Genetics,1989,123(3):585-595.
[19]Fu Y X.Statistical tests of neutrality of mutations against population growth,hitchhiking and background selection.[J].Genetics,1997,147(2):915.
[20] 赵 刚,周 剑,杜 军,等.长薄鳅(Leptobotiaelongata)线粒体DNA控制区遗传多样性研究[J].西南农业学报,2010,23(3):930-937.
[21] 曾晓芸.长江上游圆筒吻鮈遗传结构分析[D].重庆: 西南大学,2013:29-32.
[22] 袁希平,严莉,徐树英,等.长江流域铜鱼和圆口铜鱼的遗传多样性[J].中国水产科学,2008,15(3):377-385.
[23] Wright S.The interpretation of population structure by F-Statistics with special regard to systems of mating[J].Evolution,1965,19(3):395-420.
[24] Larson W A,Lisi P J,Seeb J E,et al.Major histocompatibility complex diversity is positively associated with stream water temperatures in proximate populations of sockeye salmon[J].J Evol Biol,2016,29(9):1846-1859.
[25]Corrigan L J,Lucas M C,Winfield I J,et al.Environmental factors associated with genetic and phenotypic divergence among sympatric populations of Arctic charr (Salvelinusalpinus)[J].J Evol Biol,2011,24(9):1906-1917.
[26]May-Mcnally S L,Quinn T P,Woods P J,et al.Evidence for genetic distinction among sympatric ecotypes of Arctic char (Salvelinus alpinus) in south‐western Alaskan lakes[J].Ecol Freshwat Fish,2015,24(4):562-574.
[27]范 启,何舜平.长江流域种群遗传多样性和遗传结构分析[J].水生生物学报,2014(4):627-635.
[28] 田辉伍,段辛斌,汪登强,等.长江上游长薄鳅Cyt b基因的序列变异与遗传结构分析[J].淡水渔业,2013,43(6):13-18.
[29]刘红艳,陈大庆,刘绍平,等.长江上游中华沙鳅遗传多样性研究[J].淡水渔业,2009,39(3):8-13.
[30] 李小兵,唐琼英,俞 丹,等.长江上游干流及赤水河蛇(鮈)遗传多样性与种群历史分析[J].动物学杂志,2016,51(5):833-843.
[31] 田辉伍,何 春,刘明典,等.长江上游干流三层流刺网渔获物结构研究[J].淡水渔业,2016,46(5):37-42.
(责任编辑:张红林)
Genetic diversity ofLeptobotiarubrilabrisin the upper Yangtze River inferred from mitochondrial control region
SHEN Shao-yi1,TIAN Hui-wu2,WANG Deng-qiang2,CHEN Da-qing2,LIU Shao-ping2
(1.KeyLaboratoryofFreshwaterFishReproductionandDevelopment,MinistryofEducation;theKeyLaboratoryofAquaticScienceofChongqing;SchoolofLifeScience,SouthwestUniversity,Chongqing400715,China;2.ScientificObservingandExperimentalStationofFisheryResourcesandEnvironmentintheUpperandMiddleReachesoftheYangtzeRiver,MinistryofAgriculture;YangtzeRiverFisheriesResearchInstitute,ChineseAcademyofFisheryScience,Wuhan430223,China)
Leptobotiarubrilabrisis a small freshwater fish,endemic in Upper Yangtze River,and its natural resource was decreased in recent years.In this study,mitochondrial control region sequences(896 bp)were used to analysis genetic diversity and structure ofL.rubrilabrispopulation.A total of 119 individuals collected from 3 sites as Jiangjin,Nanxi,Juexi in the Upper Yangtze River were used here.The results showed that 58 haplotypes were identified among sapmles and haplotype diversity and nucleotide diversity were 0.967 and 0.006 7,respetively,indicating moderately high genetic diversity forL.rubrilabrispopulation.Network diagram and phylogenetic NJ tree of haplotype showed that haplotypes were not clustered as the geographical samples,but divided into two lineages,which indicated that there was sympatric genetic divergence in population ofL.rubrilabris.The molecular variance analysis (AMOVA ) showed that there was no genetic differentiation between populations of sampled sites and time.The neutral test ,nucleotide mismatch distribution and Bayesian skyline plot (BSP) analysis showed that historical population expansion had occurred in Lineage 1 ofL.rubrilabris,and the expansion time was deduced in 0.007 5~0.055 Ma before.
Leptobotiarubrilabris;the upper reaches of the Yangtze River;mitochondrial control region;genetic diversity
2017-03-09;
2017-05-25
中国长江三峡集团公司资助项目(No.0799570,No.0799574)
申绍祎(1994- ),女,硕士研究生,专业方向为动物生态学。E-mail: ssy144580@163.com。
刘绍平。E-mail: lsp@yfi.ac.cn
S917.4;Q953
A
1000-6907-(2017)04-0083-08

