中华鲟促性腺激素释放激素受体基因cDNA序列的克隆及表达分析
王桂苹 杜合军 冷小茜 李创举 曹 宏
(1. 中国科学院水生生物研究所 淡水生态与生物技术国家重点实验室, 武汉 430072; 2. 三峡工程鱼类资源保护湖北省重点实验室, 中国长江三峡集团公司中华鲟研究所, 宜昌 443100; 3. 中国水产科学院长江水产研究所, 武汉 430223)
促性腺激素释放激素受体(GnRH-R)的cDNA序列最早是在小鼠(Mus musculus)中被克隆鉴定出来[1], 随后在非洲爪蟾(Xenopus laevis)的卵母细胞中进行了功能验证[2]。在鱼类中, 促性腺激素释放激素(GnRH)参与了包括繁殖在内的多个生理学进程, 和哺乳动物一样, 通过其与GnRH-R相结合, 合成并释放促性腺激素, 从而促进性腺的成熟[3]。目前,GnRH-R基因序列已在多种鱼类上有相关报道,包括虹鳟(Oncorhynchus mykiss)[4]、伯氏朴丽鱼(Haplochromis burtoni)[5]、真鲷(Pagrus major)[6]、斑纹隐小鳉(Kryptolebias marmoratus)[7]、日本鳗鲡(Anguilla japonica)[8]、欧洲舌齿鲈(Dicentrarchus labrax)[9]和革胡子鲶(Clarias gariepinus)[10]等。
中华鲟(Acipenser sinensis)是我国的一级保护动物, 其隶属于硬骨鱼纲、软骨硬鳞总目、鲟形目、鲟科、鲟属鱼类, 国际自然保护联盟(IUCN)濒危级(Endangered, EN)物种, 国际濒危动植物种贸易公约(CITES)附录Ⅱ保护物种。其寿命长, 体型大, 初次性成熟年龄大(雌性: 14—26年; 雄性:8—18年), 产卵间隔期长(约2—7年); 而且, 中华鲟是1种溯河洄游性鱼类, 栖息空间跨度大, 种群一旦遭到破坏, 就很难恢复。近几十年来, 由于人类活动的影响(如过度捕捞、航运、污染和水利工程等), 造成中华鲟的栖息地和产卵场遭到严重破坏,尤其是长江中游葛洲坝水利工程和三峡工程的修建阻断了中华鲟的产卵洄游路线, 导致中华鲟产卵量急剧减少, 野生资源量持续下降[11]。因此, 自20世纪80年代起, 我国学者对中华鲟进行了资源动态[11,12]、遗传多样性[13]、人工繁殖及幼鱼的培育[14,15]等理论和应用研究, 经过30余年的不懈努力,目前, 已成功实现中华鲟全人工繁殖[16]。但是, 人工养殖成鱼和亲鱼前期培育所需要的时间长、人力、物力投入巨大。本研究通过基因同源克隆, 获得了中华鲟GnRH-R基因的cDNA序列, 并对其序列特征、同源性、组织表达特征进行了分析, 以期为进一步研究其生长生殖调控功能提供基础资料。
1 材料与方法
1.1 实验材料
用于构建垂体SMART cDNA质粒文库的野生中华鲟由中国水产科学院长江水产研究所于2005年11月(中华鲟的繁殖季节) 于长江中捕获。其性别鉴定为雌性, 年龄鉴定为24 龄, 全长303 cm, 体长247 cm, 体重192 kg。养殖中华鲟取自中国长江三峡集团公司中华鲟研究所, 选取健康无损伤的3龄子二代中华鲟3尾, 经麻醉、放血, 取心、肝、脾、肾、肠道、肌肉、精巢及脑组织, 在液氮中速冻,然后存放于-80℃冰箱备用。
1.2 垂体RNA的提取及SMART cDNA的合成
将中华鲟垂体组织取出后, 利用SV Total RNA Isolation System试剂盒(Promega)提取总RNA, 步骤详见SV Total RNA Isolation System Promega操作手册, 中华鲟垂体SMART cDNA的合成步骤参照文献[17]。
1.3 垂体库的克隆、测序及数据分析
取合成好的中华鲟垂体SMART cDNA文库,对其进行克隆, 测序分析, 具体步骤参照文献[17]。将测序得到的SMART cDNA文库中全部小于200 bp的测序结果废弃不用, 对剩下的测序结果通过blastclust方法(ftp.ncbi.nih.gov/blast)进行聚类并通过CAP3软件(http://seq.cs.iastate.edu/download.html)来进行功能汇编。并和公共数据库GenBank里的蛋白质和核酸数据进行比对分析, 只有e-value值≤10-5的才被认为是可靠的数据。
1.4 中华鲟GnRH-R基因的全长cDNA序列的扩增与序列分析
以中华鲟垂体SMART cDNA文库为模板, 所用RACE PCR引物为: 3′-GnRH-R RACE-PCR:ATATCTCGAGCTAATGAAATACAGTACGGGG GTA; 5′-GnRH-R RACE-PCR: ATATCTCGAG CTAATGAAATACAGTACGGGGGTA; UPM: CTA ATACGACTCACTATAGGGCAAGCAGTGGTA TCAACGCAGAGT; 其具体RACE PCR步骤参照文献[18]。
1.5 GnRH-R基因的序列分析及系统进化树的构建
将所得序列利用Blast软件进行同源性分析, 利用ORF Finder查找开放阅读框, 并推导其相应的氨基酸序列; 通过NetNGlyc 1.0软件来预测N连糖基化位点。从GenBank中查询出所有已有的鱼类GnRH-R基因cDNA序列, 利用Cluster X进行氨基酸序列多重比对, 并以米氏叶吻银鲛的GnRH-R为外类群(NP_001279833), 用Mega 4 软件中的邻接法(Neighbourjoining)及最大简约法(Maximum parsimony)对相关蛋白质构建系统发育树, 用自展法(Bootstrap) 进行系统树评估, 一致性指数(Consistency index,CI)来衡量分析结果的可靠性。
1.6 实时荧光定量PCR
利用SV Total RNA Isolation System试剂盒(Promega) 提取中华鲟的心、肝、脾、肾、肠道、精巢、肌肉及脑组织用于组织表达特征研究; 步骤详见SV Total RNA Isolation System Promega操作手册。所得总RNA经琼脂糖凝胶电泳和分光光度计检测, 取各组织总RNA 0.1 μg, 利用M-MLV逆转录酶及oligo (dT)18引物(Promega), 按操作说明将其合成第1链cDNA, 以此为模板。荧光定量PCR采用引物为:GnRHR-rtime-F: GAATGTCACAGTTCAGT GGCTCG;GnRHR-rtime-R: TTTCTTCTTCGCT TCGTTGATG; 内参引物为: β-actin-F: TGGACT TGGCTGGTCGTGAC; β-actin-R: CTGGCAGCT CATAGCTCTTC。其他具体步骤参见文献[18]。
2 结果
2.1 中华鲟GnRH-R基因的cDNA全长序列及其推导的氨基酸序列
中华鲟GnRH-R基因cDNA全长1530 bp, 有478 bp的5′非翻译区, 579 bp的开放阅读框和473 bp的3′非翻译区, 编码192个氨基酸, 在poly (A)尾巴前18 bp处有一个典型的加尾信号(AATAAA)。通过软件NetNGlyc 1.0进行N连糖基化位点的预测, 结果表明,GnRH-R的氨基酸序列存在5个潜在的N连糖基化位点(Asn2、Asn24、Asn 37、Asn 91和Asn 111)。该序列的NCBI Bank ID是2033544。
2.2 中华鲟GnRH-R基因氨基酸序列的同源性分析
运用BLAST软件对中华鲟GnRH-R蛋白进行了氨基酸序列同源性分析(表 1)。结果表明, 在所有序列中, 和中华鲟GnRH-R的氨基酸序列相比较, 同源性范围为 39%—76%, 其中, 与真鲷GnRH-R的同源性最高, 为76%, 而与软骨鱼类——米氏叶吻银鲛的GnRH-R的同源性最低, 为39%。
我们将中华鲟GnRH-R和其他鱼类的GnRHR的氨基酸序列进行了分析。利用Mega 4.0 软件,分别利用邻接法及最大简约法构建了进化树(图1)。 结果发现, 以银鲛目的米氏叶吻银鲛为外类群, 鳗鲡目的日本鳗鲡、鲇形目的革胡子鲶、鲑形目的虹鳟, 以及鲈形目的伯氏朴丽鱼可以聚类为一个分支; 中华鲟则与腔棘鱼目的矛尾鱼(Latimeria chalumnae)与其他鱼类如鳉形目的斑纹隐小鳉、底鳉(Fundulus heteroclitus)以及鲈形目、鲽形目、鲉形目等代表物种聚为另一个分支。从图中的结果看, 虽然两棵树之间存在一些细微差异, 主要体现在有几个支持率比较低的节点上, 但是几个关键节点的支持率还比较高, 在90%以上, 例如, 以银鲛目的米氏叶吻银鲛为外类群, 鳗鲡目的日本鳗鲡、鲇形目的革胡子鲶、鲑形目的虹鳟, 以及鲈形目的伯氏朴丽鱼可以聚类一个分支, 2棵进化树都显示这几个物种聚类为一个分支, 支持率分别为100%和97%。

表 1 中华鲟的GnRH-R基因与其他鱼类氨基酸同源性比较Tab. 1 Amino acid identities of GnRH-R of Chinese sturgeon and other fishes

图1 基于邻接法(a)和最大简约法(b)构建的鱼类GnRH-R的系统进化树Fig. 1 Unrooted phylogenetic trees [Neighbour-joining (a) & Maximum parsimony (b) of the deduced amino acid sequence of GnRH-R of Chinese sturgeon and other fishes]
2.3 中华鲟GnRH-R基因的组织表达分析
在3-龄的雄性中华鲟中对GnRH-R基因的组织表达进行分析, 发现其在精巢中大量转录, 其精巢已发育至II期; 在肌肉和脑组织中可以检测到微量的GnRH-R基因的mRNA; 而在心、肝、脾、肾及肠道等组织中几乎无法检测到其mRNA (图2)。
3 讨论
本研究通过基因同源克隆和RACE方法, 获得了中华鲟GnRH-R基因cDNA全长序列, 这也是该基因序列首次在鲟形目鱼类中的报道。中华鲟是一种原始的硬骨鱼类。通过将其GnRH-R基因的氨基酸序列与其他已登记在NCBI上的鱼类GnRH-R氨基酸序列进行序列比对后, 我们发现鲑形目, 鲇形目和鳗鲡目的物种, 其GnRH-R可以聚为一类, 而中华鲟与同属古老硬骨鱼类的矛尾鱼一起, 与其他硬骨鱼类可以聚为一类。这是否意味着硬骨鱼类的GnRH-R在进化过程中有着2个不同的演化方向, 值得在今后的工作中进行更深入的探讨。
本研究应用实时定量RT-PCR方法, 检测其在3龄雄性中华鲟中的表达特征, 经检测发现, 中华鲟GnRH-R基因在精巢中大量转录, 和我们之前的报道[18]相吻合的是, 其精巢已发育至II期, 暗示其在性腺发育特别是精子发生过程中可能起重要作用。但是其在脑组织的表达微弱, 而在其他硬骨鱼类如斑纹隐小鳉[7]中, 其GnRH-R基因在脑组织和性腺中均有较高的mRNA的表达水平。在虹鳟[4]中, 该基因则在视网膜, 大脑, 卵巢中均有较高的mRNA的表达水平。由于中华鲟物种非常珍贵, 无法获得新鲜的野生样本, 而人工养殖的中华鲟也不易取材,所以本研究所用样本数较少, 此结果还需进一步验证。目前, 养殖中华鲟个体较野生的偏小、怀卵量少, 绝对繁殖力下降明显; 性腺发育往往仅停留在Ⅱ期, 很难向Ⅲ期或者Ⅳ期过渡, 其性腺发育至Ⅲ期普遍需要10年以上时间。同时, 由于中华鲟的寿命长, 体型大, 相比较人工养殖的施氏鲟(初次性成熟年龄: 雌性: 9—11年; 雄性: 6—7年), 中华鲟初次性成熟年龄大(雌性: 14—26年; 雄性: 8—18年),产卵间隔期长(约2—7年)。因此, 急需缩短中华鲟的初次性成熟年龄和繁殖周期, 并提高怀卵量和繁殖力。GnRH与GnRH-R的主要作用是促进促性腺激素的释放, 并促使性腺发育成熟, 但是其在中华鲟这一古老物种中的具体调控机制还有待于进一步的深入研究。本研究结果为中华鲟生长发育调控、GnRH-R基因结构与功能探讨、鱼类GnRHR进化奠定了基础。
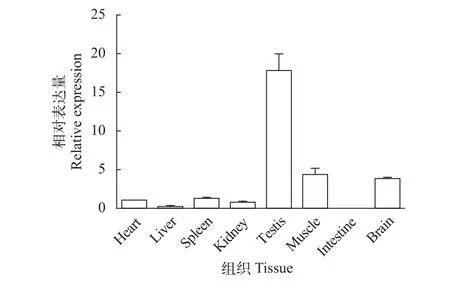
图2 GnRH-R的mRNA在中华鲟各个组织中的表达差异Fig. 2 Relative expression of GnRH-R mRNA in heart, liver,spleen, kidney, testis, muscle, intestine and brain of Chinese sturgeon
[1]Tsutsumi M, Zhou W, Millar R,et al. Cloning and functional expression of a mousegonadotropin-releasing hormone receptor [J].Molecular Endocrinology, 1992, 6(7):1163—1169
[2]Reinhart J, Mertz L, Catt K. Molecular cloning and expression of cDNA encoding the murine gonadotropin-releasing hormone receptor [J].Journal of Biological Chemistry, 1992, 267(30): 21281—21284
[3]Moncaut N, Somoza G, Power D,et al. Five gonadotropin-releasing hormone receptors in a teleost fish: isolation,tissue distribution and phylogenetic relationships [J].Molecular Endocrinology, 2005, 34(3): 767—779
[4]Madigou T, Mañanos-Sanchez E, Hulshof S,et al. Cloning, tissue distribution, and central expression of the gonadotropin-releasing hormone receptor in the rainbow trout(Oncorhynchus mykiss) [J].Biology of Reproduction,2000, 63(6): 1857—1866
[5]Robison R, White R, Illing N,et al. Gonadotropin-releasing hormone receptor in the teleostHaplochromis burtoni: structure, location, and function [J].Endocrinology,2001, 142(5): 1737—1743
[6]Okuzawa K, Tensen C, Bogerd J,et al. Molecular Cloning of a Gonadotropin-Releasing Hormone Receptor cDNA from the Red Sea Bream,Pagrus major[J].Annals of the New York Academy of Sciences, 1998, 839(1):518—519
[7]Rhee J, Seo J, Raisuddin S,et al. Gonadotropin-releasing hormone receptor (GnRHR) gene expression is differently modulated in gender types of the hermaphroditic fishKryptolebias marmoratusby endocrine disrupting chemicals [J].Comparative Biochemistry and Physiology Part C:Toxicology & Pharmacology, 2008, 147(3):357—365
[8]Okubo K, Suetake H, Usami T,et al. Molecular cloning and tissue-specific expression of a gonadotropin-releasing hormone receptor in the Japanese eel [J].General and Comparative Endocrinology, 2000, 119(2): 181—192
[9]González-Martínez D, Madigou T, Mañanos E,et al.Cloning and expression of gonadotropin-releasing hormone receptor in the brain and pituitary of the European sea bass: an in situ hybridization study [J].Biology of reproduction, 2004, 70(5): 1380—1391
[10]Tensen C, Okuzawa K, Blomenrohr M,et al. Distinct efficacies for two endogenous ligands on a single cognate gonadoliberin receptor [J].European Journal of Bioche-mistry, 1997, 243(1-2): 134—140
[11]Zhang H, Wei Q W, Kyanrd B,et al. Spatial structure and bottom characteristics of the only remaining spawning area of Chinese sturgeon in the Yangtze River [J].Journal of Applied Ichthyology, 2011, 27(2): 251—256
[12]Tao J P, Qiao Y, Yang Z,et al. Estimation on the spawning population and spawning sizes of Chinese sturgeon(Acipenser sinensis) and trend analysis of their change in recent years [J].Journal of Hydroecology, 2009, 30(2):37—43 [陶江平, 乔晔, 杨志, 等. 葛洲坝产卵场中华鲟繁殖群体数量与繁殖规模估算及其变动趋势分析. 水生态学杂志, 2009, 30(2): 37—43]
[13]Shao K, Shi F, Yan S X,et al. Allele variation of a microsatellite locus AciG-35 in individuals of wild Chinese sturgeon [J].Journal of Hydroecology, 2014, 35(1): 1—6[邵科, 史方, 闫书祥, 等. 微卫星位点AciG-35在中华鲟野生个体中不同等位基因的序列变异研究. 水生态学杂志, 2014, 35(1): 1—6]
[14]Zhang X Y, Du H, Wei Q W,et al. The post-spawned recovery of cultured Chinese sturgeonAcipenser sinensis[J].Acta Hydrobiologica Sinica, 2015, 39(4): 705—713[张晓雁, 杜浩, 危起伟, 等. 养殖中华鲟的产后康复. 水生生物学报, 2015, 39(4): 705—713]
[15]Chai Y, Gong J L, Du H,et al. Quality comparison of gamete of F1-generation and analysis of growth characteristics of F2-generation of Chinese sturgeon (Acipenser sinensis) [J].Journal of Fisheries of China, 2014, 38(3):350—355 [柴毅, 龚进玲, 杜浩, 等. 中华鲟子一代配子质量及其子二代生长特征分析. 水产学报, 2014, 38(3):350—355]
[16]Guo B F, Chang J B, Xiao H,et al. The reproductive biology of first filial generation ofAcipenser Sinensisgrowing up in the freshwater environment [J].Acta Hydrobiologica Sinica, 2011, 35(6): 940—945 [郭柏福, 常剑波,肖慧, 等. 中华鲟初次全人工繁殖的特性研究. 水生生物学报, 2011, 35(6): 940—945]
[17]Cao H, Li C J, Wei Q W,et al. cDNA clone and sequence analysis of proopiomelanocortin in Chinese sturgeonAcipenser sinensis[J].Acta Hydrobiologica Sinica,2011, 35(5): 776—782 [曹宏, 李创举, 危起伟, 等. 中华鲟阿黑皮素原cDNA序列克隆及其序列分析. 水生生物学报, 2011, 35(5): 776—782]
[18]Leng X Q, Du H J, Li C J,et al. Molecular characterization and expression pattern ofdmrt1in the immature Chinese sturgeonAcipenser sinensis[J].Journal of Fish Biology, 2016, 88(2): 567—579

