基于模式匹配算法的转基因大豆DNA快速检测方法
杨 萍,王海清,王 宇,岳江涛
(沈阳农业大学 信息与电气工程学院,沈阳 110866)
随着科技的发展,转基因食物越来越多。仅2017年,我国的转基因大豆消费量为11 196万t,除自产1 530万t外,进口量高达9 550万t。研究表明,进口转基因大豆及制品可能在30 a甚至更长时间后对人类的身体健康产生影响。虽然大豆的基因库在逐渐完善,基因序列也越来越多,但我国在转基因大豆检测方面仍存在一些不足。在此情况下,探索转基因大豆的DNA快速检测方法具有非常重要的意义。
1 转基因大豆检测方法
转基因大豆技术是通过基因工程,把其它植物的某些特定基因转移到大豆基因上,从而培育出转基因大豆,但其对人体产生的作用未知。
串的查找运算也称作串的模式匹配运算,目的是在主串中找出一个与目的串完全相同的子串序列,常见的有布鲁特-福斯 (Brute-Force,BF)算法和克努特-莫里斯-普拉特(Knuth-Morris-Pratt,KMP)算法。DNA序列包含生物的所有遗传信息,对其进行研究是基因检测领域的重要课题。转基因检测主要检测DNA的脱氧核苷酸排列顺序,即腺嘌呤,胞嘧啶,鸟嘌呤,胸腺嘧啶(分别用A,C,G,T表示)的顺序。
以抗草甘膦转基因大豆 (Roundup Ready Transgenic Soybean,RRS) 的 DNA序列为材料,在DNA序列资源有限的条件下,比较KMP算法和BF算法查找相同基因的效率和准确性,并利用KMP算法进行序列匹配,以期为转基因大豆检测提供准确高效的方法。
2 模式匹配的BF(Brute-Force)算法
BF算法是简单的模式匹配算法,其基本思路是:将主串S中某个位置i(i从0开始)起始的子串和模式串T相比,即从j=0起比较Si+j与Tj,若相等,在S中存在以i为起始位置匹配成功的可能性,继续比较(j逐步增1),直至与T中最后一个字符相等;否则,从S的下一个字符起重新进行下一轮匹配,即从i+1,j=0 开始新一轮匹配。
假设 S="ATATCATCACGAG",T="ATCAC",则模式串与目标串匹配过程如图1所示。
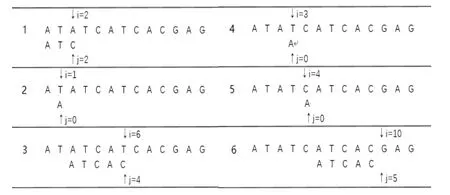
图1 BF算法匹配过程Figure 1 BF algorithm matching process
该算法比较简单,易于理解,但效率不高,主要是目标串指针回溯消耗大量时间,时间复杂度为[O(n+m),O(n*m)]。
3 模式匹配的KMP算法
回溯现象是BF算法造成效率不高的主要原因。若匹配过程中目标串指针不回溯,则整个匹配过程中的目标串指针都没有回溯现象,可大大提高算法效率,如图2所示。

图2 不回溯算法的匹配过程示例Figure 2 Matching process example of backtracking algorithm
由图2可知,在第2次匹配过程中,当i=6,j=4时,目标串和模式串对应字符不相等。若按BF算法,则下一次匹配将从i=3,j=0开始。因为目标串中第4,5 和 6 个字符是“T”,“C”,“A”,而模式串中的第一个字符为 “A”,所以仅需将模式串向右滑动3个字符,即第 3行 i=6,j=1进行字符比较。此过程中 i指针没有进行回溯,效率高。
由图2的第三行匹配可知,模式串j指针并不是从0开始的。若基因的DNA序列 (相对于模式串字符)比较多,可节省匹配时间,进一步提高效率。
设目标串s="a0a1a2…an", 模式串t="b0b1b2…bn",当ai≠bi时有

若模式串t中可相互重叠的最大真子串满足:

下一次匹配可直接从t的第k+1个字符bk起,与s的第i+1个字符ai继续进行匹配。若s中不存在"b0b1…bk-1"="bj-kbj-k+1bj-1",则下一次匹配直接从 s 的第一个字符和s的第i+1个字符开始。
令 next[j]=k,next[j]表示当 t中第 j个字符与 s中相对应的字符不等时,在t中需要重新和s中该字符进行比较的字符位置:

设模式串t=“ATAATCAC”,则next的函数值如表1所示。

表1 next的函数值Table 1 Function vablue of next
求出 next函数后,设 s="ACATAATAATCACAGT G",t="ATAATCAC",i为目标串的指针、j为模式串 t的指针,next[j]值比配过程图3所示,图4为KMP算法的演示图。

图3 KMP算法匹配过程Figure 3 Matching process of KMP arithmetic

图4 用c语言实现KMP算法功能Figure 4 The function of KMP algorithm realized by c language
KMP的核心匹配算法(基于C语言)如下:
voidgetnext(Hstring*t,int next[]
{
int j,k;
j=0;
k=-1;
next[0]=-1;
while(j<t.length)
if(t.data[j]==t.data[k]||k==-1)
{
j++;
k++;
next[j]=k;
}
else
k=next[k];
}
int KMPindext(Hstring*s,Hstring*t,int pos)
{
int next[INITSTRLEN],i,j;
getnext(t,next);
i=pos-1;
j=0;
while(i<s.length&&j<t.length)
if(s.data[i]==t.data[j]||j==-1)
{
i++;
j++;//对应字符相同,指针后移一个位置
}
else
j=next[j];//i不变,j后退,相当于模式串向右移动
if(j>=t.length)
return i-t.length+1;//匹配成功,返回第 1个匹配字符在主串中的位续
else
return 0;
4 BF与KMP算法的比较
4.1 时间复杂度
BF与KMP算法的复杂度比较见表2。

表2 BF与KMP算法的复杂度比较Table 2 The comparision of complexity of BF and KMP algorithm
由表2可知,BF算法的时间复杂度为 (N-M+1)*M=O(N*M),KMP算法的时间复杂度为 N+M=O(N+M),运算效率高于BF算法
4.2 运算速度
基于KMP算法建立一种快速高效、结果可靠的RRS转基因大豆基因检测方法。通过编写KMP算法的程序和设计界面,将待检测DNA序列导入用户数据库后,可以与转基因DNA序列进行对比,从而判别待测大豆产品是否为转基因大豆。利用Java实现BF和KMP算法,并且做成网页形式,比较BF算法和KMP算法的运算速度,初始界面如图5所示。

图5 基因检测初始界面Figure 5 Initial interface of gene detection

图6 基因检测界面Figure 6 Gene detection interface
该界面模拟转基因检测,对总基因库的文件和转基因文件中基因序列进行BF和KMP对比,能够解析出样品是否为转基因。模拟大豆总基因库有1 0000个脱氧核糖核苷酸和2 500个转基因片段,检验结果如图6所示。
计算查找相同基因的试验结果表明,KMP算法的效率比BF算法高近10倍,能够大大提高转基因大豆的检测效率。
5 结论与讨论
通过比较分析KMP算法和BF算法,确定在查找相同基因时,KMP算法用时短,检测效率高。诸多研究表明,KMP算法检测速度快,准确度高,成本低,操作简单,能够快速判断大豆的安全性,是一种值得推广的转基因大豆检测方法。
KMP算法消除了BF算法中的指针回溯问题,但目标串指针每次只能移动一个字符,应进行相应改进。因此,提出应用KMPP算法进行程序后期改进,以增加目标串指针每次移动的距离,进而提高匹配效率。目前,开发程序只能针对一种DNA序列进行检测,后期还应对程序进行改善,使其能够同时对多种转基因大豆的序列进行比对和检测。同时,还应导入基因数据库,实现数据共享,使基因数据库更加完善。

