结合卷积神经网络和模糊系统的脑肿瘤分割*
师冬丽,李 锵,关 欣
天津大学 微电子学院,天津 300072
1 引言
脑肿瘤分为良性肿瘤和恶性肿瘤两类,良性肿瘤在通过手术治疗后一般能够恢复健康,恶性肿瘤因其顽固性难以治愈,又被称为脑癌。调查显示,美国仅2015年被诊断的脑肿瘤人数新增23 000人[1]。脑肿瘤已严重危害人类生命健康,如何更好地对其进行诊断和治疗十分重要。核磁共振成像(magnetic resonance imaging,MRI)具有非侵入性,能在患者不接受高电离辐射的情况下提供形状、大小及位置等信息,且具有良好的软组织对比度[2],因此在脑肿瘤的诊断、治疗和手术引导中越来越受到人们的关注[3]。
脑肿瘤患者的MRI成像是三维多波段成像,通常包括T1、T2、T1c和FLAIR等,不同的成像方式可以提供不同的脑肿瘤信息[4]。图1显示了同一患者脑部的4种MRI成像类型及其对应的专家分割结果,其中蓝色区域为肿瘤,白色区域为囊变,红色区域为水肿。脑肿瘤分割的目的是从包括灰质、白质和脑脊液的正常脑组织中分割出包含肿瘤、囊变和水肿等非正常脑组织区域[5]。图1(a)是T1成像,因其操作简单,常用于脑肿瘤的结构分析,但成像质量差,无法提供更为详尽的肿瘤特征信息。图1(b)是T1c成像,由于增生性脑肿瘤区血脑屏障破坏导致造影剂积聚,使得T1c成像中的脑肿瘤边界变得更明亮,很容易区分出肿瘤和囊变。图1(c)是T2成像,水肿区域相比其他成像方式更加明亮,缺点是脑脊液和肿瘤的像素特征难以区分。图1(d)是FLAIR成像,水肿区域边界明显,因此是目前分割脑肿瘤最有效的成像方式。
目前,针对MRI脑肿瘤的分割方法主要分为以下几类:基于区域的分割方法、基于模糊聚类的分割方法和卷积神经网络方法等。
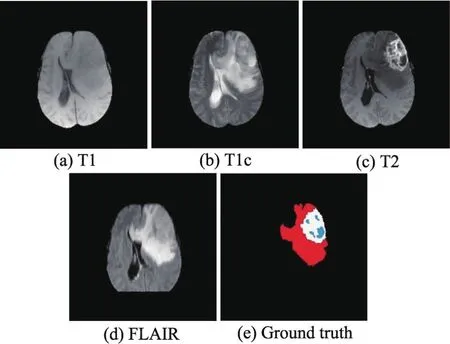
Fig.1 Four types of brain tumors and expert segmentation result图1 4种类型脑肿瘤患者MRI图像及专家分割结果
基于区域生长的分割方法首先在目标区域选中一个种子点,再将周围具有相同性质的像素点合并至种子区域,直到没有相似性质的像素点聚集为止。Weglinski等人[6]提出了利用区域生长法来进行脑肿瘤图像的分割,并通过中值滤波减少噪声对区域生长法的影响。Deng等人[7]提出了在区域生长法的基础上,利用边界的平均梯度和类内方差的特征信息,在生长过程中不断调整生长阈值以达到更好的分离精度。然而,对于区域生长法而言,普遍存在对图像阴影区域分割不理想的问题。
模糊聚类的分割方法是对图像像素按其相似度进行分类,使得同类个体间的距离较小而不同类个体间的距离较大。Khotanlou等人[8]采用模糊聚类方法进行脑肿瘤的检测与分割。Gopal等人[9]采用粒子群优化算法选择聚类中心。Logeswari等人[10]采用分级自组织映射选择聚类中心。Havaei等人[11]根据一些最低限度的用户交互信息,使用k近邻的机器学习方法,结合简单的特征向量得到较好的脑肿瘤分割结果。然而,对于聚类算法而言,通常仅考虑每个像素点的独立信息而忽略像素点之间的空间信息,最后导致算法受噪声影响较大,且无法得到连续区域的分割结果。
1998年,Lecun等人[12]将卷积神经网络(convolution neural network,CNN)首次应用于图像识别领域,该网络可以通过直接输入原始图像识别视觉上的规律,避免对图像复杂的前期预处理。近年来,CNN在脑肿瘤分割中得到了广泛应用。Zhao等人[13]提出了一种多尺度CNN模型,该模型可以自动检测图像的最优尺寸信息,并组合不同尺度区域周围的像素信息。Havaei等人[14]使用级联的体系结构,将第一个CNN模型的输出作为一个额外的信息源提供给下一个CNN模型。Pereira等人[15]在CNN模型中使用较小尺寸的卷积核以提高神经网络的速度,更有利于提取图像的特征。此外,它还有助于防止过拟合并减少网络中权重参数的数目。然而,上述方法将多种类型脑肿瘤图像同时作为模型输入,没有充分利用各类型脑肿瘤图像自身的特征信息,最终使得输入过多的冗余信息,进而影响分割精确度。
模糊推理系统是以模糊集为基础,最终可以实现复杂的非线性映射的一种方法。2012年,Sharma等人[16]提出了一种将人工神经网络与模糊推理系统相结合的方法用于脑肿瘤图像的检测。
为了充分利用不同类型图像的特性来提高脑肿瘤图像的分割精度,本文提出了一种结合卷积神经网络和模糊推理系统的全自动脑肿瘤MRI图像分割算法。首先选取FLAIR和T2两种成像方式,分别对其建立CNN模型来预测肿瘤概率。然后根据预测错误样本点建立非线性映射。最后将两类图像像素点的映射结果作为模糊推理系统的输入,预测该像素点是否属于肿瘤区域。
本文组织结构如下:第2章简要回顾了卷积神经网络和模糊推理系统的基本原理;第3章提出了结合卷积神经网络和模糊推理系统的全自动脑肿瘤MRI图像分割算法;第4章进行仿真实验并分析实验结果;第5章总结全文。
2 基本理论
2.1 卷积神经网络
卷积神经网络是一种多层神经网络,每层由多个二维平面组成,每个平面由多个独立神经元组成。CNN通过结合局部感知区域、共享权重、空间或者时间上的下采样来充分利用数据本身包含的局部性等特征,优化网络结构,并且保证在一定程度上对位移、缩放和其他形式扭曲的不变性。一个经典的卷积神经网络LeNet-5的结构如图2所示。卷积神经网络一般包括输入层、卷积层、子采样层、全连接层和输出层。
卷积层又称为特征映射层,网络的每个计算层由多个特征映射组成,每个特征映射是一个平面,平面上所有神经元的权值相等。在卷积层,前一层的特征图和一个可学习的核进行卷积,卷积结果经过激活函数后得到这一层的特征图。一般地,卷积层的形式如下:

式中,Mj为神经元j对应的局部感受野;是第l层的神经元i的第j个输入对应的权值;为l层的第i个偏置量;为l-1层神经元i的输出;为l层神经元j的输出。

Fig.2 Structure chart of convolution neural network图2 卷积神经网络结构图
子采样层又称为特征提取层,每个神经元的输入与前一层的局部接受域相连,并提高该局部的阈值。如果输入的特征图为n个,则经过子采样层后特征图的数目不变,尺寸变小。子采样层的形式如下:

式中,down()为下采样;βl、bl分别为可训练参数和可训练偏置。
全连接层是与上一层全连接,将上一层输出结果与权重向量相乘,再加上偏置项,并将这一结果传送给sigmoid函数或者tanh函数进行计算。全连接层的形式如下:
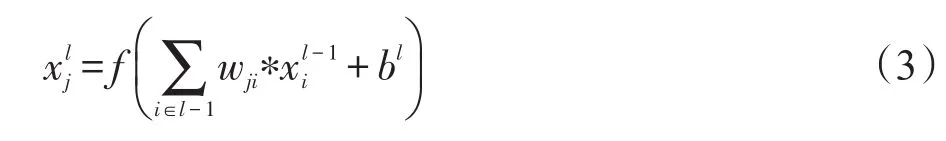
式中,wji为网络中第l-1层的第i个输入到第l层的第j个输出的连接权重;bl为第l层的可训练偏置。
输出层用于计算输入样本属于各类别的概率,输入样本归属于所属概率最大的类别,完成分类任务。
2.2 模糊推理系统
模糊推理系统作为智能计算的重要分支,以模糊集合为基础,引入模糊逻辑后以一定的程度值描述某一事件,从而体现事件更真实的情况,最终实现复杂的非线性映射关系。
模糊集是用来表达模糊性概念的集合。根据模糊集指定的规则称为模糊规则,对于一个模糊系统而言,所有的模糊规则共同组成了该系统的模糊规则集。模糊推理系统框图如图3所示。

Fig.3 Flow chart of fuzzy inference system图3 模糊推理系统框图
首先,将输入信息进行模糊化,即将输入从精确的数值根据输入变量的模糊集转化为模糊值。其次,根据建立好的模糊规则进行匹配,从而进行逻辑推理。根据逻辑推理,可以得到一个输出结果的模糊值。最终,通过输出变量的模糊集可以将输出的模糊值进行去模糊化,得到的结果为一个精确的输出数值。去模糊化的计算公式如下:

式中,μ(⋅)表示该模糊变量的隶属度函数。
3 本文算法原理
3.1 CNN模型
本文采用如图2所示的CNN模型。
将每幅FLAIR和T2图像以像素点为中心切分为若干个尺寸为35×35的图像作为原始输入。输入图像首先经过由6个6×6邻域的卷积核组成的卷积层后得到6个30×30的特征图,再经过下采样得到6个15×15的特征图;再经过由12个6×6邻域的卷积核组成的卷积层得到12个10×10的特征图,并通过下采样得到12个5×5的特征图;然后通过全连接层将得到的特征图转化为一维特征;最后,在输出层中得到输入样本是否属于肿瘤的概率。
3.2 非线性映射
通过卷积神经网络得到的概率结果通常集中在0和1,然而在大样本情况下这种过于集中的概率分布并不能较好地体现出概率相似的像素点之间的差异性。因此,需要通过非线性映射将错误样本点的概率均匀分布在0到1之间。
实验过程中,找出模型训练中所有肿瘤误判为非肿瘤的样本,将其预测概率从小到大排列并等分为10份,令其边界值分别对应于0,0.05,0.10,…,0.50。同理,找出训练样本中所有非肿瘤误判为肿瘤的训练样本,并将其预测概率从小到大排列并等分为10份,令其边界值分别对应于0.50,0.55,0.60,…,1.00。最终,对边界值和映射点进行分段线性拟合得到非线性映射函数。
3.3 模糊推理系统
针对其他方法无法充分利用不同图像特征信息的缺陷,本文根据不同类型成像方式的特点,选取FLAIR和T2两种模态图像构建模糊推理系统。通过对FLAIR和T2图像的处理,可以得到每个像素点由单独图片预测的属于肿瘤区域的概率p1和p2。模糊系统以p1和p2作为输入,输出则为最终的肿瘤概率。
对比FLAIR和T2图像可以发现,FLAIR的水肿区域边界更为明显,即在分割过程中,得到的结果相比T2图像置信度更高。因此,在模糊系统中,希望以FLAIR图像的结果作为基准,并利用T2图像进行调整。
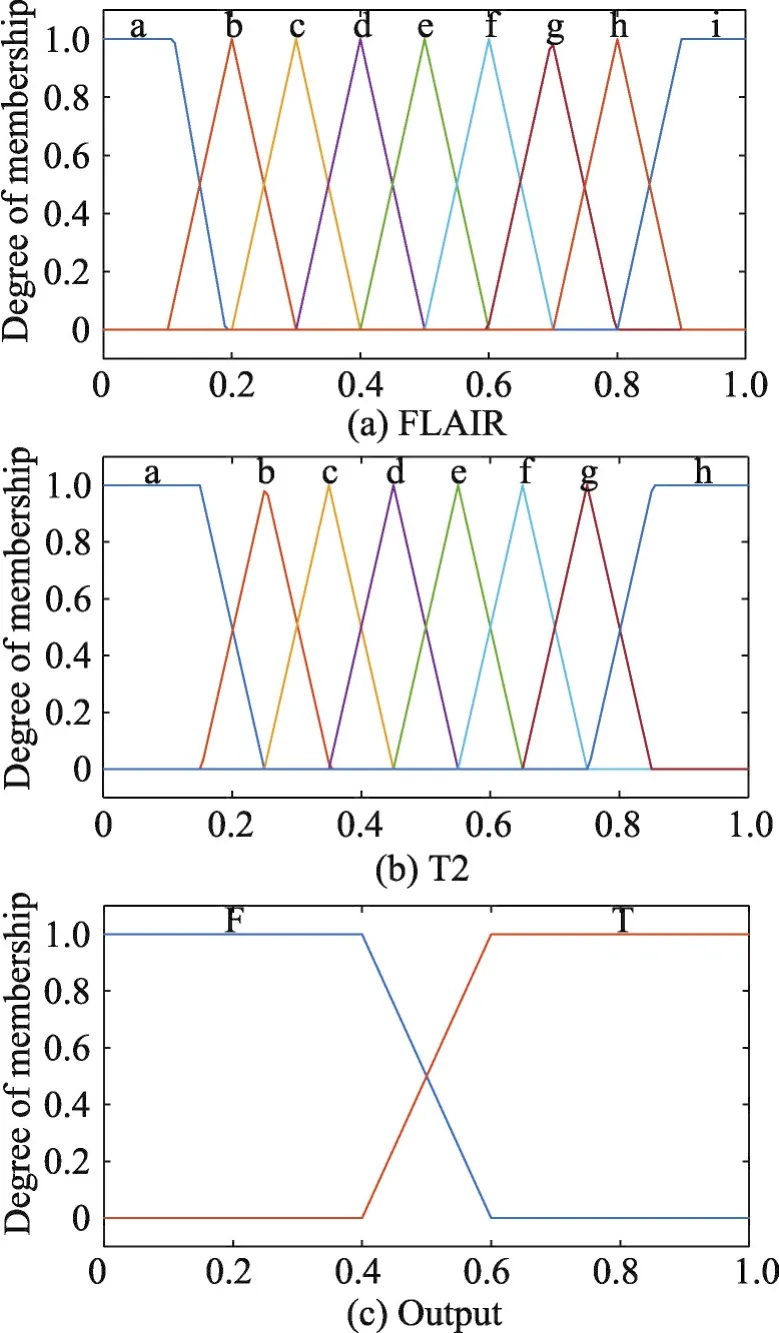
Fig.4 Input of fuzzy inference system图4 模糊推理系统的输入
由于模糊推理系统有两个输入,分别存在9种和8种状态,从而模糊规则共分为9×8=72种情况。通过对训练集样本进行分析,得到具体的推理规则,如表1所示。

Table 1 Fuzzy rule表1 模糊规则
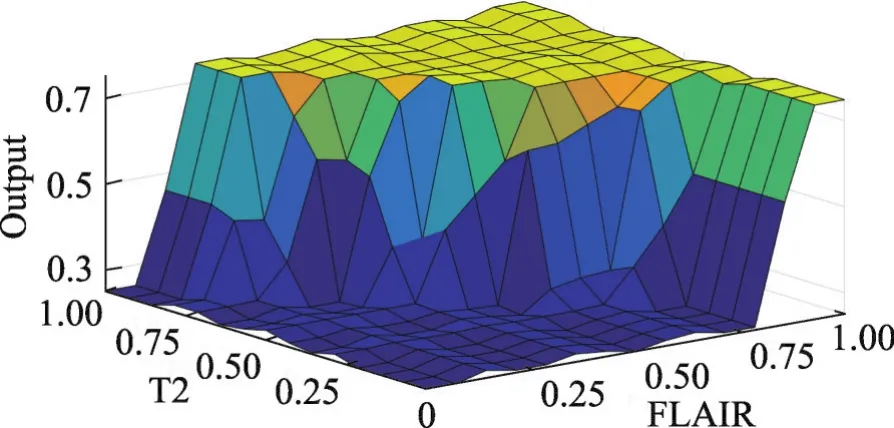
Fig.5 Relationship graph of input and output图5 输入输出关系图
该模糊系统的输出为最终的非肿瘤概率。当输出结果小于0.50时,则认为该图像对应像素点属于非肿瘤区域;而当输出结果大于0.50时,则认为该图像对应像素点属于肿瘤区域。
3.4 算法流程
在利用本文算法进行脑肿瘤分割时,原始脑肿瘤图像的对比度低,导致肿瘤区域分割困难,因此,首先需要对原始图像进行预处理。本文使用强度归一化方法,将每幅图像的灰度值均匀拉伸至0~255以增强图像对比度。
整体算法分为两阶段:训练阶段和预测阶段,如图6所示。
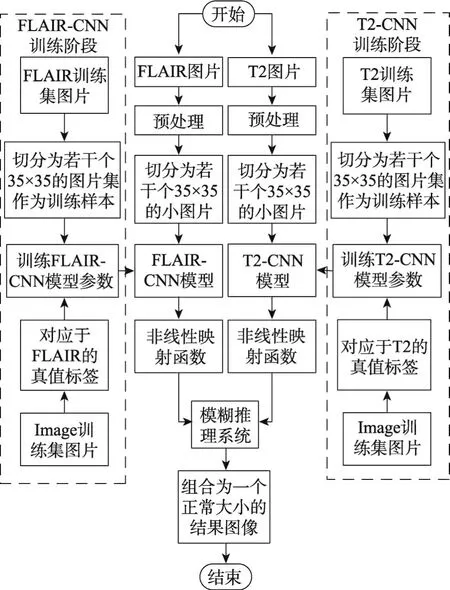
Fig.6 Flow chart of algorithm图6 算法流程图
在训练阶段,将选择好的FLAIR和T2类型的脑肿瘤图像经过预处理后分割为若干个35×35的图片集作为训练样本,并将其对应的专家分割结果图的像素点作为该训练样本的真值标签。然后,分别针对FLAIR和T2图像分割出的图片集进行训练,得到FLAIR-CNN模型和T2-CNN模型。最后,分别针对FLAIR和T2图像进行非线性映射。
在预测阶段,将FLAIR图像和T2图像经过预处理后切分为35×35的图像块,对于每个图像块,分别利用对应的CNN模型进行预测得到初始预测概率。然后,将初始预测概率经过非线性映射函数作为模糊推理系统的输入,当模糊推理系统的输出大于0.50时,认为该点属于肿瘤区域,否则属于非肿瘤区域。最后,通过对所有图像块的处理,将结果组合成为一个正常大小的预测图像,其中白色表示肿瘤区域,黑色表示非肿瘤区域。
4 实验结果与分析
为了验证本文方法的正确性和有效性,分别与卷积神经网络应用于单模态和多模态图像时脑肿瘤的分割结果进行对比实验。实验所用的MRI图像来自在线图库BRATS(brain tumor image segmentation benchmark,https://www.smir.ch/BRATS/Start2015),该图库由Menze、Jakab、Bauer等人组建,由包括ETH Zurich、University of Bern、University of Debreen 和University of Utah在内的机构提供完全匿名的患者数据[17]。图库中包含神经胶质瘤患者的4种已配准图像FLAIR、T1、T1c和T2。实验中从图库选取50例患者图像数据用于训练,另选取10例患者图像数据用于测试。实验机器采用Intel Core i7 3.5 GHz处理器,并搭载Nvidia GeForce GTX1070的GPU。
为了定量评估新算法的性能,本文选用相似性系数(dice similarity coefficient,DSC)、灵敏度(sensitivity)和阳性预测率(predictive positivity value,PPV)[18]作为脑肿瘤分割结果的评价技术指标。其中,DSC相似性系数描述实验分割结果和真值肿瘤之间的重叠程度[19];灵敏度表示正确分割的肿瘤点占真值肿瘤的比例;PPV阳性预测率表示分割正确的肿瘤点占分割结果为肿瘤点的比例。定义如下:

其中,P表示模型预测结果的肿瘤区域;T表示专家标定的肿瘤区域。
4.1 模型训练与非线性映射函数的确定
实验中,从图库选取50例患者图像数据进行训练。
首先,分别对FLAIR和T2图像进行切分,并将切分后的35×35大小的图像块作为卷积神经网络的训练集。训练完成后分别得到针对FLAIR图像的FLAIR-CNN模型和针对T2图像的T2-CNN模型。
然后,分别将针对FLAIR图像和T2图像的训练集作为FLAIR-CNN模型和T2-CNN模型的输入,对比模型预测值和真实值,并统计出所有错误预测点的预测概率分布。对于FLAIR-CNN模型和T2-CNN模型而言,预测错误点的预测分布概率如图7所示。
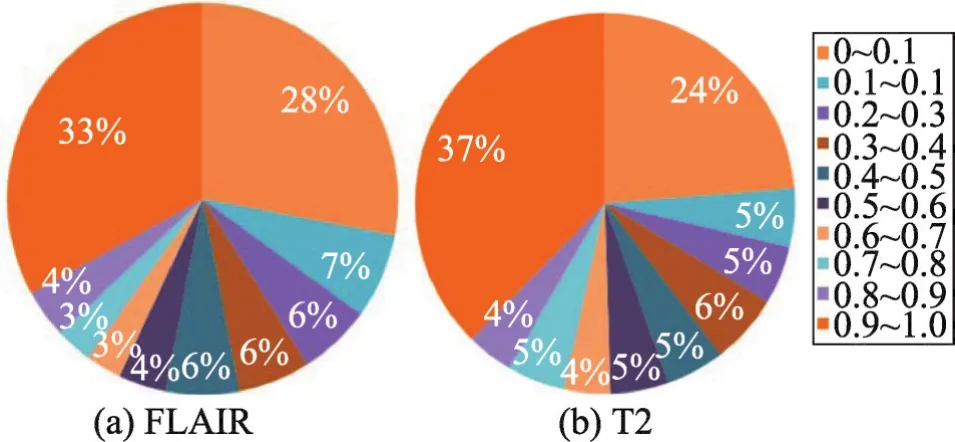
Fig.7 Error prediction point distribution图7 错误预测点概率分布图
图7(a)表示对于FLAIR-CNN模型所有预测错误点的概率分布图,图7(b)表示对于T2-CNN模型所有预测错误点的概率分布图。从图7中可以发现,绝大部分的预测错误点的概率集中在0~0.1区域和0.9~1.0区域。过于集中的概率分布并不利于针对不同概率来建立模糊推理系统进行推断。因此,本文分别对通过FLAIR-CNN模型和T2-CNN模型得到的预测概率进行非线性映射,并将其结果作为模糊推理系统的输入。为了将如图7所示的概率分布进行均衡化,使得其在每个区域的概率相似,构建了如图8所示的非线性映射函数。
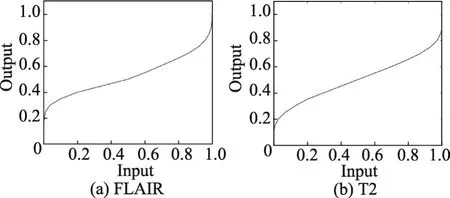
Fig.8 Nonlinear mapping function图8 非线性映射函数
图8(a)表示针对FLAIR-CNN模型的非线性映射函数,图8(b)表示针对T2-CNN模型的非线性映射函数。经过非线性映射后,FLAIR-CNN模型和T2-CNN模型所有错误预测点的分布图如图9所示。
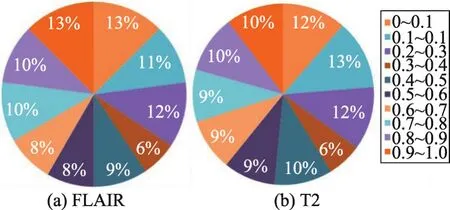
Fig.9 Error prediction point distribution after nonlinear mapping图9 经过非线性映射后错误预测点分布图
4.2 与单模态MRI图像分割的比较
核磁共振为患者提供4种模式的脑肿瘤成像,不同模态成像侧重的信息有很大区别。对于包括水肿在内的脑肿瘤分割,FLAIR图像和T2图像的轮廓更为明显。为了验证本文算法的有效性,将其与单独使用FLAIR图像或T2图像训练得到的单模态脑肿瘤MRI分割模型进行对比,实验结果如图10所示。

Fig.10 Compared with single mode MRI segmentation method图10 与单模态MRI图像分割方法比较结果图
图10中,(a)为患者的FLAIR成像,(b)为同一患者的T2成像,(c)和(d)分别表示单独使用FLAIR图像和T2图像得到的训练模型的分割结果,(e)为专家标定的分割结果,(f)为本文算法的分割结果。从图中可看出,单独使用FLAIR分割得到的结果基本轮廓清晰,精确度较高,但是预测肿瘤内部有多个漏判点,且在非肿瘤区域也存在部分较为集中的错判区域。而单独使用T2图像进行训练得到的模型分割结果虽然肿瘤区域分割良好,但在非肿瘤区域存在多处错误判别区域。本文结合两种单模态图像的训练模型分割结果,一方面克服了非肿瘤区域过多的错判点,另一方面避免了肿瘤区域内部的漏判,最终达到了更好的分割结果。
FLAIR和T2两种单模态分割方法和本文算法的分割比较结果如表2所示。

Table 2 Compared with single mode segmentation method表2 与单模态分割方法比较结果
从表2中可以看出,相比T2,单独使用FLAIR图像预测的分割精确度和阳性预测率更高,而灵敏度略差。对比FLAIR和T2两种单模态MRI图像分割结果可以看出,本文算法在相似性指数、灵敏度和阳性预测率上都有很大提升。
4.3 与多模态MRI图像分割的比较
为了定量评估算法的性能,将本文算法同Havaei[11]和Pereira[15]所提算法分别进行比较,分割结果如图11所示。
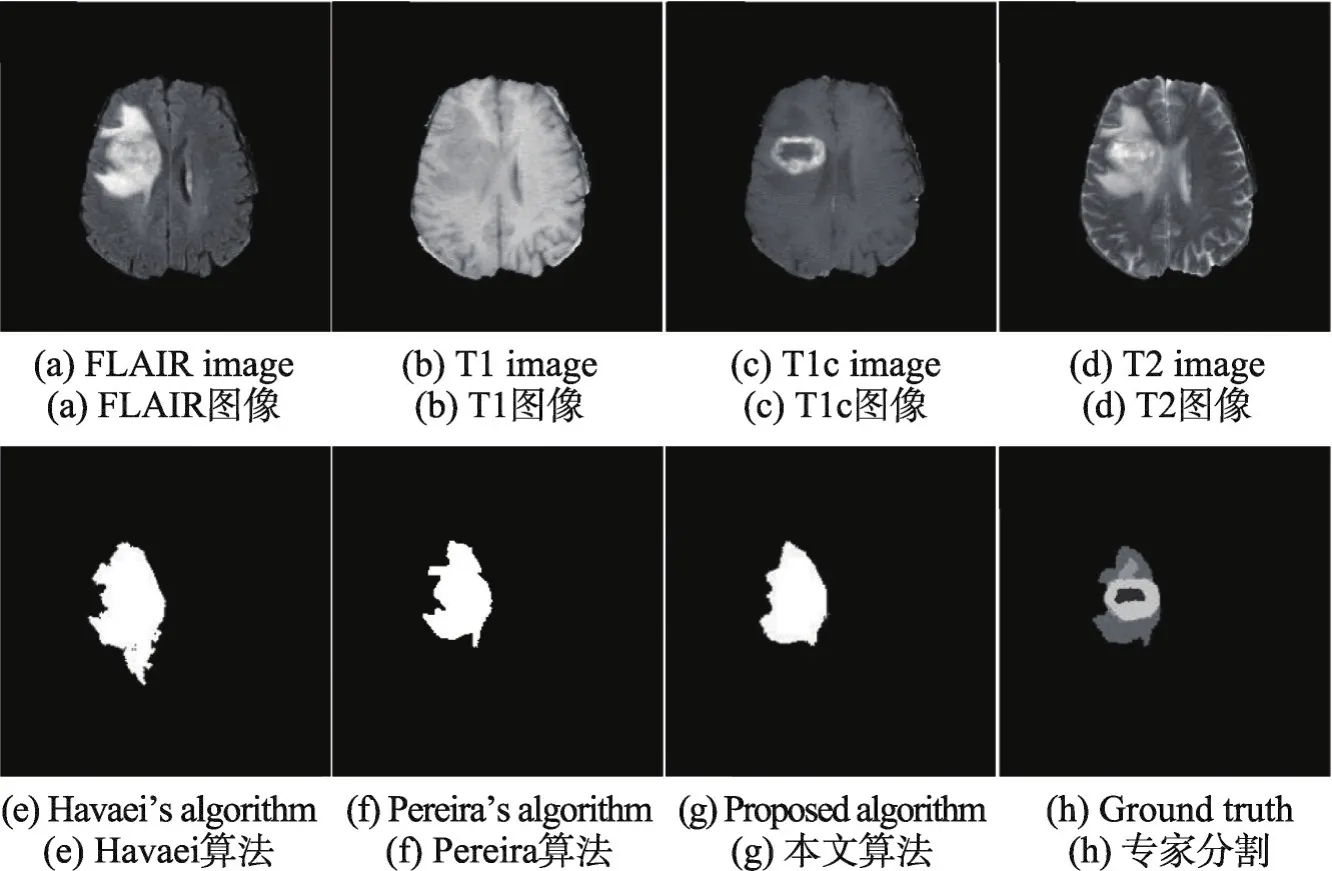
Fig.11 Compared with multi-modal MRI segmentation method图11 与多模态MRI图像分割方法比较结果图
图11中,(a)、(b)、(c)、(d)分别为脑肿瘤MRI图像的FLAIR、T1、T1c和T2图像。(e)为k近邻机器学习方法分割结果,(f)为小卷积核下的卷积神经网络方法分割结果,(g)为本文算法的脑肿瘤分割结果,(h)为专家标定的脑肿瘤分割结果。对比图(e)、(f)、(g)可以看出,(e)的分割结果相对较差,而(f)和(g)的分割结果相对较好。
为了进一步定量比较3种不同脑肿瘤分割算法的分割性能,针对10例测试图像,分别用3种脑肿瘤分割算法进行分割,并用10例测试图像的DSC、Sensitivity和PPV的平均值来评估算法性能,结果如表3所示。

Table 3 Segmentation performance evaluation of 3 segmentation methods表3 3种分割方法的分割性能评估
本文算法分割一幅完整脑肿瘤图像平均时间约1.2 min。对比Havaei的算法结果,尽管耗时略长,但在Sensitivity指标上提高了12%,DSC和PPV也同时提高了4%,使最终分割结果有了显著提升。对比Pereira提出的算法,本文降低了神经网络结构的复杂度,使得分割速度提升近7倍。此外,通过引入模糊推理系统对FLAIR和T2图像特征的结合,使得分割结果相比Pereira提出的算法在DSC和Sensitivity指标上分别提升1%和3%。
5 总结
针对MRI脑肿瘤图像分割,本文提出一种结合卷积神经网络和模糊推理系统的全自动脑肿瘤MRI图像分割算法。本文算法首先通过卷积神经网络分别处理FLAIR和T2图像,然后将预测概率经过非线性变换作为模糊推理系统的输入,最后对其输出以0.50为阈值得到像素点分类结果,并将全部像素点组合完成脑肿瘤分割。本文算法充分利用了脑肿瘤不同类型图像的特性。实验结果证明,本文算法在分割性能上相比传统方法有了一定程度的提升。
[1]Álvarez J M,Gevers T,Lecun Y,et al.Road scene segmentation from a single image[C]//LNCS 7578:Proceedings of the 12th European Conference on Computer Vision,Florence,Oct 7-13,2012.Berlin,Heidelberg:Springer,2012:376-389.
[2]Liang Zhipei,Lauterbur P C.Principles of magnetic resonance imaging:a signal processing perspective[M].Washington:SPIE,2000.
[3]Wen PY,Macdonald D R,Reardon D A,et al.Updated response assessment criteria for high-grade gliomas:response assessment in neuro-oncology working group[J].Journal of Clinical Oncology:Official Journal of the American Society of Clinical Oncology,2010,28(11):1963-1972.
[4]Liu Jin,Li Min,Wang Jianxin,et al.A survey of MRI-based brain tumor segmentation methods[J].Tsinghua Science and Technology,2014,19(6):578-595.
[5]Corso J J,Sharon E,Dube S,et al.Efficient multilevel brain tumor segmentation with integrated Bayesian model classification[J].IEEE Transactions on Medical Imaging,2008,27(5):629-640.
[6]Weglinski T,Fabijanska A.Brain tumor segmentation from MRI data sets using region growing approach[C]//Proceedings of the 7th International Conference on Perspective Technologies and Methods in MEMS Design,Polyana,May 11-14,2011.Piscataway:IEEE,2011:185-188.
[7]Deng Wankai,Xiao Wei,Deng He,et al.MRI brain tumor segmentation with region growing method based on the gradients and variances along and inside of the boundary curve[C]//Proceedings of the 3rd International Conference on Biomedical Engineering and Informatics,Yantai,Oct 16-18,2010.Piscataway:IEEE,2010:393-396.
[8]Khotanlou H,Colliot O,Atif J,et al.3D brain tumor segmentation in MRI using fuzzy classification,symmetry analysis and spatially constrained deformable models[J].Fuzzy Sets&Systems,2009,160(10):1457-1473.
[9]Gopal N N,Karnan M.Diagnose brain tumor through MRI using image processing clustering algorithms such as fuzzy C means along with intelligent optimization techniques[C]//Proceedings of the 2010 IEEE International Conference on Computational Intelligence and Computing Research,Coimbatore,Dec 28-29,2010.Piscataway:IEEE,2010:1-4.
[10]Logeswari T,Karnan M.An improved implementation of brain tumor detection using segmentation based on soft computing[J].Journal of Cancer Research and Experimental Oncology,2010,2(1):6-14.
[11]Havaei M,Jodoin P M,Larochelle H.Efficient interactive brain tumor segmentation as within-brainkNN classification[C]//Proceedings of the 22nd International Conference on Pattern Recognition,Stockholm,Aug 24-28,2014.Washington:IEEE Computer Society,2014:556-561.
[12]Lécun Y,Bottou L,Bengio Y,et al.Gradient-based learning applied to document recognition[J].Proceedings of the IEEE,1998,86(11):2278-2324.
[13]Zhao Liya,Jia Kebin.Multiscale CNNs for brain tumor segmentation and diagnosis[J].Computational&Mathematical Methods in Medicine,2016(7):8356294.
[14]Havaei M,Davy A,Warde-Farley D,et al.Brain tumor segmentation with deep neural networks[J].Medical Image Analysis,2017,35:18-31.
[15]Pereira S,Pinto A,Alves V,et al.Brain tumor segmentation using convolutional neural networks in MRI images[J].IEEE Transactions on Medical Imaging,2016,35(5):1240-1251.
[16]Sharma M,Mukharjee S.Artificial neural network fuzzy inference system(ANFIS)for brain tumor detection[EB/OL].(2012-10).https://arxiv.org/abs/1212.0059v1.
[17]Menze B H,Jakab A,Bauer S,et al.The multimodal brain tumor image segmentation benchmark(BRATS)[J].IEEE Transactions on Medical Imaging,2015,34(10):1993-2024.
[18]Lyksborg M,Puonti O,Agn M,et al.An ensemble of 2D convolutional neural networks for tumor segmentation[C]//LNCS 9127:Proceedings of the 19th Scandinavian Conference on Image Analysis,Copenhagen,Jun 15-17,2015.Berlin,Heidelberg:Springer,2015:201-211.
[19]Dice L R.Measures of the amount of ecologic association between species[J].Ecology,1945,26(3):297-302.

