基于线粒体COⅠ和16S rRNA基因序列比较分析东海带鱼群体遗传多样性
卞光明 王娜泠 胡则辉 王跃斌 胡成硕 柴学军
(1. 浙江省海洋水产研究所, 浙江海洋大学海洋与渔业研究所, 浙江省海水增养殖重点实验室, 舟山 316021;2. 江苏省射阳县长荡镇农业技术推广服务中心, 盐城 224322)
带鱼(Trichiurus lepturus)隶属鲈形目(Perciformes), 带鱼科(Trichiuridae), 带鱼属(Trichiurus),是东海区最重要的渔获对象, 其捕捞产量居于经济鱼类产量之首。由于捕捞强度日益增大以及环境因子变化, 东海带鱼资源面临巨大压力, 出现渔获质量较差、群体组成小型化、性成熟提前以及年龄结构失衡等现象, 开展带鱼种质资源研究工作刻不容缓。
带鱼种群的适应能力、生存能力以及进化潜力都与其遗传多样性有关联, 所以群体遗传多样性分析是开展种质资源保护工作的前提和基础。线粒体DNA分子量小、进化速度快, 突变形成的多态位点可反映出群体遗传特征、种群分化等特点, 是研究群体分化以及种质资源评估的理想手段[1]。线粒体COⅠ基因进化速率快, 包含遗传进化信息量大, 16S rRNA基因结构序列较为保守, 可以简便地使用通用引物进行PCR扩增, 因此,COⅠ与16S rRNA基因在生物群体遗传学中应用广泛[2—7]。
目前东海带鱼的研究主要集中在摄食[8]、资源评估模型应用[9]、繁殖[10]、资源动态[11]等方面, 关于东海带鱼分子标记群体分析, 仅见郑文娟等[12]基于线粒体控制区分析3个舟山海域带鱼群体多样性。本文基于线粒体COⅠ和16S rRNA基因对东海海域3个带鱼群体进行比较分析遗传距离, 构建分子系统树, 探讨东海带鱼不同群体基因交流情况,以期从线粒体水平上了解东海带鱼资源的遗传背景, 为带鱼资源的合理开发提供参考数据。
1 材料与方法
1.1 实验材料
本研究所用的东海带鱼样品由双拖船取自中国东海海域不同的3个群体, 现场取其背部肌肉置于酒精中保存, 并编号A、B、C, 每群体样本数、采集地点、采集时间分别为群体A (19、122°32′E 29°55′N、2015年10月)、群体B (34、123°30′E 26°75′N、2016年4月)、群体C (19、124°24′E 27°26′N、2016年4月)置于超低温冰箱(-70℃)保存备用, 具体采样情况见图 1(坐标系为WGS84)。
1.2 实验方法
基因组DNA的提取采用TIANamp Marine Animals DNA Kit试剂盒推荐的方法提取带鱼基因组总DNA, 通过0.8%的琼脂糖凝胶电泳检测其完整性, 并用Nanodrop 2000测DNA的质量与浓度, 置于-20℃保存。
线粒体COⅠ基因扩增及测序从GenBank中下载带鱼线粒体COⅠ基因序列[13], 运用Oligo7软件[14]设计COⅠ基因PCR扩增引物:COⅠ-F: 5′-CGTGGCAATTACCCGTTGATTCTTCT-3′和COⅠ-R: 5′-GACTGCACTAAGACAAAGGCTGG TT-3′(上海生工合成), 目的基因片段大小约1500 bp。PCR扩增体系为25 μL, 包含2×TaqMasterMix(康维世纪)12.5 μL, 模板DNA 100 ng, 10 μmol/L的引物各1 μL, ddH2O补足至25 μL。PCR反应条件为:94℃预变性5min; 94℃变性1min、50℃退火90s、72℃延伸45s, 共35个循环; 最后72℃延伸10min。PCR产物经1.2%琼脂糖凝胶电泳检测验证条带的大小后, 送往上海杰李生物技术公司进行纯化和双向测序。
线粒体16S rRNA片段扩增及测序所用引物序列[15]为16S-F:5′-GCCTGTTTATCAAAAAC AT-3′和16S-R:5′-CCGGTCTGAACTCAGATCA CGT-3′(上海生工合成), 目的片段大小约600 bp。PCR反应总体积为25 μL, 含有模板DNA 100 ng,0.5 μL上、下游引物(10 μmol/L), 最后补充去离子水至25 μL, 反应程序为: 94℃预变性2min, 然后进行35个循环, 每个循环包括94℃ 45s, 50℃ 1min,72℃ 1min, 在最后一个循环结束后在72℃延伸10min。PCR产物经1%琼脂糖凝胶电泳检测后委托上海杰李生物公司进行正向测序。
1.3 数据处理
用ContigExpress Project[16]对测序结果进行拼接, 然后使用BioEdit[17]对所得序列进行编辑及人工核查。利用DnaSP 5.0软件[18]计算群体的单倍型(H)、核苷酸多态位点数(S)、平均核苷酸多态性(K)、核苷酸多样性指数(π)、遗传分化系数(Gst)与固定系数(Fst), 进行Tajima中性检验[19]。用MEGA 5.0软件[20]分析序列碱基组成, 并使用Kimura two-Parameter模型计算带鱼群体间的遗传距离, 利用邻接法(Neighbor-Jioning, NJ)构建个体间的分子发育进化树, 并采用Bootstrap 1000检验系统树各分支的置信度。

图 1 采样点地理位置Fig. 1 Locations of the sampling stations
2 结果
2.1 线粒体COⅠ和16S rRNA基因片段序列特征分析
测序得到的目的片段经BioEdit比对及人工校正去除序列端部不稳定部分, 并通过BLAST分析比较, 确认得到1130 bp的COⅠ基因片段和554 bp的16S rRNA基因片段。通过MEGA 5.0软件计算72个带鱼样品的COⅠ和16S rRNA序列的碱基组成,COⅠ基因序列碱基T、C、A、G和A+T的平均含量分别为29.0%、28.9%、24.4%、17.7%和53.4%;16S rRNA基因序列的碱基T、C、A、G和A+T的平均含量分别为22.7%、27.6%、28.0%、21.7%和50.7%, A+T含量均略高于C+G, 符合硬骨鱼类线粒体DNACOⅠ和16S rRNA基因片段的碱基组成特征。
16S rRNA基因片段共检测到8种单倍型(Hap 1—Hap 8), 8个多态位点, 约占总序列的1.44%, 包括5个单一变异位点和3个简约信息位点(表 1)。其中, 转换位点7个, 包含1个A/G转换, 位于241位点;6个C/T转换, 分别位于169、170、232、256、326以及329位点。1个A/T颠换, 位于320位点, 转换/颠换率R为7。群体A、群体B、群体C共用3个单倍型, 其余单倍型均为每个群体独享, 单倍型1在群体中分布频率为79% (72个个体)。COⅠ基因序列检测到43种单倍型, 多态位点49个, 约占总序列的4.34%, 包括20个单一变异位点和简约信息位点29个(表 2)。其中, 转换位点42个, 颠换位点7个, 无插入/缺失位点, 转换/颠换率R为6。
2.2 群体遗传多样性分析
本实验将16S rRNA基因片段和COⅠ基因片段合并为一条总长度为1684 bp (不包括插入/缺失)的片段, 以用于群体遗传多样性分析。通过DnaSP 5.0软件统计带鱼群体16S rRNA、COⅠ和联合基因序列遗传多样性参数(表 3)。
基于COⅠ基因片段, 群体A单倍型多样性(Hd)最高(0.9983), 群体B与群体C的单倍型多样性相近。群体A的平均核苷酸差异数(K)和核苷酸多样性指数(Pi)在3个群体中均最高, 分别为7.111和0.0063, 其次为群体C (5.766和0.0051)、群体B(5.111和0.0045)。基于16S rRNA基因片段, 群体A的单倍型多样性(Hd)最高为0.4678, 群体B、群体C相近。群体A平均核苷酸差异数(K)和核苷酸多样性指数(Pi)分别为0.632和0.0011, 在3个群体中均处于最高值, 群体C(0.596和0.0011)、群体B(0.512和0.0009)次之。根据16S rRNA和COⅠ联合基因片段, 群体A单倍型多样性(Hd)最高(0.9992), 群体B与群体C的单倍型多样性接近。群体A的平均核苷酸差异数(K)和核苷酸多样性指数(Pi)在3个群体中均最高, 分别为9.014和0.0076, 其次为群体C (7.021和0.0066)、群体B (6.732和0.0058)。根据各遗传多样性参数比较3个群体, 线粒体单基因和联合基因片段得出的结果一致。
2.3 遗传分化分析
群体内遗传分化基于带鱼COⅠ和16S rRNA基因序列, 用MEGA 5.0构建NJ系统树(图 2、图 3),拓扑图显示, 同一群体内大部分个体首先聚在一起,存在不同群体间带鱼个体聚合, 这个现象与带鱼群体内和群体间的遗传距离差异较小以及群体间的遗传分化系数较低相一致。
群体间遗传分化基于COⅠ基因序列片段,3个群体间, 群体A与群体C遗传距离最大(0.0061),群体B与群体C种间遗传距离最小(0.0048), 这与群体地理位置远近相符合, 群体A遗传距离最大, 其次为群体B、群体C, 分别为0.0063、0.0051和0.0046(表 4)。基于16S rRNA和联合16S rRNA和COⅠ基因的遗传距离比较情况与线粒体DNACOⅠ基因一致。

表 1 带鱼单倍型16S rRNA基因序列片段及变异位点分布Tab. 1 Variable sites of 16S rRNA gene sequence from different haplotypes of T. lepturus
根据3个群体间遗传距离, 以南海带鱼[21]为外群, 构建群体间的UPGMA系统树, 据图 4、图 5显示, 基于COⅠ基因序列, 群体B与群体C首先聚成一支, 接着与群体A聚类, 南海带鱼群体独立为一支。基于16S rRNA基因序列聚类情况与COⅠ基因类似, 但群体A与群体B和群体C的分化程度低, 3个群体几乎聚在一起, 南海带鱼群体独立为一支。
通过DNAsp 5.0软件统计结果显示,COⅠ基因片段和16S rRNA基因片段的总群体遗传分化系数(Gst)为-0.00183和-0.01899, 群体总固定系数(Fst)为0.04014和-0.02676, 基因流(Nm)为5.98和-9.59。如表 5所示, 基于COⅠ基因片段, 3个群体间的遗传分化系数(Gst)在-0.00787—0.00471, 平均为-0.00043;固定系数(Fst)范围-0.00750—0.06544, 均值为0.03718; 总群体的Tajima’sD检测为负值, 不显著(P>0.10); 基于16S rRNA基因片段, 遗传分化系数(Gst)、固定系数(Fst)的范围分别-0.01672—(-0.1032)、-0.03144—(-0.02283), 均值分别为-0.01399、-0.02683; 总群体的Tajima’sD检测为负值, 不显著(0.10>P>0.05)。
表 2 带鱼单倍型COⅠ基因序列片段及变异位点分布Tab. 2 Variable sites of COⅠ gene sequence from different haplotypes of T. lepturus
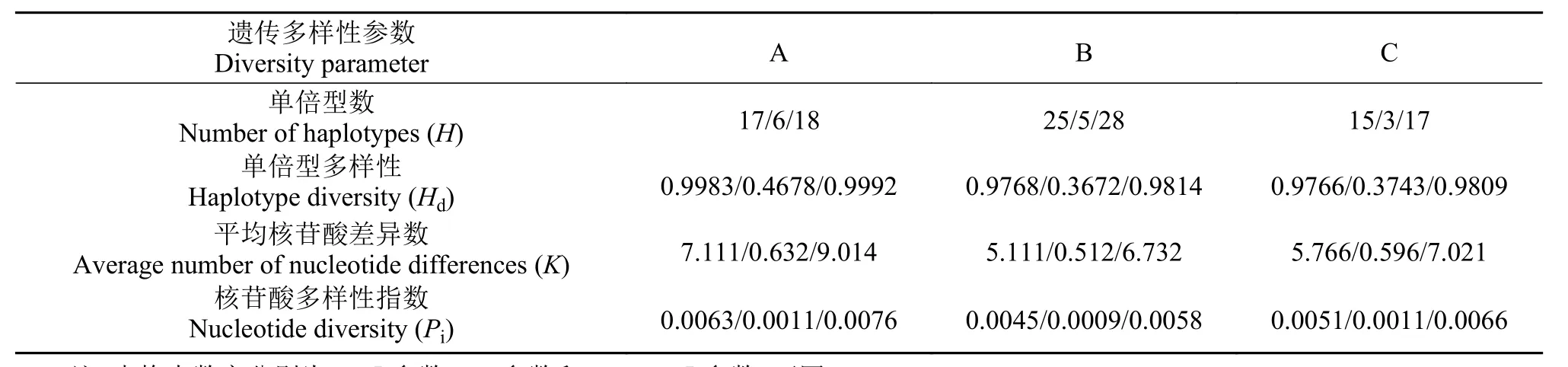
表 3 带鱼不同群体内线粒体DNA单倍型多样度Tab. 3 Haplotype diversity (H) of mtDNA in three populations of T. lepturus

图 2 基于COⅠ基因片段的NJ系统进化树Fig. 2 The construction of phylogenetic trees based on Neighbour-Joining (NJ) method for T. lepturus
3 讨论
3.1 线粒体COⅠ和16S rRNA基因片段序列比较分析
图 3 基于16S rRNA基因片段的NJ系统进化树Fig. 3 NJ phylogenetic tree based on 16S rRNA gene sequences of T. lepturus
碱基组成分析表明, 带鱼COⅠ和16S rRNA基因片段的A+T含量均微高于G+C含量, 符合硬骨鱼相应序列特征, 与蒙子宁[22]、李鹏飞等[23]、肖永双[24]研究结果基本一致。本研究的带鱼群体COⅠ基因序列共检测出49个变异位点, 占比对位点的4.34%,变异程度高于16S rRNA基因序列检测到的8个变异位点(1.44%), 这多是由于海洋动物线粒体DNA不同区域核苷酸的突变速率或不同基因片段的选择压力不同, 使得线粒体不同片段的核苷酸序列存在差异[25]。
3.2 遗传多样性分析
3个带鱼群体的COⅠ和16S rRNA基因片段的单倍型多样性(Hd)、平均核苷酸差异数(K)、核苷酸多样性指数(Pi)分别为0.97692和0.38772、5.96088和0.55595、0.00528和0.00101。蒙子宁[22]基于线粒体DNA 16S rRNA标记(500 bp)对黄海带鱼(n=12)多样性检测分析, 结果显示, 只检测到3个单倍型,遗传距离(D)为0.001, 单倍型多样性指数(Hd)为0.25, 核苷酸多样性指数(π)是0.00068; 李鹏飞等[23]对浙江近海水域带鱼群体(n=15)线粒体COⅠ基因序列(609 bp)变异分析, 发现15个序列中有10个单倍型, 转换/颠换率为5.5, 核苷酸多样性指数(π)为0.00513。本实验对比于蒙子宁[22]、李鹏飞等[23]所得的遗传距离以及核苷酸多样性指数相差无几, 平均核苷酸多样性差异略高, 推测可能由于两研究的试验样品数量不多。肖永双[24]对带鱼3个地理群体[南海(n=20)、温州(n=20)、北海(n=14)]的D-loop区序列(556 bp)测序分析, 在54个个体中检测到40个单倍型, 单倍型多样性指数(Hd)和核苷酸多样性指数(Pi)分别为0.98±0.01和0.008±0.005; 郑文娟等[12]研究舟山海域带鱼(n=54)线粒体DNA D-loop区序列(527 bp), 共检测到44个单倍型, 单倍型多样性指数(Hd)是0.991, 核苷酸多样性指数(Pi)为0.012。肖永双[24]、郑文娟等[12]得出的群体单倍型指数(Hd)和核苷酸多样性指数(Pi)均明显高于本实验, 主要是由于线粒体D-loop区进化速率高于线粒体DNACOⅠ基因序列和16S rRNA基因序列。
如表 6所示, 与中国近海其他常见鱼类相比, 基于COⅠ基因东海带鱼群体单倍型多样性以及核苷酸算多样性水平较高。鉴于一般海洋鱼类线粒体遗传信息单倍型多样性(Hd)、核苷酸多样性指数(Pi)分别处于0.11—1.00、0.0008—0.0320[32], 基于16S rRNA基因分析, 东海带鱼群体属于低单倍型、低核苷酸多样性(Hd<0.5,Pi<0.005), 基于COⅠ基因带鱼群体单倍型较高, 核苷酸多样性仍较低, 综合单倍型多样性(Hd)、平均遗传距离(D)、群体遗传多样性指数(Pi)等参数, 基于线粒体COⅠ和16S rRNA基因发现东海带鱼遗传多样性水平不高。

表 4 带鱼群体间和群体内平均遗传距离Tab. 4 Genetic distances among and within populations of T. lepturus
图 4 基于COⅠ基因序列带鱼群体间UPMGA系统树Fig. 4 Construction of a distance tree with the Unweighted Pair Group Method with Arithmatic Mean (UPGMA) among populations of T. lepturus based on COⅠ gene sequences
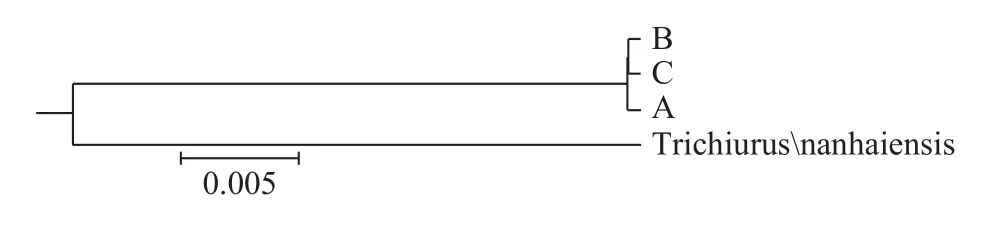
图 5 基于16S rRNA基因序列带鱼群体间UPMGA系统树Fig. 5 UPMGA phylogenetic tree among populations of T.lepturus based on 16S rRNA sequences

表 5 带鱼群体遗传分化系数Gst (上三角)与固定系数Fst (下三角)Tab. 5 Gst values (above the diagonal) and Fst values (below the diagonal) among populations of T. lepturus

表 6 海洋鱼类COⅠ基因遗传多样性Tab. 6 The genetic diversity of marine fish in COⅠ gene
3.3 群体间遗传结构
鉴于Shaklee等[33]提出的鱼类在属、种和种群三级水平上遗传距离分别是0.9、0.30和0.05的分类依据, 3个带鱼群体间均未达到种群的分化标准。3个带鱼群体与南海带鱼的COⅠ和16S rRNA基因序列的遗传距离分别0.11412和0.04907, 结果显示COⅠ基因能够较好区分东海带鱼和南海带鱼, 而线粒体16S rRNA基因序列标记则未能区分。
基于COⅠ基因片段, 群体B与群体C之间的遗传分化系数(Fst)出现负值, 说明群体B与群体C内部差异大于群体间差异, 群体B与群体C之间未发生遗传分化, 推测可能由于2个群体是随机交配的群体, 群体B虽处于闽东渔场, 但距离群体C(温台渔场)较近, 且群体B与群体C均处于东海带鱼索饵场与越冬场重叠区内[34], 两群体间交流较为频繁造成。基于16S rRNA基因片段, 群体间的遗传分化系数均为负值, 推测可能由于3个带鱼群体的线粒体16S rRNA基因序列较为保守、变异率低, 群体间分化程度较低导致。郑文娟等[12]、肖永双[24]分析带鱼群体线粒体控制区遗传多样性也得到群体间遗传分化系数为负值的结果。基于2个基因片段各群体遗传参数比较, 群体A在3个群体中遗传学背景最为丰富, 群体B遗传最稳定, 主要原因可能是群体A处于舟山渔场, 距离东海中部外侧中心越冬场较近, 且每年春季, 东海南部越冬带鱼群体向东北方向洄游至舟山渔场产卵, 是不同带鱼群体的交汇区域。带鱼分布广泛、长距离迁移能力强, 产卵、索饵、越冬均产生洄游, 一定程度上会缩小了群体间的差异。此外, 东海带鱼属于多次性产卵类型、鱼卵具有漂浮型, 产卵期间可排卵2—3次, 一般第1次与第2次排卵时间间隔约1个月[35], 这为带鱼群体间基因交流提供了时间和空间上的便利, 造成群体间遗传分化程度不显著。
东海带鱼渔获量一直位于我国海洋捕捞鱼类首位, 因带鱼资源被过度开发, 产量急剧下降。随着执行伏季休渔制度、启动东海带鱼国家级水产种质资源保护区、开展“一打三整治”工作、引进TAC及ITQ制度等一系列措施的实施, 东海带鱼产量逐渐稳定在年产8×108kg, 但根据东海区渔业资源动态监控网浙江站近年(2012—2015年)渔业资源动态调查监控资料来看[36], 东海带鱼资源量波动剧烈, 远未恢复到常年水平。故而, 在今后带鱼的资源管理和利用中, 继续以保护带鱼群体的遗传资源为中心, 合理控制捕捞力度, 避免人为基因渗透导致带鱼群体遗传多样性的丢失, 实现带鱼种质资源的可持续利用。除此之外, 采用其他分子技术或选择DNA其他基因区域获取多组序列数据进行全面评估东海带鱼群体的遗传多样性水平以及开展人工繁育等研究对于带鱼资源的持续利用也尤为重要。

