黄麻全基因组SSR鉴定与特征分析
姚嘉瑜 张立武 赵 捷 徐 益,2 祁建民 张列梅,*
黄麻全基因组SSR鉴定与特征分析
姚嘉瑜1,**张立武1,2,**赵 捷1徐 益1,2祁建民1张列梅1,*
1福建农林大学作物遗传育种与综合利用教育部重点实验室 / 福建省作物设计育种重点实验室 / 作物科学学院, 福建福州 350002;2福建农林大学海峡联合研究院基因组与生物技术中心, 福建福州 350002
黄麻是世界上重要的天然韧皮部纤维作物之一。然而, SSR标记的缺乏限制了黄麻的遗传改良。本研究从圆果种黄麻测序品种CVL-1的基因组、基因、CDS和cDNA中挖掘SSR信息, 利用SSR Primer软件查找SSR位点, 并分析其分布特征。结果表明, 基于基因组序列共开发了153,242个基因组SSR, 平均密度为467.20个SSR Mb–1; 基于cDNA序列开发了10,747个SSR, 平均密度为260.85 SSR Mb–1。大部分重复基元为二至四核苷酸, 占76.91%, 其中cDNA序列SSR中三核苷酸重复基元数量较多而基因组SSR中二核苷酸重复基元数量较多。对于不同类型的SSR重复基元, 随着重复单元数量的增加, 其基因组和cDNA的SSR分布频率呈现逐步降低特征。黄麻全基因组SSR标记鉴定, 不仅可以丰富黄麻分子标记的数量, 而且为剖析黄麻重要农艺性状的遗传机制奠定基础。
黄麻; 基因组; cDNA; SSR
黄麻为锦葵科(Malvaceae)黄麻属(spp.)一年生次生韧皮部纤维作物, 其纤维产量占世界麻类纤维总产量的80%。黄麻属有100多个种, 具有栽培价值的有圆果种()和长果种(), 两者具有不同生长习性, 存在生殖隔离, 皆为二倍体(2= 14), 主要在孟加拉、印度和中国等国家种植[1-3]。在世界范围内, 黄麻的产量和种植面积仅次于棉花, 是麻纺工业的重要原料, 每年黄麻生产产值达到23亿美元[3]。
开发重复性和稳定性好的SSR分子标记, 对于黄麻遗传多样性研究、重要性状的遗传基础剖析等具有重要意义。然而, 相对于水稻等模式作物, 黄麻分子标记开发与其他作物差距很大, 目前国内外报道较少[2-6]。这些研究报道主要集中在RAPD、AFLP、SSR、ISSR等分子标记, 少量基于二代测序获得的RAD[7-8]或SLAF[9]等分子标记。如Nishat等[5]用抗寒性有差别的2个亲本O-9897和Acc.1805, 创建了F2代作图群体, 用8个ISSR引物构建了总长度为87.3 cM的包括3个连锁群的长果种黄麻遗传连锁图谱, 3个连锁群的大小在4.8~ 52.9 cM之间, 相邻标记平均距离为8.73 cM, 该图谱远远不能满足基因定位、基因克隆等研究的要求。Das等[6]用纤维品质有差别的2个亲本JRO524和PPO4, 创建了F6代RIL作图群体, 用36个SSR标记构建长果种黄麻遗传连锁图谱, 但其只有6条连锁群, 标记密度达21.8 cM。由于SSR标记具有高效、高共显性等优点, 尤其是重复性好, 不同研究间可互比较, 是物种的起源与进化、遗传连锁图谱构建等研究优先考虑的分子标记[10]。国内外学者开发了水稻[11]、大麦[12]、玉米[13]、油菜[14-16]等作物的SSR标记, 为研究该作物的遗传多样性、遗传连锁图谱构建、关联分析等打下了基础。
最近, Islam等[3]发表了黄麻基因组草图(draft)。该研究利用二代测序技术Roche/454对黄麻基因组测序, 经组装发现, 圆果种CVL-1基因组大小为338 M (13.69 Gb raw data, Scaffold N50为4.1 Mb)。这表明已获得黄麻基因组草图, 但还没有组装到染色体水平。然而, 对于黄麻全基因组SSR鉴定尚未见报道。本研究旨在分析圆果种黄麻CVL-1基因组、基因、CDS和cDNA中SSR信息, 发掘SSR位点并分析其特征, 为黄麻遗传育种研究提供SSR信息。
1 材料与方法
通过NCBI公共数据库(https://www.ncbi.nlm. nih.gov/)获得圆果种CVL-1基因组、基因、CDS和cDN A序列(Sequence Read Archive [SRA]: SRP053213)。利用SSR Primer工具(http://hornbill. cspp.latrobe.edu.au/ ssrdiscovery.html)搜索这些序列所包含的SSR。筛选SSR的标准为, 二核苷酸重复次数>6次, 三核苷酸重复次数>4次, 四核苷酸重复次数>3次, 五核苷酸重复次数>2次。利用Primer 3软件查找SSR位点。
2 结果与分析
2.1 黄麻SSR分布的总体特征
分析了黄麻重复基元为2~6 bp, 且重复长度不短于12 bp的SSR分类和分布情况, 如表1所示。黄麻全基因组大小为338 Mb, 共开发了153,242个完整的SSR, 平均密度为467.20个SSR Mb–1。其中, I类(重复序列长度 ≥ 20 bp) SSR有38,917个(25.40%), 平均密度为118.65个SSRs Mb–1; 而II类(重复序列长度 ≥ 12 bp和<20 bp) SSR有114,325 (74.60%), 平均密度为348.55个SSR Mb–1。黄麻所有基因的cDNA大小为41.20 Mb, 共开发了10,747个SSR, 平均密度为260.85 SSR Mb–1, 包括1251个 (11.64%) I类SSR和9496 (88.36%) II类SSR, 其平均密度分别为30.36个SSR Mb–1和230.49个SSR Mb–1。
经过比较可以发现: 与基因组序列相比, cDNA序列具有较低的SSR密度, 而且具有较高的GC含量(57.79%)。I类SSR分布密度低于II类SSR。I类SSR在基因组、基因、CDS和cDNA中平均密度为118.65、81.67、19.87和30.36个SSR Mb–1, 而II类SSR相应的平均密度显著偏高, 分别348.55、295.20、163.15与230.49个SSR Mb–1。这表明I类SSR分布密度低于II类SSR。
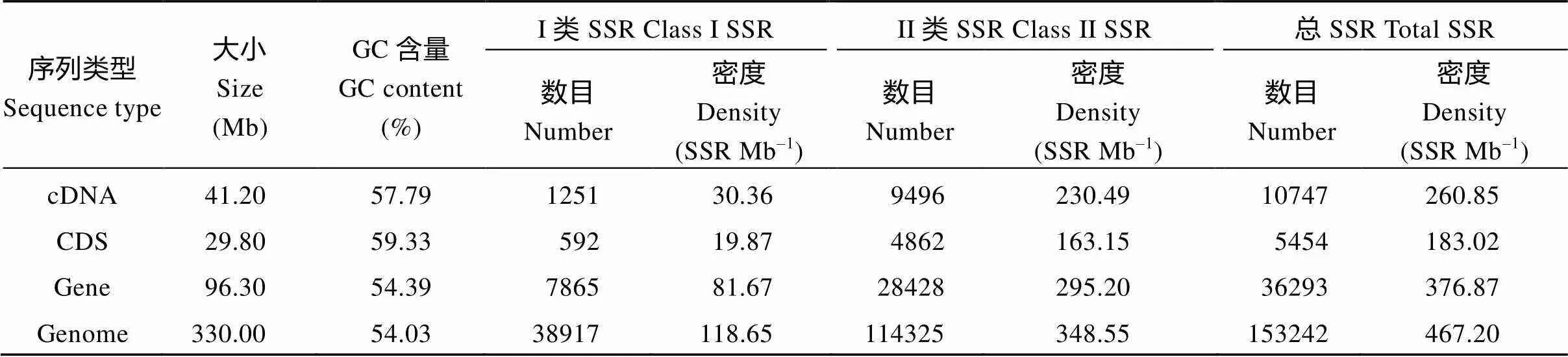
表1 黄麻SSR的分布特征
I类SSR指重复序列长度大于20 bp, 而II类SSR指重复序列长度大于12 bp且小于20 bp。
SSRs of class I mean repeat length ≥ 20 bp, SSRs of class II mean repeat length ≥ 12 bp and < 20 bp.
2.2 黄麻SSR不同重复类型的频率分布
对于II类SSR来说, 全基因组和cDNA中比例最多的SSR重复序列平均长度是12 bp, 分别占全基因组和cDNA中的45.98%和38.14% (图1-a), 占全基因组和cDNA中II类SSR的52.03%和48.19% (图1-b)。比例较多的SSR重复序列平均长度是15 bp, 分别占全基因组和cDNA中的18.90%和14.91%, 占全基因组和cDNA中II类SSR的21.39%和18.83%。
与II类SSR相比, 全基因组和cDNA中I类SSR的比例最多重复序列平均长度表现不同(图1-c)。全基因组中I类SSR比例最多的重复序列平均长度是20 bp, 具有7646个SSR; 而cDNA序列中I类SSR比例最多的重复序列平均长度是24 bp, 占29.58%, 其次是21 bp重复序列平均长度。
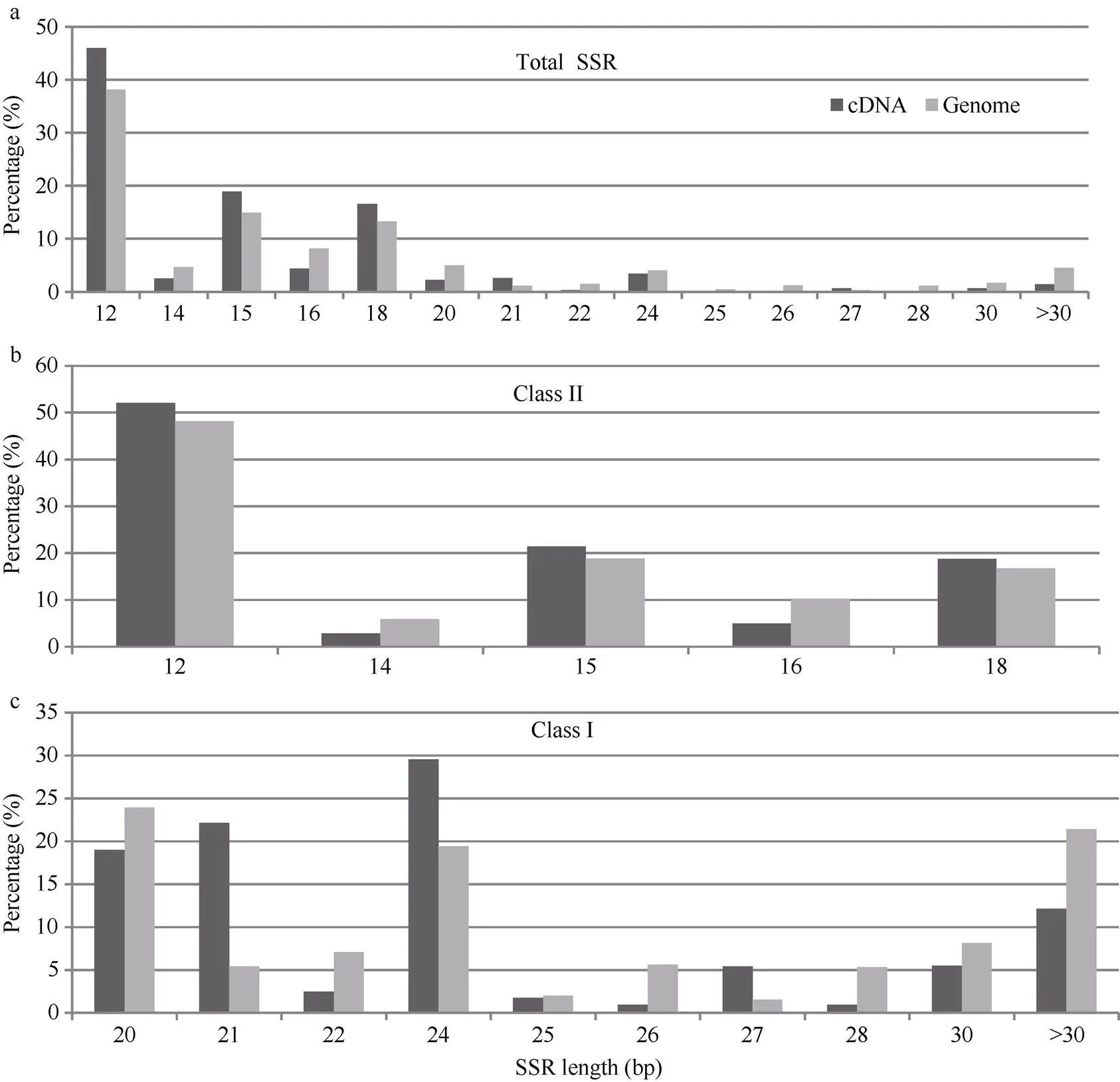
图1 黄麻SSR重复序列长度的频率分布
SSR重复基元在全基因组中比例较多的SSR重复基元是二核苷酸、三核苷酸、四核苷酸, 分别占22%、19%和23%。经比较, 不同重复基元的频率高低与核苷酸数量没有严格的相关性。如全基因组中四核苷酸重复基元的SSR总数略多于三核苷酸。同样地, 在cDNA中, 三核苷酸重复所占比例最多, 为43%, 其次是四核苷酸重复, 占13.3%, 二核苷酸重复和五核苷酸重复比较少, 分别占9.7%和7.1% (图2-a)。
按不同类型SSR来统计各重复基元的分布频率(图2-b和图2-c)。cDNA中I类SSR, 三核苷酸重复基元远远高于其他类型; cDNA中II类SSR呈现类似的特点, 推测作为蛋白质遗传密码子模板的cDNA, 三核苷酸重复基元在cDNA_SSR中数量比较突出。而基因组中I类SSR, 二核苷酸重复基元远远高于其他类型; 基因组中II类SSR, 二、三、四核苷酸重复基元均较多。
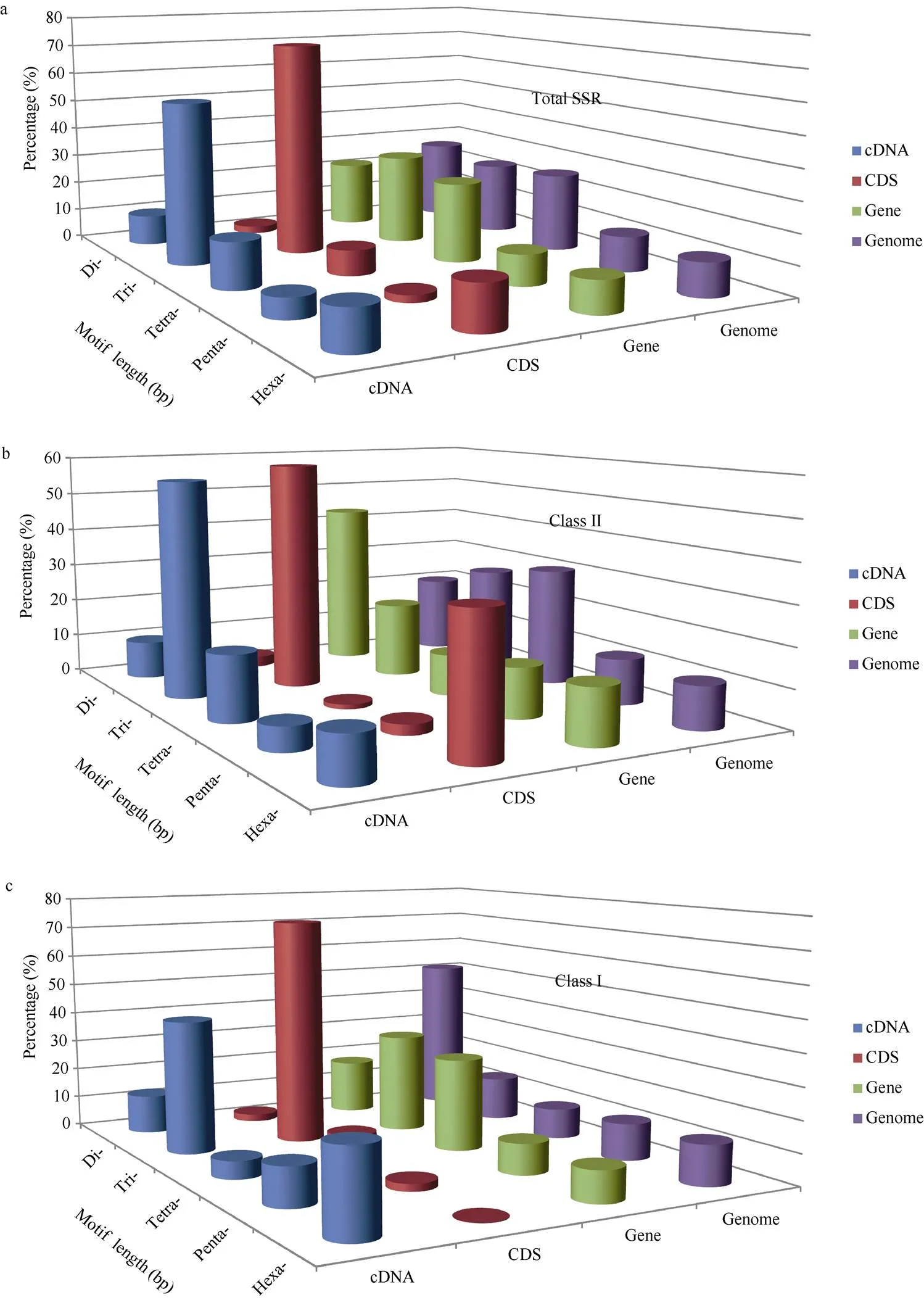
图2 黄麻基因组、基因、CDS和cDNA的SSR重复基元的频率分布
图3列出了黄麻SSR重复基元及平均重复序列长度的频率分布。随着分析的五种重复基元(2~6 bp)的数量增加, SSR分布的频率逐步降低, 特别是对于较长重复基元(如四至六核苷酸), 其频率降低最显著。
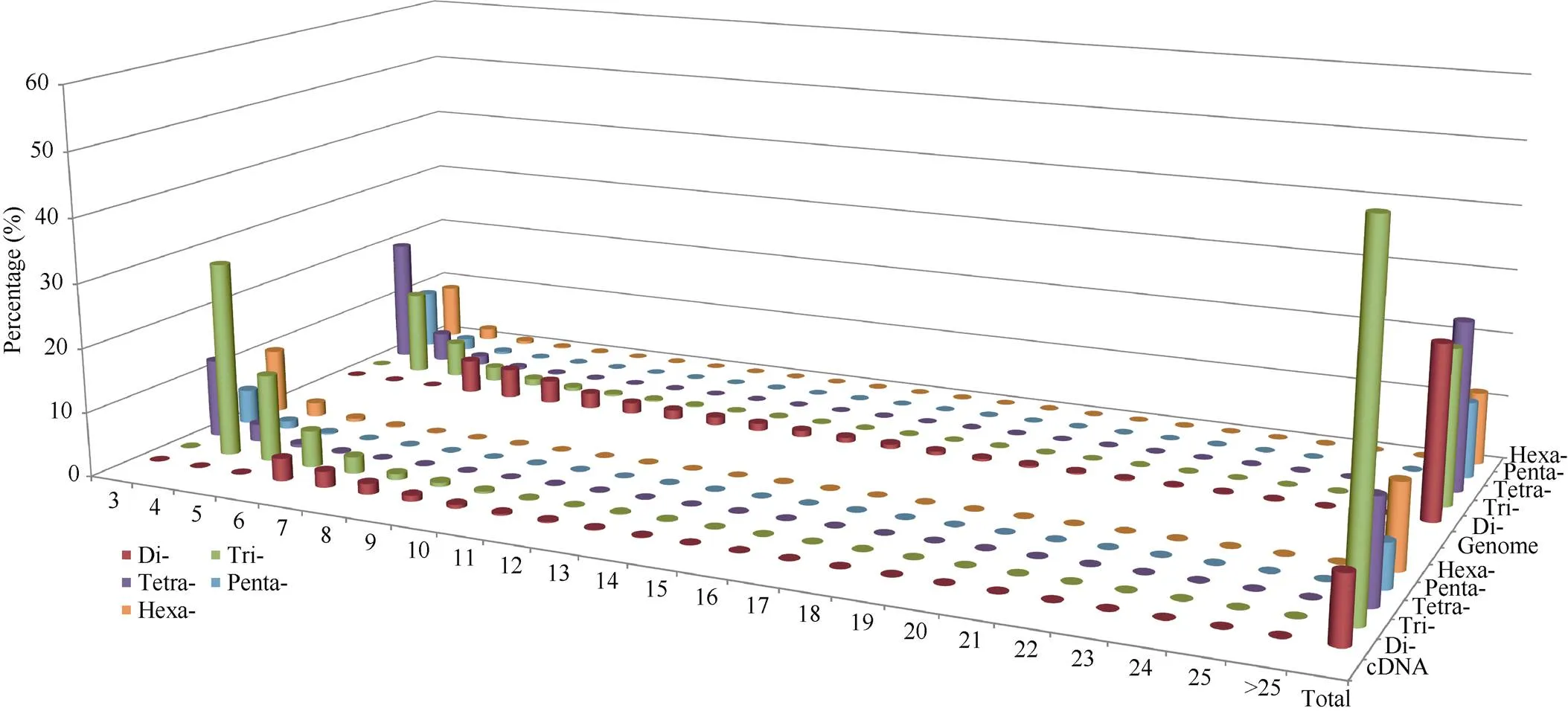
图3 黄麻基因组和cDNA的SSR重复基元及平均重复序列长度的频率分布
2.3 黄麻SSR重复基元种类及其频率分布
如表2所示, 重复基元的重复次数对于不同重复基元种类而言是不同的, 如三核苷酸重复基元的重复次数在基因组和cDNA中分别是二核苷酸的0.9倍和5.4倍, 分别是四核苷酸的0.9倍和3.5倍, 体现出三核苷酸重复基元在cDNA中较频繁地发生。对比黄麻SSR重复基元在全基因组和cDNA中的分布频率, 可以发现, SSR在基因组中, 二、三核苷酸的平均重复序列长度比其他种类要长, 显示出二、三核苷酸重复在黄麻基因组SSR中较频繁地发生, 且其重复基元数量在基因组SSR中比例也最多。统计黄麻SSR, 黄麻最长的平均重复序列长度为三核苷酸重复基元。其中, 重复基元AAT重复次数达2511, 重复序列长度达到7533 bp; 其次重复基元AAG, 重复次数达2246。Karaoglu等[17]研究显示, 较长的重复序列长度通常倾向于具有较高的突变率, 这与SSR的多态性息息相关。
考虑到二、三核苷酸重复在黄麻基因组SSR中比重大, 进一步分析其优势重复基元, 如图4所示。二核苷酸重复基元的SSR在基因组中, 以AT为主, 占75.88%。而CG重复基元则相当罕见, 仅占0.04%; 在cDNA中, 以AT和AG重复基元较多, 分别占32.9%和37.6%。三核苷酸重复基元的SSR在基因组中, AAT、AAG、ATA和ATT所占比例较多, 分别占13.4%、11.8%、11.7%和11.1%; 在cDNA中, AAG、AGA、CTT所占比例较多, 分别占13.4%、11.5%、9.4%和11.5%。
2.4 不同物种间SSR重复类型的频率分布比较
为了比较不同物种间SSR重复类型的频率分布特征, 我们选取基因组大小差不多的5个物种, 分别为2个单子叶植物(水稻和高粱)和3个双子叶植物(拟南芥、黄瓜L.和葡萄)。应用相同的标准来研究这6个物种基因组SSR分布特征, 即统计重复基元为2~6 bp, 重复长度不短于12 bp的SSR。表2列出黄麻和这5个物种SSR分布的总体特征。从SSR分布密度来看, 这6个物种的SSR分布密度变异范围从315.5 SSR Mb–1到738.5 SSR Mb–1。黄麻在分析的6个物种中SSR分布密度中等(467.2 SSR Mb–1)。高粱的SSRs密度最低, 仅为315.5 SSR Mb–1, 略低于拟南芥(364.1 SSR Mb–1)。
3 讨论
SSR标记对植物的遗传学、生态学、分类学、进化等研究至关重要。分析黄麻SSR在基因组、基因、CDS和cDNA中不同层面的重复基元分布频率和平均重复序列长度等信息, 有助于黄麻基因组的遗传研究, 并提供SSR位点信息。
3.1 黄麻SSR的分类及其频率分布
本研究从公共数据库获得黄麻基因组草图序列, 共开发了153,242个基因组SSR; 基于cDNA序列开发了10,747个SSR。结果表明, 大部分重复基元为二至五核苷酸, 其中cDNA序列中三核苷酸重复基元数量较多而基因组序列中二核苷酸重复基元数量较多。这与在大多数植物基因组中三核苷酸重复的分布频率较高的结果是一致的[14-16]。Hong等[19]分析白菜中SSR重复基元发现, 三核苷酸重复出现的频率是37%。Goff等[20]在水稻中, 也发现基因组SSR中三核苷酸重复占59%。本研究中, 黄麻SSR的分析结果显示, 二、三、四核苷酸中所有的重复基元类型都有, 但不同重复基元的比例存在显著差异。其中二核苷酸重复中AT类型最丰富, 三核苷酸重复中AAT和AAG最多。二核苷酸重复基元的SSR在基因组中, 以AT为主, 这与课题组前期利用公共数据库数据得出的结论类似[18]。
Temnykh等[11]研究发现, SSR长度大于或等于20 bp的SSR具有较高的多态性。为此, 将SSR划分为两类, 即I类SSR指重复序列长度大于20 bp, 而II类SSR指重复序列长度大于12 bp且小于20 bp。在黄麻全基因组和cDNA中, 较短的II类SSR比I类更为丰富。这种趋势可能是由于较长的I类重复是由较短的II类重复不对称配对产生的, 也可能是较长的I类重复具有不稳定性, 而较短的II类重复可能更耐受突变和保留。
3.2 黄麻SSR重复基元类型及其平均重复序列长度
在黄麻基因组中, 大多数SSR是二、三和四核苷酸重复序列, 它们占所有SSR的76.91%, 且三者的量相对比较平均。对于不同类型的SSR重复基元, 随着重复单元的数量增加, 其在基因组和cDNA的SSR分布频率呈现逐步降低; 随着重复次数的增加, 较长的重复基元, 五核苷酸和六核苷酸的分布频率会显著降低。
3.3 黄麻与不同物种间SSR特征比较
Morgante等[21]报道, 基因组大小与SSR密度或SSR数量之间呈现负相关。研究黄麻和另外5个物种基因组SSR分布特征印证这一点, 具有最大基因组(739 Mb)的高粱, SSR密度最低。在大豆(1115 Mb)和玉米(2365 Mb)基因组中也观察到类似的趋势[22]。在这6种植物的cDNA中, 三核苷酸SSR是最丰富的, 其次是四核苷酸和二核苷酸。作为蛋白质遗传密码子模板的cDNA, 三核苷酸重复基元在SSR中数量比较突出, 较频繁地发生, 可能具有类似于简并密码子的作用。黄麻基因组中的SSR密度高于基因组的转录区域cDNA, 这种现象与黄瓜和葡萄相一致, 而与拟南芥、高粱相反。
4 结论
黄麻全基因组大小约为338 Mb, 开发了153,242个SSR, 平均密度为467.20个SSR Mb–1。黄麻所有基因的cDNA大小为41.20 Mb, 开发了10,747个SSR, 平均密度为260.85 SSR Mb–1。大部分重复基元为二至四核苷酸, 占76.91%, 其中cDNA序列SSR中三核苷酸重复基元数量较多而基因组SSR中二核苷酸重复基元数量较多。不同物种间, 黄麻具中等SSR分布密度(467.2 SSR Mb–1)。
[1] 熊和平. 麻类作物育种学. 北京: 中国农业科学技术出版社, 2008. pp 208–296. Xiong H P. Breeding Sciences of Bast and Leaf Fiber Crops. Beijing: China Agricultural Science and Technology Press, 2008. pp 208–296 (in Chinese).
[2] 祁建民, 李维明, 吴为人. 黄麻的起源与进化研究. 作物学报, 1997, 23: 677–682. Qi J M, Li W M, Wu W R. Studies on origin and evolution of jute., 1997, 23: 677–682 (in Chinese with English abstract).
[3] Islam M S, Saito J A, Emdad E M, Ahmed B, Islam M M, Halim A, Hossen Q M M, Hossain M Z, Ahmed R, Hossain M S, Kabir S M T, Khan M S A, Khan M M, Hasan R, Aktar N, Honi U, Islam R, Rashid M M, Wan X H, Hou S B, Haque T, Azam M S, Moosa M M, Elias S M., Hasan A M M, Mahmood N, Shafiuddin M, Shahid S, Shommu N S, Jahan S, Roy S, Chowdhury A, Akhand A I, Nisho G M, Uddin K S, Rabeya T, Hoque S M E, Snigdha A R, Mortoza S, Matin S A, Islam M K, Lashkar M Z H, Zaman M, Yuryev A, Uddin M K, Rahman M S, Haque M S, Alam M M, Khan H, Alam M. Comparative genomics of two jute species and insight into fibre biogenesis., 2017, 3: 16223–16230.
[4] Basu A, Ghosh M, Meyer R, Powell W, Basak S L, Sen S K. Analysis of genetic diversity in cultivated jute determined by means of SSR markers and AFLP profiling., 2004, 44: 678–685.
[5] Nishat S, Haseena K, Nadim A, Sharkar M T K. Construction of an intraspecific linkage map of jute., 2006, 5: 758–762.
[6] Das M, Banerjee S, Dhariwal R, Vyas S, Mir R, Topdar N, Kundu A, Khurana J, Tyagi A, Sarkar D, Sinha M, Balyan H, Gupta P. Development of SSR markers and construction of a linkage map in jute., 2012, 91: 21–31.
[7] Biswas C, Dey P, Karmakar P G, Satpathy S. Discovery of large-scale SNP markers and construction of linkage map in a RIL population of jute ()., 2015, 35: 1–10.
[8] Kundu A, Chakraborty A, Mandal N, Das D, Karmakar P G, Singh N K, Sarkar D. A restriction-site-associated DNA (RAD) linkage map, comparative genomics and identification of QTL for histological fibre content coincident with those for retted bast fibre yield and its major components in jute (L., Malvaceae s. l.)., 2015, 35: 1–17.
[9] Tao A, Huang L, Wu G, Afshar R K, Qi J M, Xu J T, Fang P P, Lin L H, Zhang L W, Lin P Q. High-density genetic map construction and QTLs identification for plant height in white jute (L.) using specific locus amplified fragment (SLAF) sequencing., 2017, 18: 355–376.
[10] Zhang L W, Li S P, Chen L, Yang G S. Identification and mapping of a major dominant quantitative trait locus controlling seeds per silique as a single Mendelian factor inL., 2012, 125: 695–705.
[11] Temnykh S, DeClerck G, Lukashova A, Lipovich L, Cartinhour S, McCouch S. Computational and experimental analysis of microsatellitess in rice (L.): frequency, length variation, transposon associations, and genetic marker potential., 2001, 11: 1441–1452.
[12] Russell J, Fuller J, Macaulay M, Hatz B, Jahoor A, Powell W, Waugh R. Direct comparison of levels of genetic variation among barley accessions detected by RFLPs, AFLPs, SSRs and RAPDs., 1997, 95: 714–722.
[13] Pejic I, Ajmone-Marsan P, Morgante M, Kozumplick V, Castiglioni P, Taramino G, Motto M. Comparative analysis of genetic similarity among maize inbred lines detected by RFLPs, RAPDs, SSRs and AFLPs., 1998, 97: 1248–1255.
[14] Cheng X, Xu J, Xia S, Gu J, Yang Y, Fu J, Qian X, Zhang S C, Wu J S, Liu K. Development and genetic mapping of microsatellite markers from genome survey sequences in., 2009, 118: 1121–1131.
[15] Li H, Chen X, Yang Y, Xu J S, Gu J X, Fu J, Qian X J, Zhang S C, Wu J S, Liu K. Development and genetic mapping of microsatellite markers from whole genome shotgun sequences in., 2010, 28: 585–596.
[16] Xu J, Qian X, Wang X, Li R, Cheng X, Yang Y, Fu J, Zhang S C, King G J, Wu J S, Liu K. Construction of an integrated genetic linkage map for the A genome ofusing SSR markers derived from sequenced BACs in., 2010, 11: 594–603.
[17] Karaoglu H, Lee C M Y, Meyer W. Survey of simple sequence repeats in completed fungal genomes., 2005, 22: 639–649.
[18] 张立武, 袁民航, 何雄威, 徐建堂, 祁建民, 刘星, 方平平, 林荔辉, 陶爱芬. 基于GenBank黄麻EST-SSR标记的开发及其通用性评价. 作物学报, 2014, 40: 1028–1034. Zhang L W, Yuan M H, He X W, Xu JT, Qi J M, Liu X, Fang P P, Lin L H, Tao A F. Development and universality evaluation of EST-SSR markers in jute (spp.) from GenBank database., 2014, 40: 1028–1034.
[19] Hong C, Piao Z, Kang T, Batley J, Yang T, Hur Y K, Bhak J, Park B S, Edwards D, Lim Y P. Genomic distribution of simple sequence repeats in., 2007, 23: 349–356.
[20] Goff S, Ricke D, Lan T, Presting G, Wang R, Dunn M, Glazebrook J, Sessions A, Oeller P, Varma H, Hadley D, Hutchison D, Martin C, Katagiri F, Lange M, Moughamer T, Xia Y, Budworth P, Zhong J, Miguel T, Paszkowski U, Zhang S, Colbert M, Sun W, Chen L, Cooper B, Park S, Charles T, Mao L, Quail P, Wing R, Dean R, Yu Y, Zharkikh A, Shen R, Sahasrabudhe S, Thomas A, Cannings R, Gutin A, Pruss D, Reid J, Tavtigian S, Mitchell J, Eldredge G, Scholl T, Miller R M, Bhatnagar S, Adey N, Rubano T, Tusneem N, Robinson R, Feldhaus R, Macalma T, Oliphant A, Briggs S. A draft sequence of the rice genome (L. ssp.)., 2002, 296: 92–100.
[21] Morgante M, Hanafey M, Powell W. Microsatellites are preferentially associated with nonrepetitive DNA in plant genomes., 2002, 30: 194–200
[22] Huo N, Lazo G, Vogel J, You F M, Ma Y, Hayden D M, Coleman-Derr D, Hill A, Dvorak J, Anderson O D, Luo M C, Gu Y Q. The nuclear genome of: analysis of BAC end sequences., 2008, 8: 135–147.
Evaluation and characteristic analysis of SSRs from the whole genome of jute ()
YAO Jia-Yu1,**, ZHANG Li-Wu1,2,**, ZHAO Jie1, XU Yi1,2, QI Jian-Min1, and ZHANG Lie-Mei1,*
1Key Laboratory for Genetics, Breeding and Multiple Utilization of Crops of Ministry of Education / Fujian Key Laboratory for Crop Breeding by Design / College of Crop Science, Fuzhou 350002, Fujian, China;2Center for Genomics and Biotechnology of Haixia Institute of Science and Technology, Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian, China
Jute is one of the most important natural bast fiber crops worldwide. However, the lack of SSR markers limits the genetic improvement of jute. In this study, simple sequence repeats (SSRs) were identified from the genome, genes, CDS and cDNA of CVL-1, a sequenced variety in. SSR loci was called using SSR Primer software and the characteristics of SSR loci were analyzed. The 153,242 genomic SSRs were called based on the genomic sequence with an average density of 467.20 SSRs Mb–1. Based on the cDNA sequence, we called 10,747 SSRs were developed with an average density of 260.85 SSRs Mb–1. The majority of repeat types were di- to tetra-nucleotides, accounting for 76.91%. Among them, the tri-nucleotide repeat types were the highest abundance repeat types in the cDNA_SSRs while the di-nucleotide repeat types were the highest abundance repeat types in the genomic SSRs. For different SSR repeat types, the genomic and cDNA-SSR frequency decreased dramatically as repeat times increased. Identification of SSR markers in the whole genome can not only enrich the number of molecular markers, but also lay a foundation for the analysis of genetic basis of important agronomic traits in jute.
jute (); genome; cDNA; SSRs
2018-05-18;
2018-08-20;
2018-09-14.
10.3724/SP.J.1006.2019.84072
通信作者(Corresponding author): 张列梅, E-mail: zhangliemei@126.com
**同等贡献(Contributed equally to this work)
姚嘉瑜, E-mail: 3533551417@qq.com; 张立武, E-mail: lwzhang@fafu.edu.cn, zhang_liwu@hotmail.com
本研究由国家自然科学基金项目(31771369), 国家现代农业产业技术体系建设专项(CARS-19-E06), 农业部东南黄红麻实验观测站(农科教发2011)和福建省麻类种质资源共享平台(2010N2002)资助。
This study was supported by the National Natural Science Foundation of China (31771369), the China Agriculture Research System (CARS-19-E06), the Experiment Station of Jute and Kenaf in Southeast China (Nongkejiaofa 2011), and the Construction of Germplasm Resources Platform for Bast Fiber Crops in Fujian, China (2010N2002).
URL:http://kns.cnki.net/kcms/detail/11.1809.s.20180912.1326.002.html

