p62基因敲除后食管鳞状细胞癌EC109细胞生物学行为变化
陈秀英,于莉,周君阳,丁妍,谭玉洁,4
1 贵州医科大学,贵阳550004;2 三峡大学;3湖北医药学院 胚胎干细胞研究湖北省重点实验室;4 贵州医科大学附属医院
食管癌是全球常见的恶性肿瘤之一,也是导致我国癌症患者死亡的第四大原因[1]。2018年,我国大约有28.3万人死于食管癌[2]。食管癌具有起病隐匿、进展迅速、治愈率差等特点。食管癌主要分为食管鳞状细胞癌和食管腺癌两种亚型,其中以食管鳞状细胞癌最常见[3]。因此,寻找抑制食管鳞状细胞癌生长的有效基因靶点并进行深入的功能研究,对于提高食管癌患者的生存率具有重要意义。p62是由SQSTM1基因编码的信号枢纽蛋白,具有多功能结构域,能与多种信号通路关键因子结合,在信号网络中发挥着重要作用,调控细胞的增殖、存活、死亡和炎症反应、肿瘤发生发展、氧化应激[4~6]。然而,在食管鳞状细胞癌中p62的功能研究较少。2018年1月~2019年9月,我们使用CRISPR/Cas9技术构建p62敲除的食管鳞状细胞癌EC109细胞,观察p62基因敲除后对EC109细胞增殖、迁移、集落形成能力及细胞周期等生物学行为的影响。
1 材料与方法
1.1 材料 食管癌EC109细胞由湖北医药学院实验室冻存,pGK1.2质粒购自美国Invitrogen公司;DMEM培养基购自美国Gibco公司,胎牛血清(FBS)购自杭州四季青公司,0.25%的胰酶购自上海碧云天生物技术有限公司;基因组提取试剂盒、凝胶纯化/回收试剂盒、质粒提取试剂盒购自天根生化科技(北京)有限公司,细胞周期试剂盒购自南京凯基生物有限公司,T4连接试剂盒、T7E1核酸内切酶试剂盒购自北京唯尚立德公司;卡那霉素购自生工生物工程(上海)股份有限公司,琼脂糖购自上海博亚公司;抗体购自北京博奥森公司,DH5α感受态细菌由本实验室保存,引物由生工生物工程(上海)股份有限公司设计。
1.2 实验方法
1.2.1 质粒的构建 采用CRISPR Design在线工具在靶标DNA区域中设计Oligo DNA序列,在引物合成靶序列头部添加额外的碱基,以便pGK1.2载体形成黏性末端;正向引物添加CACC,反向引物添加AAAC,分别设计两组Oligo DNA。Oligo1-SQSTM1上游引物5′-CACCGCACCGTGAAGGCCTACCTTC-3′、下游引物5′-AAACGAAGGTAGGCCTTCACGGTGC-3′,Oligo2-SQSTM1上游引物5′-CACCGACCGTGAAGGCCTACCTTCT-3′、下游引物5′-AAACAGAAGGTAGGCCTTCACGGTC-3′。将Oligo DNA退火形成引物二聚体,再与经BBSⅠ酶切的线性化pGL3-U6-sgRNA-PGK-puromycin载体连接,转化感受态细胞;挑菌落测序,测序正确的质粒进行后续实验。
1.2.2 细胞培养与质粒转染 将EC109细胞培养于DMEM高糖培养基(含10% FBS、1% 100 mg/L青-链霉素),置于37 ℃、5% CO2的培养箱中培养,在倒置显微镜下每天观察细胞生长状态;每2~3 d使用0.25%胰酶消化传代,传代比例为1∶3。敲除组转染前接种于25 cm2细胞培养瓶预先培养24 h,再用0.25%胰酶消化细胞;将上述质粒电转入细胞,置于37 ℃、5% CO2的培养箱中培养48 h,加入2 μg/mL嘌呤霉素筛选稳定转染细胞株;同时,设未转染质粒细胞作为对照组。筛选3 d,待对照组细胞死亡后停药。
1.2.3 单克隆细胞的挑选及打靶鉴定 待细胞长到对数期时,用胰酶消化;挑取单个细胞置于96孔板中培养15 d,再转移至6孔板中培养。待细胞融合率达80%左右,收集细胞,提取细胞基因组DNA。SQSTM1上游引物5′-AAGAGAGGGGTCAGCAATGA-3′,下游引物5′-TTATCTGAACCCACCACCTGG-3′。采用普通PCR扩增仪(BIO-RAD)扩增打靶位点上下游共约870 bp片段,扩增体系为20 μL。反应条件: 95 ℃ 5 min,95 ℃ 30 s,58 ℃ 30 s,72 ℃ 30 s进行34个循环,72 ℃ 10 min。采用T7E1核酸内切酶检测打靶效果:T7E1检测体系11 μL(DNA PCR产物5 μL,10×T7E1 buffer 1.1 μL,ddH2O 4.4μL),使用普通PCR仪95 ℃ 5 min加热变性,自然退火冷却至室温;在上述体系中加入0.5 μL T7E1酶,37 ℃反应30 min后跑1%琼脂糖胶进行检测。随机选取p62敲除后的单克隆细胞进行后续实验。
1.2.4 p62蛋白检测 采用Western blotting法。随机选取T7E1酶切检测为阳性的克隆进一步扩大培养,收集细胞。加入100 μL蛋白裂解液(含1∶100的蛋白酶抑制剂),BCA法定量测定蛋白浓度。配置12% SDS-PAGE电泳分离蛋白,将蛋白转移至硝酸纤维素膜上,用5%脱脂奶粉室温封闭2 h;加入一抗(兔抗人p62抗体、兔抗人GAPDH抗体),于4 ℃孵育过夜;TBST洗3×10 min,加入二抗(HRP标记的抗兔IgG抗体)室温孵育2 h;TBST洗3×10 min,采用ECL化学发光法显色。用Image J软件分析条带的灰度值,以目的蛋白与内参灰度比值表示p62蛋白的相对表达量。实验重复3次。
1.2.5 细胞形态观察 将适量的两组细胞种于6孔板中,使其次日细胞单层融合度达70%左右。在倒置显微镜下,观察细胞的形态变化。在100倍镜下,随机计数5个视野下的合胞体数量,取其平均值。
1.2.6 细胞增殖能力观察 采用实时无标记动态细胞分析技术(RTCA)。取两组生长状态良好的细胞,调节细胞密度为1×104/mL。以200 μL/孔接种至E-plate板内,每组8个重复。置于RTCA培养系统中,于37 ℃、5% CO2培养箱内培养80 h,绘制细胞增殖曲线。RTCA仪器通过微电子生物感应芯片测得的阻抗值经标准公式推算获得细胞指数(CI),细胞数量越多,CI 值越高。
1.2.7 细胞隆形成能力观察 采用单克隆实验。用0.25%胰蛋白酶消化两组细胞,以含10% FBS的高糖DMEM培养液分别将其配成细胞悬液。细胞计数后,以5×102/孔接种6孔板,每组设3个复孔;置于37 ℃、5% CO2恒温培养箱中培养,3~5 d换液1次。培养15 d后肉眼可见集落形成,终止培养;吸弃培养液,以1×PBS洗涤3次;用10%甲醇固定细胞,0.1结晶紫染色20 min;再以流水洗去染色液,空气干燥后拍照,用Image-Pro Plus6.0对细胞克隆数量进行计数。
1.2.8 细胞迁移能力观察 采用划痕实验。预先在6孔板底面绘制数条参考线,用于定位划痕位置。在6孔板中分别接种两组细胞(5×105/孔),每组设3个平行孔;培养24 h后,使用200 μL枪头进行划痕处理;PBS洗涤3次,将细胞在无血清培养基中培养。用装有数码相机(德国Zeiss公司)的倒置显微镜,观察并拍摄0、48、96 h划痕照片,使用Image Pro Plus 6.0软件读取划痕面积。划痕愈合率=(0 h划痕面积-24 h划痕面积)/0 h划痕面积×100%。实验重复3次,取平均值。
1.2.9 细胞周期分布检测 取两组对数生长期细胞,接种于T75培养瓶中;待细胞融合率达90%时,使用胰酶消化,离心收集细胞;PBS清洗1次,用含70%乙醇的PBS在4 ℃冰箱固定过夜。分析前用PBS离心洗涤细胞,加入10 mg/L的RNaseA 1 μL,37 ℃孵育30 min;PI(终浓度为50 μg/mL)浸染15 min后,300目滤网过滤细胞悬液,上流式细胞仪检测。实验重复3次。

2 结果
2.1 T7E1酶切验证结果 敲除组挑取的9、11、13、20号单克隆细胞在800 bp左右有敲除后的特异性条带,对照组仅在870 bp左右有一条带。12、16号单克隆细胞p62基因未敲除。

注:12、9、11、13、20、16为敲除组p62基因敲除后T7E1的酶切情况。
图1 T7E1酶切法验证p62敲除情况
2.2 两组细胞p62蛋白表达比较 敲除组p62蛋白相对表达量为0.245±0.102,低于对照组的1.157±0.043(P<0.01),表明食管癌EC109细胞p62敲除稳转细胞株模型构建成功。
2.3 两组细胞形态比较 敲除组细胞合胞体数量为(47.6±7.5)个/视野,多于对照组的(12.2±2.9)个/视野(P<0.01)。敲除组细胞形态与对照组相比,细胞较圆,且细胞边界不清。

注:箭头所指为细胞合胞体。
图2 两组细胞形态比较(×200)
2.4 两组细胞增殖能力比较 与对照组比较,敲除组10 h后各时点CI值均低于对照组(P<0.05)。见表1。
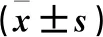
表1 两组不同时点细胞增殖CI值比较
注:与对照组比较,*P<0.05。
2.5 两组细胞克隆形成能力比较 敲除组细胞克隆数为(216±24)个,低于对照组的(363±22)个(P<0.05)。
2.6 两组细胞迁移能力观察 敲除组48 h、96 h划痕愈合率分别为33.11%±2.88%、54.59%±0.66%,分别低于对照组同时点的35.94%±3.53%、63.25%±0.14%,但96 h划痕愈合率差异有统计学意义(P均<0.05)。
2.7 两组细胞周期分布情况比较 敲除组阻滞于G0/G1期的细胞比例为62.19%±1.27%,高于对照组细胞的47.63%±3.53%(P<0.05)。
3 讨论
据统计,2012年全球食管癌新增病例大约为455 800例[7],食管鳞状细胞癌是其最常见的类型[3]。临床实践中,食管癌患者5年生存率仅有10%~25%[8]。因此,探索食管鳞状细胞癌发生发展的分子机制对食管癌的诊断、治疗具有重要意义。
p62是一种多功能结构域的枢纽蛋白[5],包含了如PB1结构域、锌指结构域、TB结构域、UBA结构域等一系列的结构域。这些结构域通过调控转录因子的活性,调控细胞的增殖、分化、转移、死亡[9]。近年来,有研究表明,在多种肿瘤组织中如胃癌[10]、肝癌[11]、肾癌[12]中p62异常高表达;在异种移植模型中发现,p62敲除后能抑制肿瘤细胞侵袭和生长[13],进一步说明p62在肿瘤的发生发展中具有重要作用。研究发现[14],在食管鳞状细胞癌中,p62表达异常增高,并且p62高表达与分化程度相关,说明p62在食管鳞状细胞癌的发生发展中发挥重要作用。然而,p62对食管鳞状细胞癌的影响如何,仍不明确。为此,我们进行了相应的研究与探讨。
在本研究中,我们使用食管鳞状细胞癌EC109细胞,进行p62基因敲除处理,建立EC109 p62基因敲除稳转细胞株,观察p62基因敲除后EC109细胞形态、增殖、迁移能力及细胞周期的改变。培养过程中发现,p62敲除后细胞形态发生了改变,敲除组细胞可见较多的类似于一层细胞膜包绕多个核的一团细胞质,即合胞体;敲除组细胞形态与对照组相比,细胞较圆,且细胞边界不清。在细胞增殖实验中,p62敲除后EC109细胞CI值低于对照组,说明p62敲除后抑制了EC109细胞的增殖能力;在单克隆形成实验中,p62敲除后EC109细胞克隆形成数量显著减少,且克隆相对细小,说明p62敲除后细胞的生存活力受到抑制;在划痕实验中,敲除组细胞96 h划痕愈合率低于对照组,说明p62敲除后抑制了EC109细胞的迁移能力;在细胞周期检测中,敲除组中G0/G1期细胞比例高于对照组,说明p62敲除后将细胞阻滞于G0/G1期。由此可见,p62基因敲除后抑制了食管癌EC109细胞的恶性生物学行为。这与近年来的相关文献报道一致[13~15]。同时也有研究报道,p62可以调控细胞的生存、增殖、分化、转移以及死亡[9,16,17]。当我们在EC109细胞中敲除p62后,细胞形成较多的合胞体,且细胞的恶性行为显著受到抑制。合胞体主要是由于细胞融合或一系列不完全细胞分裂周期所致,细胞的形态发生改变,而形态又与功能密切相关。在我们的研究中,p62敲除后,细胞的形态发生了改变,细胞周期阻滞。于是,我们推测p62敲除后,在食管癌EC109细胞周期中可以促进细胞不完全分裂,使细胞周期阻滞,导致合胞体出现,从而改变食管鳞状细胞癌的细胞形态及生物学功能。但是,p62是如何调节细胞周期、改变细胞形态,其具体机制如何,尚有待进一步研究探索。
综上所述,在我们的研究中显示,p62在食管癌EC109细胞的增殖、转移、周期分布方面具有重要调控作用,可将其作为食管癌预测基因进行更加深入的研究,以期为食管癌的早期诊断、治疗及预后提供科学合理的依据。

