小麦条锈菌产孢转录组分析
张贺平
(中化化肥有限公司河北分公司,河北石家庄050031)
由条形柄锈菌小麦专化型(Puccinia striiformis f.sp.tritici,Pst)真菌引起的小麦条锈病是世界性禾谷类作物的重要病害之一,全球每年造成小麦产量直接经济损失近10亿美元[1],在我国曾发生多次大流行,严重危害小麦生产安全[2]。经过长期不懈的努力,我国在小麦条锈病治理中取得了重大进展[3],但由于Pst极强的变异性新型致病型不断涌现,防治小麦条锈病的任务依然艰巨。
虽然不同病原菌侵染寄主所用策略不同,但最终目的都是为了获取源自寄主的营养。Pst作为活体营养专性寄生菌,既要利用多种效应因子逃避或抑制小麦的免疫反应,又要同小麦竞争吸收糖类等营养物质以供应菌体生长繁殖[4]。小麦条锈菌毒性变异丰富,目前对不同毒力小麦条锈菌在侵染小麦寄主过程中差异表达基因尚未进行深入分析。
本研究选择不同毒力小麦条锈菌(CY31、CY32、CY33和V26菌系的单孢子堆分离物),旨在通过RNA-seq技术测定Pst产孢时转录组并分析其转录组序列,并对差异表达基因进行探讨。
1 材料和方法
1.1 材料
供试小麦条锈菌为CY31、CY32、CY33和V26菌系的单孢子堆分离物;供试小麦品种为铭贤169。
1.2 方法
1.2.1 RNA提取、文库构建及测序采用Trizol法(Invitrogen,USA)分别提取小麦条锈菌各分离物的总RNA,提取过程中所用器具和耗材均经过DEPC处理。DNaseⅠ消化样品中的DNA,然后用Nanodrop检测RNA的纯度和浓度,采用Agilent 2100分析仪(安捷伦公司)检测RNA完整值(RIN),以确定RNA质量。
RNA检测合格后,用带有Oligo(dT)的低吸附磁珠通过A-T互补配对与mRNA的ployA尾结合的方式富集总RNA中的mRNA,随后加入打断试剂,按照Truseq RNA Sample Prep Kit v2说明书构建双末端cDNA文库。由诺禾致源公司采用Illumina HiSeqTM2000进行转录组测序。
1.2.2 测序数据质量控制首先检查测序原始读长每个碱基的测序错误率,采用测序Phred数值(Q值)估算碱基判别的错误率(通常Q10、Q20和Q30对应的碱基测序错误率分别为10.0%、1.0%和0.1%);其次,为保证分析质量,必须对测序得到的原始读长进行过滤:去除带接头的读长、片段内无法确定碱基信息的比例>10%的读长、Q值<20的碱基数占整个读长50%以上的低质量读长,得到过滤后读长,后续分析均基于过滤后读长进行。
1.2.3 与参考基因组序列的比对采用Tophat 2.0软件将过滤后读长比对到小麦条锈菌基因组(PST-78,GenBank:AJIL00000000.1)上,比对过程中允许≤2个碱基对的错配。统计可定位到参考基因组上的读长数量,其中包括在参考基因组上有多个比对位置的读长数量以及在参考基因组上有唯一比对位置的读长数量。如果参考基因组选择合适,而且相关实验不存在污染,正常情况下,定位到参考基因组上的读长会高于70%,而有多个比对位置的读长占总体的比例低于10%。根据过滤后读长在参考基因组上的分布情况,总体评估获得的测序数据在小麦条锈菌基因组上的覆盖情况。比对到某基因的读长若至少有1条与参考基因匹配,则将该参考基因定义为一个表达基因。
1.2.4 基因表达谱分析和差异基因注释基因表达水平与其转录本的丰度相关,通常转录本丰度越高,则基因表达水平越高。读长数量除了与基因的真实表达水平相关外,还受测序深度和基因长度的影响。为减小这种影响,转录组基因表达量以FPKM(Fragments Per Kilobase per Millions base pairs sequenced)值来衡量。FPKM同时考虑了测序深度和基因长度对读长数量的影响,是目前常用的基因表达水平估算方法。
由于本试验没有设置生物学重复,因此,先采用DEGseq软件的TMM程序对读长计数的数据进行标准化处理,然后用cufflinks软件统计小麦条锈菌两两样品间基因的表达差异状况,再通过多重假设检验(False discovery ratio,FDR)对计算出的每一差异表达基因的P值进行校正。为消除生物学变异,阈值设定为样品间FPKM比值2倍以上且FDR<0.005,同时满足差异倍数和显著水平的基因作为差异表达基因。将差异表达基因和NCBI nr数据库进行比对,用nr注释库中同源基因的注释信息代表差异表达基因的注释信息。采用KOBAS软件将差异表达基因与KEGG(Kyoto encyclopedia of genes and genomes)数据库进行比对,获得差异表达基因相对应的路径注释信息。
2 结果与分析
2.1 RNA和测序数据质量评估
用Trizal法提取小麦条锈菌CY31、CY32、CY33和V26菌系的RNA后,质量检测显示,OD260/OD280值在1.902~1.942,RIN值在9.5~9.7,25S/18S比值均为1.6,浓度范围在468~540 ng/μL。表明所用样品的RNA纯度和完整性较好,可满足转录组测序要求。
分析原始读长碱基的测序错误率和Q值分布,可以检测测序数据的质量。测序错误率受多因素影响,转录组的测序错误率具有2个特点:一是随着测序读长长度增加测序错误率升高;二是读长端的前6个碱基位置测序错误率较高。前者是由于测序过程中化学试剂消耗而导致的,后者是因为随机引物和RNA模板不完全结合,而这6个碱基位置恰好是文库构建时反转录中随机引物的位置。一般样品的读长中每个碱基测序错误率应该低于0.5%。CY31、CY32、CY33和V26菌系测序过程碱基错误率均低于0.02%(表1)。除了碱基的测序错误率外,碱基的质量也是RNA-Seq测序质量评估的一个重要指标,一般Q30在80%以上的测序质量非常可靠。本研究4个样品的Q20均在95.63%以上,Q30在89.79%以上(表1)。表明转录组测序得到的质量结果可靠,可用于后续分析。

表1 小麦条锈菌转录组测序数据评估统计
小麦条锈菌CY31、CY32、CY33和V26菌系转录组共产生396 711 824条原始读长,经过滤后获得382 125 418条过滤后读长,每个样品均获得了13.23 Gb以上的碱基(表1)。将过滤后读长与小麦条锈菌PST-78菌系参考基因组分别比对,4个样品序列的71.15%以上读长可以比对到参考基因组(PST-78,GenBank:AJIL00000000.1)(表2)。其中,CY31、CY32、CY33和V26菌系唯一比对到参考基因组的读长占总过滤后读长的比例依次为73.03%、70.14%、73.13%和70.16%,多处比对到参考基因组的读长占总过滤后读长的百分比在0.99%~1.16%(表2),远低于10%。此外,定位到参考基因组外显子区及内含子区的读长数量占定位到参考基因组上的读长的比例显示,定位到外显子区的读长的比例最高,CY31、CY32、CY33和V26菌系对应的比例分别是60.8%、73.9%、57.5%和76.4%,定位到内含子区域的读长的比例最低,4个样品对应的比例均低于0.3%,从基因覆盖度的角度也证实,转录组数据已覆盖参考基因组上大部分基因。本研究所得转录组测序数据结果能反映小麦条锈菌菌系的整个转录组,这些测序数据的质量、数量评估为后续的基因表达差异分析奠定了基础。

表2 小麦条锈菌转录组数据与参考基因组比对统计 条
2.2 基因表达谱及差异表达基因分析
转录组的读长数量可以定性地评估基因的表达情况,通过转换计算出FPKM值则可以对多个基因表达水平进行比较。统计CY31、CY32、CY33和V26菌系不同表达水平的基因数量,结果显示,转录组FPKM值小于10的基因数分别占对应文库的74.73%、75.86%、74.94%和75.07%(表3),10<FPKM值<100时,基因数分别占21.57%、20.63%、21.32%和21.37%,其中,FPKM值大于60的基因数所占比例均低于7.0%,而FPKM值大于10 000的基因在CY31、CY32、CY33和V26菌系样品文库中分别有7、8、7、7个。为了检测不同毒力小麦条锈菌基因的表达差异状况,将|log2Fold change|≥1且FDR<0.005作为显著差异表达基因的2个维度,从差异倍数和显著水平2个水平评估小麦条锈菌样品间差异表达基因,小麦条锈菌CY31、CY32、CY33和V26菌系间差异表达基因数目如表4所示,小麦条锈菌V26 vs.CY31、V26 vs.CY32、V26 vs.CY33间差异表达基因数量依次为35、4、52个,CY32 vs.CY31、CY32 vs.CY33、CY33 vs.CY31间差异表达基因数量最大不超过10个。各基因表达的差异倍数比较均一,差异水平变化主要集中在6倍以内,差异倍数最大的一个基因为13倍(V26 vs.CY33中的差异基因)。分析发现,小麦条锈菌CY31、CY32、CY33、V26菌系产孢时不但差异表达基因较少,而且在不同菌系间往往出现相同的差异表达基因。例如,V26 vs.CY31发现35个差异表达基因,其中,上调基因25个、下调基因10个,上调基因中16个与V26 vs.CY33的上调基因相同,下调基因中8个与V26 vs.CY33的下调基因相同;V26 vs.CY32发现有4个差异表达基因(均为上调表达)与V26 vs.CY33间的上调基因相同,其中的一个差异表达基因(PSTG_14209)在V26 vs.CY31、V26 vs.CY32、V26 vs.CY33的上调差异基因中均被发现。CY31 vs.CY33发现的3个差异表达基因(2个上调、1个下调)与CY32 vs.CY31的相同。而CY32 vs.CY33的差异表达基因有所不同,发现的3个差异表达基因与CY32 vs.CY31、CY33 vs.CY31的差异表达基因均不同。
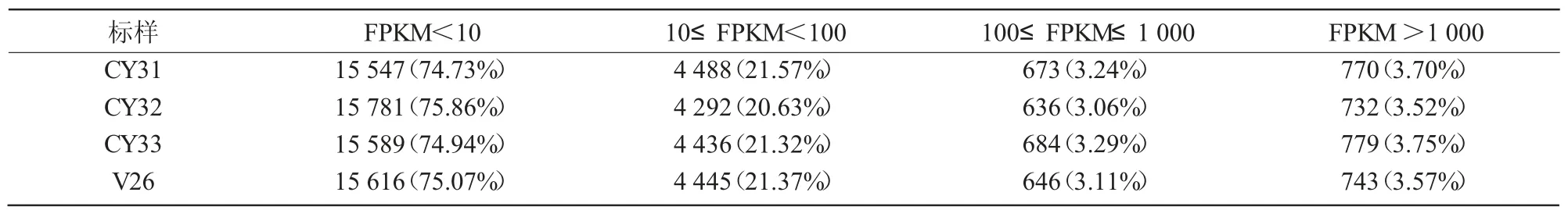
表3 小麦条锈菌不同表达水平区间的基因数量 个

表4 小麦条锈菌菌系间产孢时差异表达基因数量 个
将已注释的差异表达基因与KEGG数据库比对,结果表明,共有53个基因被注释到了KEGG中的36个通路中,其中,显著富集通路(P<0.05)有5个(表5)。显著富集通路主要参与到氧化磷酸化(Oxidative phosphorylation)、RNA降解(RNA degradation)、真核生物核糖体合成(Ribosome biogenesis in eukaryotes)、淀粉和糖代谢(Starch and sucrose metabolism)、内质网蛋白加工(Protein processing in endoplasmic reticulum)等通路中。可见,小麦条锈菌CY31、CY32、CY33和V26菌系间产孢时在糖类的代谢、磷酸化等方面变化明显,说明小麦条锈菌产孢受糖代谢系统的调节。

表5 小麦条锈菌差异基因注释
3 结论与讨论
本研究结果表明,4个小麦条锈菌分离物间产孢时差异表达基因数量较少,而且在不同菌系间往往出现相同的差异表达基因。例如,V26 vs.CY31发现35个差异表达基因(上调表达25个、下调表达10个),其上调表达基因中有16个与V26 vs.CY33的上调基因相同,下调基因中有8个与V26 vs.CY33的下调基因相同。
功能注释显示,Pst分离物间差异表达基因主要集中在糖和淀粉代谢及氧化磷酸化路径中。本研究发现,编码糖原脱支酶和磷酸葡萄糖变位酶的直 系同源基因PSTG_05529、PSTG_09692在4个Pst分离物与小麦亲和互作中表达差异显著。糖原脱支酶催化糖原分解并释放1分子葡萄糖,而磷酸葡萄糖变位酶是糖原分解及其逆反应糖原合成中共存的催化酶,可以催化葡萄糖-1-磷酸和葡萄糖-6-磷酸相互转化,2种酶在糖代谢中具有重要的作用。
比较基因组发现,病原菌基因组编码产物的类型和数量与其寄生方式紧密相关,例如,活体寄生菌中编码细胞壁降解酶的基因比其他营腐生类型的少,小麦条锈菌基因组的特征更加显著,其基因组中缺乏编码同化N、S必需酶的基因,而编码糖类和氨基酸转运蛋白的基因数量极大增加[5-7],基因组结构表明,小麦条锈菌生长依赖吸收源自寄主的糖类等营养。糖类既能提供能量,又能作为信号分子影响糖运输方向、寄主抗病性等广泛的细胞反应。
多项研究结果表明,成功侵染的细菌、真菌和卵菌等病原菌调节寄主植物的SWEET基因大量表达,增加外流到受侵染寄主质外体中的糖含量以供病原菌吸收。病原菌操纵寄主的糖转运蛋白,只是稳定获取源自植物糖类的第1步,随后需要将糖类从植物质外体吸收进入菌体的细胞质,这一过程可能需要病原菌自身编码的蔗糖酶类分解酶和糖转运蛋白的参与。蔗糖酶主要功能是将蔗糖水解成葡萄糖和果糖,例如,青杨叶锈菌(Melampsora larici-populina)和小麦秆锈菌(Puccinia graminis f.sp.tritici)侵染各自的寄主过程中蔗糖酶基因均上调表达[7]。最近发现的Pst基因组中编码蔗糖酶的PsINV基因可以高效分解蔗糖,并且在Pst与小麦的亲和与非亲和反应中均大量表达[8]。侵染过程中蔗糖酶基因表达量增加,可能将质外体中的蔗糖分解成容易吸收的葡萄糖和果糖等己糖,同时表明,这些病原菌编码的糖转运蛋白对己糖底物亲和力比较高。此外,多项研究表明,病原菌自身编码的糖转运蛋白在与寄主糖转运蛋白争夺糖的过程中发挥着重要作用。例如,蚕豆锈菌(Ustilago fabae)编码的HXT1转运蛋白专化性吸收葡萄糖和果糖[9],灰霉菌(Bortrytis cinerea)编码的Frt1转运蛋白对果糖高亲和。在玉米黑粉菌(Ustilago maydis)中发现了19个糖转运蛋白,其中,己糖转运蛋白Hxt1底物较广泛,与玉米竞争糖类时对葡萄糖具有高亲和力,也可以转运果糖和甘露糖[10-11]。蔗糖转运蛋白UmSrt1具有底物专一性,其对蔗糖亲和力比玉米的蔗糖转运蛋白ZmSUT1高200倍,敲除UmSrt1极大降低了病原菌的毒性,同时敲除UmSrt1和Hxt1的菌株致病性和寄生适合度均强烈降低,表明Ustilago maydis侵染过程需要同时吸收果糖和葡萄糖,Ustilago maydis既能吸收蔗糖也能吸收己糖,对在多种生境下与玉米争夺糖类具有极大优势[10,12-13]。寄主植物在和病原菌竞争糖类的过程中进化出相反的糖转运机制,寄主植物在免疫系统激发下调节STP类己糖转运蛋白将糖从质外体回收,降低质外体中糖含量限制病原菌获取糖类。例如,拟南芥的己糖转运蛋白基因STP13受Pseudomonas syringae、Bortrytis cinerea或白粉菌侵染激活表达,调节受侵染拟南芥质外体中己糖转运到细胞质,从而限制侵入到拟南芥质外体中的病原菌获取糖类营养[14-15]。葡萄受到活体专性寄生菌白粉菌和霜霉菌侵染时,己糖转运蛋白STP13类直系同源基因VvHT5上调表达,致使质外体中糖含量降低,限制了病原菌扩散[16]。这些结果说明植物的己糖转运蛋白在植物防御中具有重要作用。
本研究在Pst基因组中发现预测编码葡萄糖转运蛋白的基因,但未发现预测编码果糖转运蛋白的基因。一方面显示糖转运蛋白对Pst获取糖类具有重要影响,另一方面表明Pst吸收糖类机制与已知的其他活体寄生菌有显著不同[17]。
转录数据提供的仅是相关线索,如果要明确差异表达基因的确切功能,需要进行遗传、分子以及生理功能等的综合深入分析。

