米象表皮蛋白的全基因组注释及其系统发育分析
(陈二虎 侯秋莉
(南京财经大学食品科学与工程学院;江苏省现代粮食流通与安全协同创新中心;江苏高校粮油质量安全控制及深加工重点实验室1,南京 210023)
(扬州大学园艺与植物保护学院2,扬州 225009)
表皮蛋白(CPs)是昆虫表皮的重要结构物质,与几丁质相互交联形成复杂稳定的结构,以维持表皮弹性和其他物理性质[1]。大量研究已经证实CPs在昆虫生长发育、表皮骨化、爬行能力、体型塑造、先天免疫、药剂抗性等方面均发挥着重要的生理功能[2-6]。此外,CPs只存在于昆虫等无脊椎动物中,且不同昆虫间序列差异大,因此是开发高靶向性杀虫剂的理想靶标。米象(Sitophilusoryzae)是当前世界范围内危害最为严重的储粮害虫之一,其日益突出的磷化氢抗性问题严重威胁粮食安全[7]。针对上述现状,挖掘新的防效好、特异性强、安全性高的杀虫靶标则显得尤为亟需。
伴随高通量测序技术和生物信息学的快速发展,迄今为止已从不同昆虫的基因组或转录组数据库中鉴定获得大量CPs,包括赤拟谷盗(Triboliumcastaneum)(151个)[8]、马铃薯甲虫(Leptinotarsadecemlineata)(175个)[9]、家蚕(Bombyxmori)(226个)[10]、黑腹果蝇(Drosophilamelanogaster)(174个)[11]、东亚飞蝗(Locustamigratoria)(81个)[12]、等。依据氨基酸序列的保守结构域,昆虫CPs被划分为10余个家族类群,包括CPR[13,14]、CPAP1(Cuticular Proteins Analogous to Peritrophin 1)[5]、CPAP3(Cuticular Proteins Analogous to Peritrophin 3)、CPLCA、CPLCG、CPLCP、CPLCW[15]、Tweedle、CPF[16,17]、CPFL、CPCFC、CPG[18,19]等。
鉴于昆虫CPs数量众多(约占昆虫基因数量的1%),而且不同昆虫类群间表皮蛋白基因的数量和种类差异显著,因此昆虫CPs的鉴定、序列分析和家族分类是开展后续功能研究的重要前提。目前,虽然米象全基因组数据已公布,但是目前有关米象表皮蛋白的研究仍相对滞后,米象CPs的基因信息尚属未知。因此,本研究拟基于米象基因组数据库,开展CPs的鉴定、序列特征及其系统发育分析,研究结果可为后续的功能研究奠定基础,进而为米象的防治提供新的思路和方法,具有重要的理论和应用参考价值。
1 材料与方法
1.1 昆虫基因信息数据来源
米象基因组数据源于National Center for Biotechnology Information(NCBI)公共数据库(NCBI Assembly: GCF_002938485.1)。其他昆虫CP基因信息均从NCBI的GnenBank数据库(http://www.ncbi.nlm.nih.gov/)或者VectorBase数据库(https://www.vectorbase.org/)下载。
1.2 米象表皮蛋白基因的全基因组鉴定
基于米象基因组数据信息,通过同源序列比对方法鉴定获得米象CPs序列(E-value < 1×10-5)。CPR家族以扩展保守基序R&R(pfam00379)作为参考序列进行BLASTP比对[20, 21],运用CuticleDB网站http://bioInformatics2.biol.uoa.gr/CuticleDB/index.jsp将CPR家族基因划分为RR-1和RR-2亚家族[11];关于CPAP家族,基于GenBank数据库下载的赤拟谷盗和马铃薯甲虫CPAP1和CPAP3序列,通过与米象基因组比对鉴定得到相应CP家族基因[22];基于昆虫保守的Tweedle基序(PF03103)识别米象同源的Tweedle家族[3];通过与黑腹果蝇同源序列比对获得米象CPLCG和CPLCP家族序列[15];以CPF家族蛋白特征序列中最保守部分(VSxYSKAVDTPFSSVRKxDxRIVNxA)鉴定获得米象CPF家族基因[17];利用赤拟谷盗和马铃薯甲虫CPs序列,经同源比对获得米象CPFL家族基因序列[9];依据保守YPAGVNPAACPNYPYCD基序识别米象CPCFC家族[19]。
1.3 信号肽及保守结构域分析
使用SignalP 4.0(http://www.cbs.dtu.dk/services/SignalP-4.0/)[23]预测米象CPs信号肽。分别提取CPs家族保守结构域序列,运用ClustalW软件进行多重序列比对,并结合在线工具WebLogo(http://weblogo.berkeley.edu/logo.cgi)绘制保守结构域LOGO图,分析CPs各家族序列特征及保守氨基酸的出现频率。
1.4 系统发育分析
分别获取昆虫(米象、赤拟谷盗、马铃薯甲虫、黑腹果蝇、家蚕等)CPR家族保守R&R基序,CPAP1和CPAP3家族保守ChtBD2结构域,以及Tweedle、CPLCG、CPLCP、CPCFC、CPF和CPFL家族不包含信号肽的全长氨基酸序列。运用ClustalW对上述对应氨基酸序列进行多重比对。利用Mega 6.0软件中的邻接法(Neighbor-Joining)分别开展昆虫不同家族CPs氨基酸序列的系统发育树构建,其中各树分支都进行1 000次重复抽样检验。
2 结果与分析
2.1 米象CP基因的全基因组鉴定
基于米象基因组共鉴定获得135个CP基因,隶属9个家族:CPR(RR1,RR2和CPRNC)、Tweedle、CPAP1、CPAP3、CPLCG、CPLCP、CPF、CPFL和CPCFC,其中有85.93%(116个)的CPs包含信号肽。通过与鞘翅目昆虫赤拟谷盗和马铃薯甲虫的CPs比较分析发现,不同昆虫间CPs的数量存在较大差异,马铃薯甲虫(175个)CPs个数最多,赤拟谷盗(151个)次之,米象(135个)最少,其中不同昆虫间尤以CPR家族基因的数量差异最为明显(表1)。此外,本研究发现马铃薯甲虫的CPs缺少CPLC家族,米象则无CPLCA家族基因(表1)。
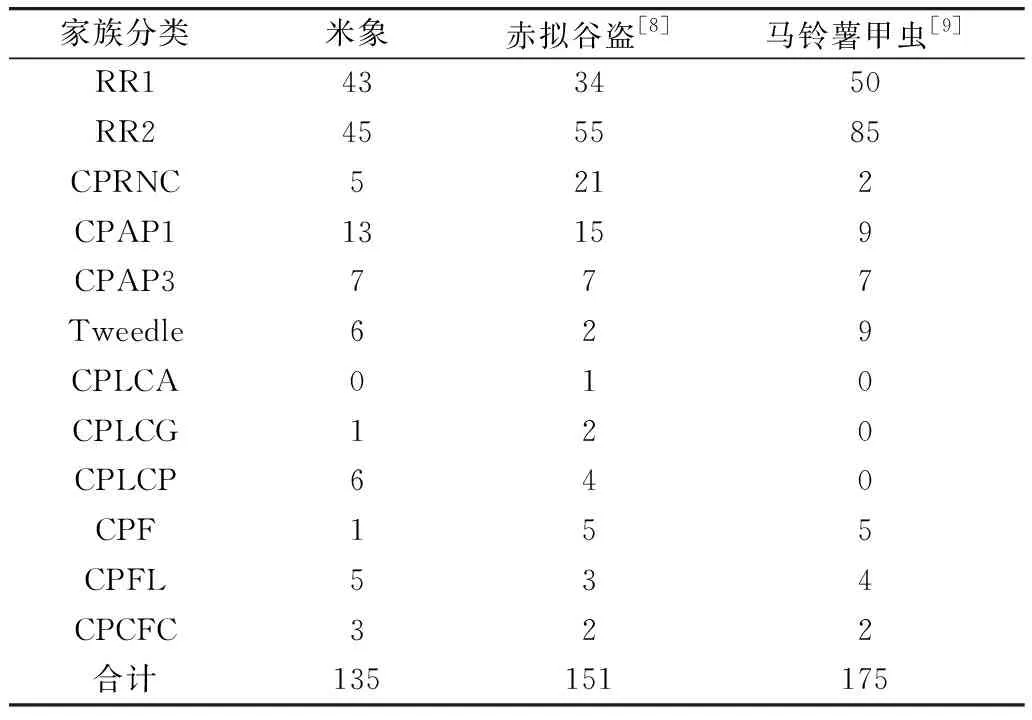
表1 三种鞘翅目昆虫表皮蛋白基因汇总表
2.2 CPR家族基因
本研究共鉴定获得含保守R&R基序的CPR家族基因93个,占米象CP基因总数的68.89%。依据CuticleDB运算,米象CPR家族分别包含43个RR1亚家族和45个RR2亚家族成员,另有5条基因序列被定义为CPRNC(未分类CPR)(表1)。此外,系统发育分析结果显示RR1、RR2和CPRNC各自聚为一支,证实上述CPR亚家族分类的准确性(图1a)。有趣的是,CuticleDB数据库分类结果显示XP_030753085.1蛋白隶属于RR1亚家族,而进化树分析结果则与CPRNC蛋白聚为一支,暗示传统CPR亚家族分类方法与系统发育关系存在不一致性(图1a)。米象RR1和RR2家族蛋白的R&R保守基序分别如图1b和图1c所示,其中RR1家族蛋白拥有8个保守氨基酸识别位点(YTADENGF),RR2家族蛋白则含有2个保守识别序列(EERDGDVVKG;3个G-x(3)-VV重复序列)。为进一步探究昆虫RR1和RR2蛋白的进化关系,本研究以米象、赤拟谷盗、黑腹果蝇和家蚕的R&R基序进行系统发育分析,分别构建不同昆虫RR1和RR2家族蛋白系统发育树(图2和图3),结果表明上述不同昆虫均形成相应物种特异性基因簇,米象RR1蛋白包含4个基因聚类(图2),RR2蛋白则拥有8个独立基因簇(图3)。
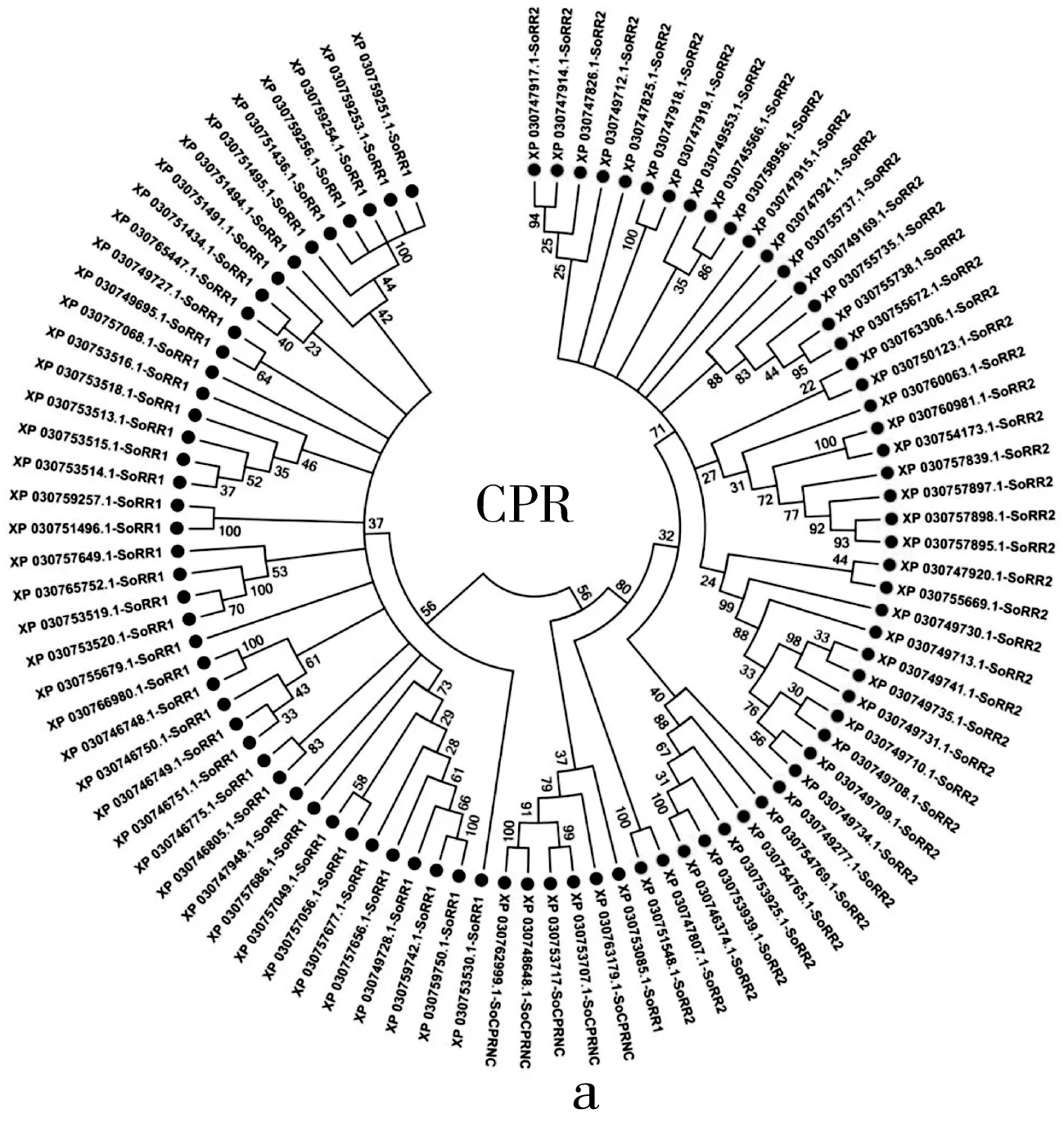
注:a 米象CPR家族蛋白系统发育分析(邻接法);b 米象RR1蛋白R&R基序分析;c 米象RR2蛋白R&R基序分析。
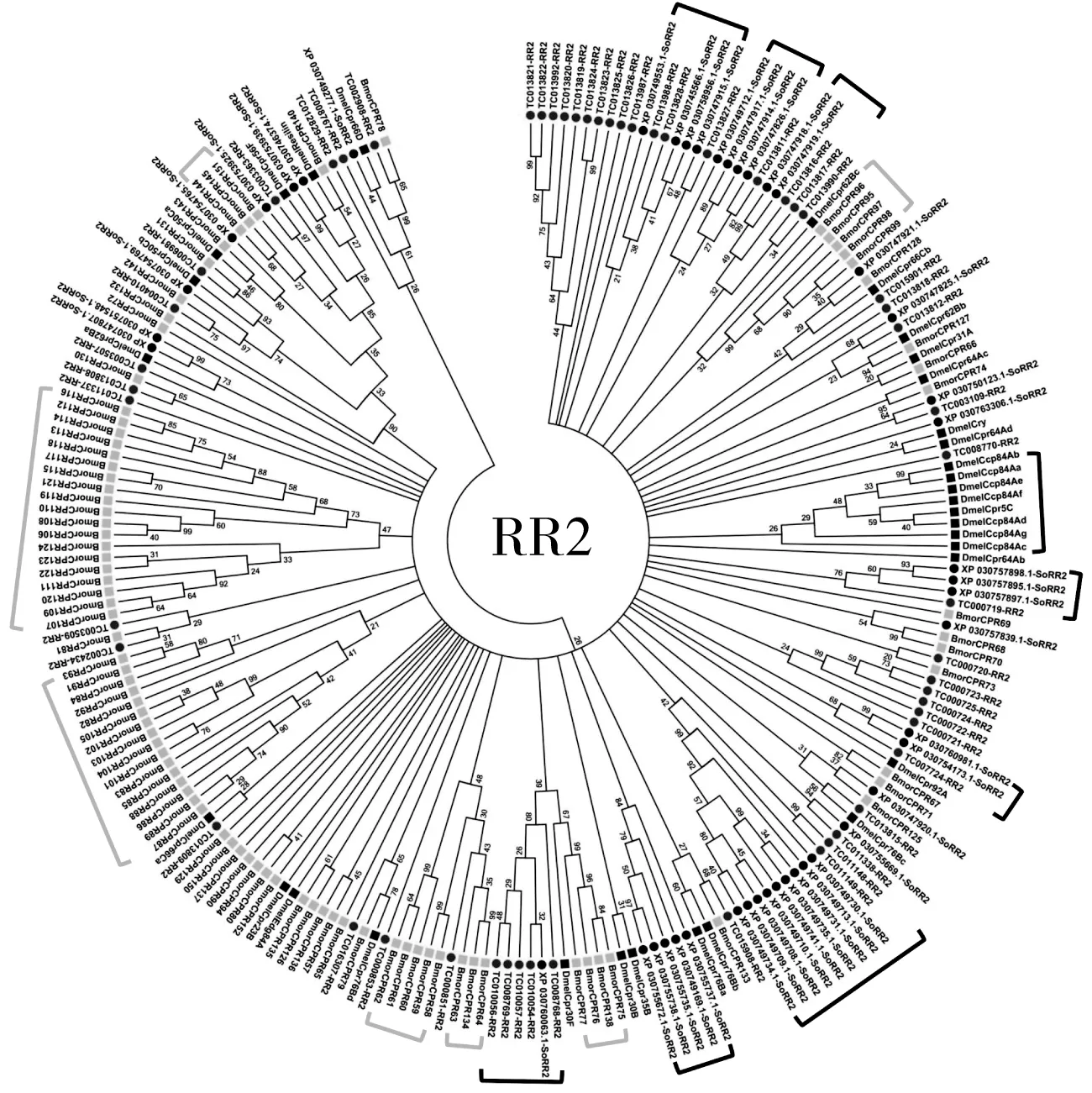
注:米象(So),赤拟谷盗(Tc),黑腹果蝇(Dm),家蚕(Bm);黑括号表示基因聚为一簇。
2.3 CPAP1和CPAP3家族基因
在米象基因组中共鉴定获得13个CPAP1和7个CPAP3家族蛋白基因(表1)。系统发育分析结果表明,不同昆虫CPAP1和CPAP3家族蛋白各自形成两个不同分支的基因簇,且米象与赤拟谷盗亲缘关系较高,两个物种的CPAP基因在进化树中均成对出现(图4a)。蛋白序列结构分析结果显示,米象CPAP1和CPAP3家族蛋白序列分别拥有1个和3个保守ChtBD2型几丁质结合域,且多数蛋白均含有1个N末端信号肽(除SoCPAP1-B、F、J和M)(图4b和图4c)。此外,米象CPAP1序列长度差异较大,氨基酸长度介于170和169 3 aa之间(图4b);CPAP3氨基酸序列长度的变异则较小,其范围为230~281 aa(图4c)。与此同时,米象CPAP1和CPAP3蛋白家族间的几丁质结合域数量和位置存在显著区别,即SoCPAP3蛋白的3个几丁质结合域主要靠近N端,而SoCPAP1蛋白几丁质结合域在N端、C端和中间位置均有分布(图4b和图4c)。

注:a 昆虫CPAP1和CPAP3家族蛋白系统发育分析。米象(So),赤拟谷盗(Tc),黑腹果蝇(Dm),地中海实蝇Ceratitis capitata(Cc),家蚕(Bm),烟草天蛾Manduca sexta(Ms);b CPAP1和c CPAP3蛋白结构分析。
2.4 Tweedle家族基因
在米象基因组中共鉴定得到6个Tweedle蛋白基因,根据米象、赤拟谷盗等11种不同昆虫的氨基酸序列构建Tweedle家族蛋白的系统进化树。结果显示,昆虫Tweedle家族蛋白可形成三个并列基因簇,分别包括蝇虫类(地中海实蝇和黑腹果蝇)、蚊子类(尖音库蚊、埃及伊蚊和冈比亚按蚊)的特异性Tweedle基因聚类分支,以及由11种昆虫共同组成的Tweedle基因混合簇(图5a)。此外,氨基酸序列分析结果显示,米象Tweedle家族蛋白拥有4个保守氨基酸区域,且每个Tweedle家族成员中均存在一个内部重复结构(图5b)。
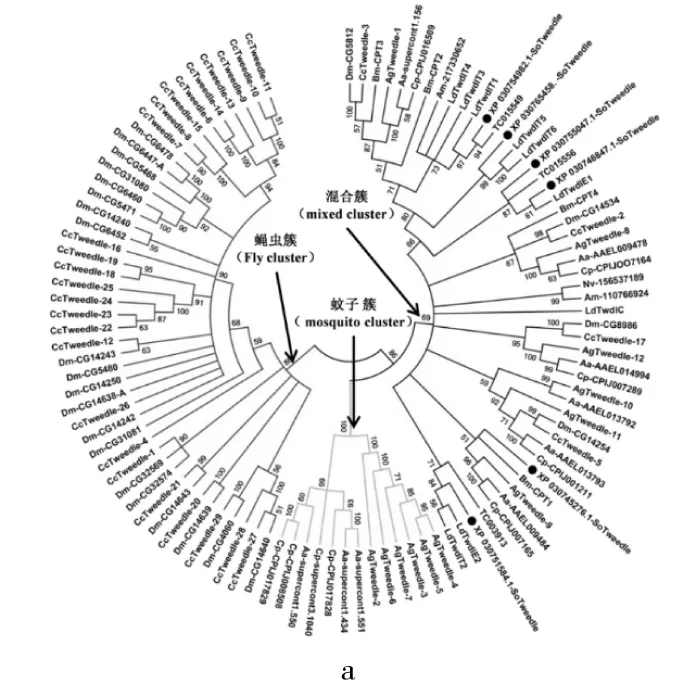
注:a 不同昆虫Tweedle家族蛋白进化关系分析,包括米象(So)、赤拟谷盗(Tc)、马铃薯甲虫Leptinotarsa decemlineata(Ld)、黑腹果蝇(Dm)、地中海实蝇Ceratitis capitata(Cc)、冈比亚按蚊Anopheles gambiae(Ag)、埃及伊蚊Aedes aegypti(Aa)、尖音库蚊Culex pipiens(Cp)、家蚕(Bm)、丽蝇蛹集金小蜂Nasonia vitripennis(Nv)和意大利蜜蜂Apis mellifera(Am);b 米象Tweedle蛋白保守序列分析。
2.5 低复杂度CP家族基因
在米象基因组中共鉴定得到米象低复杂度CP家族基因7个,包括1个CPLCG基因和6个CPLCP基因,未鉴定到CPLCA基因(表1)。不同昆虫CPLCG和CPLCP蛋白家族分别聚类形成单独分支(图6a),并且聚集形成4个蚊类基因簇(图6a),表明该家族蛋白数量在蚊子类群中得到显著扩展。氨基酸保守序列分析显示,CPLCP蛋白含有高密度脯氨酸PV和PY重复序列(图6b)。
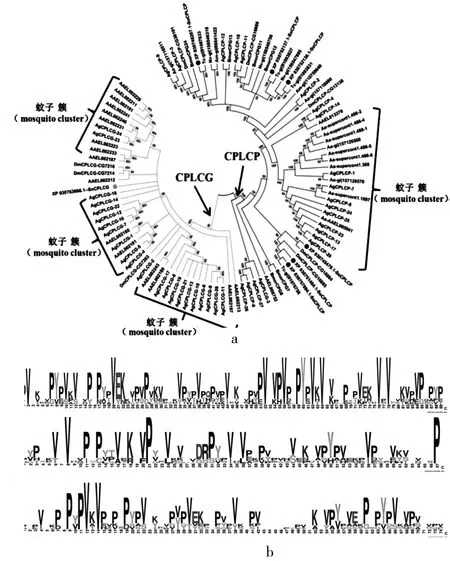
注:a 不同昆虫CPLCG和CPLCP家族蛋白进化关系分析,包括米象(So)、赤拟谷盗(Tc)、冈比亚按蚊(Ag)、埃及伊蚊(Aa)、尖音库蚊(Cp)、黑腹果蝇(Dm)、家蚕(Bm)、丽蝇蛹集金小蜂(Nv)和意大利蜜蜂(Am);b 米象CPLCP蛋白保守序列分析。
2.6 CPF和CPFL家族基因
在米象基因组共鉴定得到1个CPF和5个CPFL家族蛋白基因(表1)。依据不同昆虫CPF和CPFL的蛋白序列构建进化树,结果显示这两个家族蛋白分别聚集形成两个并列分支(图7a)。序列分析发现,鞘翅目昆虫米象、赤拟谷盗和马铃薯甲虫CPF蛋白分别含有保守的44个氨基酸和C末端序列(图7b和图7c),而CPFL家族蛋白则仅拥有与CPF蛋白同源性较高的C末端保守序列(图7d)。

注:a 昆虫CPF和CPFL家族蛋白进化关系分析,包括米象(So)、赤拟谷盗(Tc)、马铃薯甲虫(Ld)、黑腹果蝇(Dm)和地中海实蝇(Cc);(b和c)米象、赤拟谷盗和马铃薯甲虫CPF保守结构域分析;d 米象、赤拟谷盗和马铃薯甲虫CPFL保守结构域分析。
2.7 CPCFC家族基因
本研究在米象基因组中共注释得到3个CPCFC家族蛋白基因(表1)。为明确CPCFC蛋白的系统发育关系,共选择9种双翅目和3种鞘翅目昆虫的CPCFC蛋白序列开展系统发育分析,结果表明这两大类昆虫的CPCFC蛋白各自聚集形成一簇(图8a)。米象、赤拟谷盗和马铃薯甲虫)3种鞘翅目昆虫的CPCFC氨基酸序列分析结果显示,该家族蛋白具有共同的保守结构域,即两个重复的C-X5-C基序(图8b)。
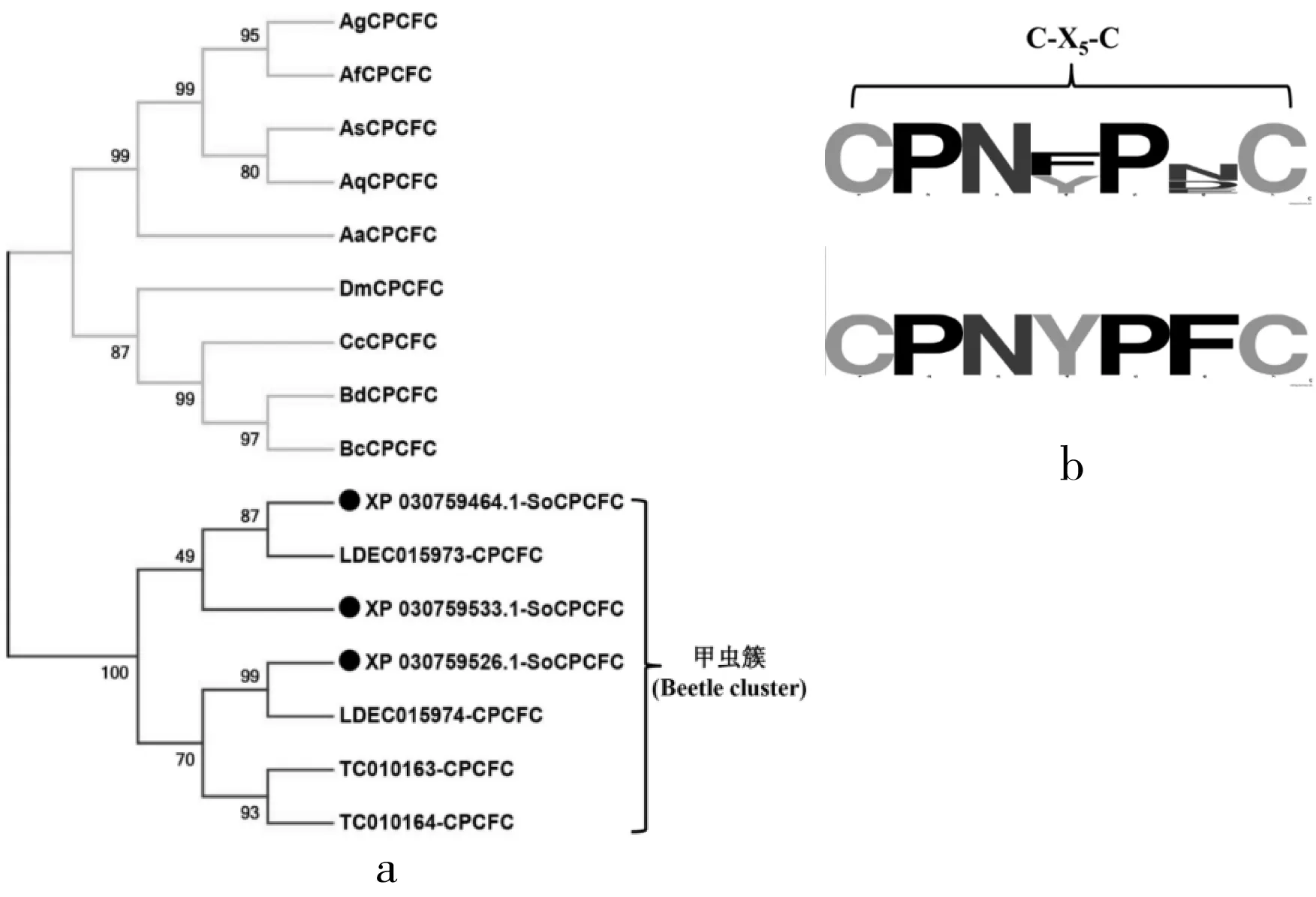
注:a 昆虫CPCFC家族蛋白进化关系分析,包括米象(So)、赤拟谷盗(Tc)、马铃薯甲虫(Ld)、桔小实蝇Bactrocera dorsalis(Bd)、瓜实蝇Bactrocera cucurbitae(Bc)、地中海实蝇(Cc)、黑腹果蝇(Dm)、埃及伊蚊(Aa)、冈比亚按蚊(Ag)、不吉按蚊Anopheles funestus(Af)、中华按蚊Anopheles sinensis(As)、四斑按蚊An.quadrimaculatus(Aq);b 米象、赤拟谷盗和马铃薯甲虫CPCFC保守结构域分析。
3 讨论
作为昆虫表皮重要结构物质,CPs不仅种类数量众多,而且在昆虫生长发育和环境适应等方面均发挥关键作用,是开发新型害虫控制剂的理想靶标[24, 25]。本研究运用生物信息学手段从米象基因组数据中共鉴定得到135个CPs基因,隶属于9个家族,包括CPR、Tweedle、CPAP1、CPAP3、CPLCG、CPLCP、CPF、CPFL和CPCFC。
本研究发现同属鞘翅目昆虫的米象、赤拟谷盗和马铃薯甲虫的CPs数量差异明显。已有研究证实CPs基因的存在可能与昆虫应对外界复杂环境密切相关,因此造成物种间CPs数量和种类巨大差别的可能原因是不同种类昆虫所处生存环境的差异[26,27]。由于米象和赤拟谷盗均为储粮害虫,其活动范围主要集中在环境稳定的粮仓内,相比而言马铃薯甲虫的生存环境则更为复杂,因此其拥有CPs数量也较多。此外,与赤拟谷盗相比,米象幼虫期和蛹期均于稻谷内部进行发育,其相对稳定和受保护的生长环境可能导致该昆虫进化出的CPs数量少于赤拟谷盗。
米象CPs另外一个重要特点是诸多CPR家族蛋白在系统发育上呈现聚类现象,即在进化树上形成许多相应物种的特异性CPs基因簇,这意味着聚集的CPs基因具有较高的序列相似度和协调一致的进化模式。前人亦分别在家蚕、斜纹夜蛾、马尾松毛虫、冈比亚按蚊等昆虫中发现类似的物种特异性CPR基因聚类,经证实这些基因均来源于染色体上的同一个基因簇,暗示其可能是从同一个祖先基因进化而来,且在各物种中分别进行扩增,结果进一步证明CPs基因的复制是在各昆虫类群中独立发生[10, 15, 27-29]。
除CPRs外,CPAPs是昆虫第二大类几丁质结合蛋白,其依据氨基酸序列所包含几丁质结合域(ChtBD2)数量,被进一步分为CPAP1(包含1个ChtBD2)和CPAP3(3个ChtBD2)两个家族[22]。然而,本研究发现昆虫CPAPs与CPR蛋白之间的系统发育关系存在巨大差异,即不同昆虫CPAP1和CPAP3同一家族成员间的亲缘关系要强于同一物种各个蛋白间的关联度,显示出这些蛋白是依据某些特定保守结构域进化而来,暗指CPAPs发展成为多基因家族的过程要早于不同种类昆虫的分化过程。之前的研究发现昆虫CPAP1家族基因的数量在不同物种间变异较大(9-17个)[30],本论文中米象较赤拟谷盗缺少CPAP1-D和CPAP1-E基因。
与其他CPs相比,不同种类昆虫间CPAP3家族基因的数量、种类及其氨基酸序列结构最为保守[30,31],并且已有研究证实CPAP3蛋白与几丁质均有较高的结合能力[32],暗示该家族蛋白可能通过与表皮中的几丁质结合从而在节肢动物的生长发育过程中发挥重要作用。例如,果蝇CPAP3-A和CPAP3-E蛋白通过与几丁质相互作用来控制昆虫表皮完整性、表皮外骨骼物理性质以及幼虫体型[33, 34]。赤拟谷盗CPAP3家族蛋白则承担着多样的生理功能,利用RNA干扰技术有效沉默该家族不同基因会对昆虫鞘翅、表皮角质层、肠道、卵巢、脂肪体、足、胚胎等造成严重发育缺陷,从而导致死亡表型[5]。这些研究结果证明昆虫特定的关键CPs,如CPAP3家族基因可成为基于dsRNA控制米象的潜在靶位点,后续可进一步针对该家族基因开展功能研究,验证其在米象生理发育过程中的重要功能。
4 结论
表皮覆盖于虫体外表面,承担昆虫外骨骼的功能,在昆虫生长发育过程中发挥重要作用,并可抵御外界不良环境,如病原体、杀虫剂等,是昆虫的一道天然保护屏障。几丁质和不同种类的CPs的比例是决定昆虫表皮结构、特性和机械性能的关键因素。因此,本论文针对米象CPs的全基因组鉴定研究不仅可以丰富昆虫CP超基因家族数据,还可为开展米象CP基因功能及生物学研究提供基础的基因序列信息,最终为米象等储粮害虫的新型防控策略奠定重要基础。

