基于线粒体基因组全序列的鲟形目鱼类(Pisces:Acipenseriformes)的分子系统发育重建
程佩琳 俞 丹 刘焕章 杜 浩 危起伟
(1. 中国水产科学研究院长江水产研究所, 农业农村部淡水生物多样性保护重点实验室, 武汉 430223;2. 中国科学院水生生物研究所水生生物多样性与保护重点实验室, 武汉 430072)
鲟形目鱼类(Acipenseriformes), 隶属于辐鳍鱼亚纲(Subclasss Actinopterygii)、软骨硬鳞鱼下纲(Chondrostei), 是现生软骨硬鳞鱼类的代表类群[1]。除已绝迹的2个化石科类群(Chondrosteidae和Peipiaosteidae)外, 现生鲟形目鱼类总计有2科6属27个有效种[2—4]。作为最古老的鱼类之一, 鲟类的化石记录最早可追溯到2亿年前的白垩纪时代, 因此鲟类在鱼类乃至整个脊椎动物进化史上都占有极其重要的地位, 被誉为鱼类的“活化石”[5,6]。
在过去的20多年中, 基于形态学特征及分子数据对鲟形目鱼类的系统进化关系进行了广泛探讨,但仍未取得一致意见。特别是, 鲟科的内部系统发育关系及鲟科的各个属的单系性仍存有许多争议。Birstein等[7]基于3个线粒体基因(Cytochromeb、12S rRNA和16S rRNA)首次较为完整地分析了鲟形目20个物种的系统发育关系, 发现鲟形目鱼类的2个科, 匙吻鲟和鲟科是单系群。但鲟科内部关系复杂, 鲟属和鳇属不是单系群; 铲鲟属是鲟科最原始的基部类群[7,8]。张四明等[9,10]采用2个线粒体基因(ND4L和ND4)对包括中国特有鲟类在内的12种鲟形目鱼类进行了分子系统分析, 指出环太平洋地区的鲟科鱼类可能有共同的起源。Ludwig等[11,12]基于22种鲟类的线粒体细胞色素b基因序列进行了分子系统发育构建, 显示铲鲟属和尖吻鲟类为并系, 位于鲟科的基部位置; 并发现鲟科的部分鱼类按生物地理分布区域进行聚类, 首次提出鲟科内部可以划为大西洋鲟类和太平洋鲟类2个大类群。这一划分得到之后的许多鲟类分子系统发育研究的支持[5,13]。Fontana等[14]回顾了早期的鲟类染色体核型和分子系统发育的研究结果, 基于18种鲟类的线粒体基因细胞色素b基因序列进行了系统发育重建, 确认了尖吻鲟类(大西洋鲟和尖吻鲟组成的单系群)是鲟科的基部类群。但Krieger等[15]基于25个鲟物种的5个线粒体基因(Cytochromeb、12S rRNA、Cytochromecoxidase subunit II、tRNA Asp和tRNA Phe)联合数据, 结果显示鲟科鱼类的基部类群仍无法确定, 可能是铲鲟属, 也可能是尖吻鲟类。Luo等[16]基于16种鲟类的30个单拷贝核基因序列联合数据, 分子系统分析认为, 鲟科可以划分为3个类群: 尖吻鲟类、太平洋鲟类和大西洋鲟类; 并确认了尖吻鲟类为鲟科的基部类群。尽管早期鲟形目鱼类形态分类与分子系统发育结果的差异很大, 但近些年的鲟形态分类学的研究也得到了新的进展。Hilton等[17,18]对鲟类化石物种和现生种进行了物种水平的形态学比较研究, 证据支持鲟属不是单系群, 并定义了鲟科的2个新类群: 鳇科(Husinae)及拟铲鲟科(Pseudoscaphirhynchinae)。因此, 鲟科的基部类群究竟是铲鲟属还是尖吻鲟类?鲟科的鲟属和鳇属的单系性是否有效? 及鲟科内部的系统发育关系如何? 都是尚未解决的问题。
鱼类线粒体基因组是一个15—20 kb的环状双链DNA分子, 通常编码37个基因, 即13个蛋白质编码基因(PCGs)、22个转运RNA基因(tRNAs)和2个核糖体RNA基因(rrnL和rrnS)[19]。此外, 线粒体基因组通常还有2个负责复制和转录的非编码区(OL和OH)。由于线粒体基因组具有母系遗传、多拷贝、缺少遗传重组及进化速率快等特点, 已广泛应用到系统发育重建、比较基因组学、谱系地理学及种群遗传学等研究领域[20,21]。在过去的10年中, 鱼类线粒体基因组研究受到了高度关注, 并由于高通量测序技术的广泛应用而使得已测线粒体基因组的鱼类种类呈现快速增长的趋势。目前, 线粒体基因组数据广泛用于硬骨鱼类不同分类阶元的系统进化关系研究[22,23]。然而, 鲟类中基于线粒体基因组数据的研究很少。一方面, 鲟类作为全球珍稀保护鱼类, 广泛分布在北半球, 样本珍贵且难以收集; 另一方面, 不同的线粒体基因数据集的构成, 不同的系统发育分析方法对系统发育的构建的结果也存在影响。因此, 获取更多的鲟线粒体基因组数据, 特别是鲟属鱼类, 并比较分析不同系统发育分析方法在构建鲟形目鱼类的系统进化关系方面的价值和潜力, 显得非常必要。
本研究新测定了中华鲟(Acipenser sinensis)、长江鲟(A. dabryanus)、短吻鲟(A. brevirotrum)、纳氏鲟(A. naccarii)、鳇(H. dauricus)和匙吻鲟(Polyodon spathula), 共计3属6个种的线粒体基因组, 进一步丰富了鲟形目鱼类的线粒体基因组数据库。在注释这6种鲟的线粒体基因组基础上, 联合已有的17个鲟类的线粒体基因组, 基于5个线粒体基因组数据集(Pro、AA、2rRNA、Pro_2rRNA和Com_Mito), 采用2种方法(最大似然法和贝叶斯法)重新构建了鲟形目的系统发育关系, 并采用似然值检验对不同的树拓扑结构进行了评价。
1 材料与方法
1.1 基因组DNA提取、NGS技术测序组装
本研究样本均由中国水产科学研究院长江水产研究所荆州太湖基地提供, 包括3属6种鲟形目鱼类样本(中华鲟、长江鲟、鳇、短吻鲟、纳氏鲟和匙吻鲟)。本研究所涉及的动物实验均得到中国水产科学研究院长江水产研究所实验动物福利伦理委员会(编号: YFI-001)批准。收集样本的新鲜血液或鳍条送至武汉天一辉远生物科技有限公司进行测序。采用DNA提取试剂盒(天根生物, 北京)在提取总DNA后, 用Thermo Scientific NanoDrop 2000测定DNA浓度, 采用琼脂糖电泳和Agilent 2100 Bioanalyzer检测DNA完整性。采用全基因组鸟枪法(Whole Genome Shotgun, WGC)策略, 构建400 bp文库, 利用第二代测序技术(Next Generation Sequencing, NGS), 基于Illumia Miseq测序平台进行双末端(Paired-end, PE)测序。采用A5-miseq v20150522[24]和SPAdes v3.9.0[25]对高质量的二代测序数据进行从头拼装, 构建contig和Scaffold序列。根据拼接序列的测序深度提取序列, 将高测序深度的序列同NCBI(National Center for Biotechnology Information)的nt库进行blastn(BLAST v 2.2.31)比对, 挑出各拼接结果的线粒体序列。将得到的线粒体拼接结果利用mummer v 3.1[26]软件进行共线性分析, 确定contig间的位置关系, 进行contigs间gap填补。使用pilon v 1.1.8[27]软件对结果进行校正得到最终的完整线粒体基因组全序列。将拼接得到的完整线粒体基因组序列上传至MITOS网页服务器(http://mitos.bioinf.uni-leipzig.de/)进行编码基因, RNA和非编码区的注释[28]。密码子选择脊椎动物数据库,其余为默认参数。采用软件tRNAscan-SE 1.2.1[29]鉴定tRNA基因, 并预测其二级结构。采用Mega 6.06[30]分析碱基组成和密码子使用, 并计算AT-偏斜(AT-skew)和GC-偏斜(GC-skew)。
1.2 系统发育分析
采用本研究新测的6种鲟的线粒体基因组全序列, 结合NCBI中已发表的17种鲟类的线粒体基因组进行系统发育分析。通过软件CLUSTAL X version 1.83[31]进行序列的多重比对。为了明确线粒体数据集的构成差异对系统发育结果的影响, 构建了5个线粒体数据集: Pro(Combined 13 coding protein gene nucleotide sequences, 13个蛋白质编码基因核苷酸序列)、AA(Combined 13 protein coding gene amino acid sequence, 13个蛋白质编码基因氨基酸序列)、2 rRNA(Combined 2 rRNA nucleotide sequences, 2个rRNA联合序列)、Pro_2 rRNA(Combined 13 coding protein gene and 2 rRNA gene nucleotide sequence, 13个蛋白编码基因及2个rRNA联合序列)和Com_Mito(Complete mitochondrial genome nucleotide sequences, 线粒体基因组核苷酸全序列)。其中1、2、3和4数据集均由单个蛋白质编码基因或者rRNA基因单独比对。为了避免比对位点的偏倚, 排除模糊区域位点(包括蛋白质编码基因的起点和终点区域, 及rRNA序列中高度可变区域)使用软件GBlocks 0.91b[32]进行比对删除。将比对好的单个蛋白质编码基因或者rRNA基因联合在一起获得目标数据集。而5个线粒体基因组全序列则采用所有碱基无差别对待策略, 通过CLUSTAL进行序列多重比对后, 采用GBlocks去除空位及模糊位点, 最后基于比对删除后得到的23种完整线粒体基因组数据的集合。对于这5个不同类型的数据集, 分别采用IQ-TREE web server[33](http://iqtree.cibiv.univie.ac.at/)的Model Finder[34]选择序列的最佳分区及最优碱基/氨基酸替代模型检测, 并应用于后续系统发育分析。对不同数据集分别使用IQTREE的Tree Inference[35]构建最大似然(ML)树, 采用对应的最佳分区模式及进化模型, 选择超快自展法(Ultrafast bootstraping)[36]构建ML树, 节点可靠性采用1000次的bootstrap进行评估。贝叶斯BI分析采用软件Mrbayes 3.2.2[37]进行, 基于贝叶斯信息准则(BIC)选择最优碱基替换模型。根据结果设置模型参数, 然后再运行4条独立的马尔可夫链(Markov chains), 即3条热链(Hot chain)和1条冷链(Cold chain)同时运行6千万代(Generation)。每运行100代抽样1次, 当运行结束时显示“分列频率平均标准差”(Average standard deviation of split frequencies)小于0.01即认为分析趋于稳定状态。舍去25%的老化样本, 再由剩余树产生多数一致树, 并计算贝叶斯后验概率。
由于不同的数据集及树构建方法获得的鲟形目的系统发育关系存在不一致性, 因此为了明确哪种系统发育树值得信任, 进一步采用了IQ-TREE web server的Tree topology evaluation and tests进行树拓扑结构检验。对于5个数据集, 分别采用SH(Shimodaira-Hasegawa test)[38]、WKH(Weighted Kishino-Hasegawa test)[39]、WSH(Weighted SH test)[38]和AU(Approximately unbiased test)[40]共4种方法进行统计分析, 并设置1000次重复。其结果顺序在扩展名为iqtree的文件中给出, 一般认为SH-aLRT≥80%且ultrafast bootstrap value ≥95%的节点可信赖。
2 结果与讨论
2.1 六种新测鲟的线粒体基因组基本特征
通过测序组装, 我们获得了中华鲟、长江鲟、短吻鲟、纳氏鲟、鳇和匙吻鲟的完整线粒体基因组(GenBank登录号: MK078260-MK078265)。新测的6种鲟的线粒体基因组均显示是典型的闭合环状双链DNA分子, 总长度为16439—16766 bp, 编码37个线粒体基因, 即13个PCGs、22个tRNAs和2个rRNAs, 及非编码控制区(OH区)和轻链复制起始区(OL区)。只有8个tRNA和ND6基因在L链上编码,其余多数基因均在H链上编码, 且基因排序与鱼类线粒体基因的原始排序完全一致。
新测的6种鲟显示鲟类的基因组结构较为紧凑。以中华鲟为例, 统计共有15处碱基间隔区, 间隔碱基数为1—395 bp; 有9处碱基重叠区, 重叠碱基数为1—10 bp。其中有3对蛋白质编码基因的开放阅读框存在碱基重叠, 包括ATP8-ATP6、Nad4LNad4和Nad5-Nad6分别重叠10、7和4 bp。基因间既没有间隔又无重叠的基因对共15处。其余物种的线粒体基因组特征同中华鲟类似。
6种鲟的碱基组成显示具有一定的A+T偏向性,同时还呈现出较强的A碱基和C碱基偏好。以中华鲟为例, 线粒体全基因组的碱基A、C、G和T含量分别为30.17%、29.51%、16.47%和23.85%。其中A+T含量(54.02%)大于G+C含量(45.98%), 表明存在一定的A+T偏向性, 这与其他脊椎动物的线粒体基因组特征类似。而且, 除ND6基因外, 线粒体基因组基因都表现出一定的A+T偏向性, 其中ATP 8基因的A+T偏向性最高(60.12%)。因为ND6基因以L链为反义链, 所以密码子第三位G比重较大, 因而整个基因的T+G含量很高(76.82%), 这与脊椎动物的线粒体基因组的情况是相似的。6个鲟的线粒体全基因组的AT-偏斜均为正值(0.109—0.126), 而GC-偏斜为负值(–0.284—0.316), 这与其他脊椎动物的线粒体基因组核苷酸偏斜的规律一致(表 1)。在6种鲟中, 匙吻鲟的线粒体基因核苷酸偏斜值最大, AT-偏斜为0.126, GC-偏斜为–0.316。
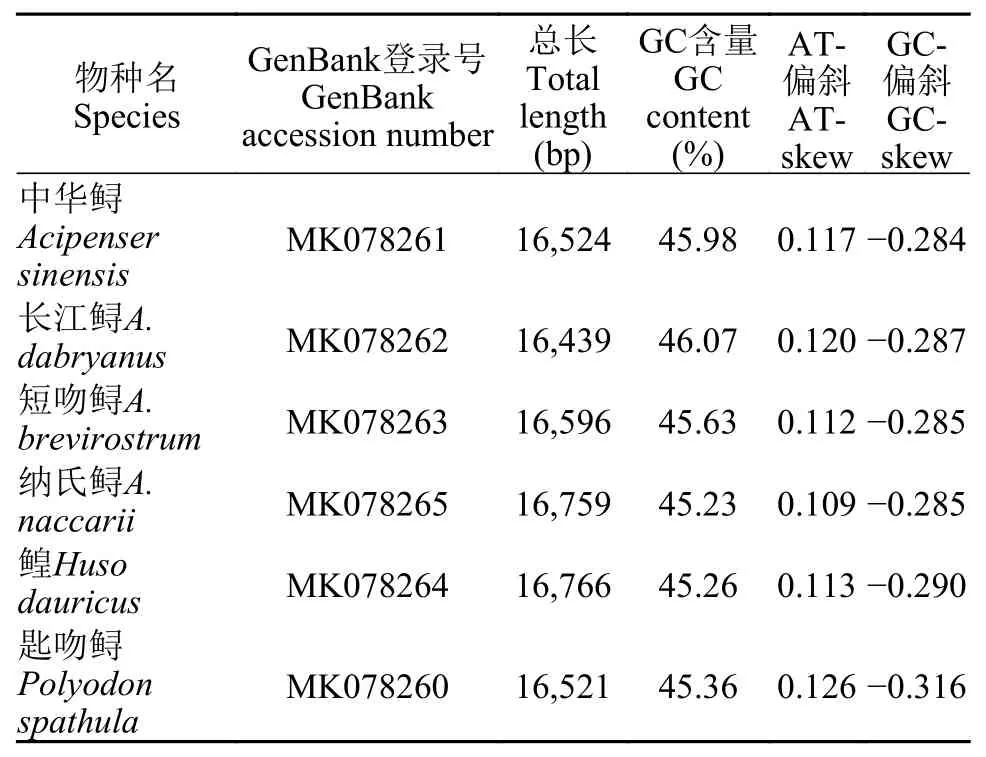
表 1 新测6种鲟形目鱼类的线粒体基因组长度及碱基组成Tab. 1 Total length and base composition of mitochondrial genomes of six sequenced Acipenseriforme fishes
6种新测鲟的线粒体基因组高的A+T含量及不同链上核苷酸的偏斜性, 也反映在蛋白质编码基因的密码子使用上。相对同义密码子使用频率(Relative synonymous codon usage, RSCU)分析表明, 所有蛋白质编码基因的密码子使用都存在较强的偏好性, NNA或NNC(即第三位点为C的密码子)的RSCU都大于1, 使用频率较高。6种鲟的基因组的这种密码子的使用模式, 与已测其他鲟的高度相似。
在6种鲟的13个蛋白质编码基因中, 起止密码子均为TAG或GTG, 其余12个蛋白质编码基因的起始密码子均以ATG开始, 仅COXI基因以GTG开始。终止密码子为TAA、TAG或T--, 其中COXII、ND4和Cytb基因以不完全密码子T--终止, 而ND1、ND2、COXI、ND3和ND6基因以完整密码子TAG终止, ATP8、ATP6、COXIII、ND4L和ND5基因以完全密码子TAA终止。不完全的终止密码子在鱼类线粒体基因组是普遍存在的特征。tRNAscan-SE分析显示, 6种鲟的22个线粒体tRNAs基因均具有经典的三叶草结构。
2.2 系统发育分析
基于5个数据集(Pro、AA、Pro_2 rRNA、2 rRNA和Com_Mito)和2种系统发育分析方法(ML和Bayes)总共获得了10个系统发育树(图 1)。尽管这10个系统发育树的拓扑结构不完全一致, 但均高度支持匙吻鲟科和鲟科的单系性(表 2)。Pro_2rRNA和Com_Mito两个数据集的BI和ML的拓扑结构, 及Pro数据集的BI树的拓扑结构完全一致, 即5个系统发育树的结果完全一致。分子系统发育重建的结果显示(图 1), 匙吻鲟科为单系(BI:1.00/MP:100), 包括匙吻鲟属和白鲟属2个单属单种。鲟科为单系(BI:1.00/MP:100), 但鲟属和鳇属的物种不构成单系群。鲟科按系统发育结果可以分为3个单系类群。鲟科的基部类群是由欧洲大西洋鲟(A. sturio)和尖吻鲟(A. oxyrinchus)构成的尖吻鲟类(A. sturio-A.oxyrinchusclade), 具有极高的节点支持率(BI:1.00/MP:100)。其余鲟科鱼类可以分为2个大的单系类群: 大西洋鲟类(Atlantic clade)和太平洋鲟类(Pacific clade)。其中大西洋鲟类由3个铲鲟属物种、欧鳇及9个鲟属物种构成。铲鲟属显示是大西洋鲟类的基部类群。太平洋鲟类由鳇和7个鲟属物种构成。3个BI树的结果完全一致, 且节点支持率极高(BI>0.95), 仅一处节点支持率较低。而2个ML树中节点支持率低于90的共计有2处。
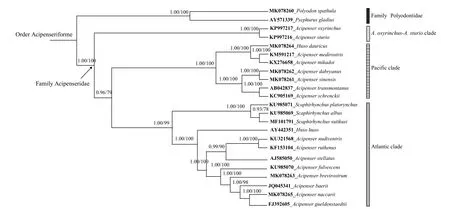
图 1 基于23种鲟形目鱼类的线粒体全基因组(Com_Mito)构建的贝叶斯法和最大似然法的系统发育树(节点前数字表示BI后验支持率及ML自展数)Fig. 1 Baysian and ML phylogenetic tree based on 23 complete mitochondrial genomes nucleotide sequences
Pro数据集的ML树仅有一处不同于BI树, 位于大西洋鲟类内部的闪光鲟(A.stellatus)位置上(BI:40)。这表明, 尽管最大似然法在系统发育分析中得到广泛应用, 但在构建某些类群的系统发育关系方面有时表现欠佳, 这与其他已有研究结果一致。2 rRNA数据集的BI和ML树的结果一致, 支持铲鲟属为鲟科的基部类群(BI:1.00/ML:100), 然后是尖吻鲟类(BI:0.52/ML:65)。其余鲟科鱼类可以分为2个单系群(BI:0.31/ML: 36)。AA数据集的BI和ML结果显示, 支持尖吻鲟类是鲟科的基部类群(BI:1.00/ML:100), 其余鲟科鱼类可以分为2个单系群(BI:0.98/ML:54)。
综合比较10个系统发育树发现(表 2), 核苷酸数据与氨基酸数据相比, 表现出更好的系统发育信息。而与13个蛋白质编码基因联合序列或者2个rRNA基因联合序列相比, 蛋白质编码基因和2个rRNA基因联合序列, 及线粒体基因组全序列2个数据集的结果具有一致且极高的节点支持率, 这表明完整的线粒体基因组数据更有助于解决鲟形目的系统发育关系。
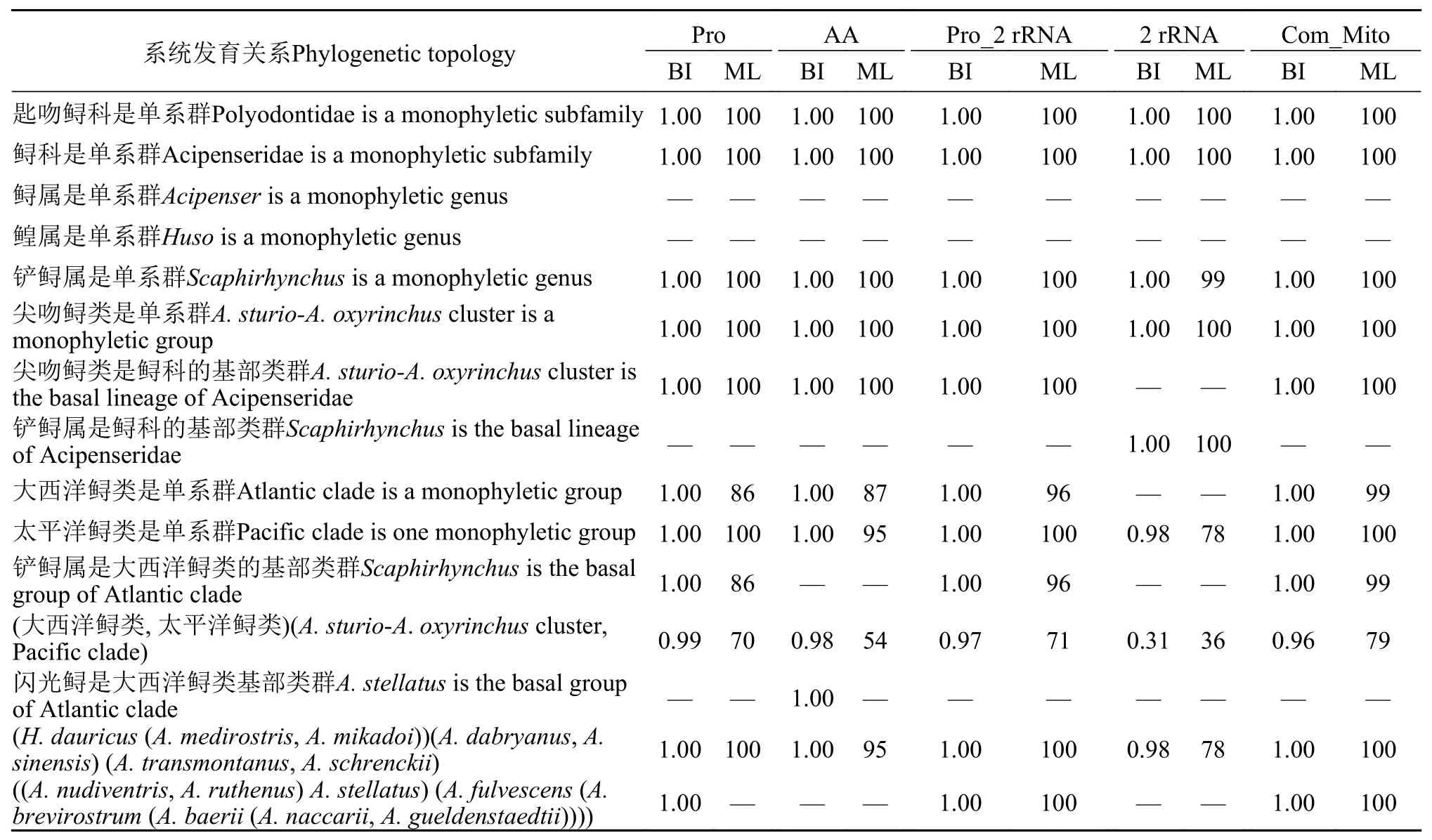
表 2 不同数据集、不同方法获得的鲟形目鱼类的系统发育结果Tab. 2 Phylogenetic relationships within order Acipenseriforme based on different dataset
2.3 树拓扑结构检验
按照鲟形目的系统发育关系, 本研究的10个系统发育树共有5种不同的树拓扑结构。树拓扑结构检验结果表明, 基于5个数据集的树拓扑结构检验,获得了高度一致的结果, 均支持Pro_2 rRNA和Com_Mito两个数据集的BI和ML的系统发育关系(表 3)。系统发育结果表明, 鲟科的基部位置有2种情况: (1)拓扑结构检验支持率最高的是尖吻鲟类为基部类群, 这与Fontana等[14]为代表的研究结果是一致的。(2)铲鲟属为鲟科的基部类群, 拓扑结构检验支持度较低, 但早期的研究如Birstein等[7,8]也曾发现该系统发育关系。基于相同数据集的SH检验结果表明, 尖吻鲟类为鲟科的基部类群; 铲鲟属与欧鳇, 及里海区鲟类共同构成大西洋鲟类。
2.4 鲟形目鱼类的系统发育关系探讨
回顾鲟形目鱼类的分子系统发育研究, 不同的研究在数据集构成, 分析方法及物种选择数量等方面存在差异, 推测这可能是导致鲟形目鱼类不同研究的分子系统发育结果不一致的主要原因。鲟形目鱼类目前现生种总计有27种, 本研究共收集23种,覆盖率达85.2%。本研究结果表明, 鲟形目鱼类包括匙吻鲟科和鲟科2个单系群。鲟科内部关系复杂,鲟属和鳇属不是单系群。鲟科的基部类群为尖吻鲟类, 其余19种鲟科鱼类可以分为大西洋鲟类和太平洋鲟类。这与多数前人的鲟类分子系统发育研究结果是一致的[11,12,14,16]。大西洋鲟类包括12个鲟科物种, 其中铲鲟属3种、鳇属1种和鲟属8种。值得注意的是, 本研究首次显示铲鲟属为大西洋鲟类的基部类群。这与Dillman等[41]的推测是吻合的。Dillman等[41]基于2个线粒体基因(Cytb和控制区片段)对铲鲟亚科进行分子系统发育重建, 结果显示铲鲟亚科单系性被拒绝, 拟铲鲟属与闪光鲟有更近的亲缘关系, 并推测铲鲟属与鲟属物种的亲缘关系更近。太平洋区类群共7个鲟科物种, 包括鳇属1种和鲟属6种。其中达乌尔鳇、中吻鲟、长江鲟、中华鲟和施氏鲟, 均属于亚洲特有物种。而分布在中国的长江鲟, 及分布在东太平洋区的高首鲟, 被认为可能是淡水陆封种。
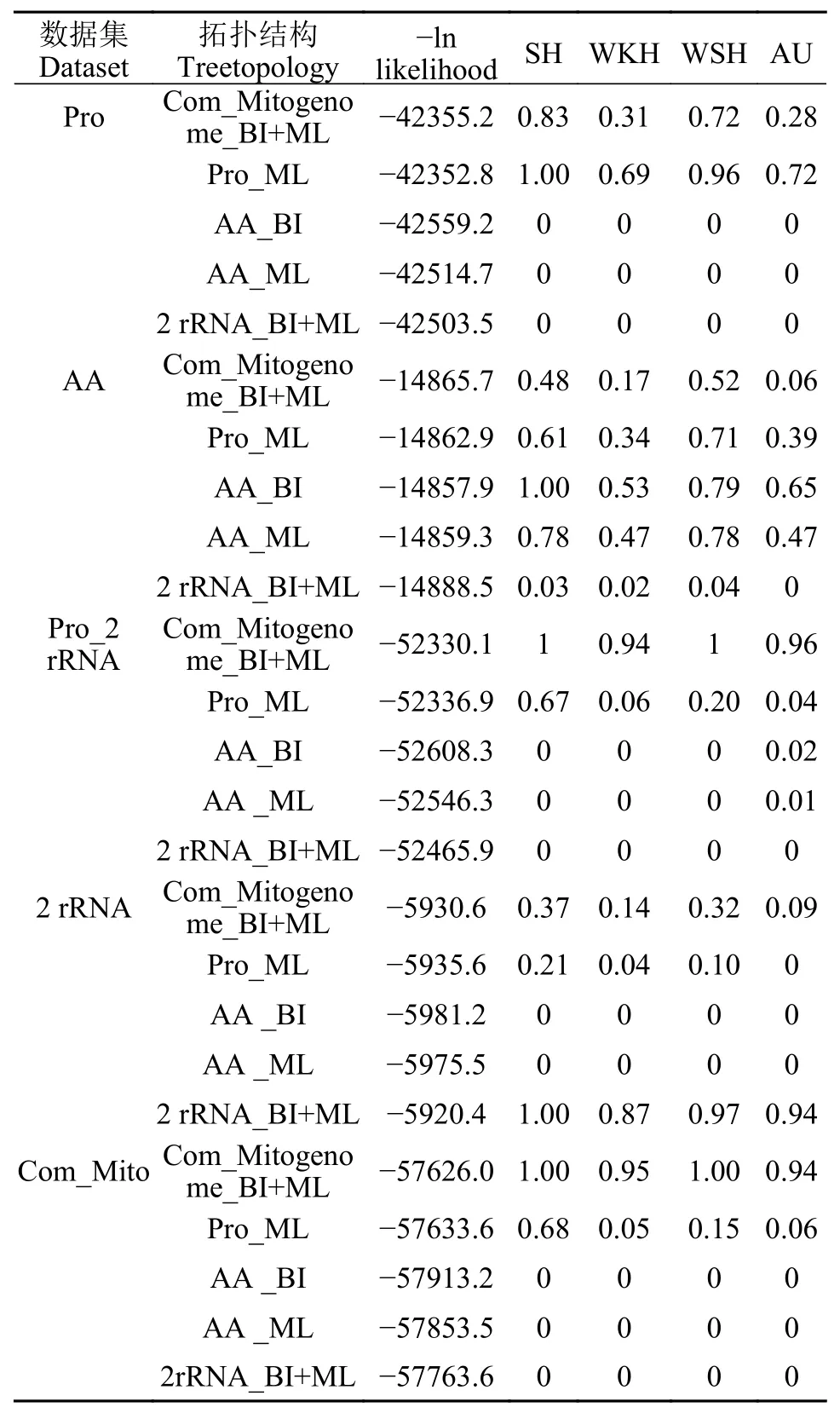
表 3 系统发育树拓扑结构检验结果Tab. 3 Results of topological tests for five phylogenetic trees
本研究没有覆盖的鲟物种有4种, 包括拟铲鲟属(Pseudoscaphirhynchus)的3个物种和鲟属的波斯鲟(A. persicus)。已有的形态学及分子生物学的证据表明, 拟铲鲟属与闪光鲟有很近的亲缘关系[16,17,41],因此建议将拟铲鲟属和闪光鲟共同列入大西洋鲟类。综合考虑到现有鲟形目鱼类的分子系统发育结果同形态学结果仍有许多不一致, 未来仍需要联合形态学数据进行综合分析, 可为鲟形目鱼类的分类, 尤其是鲟科的属的分类及属间亲缘关系提供新的认识。
3 结论
本研究新测定了包括中华鲟、长江鲟、短吻鲟、纳氏鲟、鳇和匙吻鲟在内的6种鲟的线粒体基因组全序列, 基因含量、基因排序、碱基组成、密码子使用模式及tRNA二级结构等与大部分已测鲟类高度相似。基于5个线粒体基因组数据集及2种树构建方法的系统发育分析结果表明, 鲟科的基部类群是尖吻鲟类, 其余鲟科鱼类可以划分为大西洋鲟类和太平洋鲟类2个生物地理类群。鲟科的系统发育关系为(尖吻鲟类(太平洋鲟类(铲鲟属(欧鳇,里海区鲟类))))。尽管线粒体数据集的构成及系统发育分析方法对树拓扑结构具有一定影响, 但线粒体全基因组数据在解决鲟形目鱼类的系统发育关系上显示出重要的系统发育信息价值。

