高通量测序技术在传统发酵食品微生物群落中的应用研究
夏梦雷,杨 帆,陆 锴,王 頔,郑 宇,王 敏
(天津科技大学 生物工程学院 食品营养与安全国家重点实验室 工业发酵微生物教育部重点实验室天津市工业微生物重点实验室,天津 300457)
高通量测序技术是当今基因组学研究中最为重要的方法之一,是将脱氧核糖核酸(deoxyribonucleic acid,DNA)或者互补脱氧核糖核酸(complementary DNA,cDNA)随机片段化、加接头,制备测序文库,通过对文库中数以万计的基因进行延伸反应,检测对应的信号,最终获取序列信息,现已广泛应用于传统发酵食品中,高通量测序技术的不断进步为传统发酵食品中菌群的研究提供了重要的参考。
传统发酵食品具有悠久的历史,且种类繁多,因其独特的风味和丰富的营养而深受大众欢迎。传统食品最大的特点就是区域性差异,即在复杂的地理环境和气候条件不断创新发展形成[1-2]。发酵过程一般来说是在常温常压下进行的生物化学反应,生产条件简单。传统发酵食品已成为中国食品产业中不可或缺的组成,近年来随着国内外发酵食品工业化水平的逐年提高,传统发酵食品产品结构持续优化,中高端产品不断涌现,现代科学技术已成为传统发酵食品企业纷纷借力的重要支撑[3]。
一般来说,利用微生物的作用而制得的食品都可称之为发酵食品,其自然接种的生产方式造就了微生物群落的复杂性和多样性[4-5]。深入理解发酵机制,必须对微生物的代谢机制进行研究[6-7]。传统发酵食品(如食醋、酱油、白酒等)涉及到的菌种多、菌群产物复杂,传统方法对菌群的分离纯化培养会脱离原始的菌群环境,使分析结果产生偏差,利用现有的分子生物学技术可以避免此现象的发生,同时,食品发酵的相关研究主要停留在产品和工艺的描述上,而对于大部分食品发酵背后的生化原理和微生物菌群的代谢机理研究甚浅。随着高通量测序、宏基因组学等技术的快速发展,系统分析传统发酵食品的风味特征、功能成分和菌群结构成为传统酿造食品研究的主流方向[8-11]。这些研究的不断深入,对促进传统发酵食品的科技创新、产业的现代化发挥着重要的作用。
本文聚焦以聚合酶链式反应-变性梯度凝胶电泳(polymerase chain reaction-denatured gradient gel electrophoresis,PCR-DGGE)、实时荧光定量聚合酶链式反应(real-time fluorescence quantitative-polymerase chain reaction,RTFQPCR)、宏基因组测序、宏转录组测序为代表的现代分子生物学技术,综述了其在国内外传统发酵食品功能微生物菌群研究中的最新进展,以期为传统发酵食品中功能微生物的研究提供参考依据。
1 PCR-DGGE技术
PCR-DGGE技术能够快速分析微生物的物种多样性,是分析微生物多样性和鉴定不同地区微生物群落的有效工具。与依赖培养的方法相比,PCR-DGGE方法直接从样品中提取DNA,分析速度更快,能反映样品中的整个微生物群落[12],因此被广泛应用在微生物生态学[13]和食品微生物学[14]等领域。
PCR-DGGE技术对微生物的分析如图1所示,主要包括三个步骤:①核酸提取;②目标序列的PCR扩增;③序列对比分析。总DNA的提取是整个分析过程的基础,是否能获取样品优质的总DNA决定了分析的可能性。DGGE是在聚丙烯酰胺凝胶的基础上加入甲酰胺和尿素两种变性剂,当泳道与DNA变性所需变性剂浓度一致的凝胶浓度位置时,导致双链部分变性。理想情况下,群体聚合酶链反应产物中的每个独特序列将在凝胶中的不同位点部分变性,从而得到高效分离[15-16]。
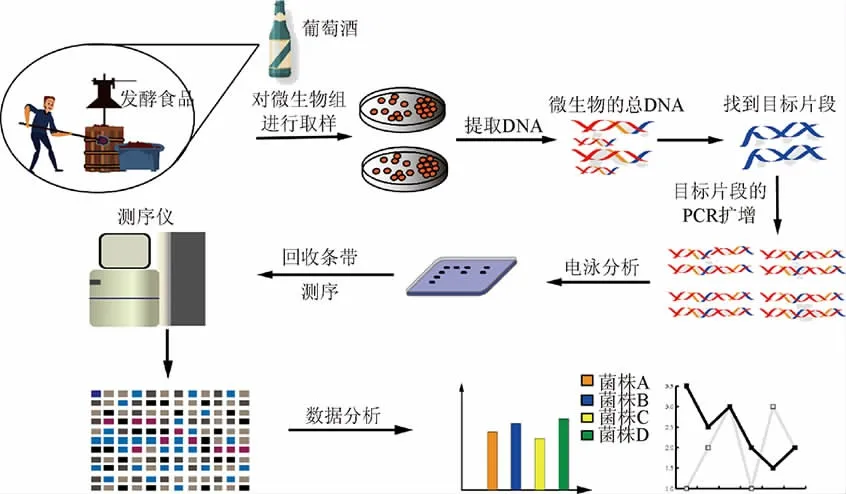
图1 PCR-DGGE技术分析微生物菌群Fig.1 Analysis of microbial community by PCR-DGGE
PCR-DGGE技术可以对生态样品中微生物菌群的构成和动态变化趋势在较短时间内做出相对客观的评价,COCOLIN L等[17]将PCR-DGGE技术初步应用于意大利发酵肠发酵过程中微生物菌群动力学研究,证实了发酵肠成熟早期的优势菌是乳酸菌。而彭杨等[18]为了探究四川麸醋和传统豆酱中的菌群,利用PCR-DGGE技术对其中的微生物群落多样性进行了探究。左丽丽等[19]采用PCR-DGGE技术分析吉林市传统发酵豆酱中细菌的多样性,结果表明,豆酱发酵过程中细菌种类较为丰富,其中乳酸菌是发酵过程的优势菌,对豆酱的风味起主要作用。张文学等[20]对白酒窖池中的糟醅样品进行了DGGE分析,并结合16S rDNA同源性比较,揭示了白酒初步发酵时期窖池的糟醅中微生物多样性的分布规律,并鉴定和筛选出主要优势菌,为掌握中国浓香型白酒发酵过程中细菌类微生物区系的形成和动态变化规律提供了理论依据。
PCR-DGGE技术的优点突出,特别是在传统培养技术无法实现的地方,此技术可以大大缩短样品的分析时间,同时不依赖于微生物培养过程而将环境样品中大部分不可培养的微生物的遗传信息显现出来[20]。但是PCR-DGGE技术的应用存在一定的局限性,因其具有很大的随机性,检测过程中菌种数量的改变和DNA含量的变化都会造成检测结果的偏差。
2 RT-FQPCR技术
实时荧光定量聚合酶链式反(RT-FQPCR)技术是通过监测PCR过程中所加入荧光基团的荧光信号,利用已知浓度的标准品绘制标准曲线,对未知样品进行定量分析的一种核酸定量技术[21]。与传统培养的方法相比,PCR更简便、更灵敏、更具有特异性,即使在没有选择性增菌培养基和存在其他种群的情况下,也能检测到亚优势种群。RT-FQPCR的出现,一举克服了常规PCR技术的诸多难题,简化了定量检测的实验过程,荧光定量可以实现精确定量,普通PCR则不行,RT-FQPCR可以看到整个扩增过程,包括扩增效率、溶解温度、标准曲线等,这些是普通PCR技术无法实现的,RT-FQPCR技术在基本实现绝对定量的同时,显著提高了工作效率。RT-FQPCR包括一系列的扩增循环,在这些循环中,模板核酸被变性,用特定的寡核苷酸引物退火,并使用耐热的DNA聚合酶延伸以产生互补链。这导致扩增产物呈指数增长,与终点PCR相比,可以使用荧光报告程序在每个周期实时进行监测[22]。随着生物芯片技术和荧光探针定量技术的结合,RT-FQPCR在微生物研究及其他领域中也会拥有更加广阔的应用前景[23]。
目前RT-FQPCR检测方法如图2所示,主要有三种:①双链DNA内插染料;②分子信标技术;③TaqMan探针[24]。双链DNA内插染料是荧光实时定量PCR中常用的方法之一。有些染料可以选择性的与双链DNA结合,同时产生强烈荧光,如SYBR Green Ⅰ,在水溶液中,染料本身的荧光强度很低,一旦插入双链DNA中,染料就会发出强烈的荧光,当染料从DNA双链上释放出来时荧光强度急剧降低[25]。因此在PCR体系中,荧光的信号强度与DNA的含量成正比。对于荧光定量检测,双链DNA内插染料是一种成本低廉的选择。分子信标技术是基于荧光共振能量转移(fluorescence reso nance energy transfer,FRET)设计的一种荧光标记核酸探针,在同一探针的两末端分别标记荧光基团和淬灭基团,该探针5'端和3'端可形成一个发卡结构,携带约8个互补碱基。当溶液中无特异性模板时,探针末端两基团邻近,无信号产生。反之,特异性模板与探针结合,上面的发卡结构,即FRET被破环。特异性模板浓度越高,溶液产生的荧光强度也越高,因此可用于PCR定量分析[26]。

图2 RT-FQPCR三种信号监测方式的原理图[24]Fig.2 Schematic diagram of three signal monitoring methods of RT-FQPCR
目前,RT-FQPCR技术已经被广泛用于传统发酵食品中微生物的定量分析。罗青春等[27]采用RT-FQPCR技术对不同年份窖泥中主要产甲烷菌进行了定量研究,王兴兴等[28]基于RT-FQPCR方法初步分析了西藏开菲尔粒中细菌与酵母菌的数量变化。同时,陶京兰等[29]利用RT-FQPCR监测镇江香醋醋酸发酵过程中微生物变化,证实了醋醅中的微生物主要为醋酸菌、乳酸菌和酵母菌,三者在细菌和真菌总和占比中超过80%。并对醋酸发酵阶段醋醅中总细菌、总真菌、醋酸菌、乳酸菌和酵母菌的动态变化进行了定量分析。对于食醋发酵过程的控制以及提高产品的风味具有重要的生产意义。
虽然RT-FQPCR技术的优势尽显无疑,但也有一定的局限性,如实验设备要求高,实验材料成本高,不能实时检测出丰度较小的PCR产物数据,实验数据处理繁琐等[30]。随着相关技术的发展,尤其是与基因芯片、高通量测序等其他生物技术的结合,RT-FQPCR技术正在经历快速的改进和完善。
3 宏基因组测序技术
自2005 年以来,随着分子技术的发展,以罗氏公司454测序技术、Illumina公司的Solexa测序技术和ABI公司的Solid 测序技术为代表的高通量测序技术相继诞生,并迅速广泛应用于传统发酵食品的微生物群落的宏基因组研究中[31-32]。与传统的16S rDNA技术相比,宏基因组测序将研究深入至基因和功能层面,能对微生物群落结构、基因功能活性、微生物之间的相互作用关系以及微生物与环境之间的关系进行全面剖析,为微生物群落的研究及微生物资源的利用提供了有效工具[33-34]。
宏基因组测序技术步骤如图3所示,主要包括:①样品准备;②DNA提取;③宏基因测序。宏基因组测序技术对抽提的DNA量和完整性要求很高,尽可能保证获得的环境微生物组DNA的质量和完整度,建立宏基因组文库,对宏基因组文库中的DNA片段进行桥式PCR扩增,扩增后通过Illumina Hiseq PE150、Roche 454焦磷酸等测序平台对DNA片段进行测序[35]。

图3 宏基因组测序技术分析微生物菌群Fig.3 Analysis of microbial community by metagenomic sequencing technology
近年来,宏基因组测序逐渐应用于食品微生物的研究,并取得了一系列研究成果。JUNG J Y等[36]利用宏基因组的方法来监测泡菜发酵过程中细菌种群、代谢潜能和微生物群落整体遗传特征的变化,其中源自宏基因组16S rRNA基因的系统发育分析表明,泡菜的微生物群落主要由明串珠菌属(Leuconostoc)、乳酸菌属(Lactobacillus)和魏斯氏菌属(Weissella)3个属组成。同时从宏基因组数据中还发现了大量噬菌体DNA序列,这些结果不仅深入揭示泡菜发酵微生物的功能,也揭示了复杂的微生物群落对发酵过程的影响。此外,研究人员通过宏基因组测序并与数据库比对进行相关的注释,分别研究可可豆[37]、普洱茶[38]、奶酪[39]和传统发酵饮料[40]等发酵食品。汤涵岚[41]则将其推广应用到食醋大曲发酵中功能微生物和酶系的研究,通过在实验室模拟食醋大曲固态发酵过程,以增温实验组(HT)和对照组(MT)两批次大曲为研究对象,结合宏基因组技术分析增温对食醋大曲发酵过程微生物菌群的影响,揭示了增温对食醋大曲发酵过程中的微生物菌群的影响,为大曲微生物及酶系形成机理提供参考依据,对传统酿造工艺改进和提升食醋品质具有重要意义。
宏基因测序技术为环境微生物群落的研究提供了有效工具,其研究的对象是特定环境中的总DNA,不是某特定的微生物或其细胞中的总DNA,不需要对微生物进行分离培养和纯化,此技术特点打破了传统微生物学基于纯培养研究的限制,这对认识和利用95%以上的未培养微生物提供了一条新的途径。
4 宏转录组测序技术
宏转录组测序技术是当今基因组学研究中最为重要的方法之一,其研究对象是自然界相对复杂的微生物群体,宏转录组测序技术可以分析活跃的转录过程,更关注于微生物群落的动态变化,填补了测序技术的空白[42]。它以微生物群落的总RNA为研究对象,提取样品总RNA,将信使核糖核酸(messenger ribonucleic acid,mRNA)反转录为互补脱氧核糖核酸(cDNA),进而对cDNA分析来反映特定时空下基因的表达情况[43]。早期转录组研究主要运用微阵列芯片技术,但是设计和构建微阵列芯片费用高而且费时,且不能检测到设计模板之外的基因的表达水平。而宏转录组测序技术较好地解决了这一困境,它针对转录产物mRNA进行高通量测序,可全面快速地获取特定样品在某一特定状态下的完整表达信息,普遍应用于差异表达基因分析、功能基因挖掘、转录图谱绘制等各个方面。与宏基因组技术相比,该技术解决了前者无法准确揭示特定时空条件下微生物群落基因的动态表达与调控的缺点。
宏转录组测序技术分析食品微生物群落的主要操作流程如图4所示,主要包括:①样品检测;②文库构建;③文库质控;④上机测序。高质量的RNA 是整个项目成功的基础,为保证测序数据准确性,需要先对样品进行检测。检测样品纯度要求RNA的OD值应在1.8~2.2之间,样品质量浓度不低于400 ng/μL,样品总量不低于15 μg。文库构建完成后,对文库质量进行检测,达到要求后方可进行上机测序,不同文库按照目标数据量利用Illumina HiSeq平台进行测序。

图4 宏转录组测序技术分析微生物菌群Fig.4 Analysis of microbial community by macro-transcriptome sequencing technology
测序技术飞速发展,通过对食品中的微生物进行转录组测序,并对数据进行分析,能够明确发酵机理和菌种的代谢过程,从而对菌种进行筛选和优化提供重要的指导。近年来,宏转录组测序在食品行业渐渐崛起,食品科学界已广泛应用高通量测序技术来监测各种发酵食品(如豆酱[44],泡菜[45],酸马奶[46]等)发酵过程中微生物的多样性。ING M等[47-48]分别对茅台酒酒醅和食醋醋醅中微生物间的相互作用进行探究,为功能微生物研究奠定了基础。牟俊[49]则利用高通量测序方法重点分析了山西老陈醋醋酸发酵阶段乳酸菌和醋酸菌的组成和演替规律,并通过转录组学的方法对其相互作用机制进行初步解析,对深入理解山西老陈醋酿造机理具有重要意义。
转录组测序包括真核转录组测序和原核转录组测序,该技术通过快速获得某一物种特定的细胞或组织,在某一状态下的大部分的转录本及基因序列,对研究对象的基因结构和基因功能、可变剪接和新转录本预测等进行系统的研究[50]。该技术的应用也会受到一些因素的影响,如RNA的降解和RNA的起始量不足都会影响测序的质量[51],但随着分子生物学的进展,宏转录组测序的相关研究也会越来越深入。
5 近年来传统发酵食品中微生物的研究方法汇总
对近年来传统发酵食品菌群研究情况进行了汇总,如表1所示。基于新技术强大的破译复杂微生物群落的能力,以上技术已逐渐被人们应用于阐明不同种类传统发酵食品中微生物群落的组成和动态,取得了一系列重要的研究成果。

表1 近年传统发酵食品中微生物的研究方法汇总Table 1 Summary of research methods for microorganisms in traditional fermented foods in recent years
6 传统发酵食品中微生物的研究方法的优缺点对比
对传统发酵食品中微生物的研究方法(PCR-DGGE、RT-FQPCR、宏基因组测序、宏转录组测序)优缺点进行了归纳和对比,如表2所示,PCR-DGGE和RT-FQPCR的优势在于菌群种类的鉴定,而宏基因组测序和宏转录组测序则深入至具体的生物学功能,因此获得的生物学信息更加的深入和全面[43]。随着检测成本的日益降低,宏基因组相关的技术将成为未来研究的主流技术。

表2 传统发酵食品中微生物的四种分析方法的优缺点对比Table 2 Comparison of advantages and disadvantages of four research methods for microorganisms in traditional fermented foods in recent years
7 展望
传统发酵食品种类繁多,但是大部分仍沿用传统的生产工艺,其质量的稳定性与多种微生物密切相关,菌群的研究可以为发酵机理和代谢过程做出重要解析。但不同菌群和同一菌群之内基因组序列均有不同程度变化,从百分之几到千分之几不等,必须测定成千上万个体的基因组序列才能为传统发酵食品提供更加全面的依据,因此需要对测序的通量提高要求。随着生物过程的研究,高通量测序技术将逐渐与生物数据库、深度学习等智能化信息技术相结合,最终实现对传统发酵机制的全方位解析,推动传统发酵过程的现代化与智能化升级,同时不同代次的测序技术依然会长期共存和发展,力求通过各自的性能优势互补不足。

