鸡TNNI2 基因的真核表达载体构建及功能生物信息学分析*
李金炜,史洪岩,谭 茗,张杉杉,张伟伟,宫晓庆,夏靖秋,孙婴宁
(1.齐齐哈尔大学 生命科学与农林学院,黑龙江 齐齐哈尔 161000;2.北京市密云区农业农村局,北京 101500)
脊椎动物肌钙蛋白I (troponin I,TnI) 基因有3 个亚型,包括TNNI1、TNNI2和TNNI3。其中TNNI2编码快骨骼肌亚型肌钙蛋白I (fast skeletal muscle troponin I,fsTnI)[1]。人们最初认为:TNNI2基因只在肌肉中表达,参与调节肌肉收缩[2-3]。随后的研究发现:TNNI2基因在多种组织细胞中表达,并且发挥重要作用。TNNI2在软骨组织中表达,该基因编码的蛋白fsTnI 能够抑制软骨形成[4]。另外,在角膜上皮中也检测到TNNI2基因的表达[5]。在乳腺癌细胞中,fsTnI 蛋白定位于细胞核,主要作为雌激素相关受体α 的活化物来发挥功能[6]。fsTnI 蛋白在哺乳动物的骨骼和肌肉发育中发挥重要的作用,TNNI2基因的错译突变或缺失是导致远端关节病的重要因素[7]。在人胃癌组织中检测到TNNI2基因的表达,该基因可作为预测胃癌腹膜转移的特异性生物标志物,有助于对患者病理的监测[8]。
随着研究的不断深入,有关TNNI2的报道已经不仅限于生理学、运动学和临床医学等方面,对于TNNI2的研究趋向于农业领域,该基因对畜禽类肉品产量和肉质方面具有重要影响[9]。研究发现:TNNI2在牛臀肌中表达量最高,在幼猪的肱二头肌和咬肌表达中无明显差异[10-11]。PARK等[12]发现:TNNI2在猪、牛和鸡脂肪组织中表达,提示该基因在畜禽脂肪组织发育过程中可能发挥重要作用。肉鸡体脂(尤其是腹脂)积蓄过多是肉鸡产业的突出问题。肥胖会明显降低饲料转化效率,降低瘦肉对脂肪组织的比例,还会影响产蛋率、受精率和孵化率[13-14]。因此控制肉鸡的体脂积蓄,进一步提高饲料的转化效率是肉鸡行业急需解决的问题。本研究以鸡脂肪组织为试验材料,成功克隆TNNI2基因,构建TNNI2的真核表达载体,并对其进行功能生物信息学分析,可为TNNI2在鸡脂肪组织中的功能研究奠定基础。
1 材料与方法
1.1 材料
1.1.1 试剂
反转录试剂盒、定向克隆试剂盒、高保真酶和LipofectaminTM2000 转染试剂均购自南京Vazyme 公司,Trizol 试剂购于大连TaKaRa 公司,胶回收试剂盒和质粒小提试剂盒(离心柱型)均购自北京天根生化科技有限公司,pCMVHA 载体购自美国Clontech 生物公司,限制性核酸内切酶购自美国NEB 生物公司。Escherichia coliDH5α 菌种购自上海生工生物工程股份有限公司,细胞培养基D-MEM/F-12 和胰酶均购自Gibco 公司,胎牛血清购自Biological Industries公司;Western-blot 试剂购自Biosharp 公司,anti-HA 标签抗体购自美国CST 公司,β-actin 内参抗体购自上海碧云天生物技术有限公司,二抗购自美国LI-COR 公司。
1.1.2 试验动物和组织
爱拔益加(Arbor Acres,AA) 白羽肉鸡购自北京爱拔益加家禽育种有限公司。取AA 白羽肉鸡腹部脂肪组织(n=5),冻存于液氮中,直至提取RNA。
1.1.3 细胞系
永生化鸡前脂肪细胞系(immortalized chicken preadipocyte cell line,ICP1)由东北农业大学农业农村部鸡遗传育种重点实验室惠赠。ICP1 细胞系由该实验室利用带有鸡端粒酶逆转录酶(chTERT)基因的逆转录病毒感染鸡原代前脂肪细胞,而后通过药物筛选培养获得[15]。鸡前脂肪细胞使用添加10%胎牛血清(Biological Industries)和1%青霉素—链霉素溶液(Beyotime) 的DMEM/F12 培养基(Gibco),于37 ℃、5% CO2细胞培养箱中培养。
1.2 细胞转染
转染按照LipofectaminTM2000 转染试剂(Vazyme)说明书进行操作,待ICP1 细胞系汇合度至70% 时进行转染。使用无血清培养基分别稀释LipofectaminTM2000 转染试剂与质粒,用移液器轻轻混匀,室温静置5 min。将质粒—无血清培养基混合物滴加到LipofectaminTM2000 转染试剂—无血清培养基混合物中,用移液器轻轻混匀,室温静置10 min 后立即转染。转染6 h 后更换1 次培养基,48 h 后收集细胞。
1.3 提取总RNA 及反转录
采用Trizol 法分别提取AA 白羽肉鸡脂肪组织(5 只鸡的RNA 提取后混合)和ICP1 细胞系的总RNA。脂肪组织取100 mg,ICP1 细胞培养在6 孔板中,分别加入1 mL RNAiso Plus (Takara),充分裂解后12 000 r/min 离心5 min。将上清液转移至新的离心管中,加入200 μL 三氯甲烷,剧烈震荡混匀后置于冰上静置5 min,12 000 r/min离心15 min。将上清液转移至新的离心管中,加入与上清液体积相等的异丙醇,颠倒混匀后置于冰上静置10 min,12 000 r/min 离心10 min。弃上清,加入1 mL 75%乙醇清洗沉淀,7 500 r/min离心5 min。RNA 沉淀于室温干燥3 min,加入30 μL DEPC-H2O 溶解RNA。
反转录按照HiScript®II Q Select RT Super-Mix for qPCR (+gDNA wiper) (Vazyme)说明书进行操作。首先去除基因组的DNA,反应体系为:总RNA 0.5 μg,4×gDNA wipe Mix 2 μL,加入RNase free ddH2O,总体系为8 μL。上述体系用移液器轻轻吹打混匀,42 ℃ 2 min。反转录时向上述反应体系中直接加入5×HiScript®Ⅱ qRT SuperMix Ⅱ 2 μL,用移液器吹打均匀,25 ℃10 min,50 ℃ 30 min,85 ℃ 5 min,置于-20 ℃冰箱中保存待用。
1.4 PCR 扩增及纯化
根据NCBI (http://www.ncbi.nlm.nih.gov)数据库中的TNNI2(NM_205417.1) 基因序列设计引物。上游引物:5′-tggccatggaggcccGAATTCGCAAGATGTCTGATGAAGAGA-3′ (下划线表示EcoRI 酶切位点);下游引物:5′-gcggccgcggtac-AGCTCGGGAGAGACCAGTGCTTAGGAC-3′ (下划线表示XhoI 酶切位点)。以反转录得到的cDNA 为模板进行PCR 反应,反应体系为:DNA Buffer 25 μL,dNTP 1 μL,cDNA 5 μL,上、下游引物(10 μmol/L)各2 μL,ddH2O 14 μL,1 个活性单位的Taq酶。反应条件为95 ℃预变性3 min,而后进行35 个循环,每个循环包括95 ℃15 s,55 ℃ 15 s 和72 ℃ 5 min,最后4 ℃保存。将上述得到的PCR 产物进行1% 琼脂糖凝胶电泳,将得到的胶条放到紫外灯下观察,找到与预期结果最相近的条带进行切胶回收。
1.5 连接与转化
胶回收产物使用定向克隆试剂盒(Vazyme)克隆到pCMV-HA 载体的酶切位点上。操作流程为:用限制性核酸内切酶EcoRI 和XhoI 对pCMVHA 载体进行双酶切,PCR 产物5′和3′末端都含有能使目的片段被引入线性化克隆载体的同源臂(15~20 bp,引物序列中小写字母表示)。反应体系为:ddH2O 12 μL,5×CE MultiS Buffer 4 μL,线性化克隆载体1 μL,纯化后的扩增产物1 μL,ExnaseMultiS 2 μL,37 ℃反应30 min。将得到的连接产物转化到DH5α 感受态细胞中,涂布在具有氨苄青霉素抗性的固体LB 培养基上,37 ℃过夜。第2 天挑取单克隆菌落至摇菌管中,将摇菌管放置于摇床中,培养8 h 以上,用质粒小提试剂盒(TIANGEN)提取质粒。
1.6 双酶切验证与测序
将提取的质粒进行双酶切鉴定,反应体系为CutSmart Buffer 5 μL,质粒6 μL,XhoI 和EcoRI各1 μL,ddH2O 37 μL。反应条件为37 ℃ 2 h 后,65 ℃ 20 min。将双酶切鉴定正确的重组质粒对应的甘油菌液送至苏州金唯智公司进行测序,完成后将测序结果与NCBI 网站的预测序列进行对比。
1.7 RT-PCR
RT-PCR 反应按照ChamQTMSYBR®qPCR Master Mix (Without ROX) (Vazyme)说明书进行操作,TNNI2引物序列为F:5′-GCTGGGCTCCAAGCACAAG-3′ 和 R:5′-CCTCCAGTCACCCACATCG-3′。以无POU 域八聚体结合蛋白(non-POU domain containing octamer-binding protein,NONO)为内参照基因,NONO引物序列为F:5′-AGAAGCAGCAGCAAGAAC-3′ 和 R:5′-TCCTCCATCCTCCTCAGT-3′。反应体系:2×ChamQ SYBR qPCR Master Mix 10 μL,cDNA 模板2 μL,上、下游引物(10 μmol/L)各0.4 μL,ddH2O 7.2 μL。反应程序:95 °C 预变性30 s,95 °C 变性10 s 及55 °C 退火延伸30 s,共40 个循环。每个样品设置3 个重复,利用2-ΔΔCt算法将原始Ct值转换为基因相对表达量。
1.8 Western-blot
将鸡前脂肪细胞接种至6 孔板,分别转染pCMV-HA-TNNI2及pCMV-HA 质粒,转染48 h后,使用RIPA 裂解液(含1% PMSF) (Beyotime)提取细 胞中总蛋白,4 °C,12 000 r/min 离 心5 min,上清即为总蛋白,加入5×蛋白上样缓冲液(Beyotime) 煮沸5 min 后进行SDS-PAGE 电泳,电泳结束后将蛋白样品转移至NC 膜(Millipore),室温封闭1 h,anti-HA 标签抗体孵育1 h,二抗孵育1 h。Odyssey 双红外激光成像系统检测HA-fsTnI 蛋白表达水平。拍照完成后,采用一抗二抗洗脱液(Beyotime) 洗脱1 h,采用内参抗体anti-β-actin 孵育1 h,二抗孵育1 h,检测内参蛋白β-actin 的表达水平。
1.9 数据库与软件分析
采用在线网站ProParam (http://web.expasy.org/protparam/)分析蛋白质的氨基酸等电点和相对分子质量等理化性质。采用在线工具ProtScale(http://web.expasy.org/protscale/)分析蛋白质的疏水性。预测蛋白结构域采用SMART (http://smart.embl-heidelberg.de)在线网站。预测蛋白质三级结构采用Phyre 在线服务(http://www.sbg.bio.ic.ac.uk/phyre2/html)。分析蛋白质功能位点采用在线软件PROSITE (https://prosite.expasy.org/)。分析预测信号肽采用在线工具SignalP 5.0 Server(http://www.cbs.dtu.dk/services/SignalP/)。预测蛋白质跨膜区域采用TMHMM-2.0 (https://services.healthtech.dtu.dk/service.php?TMHMM-2.0)。亚细胞定位分析采用在线网站WoLF PSORT (https://wolfpsort.hgc.jp/)。序列对比采用在线工具Clustal Omega (https://www.ebi.ac.uk/Tools/msa/clustalo/)。利用MEGA 5.0 (https://www.megasoftware.net/index.php)工具,采用邻接法(neighbor-joining method,NJ 法)构建系统发育树,并采用Bootstrap 对系统发育树进行评估。所有分析参数设置均采用默认值。
2 结果与分析
2.1 TNNI2 基因扩增
以AA 白羽肉鸡的cDNA 为模板,进行PCR扩增,产物进行1%琼脂糖凝胶电泳检测。结果显示:在500~750 bp 位置成功扩增出1 条特异性条带(图1),与预期大小基本一致,可以用于后续载体连接。

图1 TNNI2 基因PCR 扩增结果Fig.1 PCR amplification results of TNNI2 gene
2.2 pCMV-HA 重组载体的鉴定
将pCMV-HA-TNNI2重组载体用EcoRI 和XhoI 双酶切鉴定,酶切产物经1%琼脂糖凝胶电泳,在500~700 bp 出现1 条特异性条带(图2),大小符合预期,提示TNNI2基因已插入到pCMV-HA载体上。采用BLAST 将测序结果与数据库中的序列进行比对,并与NCBI 数据库中鸡TNNI2基因预测序列对比,克隆序列在第45、84 位存在2 个同义替换,其他碱基完全一致(图3)。

图2 重组质粒pCMV-HA-TNNI2 的鉴定Fig.2 Identification of recombinant expression plasmid pCMV-HA-TNNI2

图3 鸡TNNI2 基因克隆序列与翻译的氨基酸序列Fig.3 The nucleotide sequence of the cloned gene and amino acids sequence of chicken TNNI2
2.3 重组质粒pCMV-HA-TNNI2 在ICP1 细胞中的mRNA 表达
为了确定TNNI2的过表达效果,将pCMVHA 空载体和pCMV-HA-TNNI2分别转染至ICP1细胞,48 h 后提取RNA 进行RT-PCR 检测,结果显示:转染pCMV-HA-TNNI2载体组的ICP1细胞TNNI2的mRNA 水平显著高于对照组(图4,P<0.01)。在mRNA 水平证明TNNI2过表达载体构建成功。
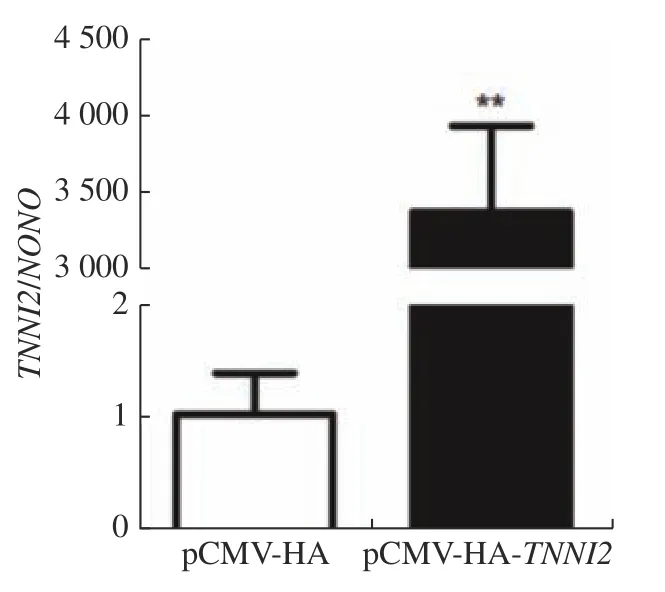
图4 qRT-PCR 检测鸡TNNI2 基因过表达效果Fig.4 Detection of overexpression of chicken TNNI2 gene by qRT-PCR
2.4 重组质粒pCMV-HA-TNNI2 在ICP1 细胞中的蛋白表达
为进一步确定fsTnI 蛋白在ICP1 细胞中的过表达效果,将pCMV-HA-TNNI2表达载体转染ICP1 细胞,48 h 后提取蛋白进行Western-blot 分析,由于pCMV-HA-TNNI2质粒N 端连有HA 标签,所以用HA 标签抗体检测重组蛋白的表达。结果显示:fsTnI 蛋白在细胞量一致的情况下(βactin 为内参),转染pCMV-HA-TNNI2质粒的ICP1细胞表达出1 条约23 ku 的特异蛋白条带,而转染pCMV-HA 空载体的细胞没有表达出23 ku 的特异带(图5),表明fsTnI 蛋白成功过表达。
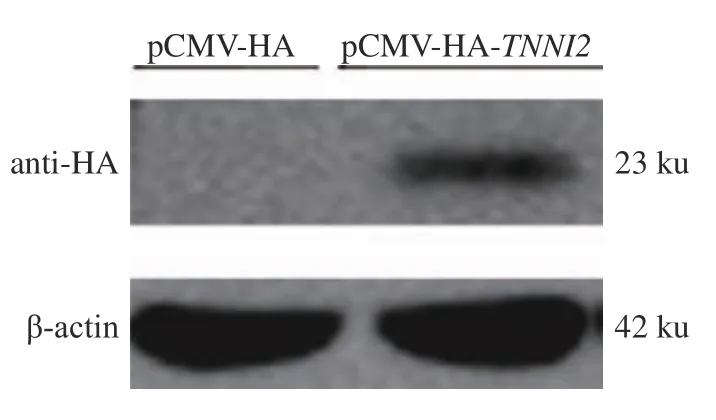
图5 Western-blot 检测鸡TNNI2 基因过表达效果Fig.5 Detection of chicken TNNI2 gene overexpression by Western-blot
2.5 生物信息学分析
2.5.1 鸡fsTnI 蛋白质一级结构信息
利用ProParam 对测序所得鸡TNNI2基因序列进行理化性质分析,结果显示:TNNI2基因编码183 个氨基酸,其中非极性疏水性氨基酸(G、A、F、P、V、I 和L) 54 个,占 29.5%;极性氨基酸(T、S、N、C、Q、Y、W 和M) 41 个,占22.4%;酸性氨基酸(D 和E) 35 个,占 19.2%;碱性氨基酸(K、R 和H) 45 个,占 24.6%。TNNI2基因编码的fsTnI 蛋白相对分子质量为21.24 ku,分子式为C913H1526N276O284S10,理论等电点值(pI)为9.19。精氨酸和赖氨酸残基总数为41,天冬氨酸和谷氨酸残基总数为35,脂肪系数为72.02,不稳定系数为65.62,属于不稳定性蛋白。
应用ProtScale 软件分析TNNI2编码的fsTnI蛋白亲疏水性,结果显示:fsTnI 蛋白质的疏水性在24 位点有最大值0.501,149 位点有最小值-2.903,平均亲水性(grand average of hydropathicity,GRAVY) 为-1.055。根据GRAVY 数值为负可以推测fsTnI 为亲水性蛋白。
2.5.2 鸡fsTnI 蛋白质结构域及三维结构预测
利用SMART 在线工具预测fsTNI 蛋白的三级结构,发现其在15~109 bp 存在1 个coiled coil 结构域。利用在线网站预测鸡TNNI2基因编码的fsTnI 蛋白三级结构。提交鸡fsTnI 蛋白的氨基酸序列,得到三级结构模型如图6 所示。有141 (77%的序列) 个残基根据100% 可信度的最高评分模板构建模型;有180 (98%的序列)个残基根据>90%的可信度的模板构建模型。
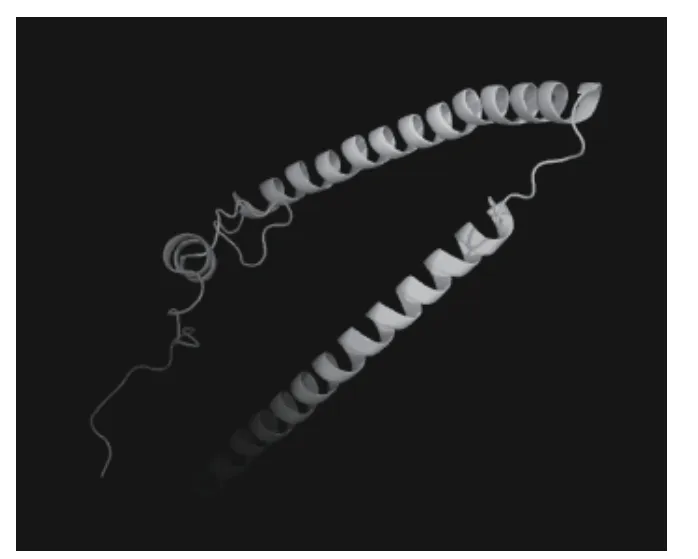
图6 fsTnI 蛋白的三级结构Fig.6 The tertiary structure of chicken fsTnI
2.5.3 鸡fsTnI 蛋白功能位点分析
利用Prosite 预测鸡fsTnI 蛋白的功能位点。发现鸡fsTnI 蛋白含有4 类功能活性位点,包括酪蛋白激酶II 磷酸化位点、蛋白激酶C 磷酸化位点、酪氨酸激酶磷酸化位点1 和酰胺化位点(表1)。

表1 fsTnI 蛋白质功能活性位点Tab.1 Fuctional active site of fsTnI protein
2.5.4 鸡fsTnI 蛋白质跨膜结构、信号肽及亚细胞定位
利用SignalP 5.0 预测TNNI2编码的fsTnI 蛋白的信号肽,结果显示:fsTnI 蛋白无信号肽。利用TMHMM 2.0 在线工具预测fsTnI 蛋白质的跨膜区,结果显示:TNNI2编码的fsTnI 蛋白不具有跨膜结构。通过在线网站WoLF PSORT 对fsTnI 蛋白进行亚细胞定位发现:fsTnI 蛋白定位于细胞核(nucl:21)。
2.5.5 鸡TNNI2基因同源性对比分析及系统发育树构建
将鸡TNNI2基因的编码区序列与鸭(XM_005019741.4)、犬(XM_022405520.1)、小鼠(NM_009405.3)、大鼠(NM_017185.1)、人(KR_7111-60.1)、猪(NM_001032359.1)、兔(NM_0010827-83.1)和羊(XM_027959866.1) 等9 个物种的序列对比并计算相似度(表2)。鸡与哺乳动物的相似性均在70%以上,与鸭的相似性为94.13%。利用MEGA 5.0 软件,采用 NJ 邻接法构建系统进化树,结果(图7)显示:鸡TNNI2基因的编码区与鸭在系统进化关系上最亲密,小鼠距离其他哺乳动物较远。基因进化比较符合物种进化关系。
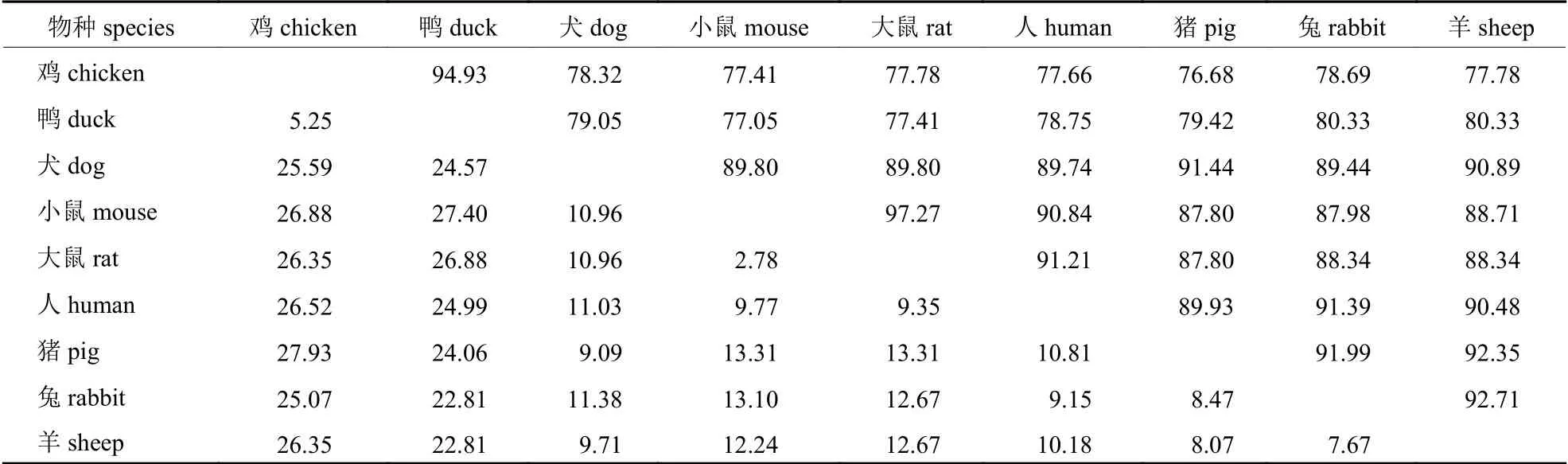
表2 9 个物种TNNI2 基因编码区序列相似性比对Tab.2 The percent identity analysis for TNNI2 gene coding region sequences of 9 species %
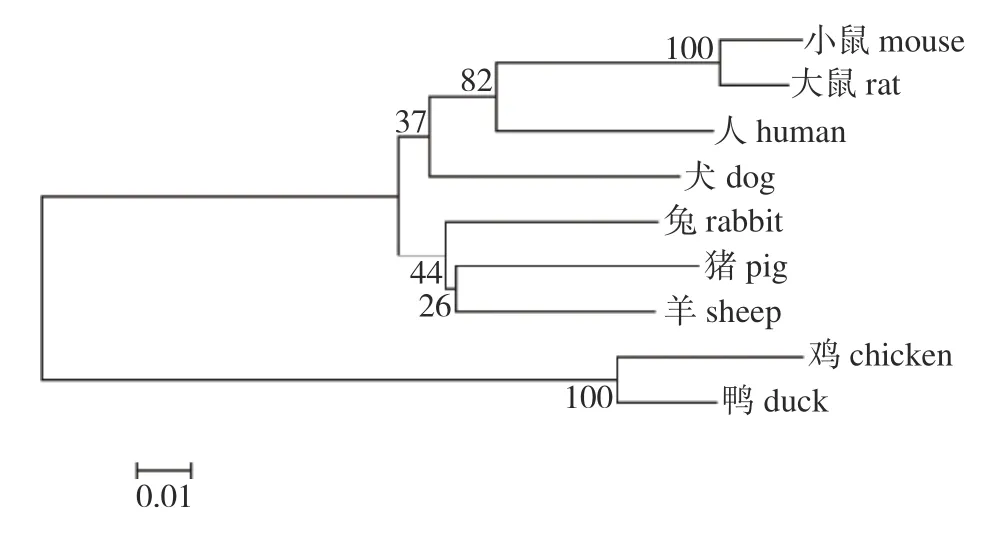
图7 TNNI2 基因系统发育树Fig.7 Phylogenetic tree based on TNNI2 gene
3 讨论
目前,肌钙蛋白已在人类[16-18]、鸭[19]、猪[1]、羊[20]和牛[10]等物种中成功克隆并开展研究。在鸡的脂肪组织中检测到TNNI2基因的表达[12],提示该基因在脂肪组织中发挥作用,但该基因在鸡脂肪组织中的结构和功能还不清楚。本研究通过PCR 扩增鸡的TNNI2基因,成功构建了真核过表达载体pCMV-HA-TNNI2,并在ICP1 细胞中进行了表达。
fsTnI 是一种快骨骼肌蛋白,属于肌钙蛋白I 基因家族,是包括肌钙蛋白T、肌钙蛋白C和肌钙蛋白I 亚基在内的肌钙蛋白复合体的1 个亚基[1]。肌钙蛋白复合物与原肌凝蛋白共同参与横纹肌收缩的钙依赖调节。fsTnI 也存在于血管平滑肌中,可能对平滑肌功能发挥起调节作用[2-3]。除了肌肉组织外,fsTnI 还存在于角膜上皮[5]和软骨中,在软骨中它是血管生成的抑制剂,以抑制肿瘤生长和转移[4]。此外,fsTnI 还作为核受体共同激活剂与人类乳腺癌细胞中的雌激素受体相关受体α (estrogen receptor-related receptor alpha,ERR α)相互作用[6],并作为转录因子通过增加Hif3a 的表达参与骨骼发育[21]。本研究亚细胞定位预测结果同样显示:在鸡前脂肪细胞系中,fsTnI 蛋白定位于细胞核,说明fsTnI 可能作为转录因子在脂肪组织中发挥作用。此外,TNNI2在脂肪组织中的表达结果显示:其mRNA在蛋鸡中的表达高于脂肪系肉鸡[22],可以推测fsTnI 蛋白在细胞核中作为转录因子对脂肪发育起到抑制作用。脂肪形成是一个复杂网络的调控过程,其中PPARγ 和C/EBPα 是脂肪形成过程中的2 个关键因子,其他的转录因子或多或少会通过影响PPARγ 和C/EBPα 的表达来影响脂肪形成[23-24]。fsTnI 可能通过影响这2 个关键因子的表达,或者在蛋白水平与PPARγ 或C/EBPα 形成复合物,来影响鸡前脂肪细胞分化。另外,fsTnI也有可能与其他转录因子形成蛋白水平的复合物,影响PPARγ 或C/EBPα 的表达,最后影响前脂肪细胞分化。总之,后续有必要开展fsTnI 在脂肪形成过程中的作用,进一步揭示fsTnI 的调控机制。
由同源性分析可知:鸡与鸭的TNNI2基因编码区差异性最小,只有28 个核苷酸有差异,鸡与哺乳类序列具有较高的相似性(均>70%),说明该基因在不同物种中可能具有相同的功能。另外,大鼠和小鼠的TNNI2基因编码区序列完全相同,说明同一基因在亲缘性相近的不同物种中具有保守性。综上所述,TNNI2基因在各物种中有较高的同源性。
4 结论
本研究成功构建了鸡TNNI2基因的真核表达载体pCMV-HA-TNNI2,在ICP1 细胞中进行了真核表达,同时对该基因序列进行了功能生物信息学预测。研究结果为TNNI2基因在鸡脂肪细胞中的进一步研究奠定基础。
致谢:感谢东北农业大学农业农村部鸡遗传育种重点实验室王宁教授提供永生化鸡前脂肪细胞系。

