重寄生拟盘多毛孢cr013菌株聚酮合酶基因的多样性分析*
孔磊,余进德,张登云,梅超,陈玉惠,李靖
(西南林业大学 生命科学学院,云南 昆明 650224)
拟盘多毛孢属(Pestalotiopsis) 真菌具有产丰富次生代谢产物的巨大潜力,迄今为止,国内外学者已从拟盘多毛孢属中分离得到了多种具有丰富活性的化合物,如生物碱类、异香豆素衍生物、香豆素类、萜类、醌类、半醌类等,是产生新的活性化合物的重要来源[1-2]。前期实验表明重寄生拟盘多毛孢(Pestalotiopsissp.)cr013菌株具有产新型聚酮类化合物的巨大潜力[3],且不同于内生拟盘多毛孢产生的化合物[4]。
聚酮合酶(polyketide synthase,PKS)是催化合成具有抗细菌、抗肿瘤、抗真菌等活性的聚酮类化合物的关键酶[5],与脂肪酸合成有相同的前体——乙酸,两者的生物合成过程相似,其共同的生物合成途径称为乙酸途径[6]。乙酸单元先被硫代酯化反应活化为乙酰辅酶A(起始单元)和丙二酰辅酶A(延长单元),酰基转移酶(acyltransferase,AT)结构域将乙酰辅酶A(acetyl Coenzyme A)的酰基转移到酰基载体蛋白(acyl carrier protein,ACP),ACP将起始单元传递到酮合酶(ketosynthase,KS)结构域,并可用于接收AT催化的扩链单元丙二酰辅酶A,最后通过一系列还原、氧化等反应,完成合成[7]。按照结构域的不同,PKS可以分为I型、II型和III型PKS,其中对I型PKS的研究较多。I型PKS分为模块化PKS(细菌)和迭代PKS(真菌),模块化PKS将它们的域排列在按顺序操作的模块中,而迭代PKS只有一组域,在每次迭代中重复使用。迭代PKS根据序列中的β位置是否存在裁剪域分为3类:(1)非还原型PKS(non-reducing PKS,NR-PKS),无还原裁剪域,其中PT是其特有的结构域[8];(2)高度还原型PKS(highly-reducing,HR-PKS)均含KR和ER;(3)部分还原型PKS(partial-reducing,PR-PKS),含酮基还原酶(ketoreductase,KR),但不含烯酰还原酶(enoylreductase,ER)[9]。
近几年来,高通量技术发展迅速,基因组测序成本降低,利用基因组挖掘更多潜在的新天然化合物,且利用基因组挖掘方法结合分析工具能够促进新化合物的发现,再通过生物信息学分析可预测潜在基因编码的产物[10]。目前,已有很多科研人员利用生物信息学挖掘基因组,获得了更多的PKS基因[11-15]。2011年,Bunet等[11]通过基因组挖掘,揭示了Streptomycesambofaciens中有10余个基因簇参与了次级代谢产物的合成,其中alp基因簇参与了II型聚酮类化合物Kinamycin的生物合成;2019年,原晓龙等[12]从蛹虫草(Cordycepsmilitaris)的基因组中,通过生物信息学分析,挖掘到14个PKS基因后,对其蛋白序列进行结构域和聚类分析来推测其功能;2020年该课题组以相同的方法从球孢白僵菌中发现了13个PKS基因,也对获得的PKS基因的结构域和功能进行了推测[13];János等[14]也通过系统发育方法对PKS蛋白序列中的结构域进行了排列和分析,以此来推测Aspergillus属中PKS基因的功能;Preetida等[15]对曲霉属(Aspergillussp.)全基因组测序,从5株曲霉菌中获得21~34个PKS基因。本研究首次对重生拟盘多毛孢cr013菌株全基因中的PKS基因组进行了挖掘,挖掘了cr013菌株基因组中的PKS基因,对其进行生物信息学分析,推测其可能的功能,通过实时荧光定量PCR(real-time quantitative PCR,qPCR)的方法,检测PKS基因在改良Fries培养基上5个不同时间段的表达情况,以此为重寄生拟盘多毛孢PKS基因的开发利用及其还原型聚酮类化合物的生物合成机制的阐明奠定理论基础和数据支持。
1 材料与方法
1.1 微生物材料
cr013菌株分离自感病的华山松(Pinusarmandii)枝干的茶藨生柱锈菌(Cronartiumribicola)锈孢子堆[16],现保存于西南林业大学生物化学教研室。
1.2 方法
1.2.1 菌株培养
挑取规格约为0.5 cm的cr013菌块接种到改良Fries培养基上(0.13 g CaCl2·2H2O,1.0 g KH2PO4,5.0 g 酒石酸铵,0.5 g MgSO4·7H2O,20.0 g 蔗糖,0.1 g NaCl,1.0 g 酵母浸膏,20.0 g琼脂,1.0 g NH4NO3, 1 000 mL蒸馏水;自然pH5.7),放置室温分别培养4、8、12、16及20 d,并收集5个不同培养时间的菌丝体,迅速放置-80 ℃冰箱保存,以供后续qPCR实验使用。
1.2.2 聚酮合酶基因的挖掘
获得cr013菌株基因组后,利用8个注释数据库(NR、KEGG、COG、SwissProt、GO、ARDB、PHI、CAZy)对该基因组中基因的功能进行注释,最后共有94%的蛋白序列注释到功能。通过生物信息学分析注释到的数据,获得cr013菌株基因组中所有完整的mRNA序列及蛋白序列。以结构和功能已知的同属PestalotiopsisficiW106-1[17](登录号:W3X7U2.1;A0A067XNI2.1)的PKS蛋白的KS保守结构域蛋白质序列为模板,对cr013菌株的基因组进行搜索,挖掘cr013菌株基因组中潜在的PKS基因,并利用Conversed Domain Database数据库分析挖掘到的PKS蛋白的结构域。
1.2.3 PKS蛋白的聚类分析
从NCBI上下载已知功能的高度还原性PKS、部分还原PKS和非还原PKS的蛋白质序列作为参考序列,以此构建系统进化树。通过Clustal W对挖掘到的28条PKS蛋白序列与所选择的57条真菌PKS的蛋白序列进行比对,用MEGA 7.0中的邻接法(neighbor-joining,NJ)构建系统进化树,自检举重复1 000次,其余参数默认。利用生物信息学分析对cr013菌株基因组挖掘到的28条PKS基因进行分类和推测其功能。
1.2.4PKS基因的表达分析
根据本课题组前期研究,cr013菌株中分离到的4种新聚酮类化合物均从改良Fries培养基分离得到的[3]。所以将cr013菌株在改良Fries培养基上室温下分别培养4、8、12、16及20 d后,总RNA的提取采用UNIQ-10柱式Trizol 总RNA抽提试剂盒,再反转录成cDNA。根据序列比对结果,分别从非还原型PKS和还原型PKS中分别选择相似性最高,且结构和功能已知的基因来进行表达分析。cr013菌株基因组获得非还原型crPKS3基因和还原型crPKS12基因的全长mRNA序列,利用Primer 5.0软件分别在两条PKS基因的保守KS片段设计特异性引物(crPKS3-F:CAAGGTGCTGTTGATGTTCC,crPKS3-R:CTGCAATGCGTTTCCTGTTC;crPKS12-F:CCGTGATTCGGACACCATT,crPKS12-R:GACTCTGACACGCCAAATGC),用设计的特异性引物检测PKS基因在5个不同时间段的表达情况。
2 结果与分析
2.1 cr013菌株基因组中PKS基因的挖掘
以真菌PKS蛋白的KS保守结构域蛋白质序列为模板,对cr013菌株基因组(基因登录号:GCA_018092615.1)进行扫描后,结果表明有28个PKS基因(编号:crPKS1~28,基因登录号:MW373474~MW373501)。利用BLASTp将获得的28个PKS基因一一进行比对,利用Conversed Domain Database分析其结构域。按照结构域组成的不同,将28个PKS基因分为3类:NR-PKS、PR-PKS和HR-PKS。其中包括8个NR-PKS(crPKS1~8);4个PR-PKS(crPKS9~10、crPKS17、crPKS19);14个HR-PKS(crPKS11~13、crPKS15~16、crPKS18、crPKS20~27);2个杂合NRPS/PKS(crPKS14、crPKS28)均为PR-PKS与NRPS的杂合,结果见表1。

表1 cr013菌株中的28个PKS基因Tab.1 28 PKS genes in the cr013 strain
通过将cr013菌株中的PKS蛋白序列的相似性和结构域组成与其他真菌的PKS蛋白序列相比,发现crPKS1与Xylariasp.BCC 1067(登录号:AAM93545.1)中编码黑色素生物合成的PKS蛋白序列有77.89%的一致性;crPKS3与PestalotiopsisficiW106-1(登录号:A0A067XNI2.1)中参与pestheic acid生物合成的PKS蛋白序列有87.20%的一致性;crPKS4和crPKS8分别与Daldiniachildiae(登录号:XP_033440273.1)和Aspergillusudagawae(登录号:GFG26526.1)中编码分生孢子黄色素生物合成的PKS蛋白序列有69.62%和56.50%的一致性;crPKS10和crPKS25分别与Fusariumproliferatum(登录号:RKL45057.1)和Fusariumoxysporum(登录号:RKK90944.1)中编码美伐他汀生物合成的PKS蛋白序列有53.01%和44.80%的一致性;crPKS11与Aspergilluslentulus(登录号:GFF99174.1)中编码洛伐他汀二酮合酶的LovF基因的蛋白质序列有47.51%的一致性;crPKS12与Daldiniachildiae(登录号:XP_033438860.1)中编码洛伐他汀二酮合酶的LovF基因的蛋白质序列有71.61%的一致性;crPKS13与Talaromycespinophilus(登录号:KAF3392115.1)中编码洛伐他汀二酮合酶的mokB基因的蛋白质序列有47.02%的一致性;crPKS16与Fusariumproliferatum(登录号:RKL38412.1)中编码洛伐他汀二酮合酶的mokB基因的蛋白质序列有60.44%的一致性;crPKS19与Valsamali(登录号:KUI66310.1)中编码洛伐他汀二酮合酶的LovF基因的蛋白质序列有50.00%的一致性;crPKS22与Alternariatenuissima(登录号:RYN89545.1)中编码洛伐他汀二酮合酶生物合成的的蛋白质序列有53.13%的一致性;crPKS23与Trichodermalentiforme(登录号:KAF3074072.1)中编码洛伐他汀二酮合酶的mokB基因的蛋白质序列有64.40%的一致性;crPKS26与Madurellamycetomatis(登录号:KXX82669.1)中编码洛伐他汀二酮合酶的LovF基因的蛋白质序列有56.08%的一致性;crPKS20与Lachnellulaarida(登录号:TVY19100.1)中编码富马霉素生物合成的蛋白质序列有65.22%的一致性;crPKS28与Trichodermaarundinaceum(登录号:RFU77043.1)中编码洛伐他汀九酮合酶生物合成的蛋白质序列有54.48%的一致性;crPKS2、crPKS5、 crPKS6、 crPKS7、crPKS9、crPKS14、crPKS15、crPKS17和crPKS18这9个PKS的终产物尚未知。
2.2 PKS蛋白序列聚类分析
将cr013菌株基因组中的PKS蛋白序列与NCBI中相似性较高、功能或化合物已知的PKS蛋白序列进行聚类分析,由系统进化树(图1)推测crPKS10、crPKS25 、crPKS27蛋白序列可能参与美伐他汀(compactin)的生物合成;crPKS19、crPKS12、crPKS26、crPKS13、crPKS11、crPKS23、crPKS28蛋白序列可能催化洛伐他汀(lovastatin)的生物合成;crPKS3蛋白序列可能参与pestheic acid的生物合成;crPKS1蛋白序列可能参与黑色素(melanin)的生物合成;crPKS4、crPKS8蛋白序列可能参与黄色分生孢子色素(conidial yellow pigment)的生物合成;crPKS20蛋白序列可能参与富马霉素(fumagillin)的生物合成;而crPKS24、crPKS15、crPKS18、crPKS22、crPKS21、crPKS16、crPKS9、crPKS14、crPKS17、crPKS6、crPKS7、crPKS5、crPKS2蛋白序列均与未知功能的聚酮类化合物聚在一起,其催化的化合物未知。
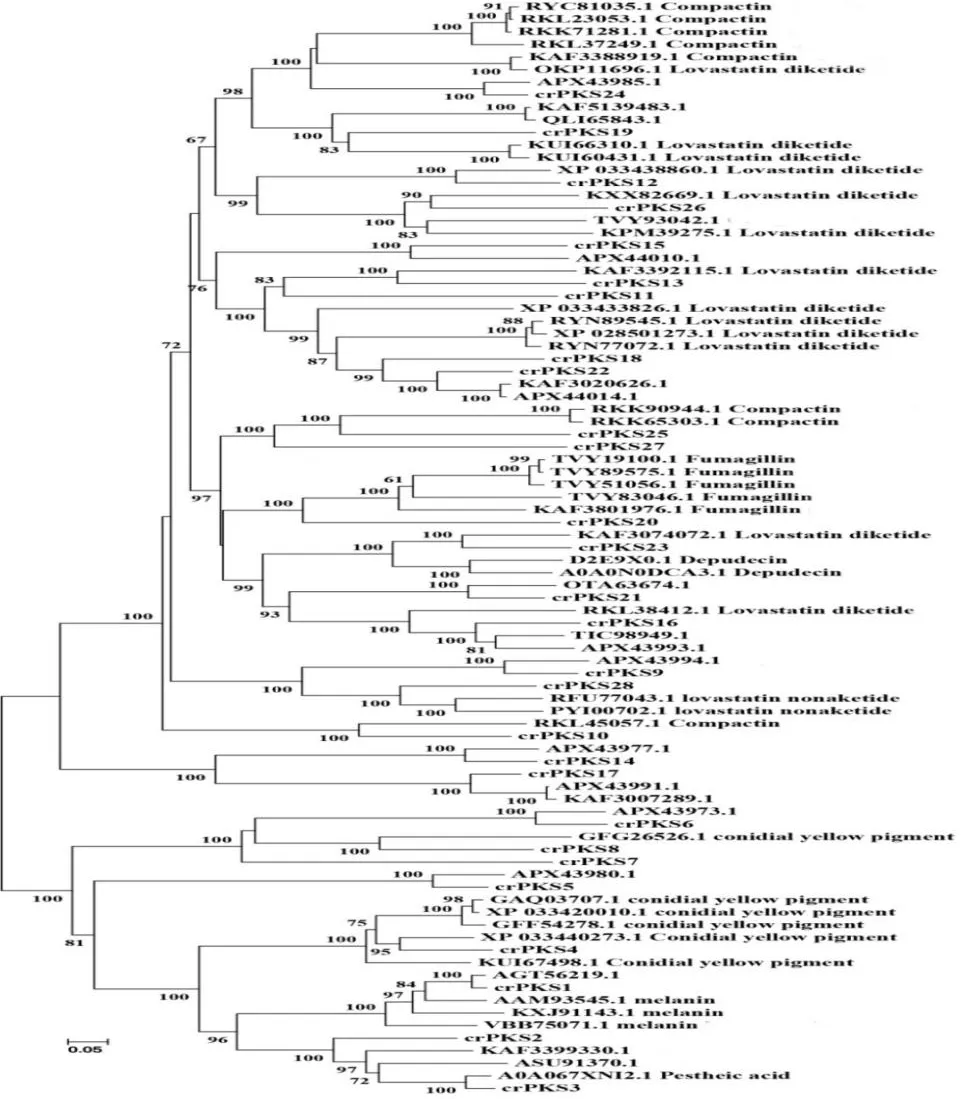
图1 cr013菌株中的PKS与其他相关真菌PKS蛋白的聚类比较Fig.1 The cluster comparison of the PKS in cr013 strain and other PKS proteins of related fungus
2.3 PKS基因的表达分析
本课题组前期研究表明,cr013菌株中分离到的4种新还原型聚酮类化合物均从改良Fries培养基分离得到[3]。所以将cr013菌株在改良Fries培养基上室温下分别培养4、8、12、16及20 d后,提取RNA,反转录成cDNA,并检测特异性引物的有效性。图2结果显示,非还原型crPKS3和高度还原型crPKS12基因的溶解曲线都是单峰,证明这两条目的基因的引物均为特异性扩增。
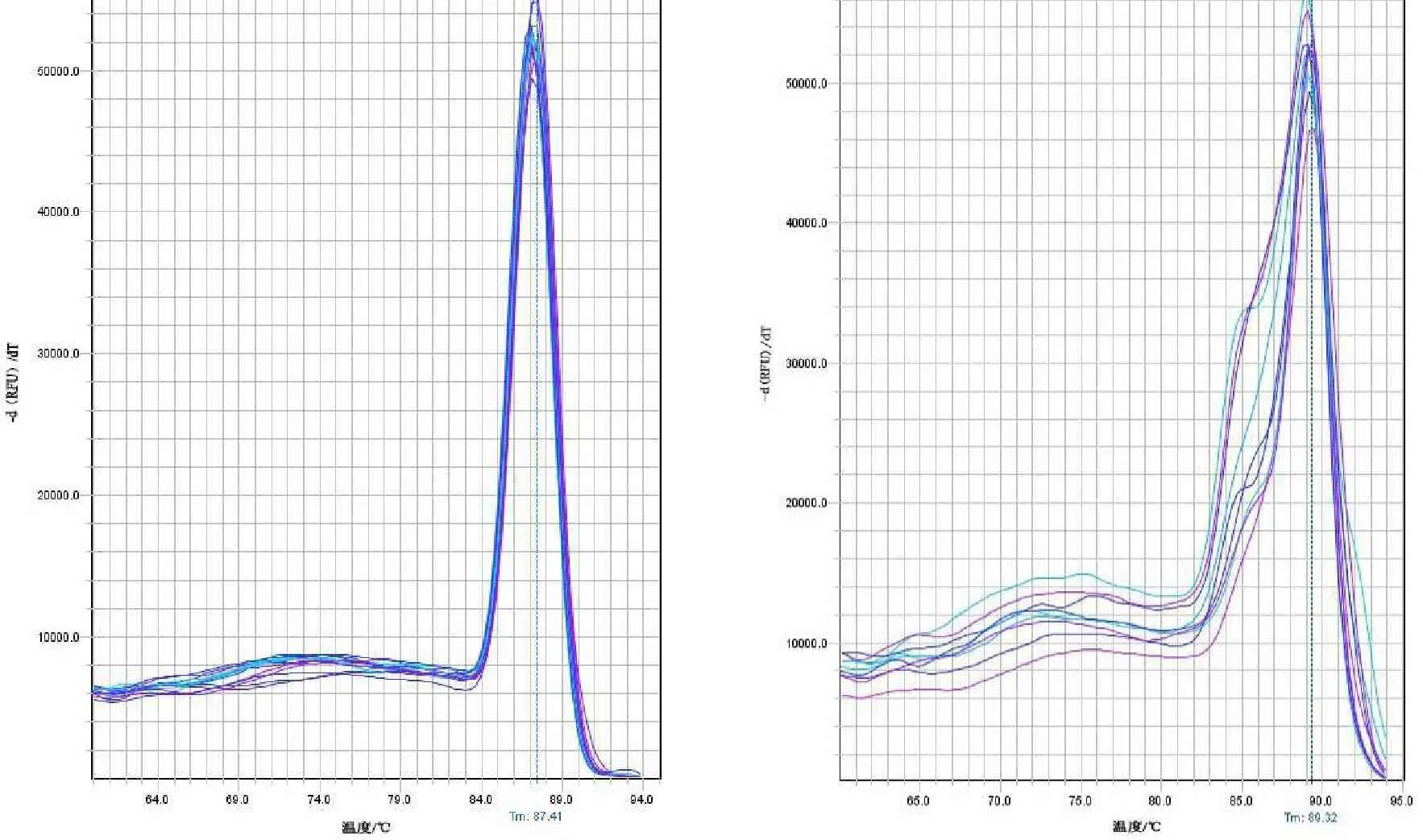
图2 检测crPKS3和crPKS12基因特异性引物的有效性
由图3结果可知,非还原型聚酮crPKS3和还原型聚酮crPKS12基因在5个不同时间段均可表达,且基因表达量差异较大。两条PKS基因均在改良Fries培养基培养第16 d时,基因表达量最高,但crPKS12的表达量为21.72,crPKS3的表达量为12.19,crPKS12的表达量远高于crPKS3。

图3 crPKS3 (左)和crPKS12(右)基因在5个不同培养时间的相对表达量Fig.3 Relative expression of crPKS3 (left)and crPKS12 (right)genes in five different culture time
3 结论与讨论
重寄生拟盘多毛孢产生的次级代谢产物区别于内生拟盘多毛孢(Pestalotiopsissp.)真菌。本课题组前期从该重寄生拟盘多毛孢真菌中分离鉴定了4个还原型聚酮类化合物,具有较强的抗肿瘤活性,该聚酮类化合物为拟盘多毛孢属真菌中首次被发现[3]。
为获得更多新颖的聚酮合酶基因,本研究通过基因组挖掘[20]和生物信息学分析,挖掘到28个PKS基因,包括14个HR-PKS、4个PR-PKS、8个NR-PKS和2个杂合NRPS/PKS,说明重寄生拟盘多毛孢cr013菌株中PKS基因多样高,也证明了该菌株具有产PKS的巨大潜力。基于基因进化过程,根据蛋白质序列相似性来推测未知功能与已知功能蛋白质序列的同源关系,以此推测未知PKS基因的功能。研究结果显示,重寄生拟盘多毛孢cr013菌株中有14条PKS蛋白序列聚类到了已知功能的分支,产生了6种可能的聚酮类化合物,其中他汀类物质最为丰富,3条PKS蛋白质序列可能参与美伐他汀或其类似物的生物合成,7条PKS蛋白质序列可能参与洛伐他汀或其类似物的生物合成。
目前,他汀类药物是重要的降脂药物,主要包括洛伐他汀、美伐他汀等药物[21]。1976年,Brown等[22]从Penicilliumbrevicompactum中首次分离到了美伐他汀,这也是第一个他汀类物质,同时也是普伐他汀和辛伐他汀的直接转化底物,其遵循聚酮生物合成途径,属于聚酮类化合物,具有十分重要的市场地位,能够为他汀类的药物发展提供原料和底物[23]。洛伐他汀是一类天然且具有重要价值的聚酮类化合物,目前,还未见通过合成方法获得大量洛伐他汀的研究报道,探讨利用发酵方式来获得大量他汀类物质具有重要的意义。关于洛伐他汀的生物合成的主要来源是土曲霉(Aspergillusterreus)和红曲霉(Monascuspurpureus),尚未发现从重寄生拟盘多毛孢真菌中分离获得他汀类物质。优化发酵工艺、优化培养基成分等均有助于提高洛伐他汀的产量[24]。在后续研究中,将进一步筛选和优化其培养条件,为新活性化合物的发现及其应用奠定理论基础。

