基于转录组测序的芒果过敏原基因鉴定及表达分析
闫慧清 吴宗敏 田旭 杨兆云 黄绒


摘要:【目的】基于芒果果实和叶片的转录组测序结果,筛选出潜在过敏原基因,并分析其在不同组织中的表达模式,为选育低过敏性的芒果品种提供理论依据。【方法】提取芒果果实和叶片总RNA,构建cDNA文库,利用Illumina HiSeq 4000进行转录组测序(测序读长为150 bp),从原始数据中筛选获得Clean reads,并利用Trinity进行组装,得到Unigenes,将其提交至NR数据库和Pfam数据库进行功能注释,从而筛选潜在的过敏原基因。通过实时荧光定量PCR (qRT-PCR)測定过敏原基因在不同组织中的表达情况。通过Cluspro分析果实中高表达过敏原与IgE抗体结合的表位区域。【结果】经组装获得230242条Transcripts和99328条Unigenes,二者的N50分别为2278和1183 bp,说明组装完整性较好。通过NR数据库和Pfam数据库共筛选到66个潜在的芒果过敏原基因,主要被注释为花粉过敏原、Bet v家族和NADPH-依赖FMN还原酶等,其中有6个不表达,其余60个均表达,其中有24个主要在果实中表达,其他36个基因在叶片中高表达。qRT-PCR检测结果显示,9个过敏原基因在芒果叶片和果实中表达量检测结果与RNA-Seq转录组表达分析结果基本相符;除几丁质酶基因c64821.graph_c0在叶片中表达量显著高于花和果实(P<0.05,下同)外,其他过敏原基因在花中的表达量显著高于叶片和果实。在RCSB PDB数据库搜索获得在果实中高表达的过敏原c61327.graph_c0的同源蛋白结构域(PDB:4Z8W),其与IgE结合表位的氨基酸序列为74-SFRQEVKTEKHGEFKVHLPFSVSEHV-99。不同物种过敏原蛋白的结合表位氨基酸序列具有高度保守性。【结论】不同芒果过敏原基因具有组织表达特异性,大部分基因在花中高表达,说明对食用芒果后产生过敏反应的人群应避免触摸芒果花等组织。c61327.graph_c0与其他物种的同源过敏原产生交叉过敏反应。
关键词: 芒果; 过敏原; 转录组; 基因鉴定; 表达分析
中图分类号: S667.703.6 文献标志码: A 文章编号:2095-1191(2021)07-1771-09
Identification and expression analysis of mango allergens according to transcriptome sequencing
YAN Hui-qing, WU Zong-min, TIAN Xu, YANG Zhao-yun, HUANG Rong
(School of Life Science,Guizhou Normal University,Guiyang 550025,China)
Abstract:【Objective】The potential allergen genes were determined and the expression profiles in different tissues were evaluated using transcriptome sequencing of mango leaves and fruits to provide a theoretical basis for breeding lower allergenic mango cultivars. 【Method】The total RNA was extracted from mango fruits and leaves. Transcriptome sequen-cing(150 bp reads) using HiSeq 4000 was performed after the cDNA libraries construction. Unigenes were obtained with Trinity assemble of clean reads. The unigenes related to potential allergen genes were annotated using NR and Pfam databases. The expression levels of allergen genes in different tissues were determined using real - time fluorescence quantitative PCR(qRT-PCR). The epitopes of the allergen highly expressed in fruits combined with IgE antibody were obtained by Cluspro analysis. 【Result】A total of 230242 transcripts and 99328 unigenes were obtained. The N50 lengths of transcripts and unigenes were 2278 and 1183 individually,indicating the high assembly quality. Identified 66 potential allergen genes using NR and Pfam databases,including pollen allergens, Bet v family and NADPH-dependent FMN reductase. Six allergens showed no expression, 60 showed expression. There were 24 allergens and 36 allergens highly expressed in fruits and leaves,respectively. The qRT-PCR test showed that the expression of nine allergen genes in mango leaves and fruits was largely consistent with the RNA-Seq transcriptome expression analysis. Except that the expression of chitinase gene c64821.graph_c0 in leaves was significantly higher than flowers and fruits(P<0.05,the same below),the others were significantly expressed in flowers than leaves and fruits. The allergen c61327. graph_c0 highly expressed in fruit was used as the template to obtain homologous protein domain(PDB:4Z8W) by searching in the RCSB PDB database, the amino acid sequences combined with IgE epitope was 74-SFRQEVKTEKHGEFKVHLPFSVSEHV-99. The amino acid sequences of binding epitopes of allergen protein from different species were highly conserved. 【Conclusion】Different mango allergen genes have tissue expression specificity. Most genes are highly expressed in flowers,suggesting that people with allergic reactions after eating mango should avoid touching mango flowers. C61327.graph_c0 has cross-allergenic reactions with homologous allergens of other species.
Key words: Mangifera indica Linn; allergens; transcriptome; gene identification; expression analysis
Foundation item: National Natural Science Foundation of China(31660554); Guizhou Joint Fund Project(Qinkehe LH〔2015〕7772)
0 引言
【研究意義】芒果(Mangifera indica Linn)属漆树科,是我国热带和亚热带重要的水果,分布在广西、海南和贵州等省(区)(Wu et al.,2012),其果实营养丰富,具有抗菌和抗癌作用(Noratto et al.,2010)。除鲜食外,芒果果实还可加工食用(Fasoli and Righe-tti,2013)。由于芒果中含有大量过敏原,不同芒果品种中过敏原的种类差异不明显,部分人群在接触或食用芒果后均会产生超敏反应(Kinder et al.,1999)。芒果引起的过敏反应不容忽视,其高发生率不仅与芒果的过敏原密切相关,还与其他食物或吸入性过敏原产生交叉反应有关。芒果的过敏原与特异性B细胞产生的IgE抗体Fc段受体I(IgE-Fc reception 1)结合形成复合体,再结合肥大细胞和嗜碱性粒细胞,导致机体处于过敏状态(Tsujimura et al.,2008)。由于相似的表位结构域具有相同的免疫活性,因此芒果的过敏原与桦树花粉、开心果、可可和木瓜等均有交叉反应(谷冬梅和李会强,2015)。通过转录组全面系统地评价芒果中潜在过敏原及转录水平的表达模式,并通过同源比对得到芒果与其他物种过敏原交叉反应的物质基础,分析主要过敏原与IgE抗体结合的过敏表位,对选育低过敏性的芒果品种及防止交叉过敏现象具有重要意义。【前人研究进展】Wellhausen等(1996)通过IgE检测方法首次从芒果中鉴定出分子量为35 kD的过敏原蛋白,且与桦树花粉过敏原Bet v 1的蛋白氨基酸序列同源性较高,可产生交叉反应。Funes等(1999)通过IgE检测方法从芒果的果肉和果皮粗提取物中鉴定出不同分子量的9个过敏原蛋白。Renner等(2008)利用芒果过敏病人的血清在芒果果肉中鉴定获得分子量为27 kD的过敏原蛋白。Tsai等(2017)通过Allergome数据库(http://www.allergome.org/index.php)搜索获得已报道的13个芒果过敏原,主要包括Man I 1、Man I 2、Man I 3和几丁质酶,前3种过敏原分别属于核糖体蛋白、NADH质体醌氧化还原酶亚基和细胞色素c血红素附着蛋白。其中,重组Man I 1已在大肠杆菌中表达纯化,可用于芒果过敏反应的免疫治疗。几丁质酶是体内防御真菌和病虫害的植物保护酶,其在人体中的含量可作为哮喘人群血清的检测指标(Goldman et al.,2012),表明几丁质酶具有免疫学特性。此外,芒果的过敏原还包括抑制蛋白(Profilin)类(闫慧清等,2017)。该类抑制蛋白主要存在于植物的花粉中,可结合骨架蛋白并控制肌动蛋白聚合从而产生口腔过敏反应综合症(Song et al.,2008),表明抑制蛋白也具有免疫学特性。芒果中的前纤维蛋白与苹果中的过敏原Mal d 4具有较高的同源性且两者的空间构象极相似,表明两者极易发生交叉反应(夏宏林等,2012)。随着基因组学的发展,Illumina测序技术为探究芒果基因的功能和表达提供了较好的平台。如Wu等(2014)以芒果品种Zill的果肉和果皮为研究材料,通过转录租测序共组装得到54000个转录本;Dautt-Castro等(2015)对未成熟和成熟芒果果实进行转录组测序,挖掘软化相关基因并分析其他营养特性的差异;Hoang等(2015)通过Illumina测序技术测定分析芒果在成熟过程中的生理过程、功能成分、采后管理和果实品质的变化;Zhao等(2017)通过Illumina测序技术分析得到菊科植物豚草(Ambrosia artemisiifolia)花粉中的过敏原。【本研究切入点】目前未见有关利用Illumina测序技术鉴定芒果中过敏原基因及其表达分析的研究报道。【拟解决的关键问题】通过Illumina测序技术鉴定得到芒果中过敏原基因并分析其转录水平的表达模式,通过实时荧光定量PCR(qRT-PCR)检测过敏原基因在叶片、花和果实中的表达量,并对果实中高表达过敏原进行表位分析,为培育低致敏性芒果品种提供理论依据。
1 材料和方法
1. 1 试验材料
供试芒果品种为台农1号。RNA提取试剂盒、反转录试剂盒、SYBR[?]Premix Ex TaqTMⅡ试剂盒及其他分子试剂均购自宝生物工程(大连)有限公司。主要仪器设备:Aglient 2100生物分析仪(美国)、Illumina HiSeq 4000(San Diego,CA,USA)和实时荧光定量PCR仪(Qiagen,Rotor-Gene Q2 PLex)。
1. 2 样品采集和RNA提取
采集芒果果实和叶片,立即置于液氮中冷冻,机械研磨成细粉,-80 ℃保存;使用RNA纯化试剂盒分离芒果果实和叶片的总RNA。首先,采用Oligo(dT)的磁珠从总RNA中分离出poly(A) mRNA,随机打断mRNA片段,并以这些打断的k-mer片段为模板合成并纯化cDNA后,连接接头。从中选择合适的片段进行PCR扩增从而构建cDNA文库,采用Agilent 2100生物分析仪对该文库进行定量鉴定。
1. 3 转录组测序及数据组装
采用Illumina HiSeq 4000进行转录组测序,测序读长为150 bp,委托北京百迈客生物科技有限公司完成。从原始数据中滤除杂质,包括5%以上未知碱基的reads和低质量reads(含50%以上Q值≤10碱基的reads),最终筛选获得Clean reads。使用Trinity组装数据,将获得的所有Unigenes提交至NR(NCBI non-redundant protein database,http://www. ncbi.nlm.nih.gov/)和Pfam(http://pfam.xfam.org/)数据库进行序列比对及功能注释。
1. 4 过敏原基因筛选及其表达分析
基于Unigenes在NR和Pfam数据库中的比对及注释结果,筛选出叶片和果实中与过敏原相关的所有基因,并对过敏原进行描述。基于RNA-Seq转录组数据,采用FPKM(Fragments per kilobase million)方法计算各过敏原基因的表达水平。
采用Trizol试剂提取芒果花、叶片和芒果果实的总RNA,并利用反转录PCR(RT-PCR)试剂盒和oligo dT-adaptor引物反转录合成cDNA,用1%琼脂糖凝胶电泳检测。以cDNA为模板,采用FastStart DNA Master SYBR Green I试剂盒进行qRT-PCR检测。过敏原基因和内参基因β-actin的引物如表1所示。扩增程序:95 ℃预变性30 s;95 ℃ 30 s,60 ℃ 30 s,74 ℃ 1 min,进行30个循环。设3次生物学重复。采用2?△△Ct方法计算过敏原基因的相对表达量,并利用SPSS 22.0进行方差分析。
1. 5 过敏原蛋白的蛋白质结构分析和过敏表位预测
根据筛选出的过敏原基因,通过ORF(https://www.ncbi.nlm.nih.gov/orffinder/)推导出过敏原的氨基酸序列,并通过PDB数据库(http://www.rcsb.org/pdb/home/home.do)搜索过敏原的蛋白结构域。使用Cluspro进行蛋白—蛋白对接,根据结构簇得分选择装配体三维结构,得到能量较低的大簇对接结构(E=0.40Erep+?0.40Eatt+600Eelec+1.00EDARS)(Kozakov et al.,2013)。利用Discovery studio 4.5获得对接蛋白序列(Swellmeen et al.,2017),并将其提交至ESPript 3.0(http://espript.ibcp.fr/ESPript/ESPript/)进行序列比对。
2 结果与分析
2. 1 芒果转录组数据分析结果
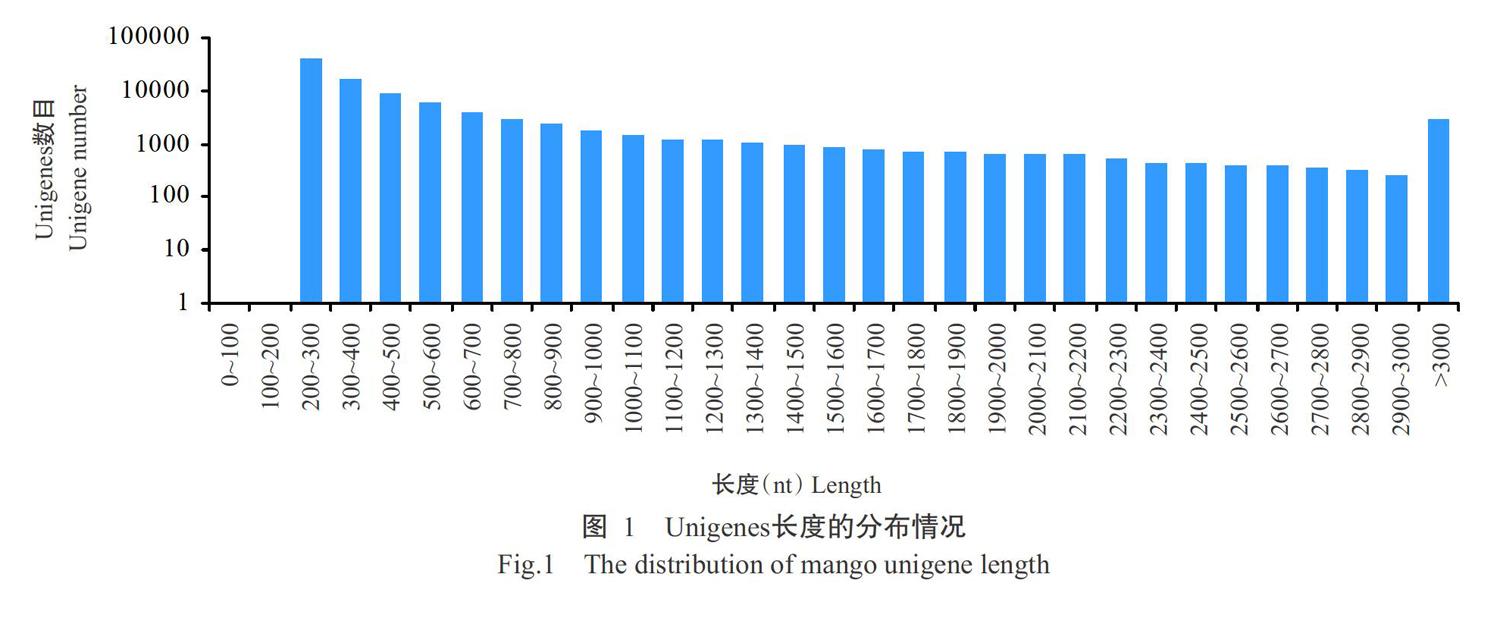
芒果果实和叶片的转录组数据已提交到NCBI的GEO数据库(No. GSE142427)。基于转录组测序结果,经过滤除杂后获得17.63 Gb Clean reads,各样品Clean reads均达8.36 Gb,Q30碱基百分比在94.58%及以上,共生成11751123个Contig,组装共得到230242条Transcripts和99328条Unigenes,二者的N50分别为2278和1183 bp,说明组装完整性较好。由图1可知,长度为200~300 bp、300~500 bp、500~1000 bp、1000~2000 bp和>2000 bp的Unigenes所占比例分别为39.75%、26.16%、17.13%、9.59%和7.37%,平均长度为680.53 bp。
2. 2 芒果果实和叶片中过敏原基因的鉴定结果
利用NR数据库和Pfam数据库对47949个Unigene进行注释,结果表明46303个(96.57%)均在NR和Pfam数据库中注释成功,通过功能注释信息,从中共筛选出66个潜在的过敏原基因,如表2所示。根据过敏原种类不同,66个过敏原基因大致可分为三大类,第一类为花粉过敏原,主要为扩展类(Expansin-like)蛋白;第二类为Bet v家族,包括致敏异黄酮还原性蛋白Bet v 6.0102(c88876.graph_c0和c82555.graph_c0)、Bet v I家族及其他病程相关蛋白,其中,Bet v I家族成员c45493.graph_c0、c51367.graph_c0、c94579.graph_c0和c40982.graph_c0,均被注释为过敏原Pru ar 1-like;第三类包括NADPH-依赖FMN还原酶、Ana o 2、Can a 1、Mal d 1、Hsp90/Hsp、Pis v 2.0201、Cla h和醛脱氢酶家族等。
2. 3 转录组表达分析结果
基于RNA-Seq转录组表达分析结果(图2)显示,66个过敏原基因中,有6个不表达,其余60个均表达,其中有24个主要在果实中表达,其他36个基因在叶片中高表达。根据FPKM值,将66个过敏原基因分为四大类,第一类有6个基因,在果实和叶片中均不表达;第二类有12个基因,仅在果实中高表达;第三类有14个基因,仅在叶片中高表达;第四类有34个基因,在果实和叶片中均表达,但存在明显差异。尤其是c61327.graph_c0、c49299.graph_c0和c44502.graph_c0基因在果实中非常高,说明对这3种过敏原过敏的人群应避免食用芒果果實。
2. 4 过敏原在不同组织中的表达分析结果
选取在芒果叶或果实中高表达的9个过敏原基因,采用qRT-PCR检测其在花、叶和果实中的表达量,结果如图3所示。c44502.graph_c0、c49299.graph_c0、c61297.graph_c0、c17261.graph_c0和c61327.graph_ c0基因在果实中的表达量高于叶片,其他基因在叶片中的表达高于果实,与上述RNA-Seq转录组表达分析结果基本相符。除c64821.graph_c0基因在叶片中表达量显著高于花和果实(P<0.05,下同)外,其他过敏原基因在花中的表达量显著高于叶片和果实。
2. 5 过敏原蛋白结构分析结果
以芒果果实中高表达的过敏原c61327.graph_c0为模板,在RCSB PDB数据库搜索获得同源蛋白结构域(PDB:4Z8W),并通过Cluspro进行c61327.graph_c0和IgE-Fc模拟对接,获得过敏原蛋白结合表位的氨基酸序列(74-SFRQEVKTEKHGEFK-VHL PFSVSEHV-99)(图4-A)。通过NCBI的BLASTp搜索其他物种的同源序列,结果发现c61327.graph_c0与可可树(Theobroma cacao)中的花粉过敏原Ole e1(GenBank登录号EOY03810.1)高度同源,氨基酸序列相似性达71%(图4-B),与土瓶草(Cephalotus follicularis)的花粉过敏原Ole e1(GenBank登录号GAV60102.1)和黄麻(Corchorus capsularis)的花粉过敏原Ole e1(GenBank登录号OMP03537.1)的氨基酸序列相似性分别是62%和49%。不同物种过敏原蛋白结合表位的氨基酸序列(图4-B中阴影部分)具有高度保守性,表明芒果过敏原c61327.graph_c0与其他物种的同源过敏原序列产生交叉过敏反应。
3 讨论
Illumina测序技术已用于葡萄(Tu et al.,2016)、甜橙(Hu et al.,2016)和菠萝(Sharma et al.,2017)等多种物种果实中功能物质基因的研究及挖掘。本研究经芒果叶片和果实的转录组测序分析,共获得17.63 Gb的Clean reads,组装后获得99328条Unigenes,其中,长度为1 kb以上的Unigenes有16848条。与武红霞等(2016)将6.16 Gb的Clean reads组装后获得54207条Unigenes相比,本研究组装得到的Unigenes数量较多,可更好地覆盖基因组信息。本研究发现,96.57%的Unigene在NR和Pfam数据库中比对成功,推测其余3.43%的转录本是由于基因组数据库注释信息的限制和芒果EST信息的缺乏而无法进行功能注释。此外,部分Unigenes被注释为“假设性蛋白”“预测性蛋白”和“假定性蛋白”,表明仍需通过进一步的分子生物学实验验证其生物学功能。
本研究将组装得到的Unigenes在Pfam和NR数据库中进行比对及注释,共鉴定出66个过敏原基因,其编码蛋白分别为花粉过敏原、致病相关蛋白Bet v I家族蛋白、抑制蛋白、几丁质酶和过敏原Alt a 1等。其中,花粉过敏原的扩展蛋白是芒果的主要过敏原,存在于许多物种的花粉中,被认为是广泛的过敏原,能引起食物和花粉之间的交叉反应;芒果过敏原致病相关蛋白Bet v I家族和几丁质酶参与植物对真菌和细菌的防御(Diaz-Perales et al.,1999);芒果过敏原抑制蛋白是一种肌动蛋白的结合蛋白,通过结合肌动蛋白而防止高等植物细胞骨架的形成(Song et al.,2008),在芒果的花粉中表达量较高且具有较高的免疫活性。芒果过敏原Alt是一种NADPH-依赖FMN还原酶,与过敏原性密切相关,推测其在芒果过敏的免疫治疗中具有潜在的应用价值(Moreno et al.,2016;Gabriel et al.,2017)。目前一些高表达的过敏原可使用IgE方法进行检测,并利用体外重组进行纯化,用于免疫治療(吴序栎等,2010)。
虽然芒果叶片和果实存在许多过敏原,但其表达水平不尽相同。本研究采用qRT-PCR检测9个过敏原基因在3种不同组织中的表达情况,其中在果实和叶片的检测结果与RNA-Seq转录组表达分析结果基本相符;除几丁质酶基因c64821.graph_c0在叶片中表达量显著高于花和果实外,其他过敏原基因均在花中的表达量显著高于果实和叶片,表明对食用芒果后产生过敏反应的人群应避免触摸芒果的花组织。此外,本研究通过对芒果果实中高表达过敏原c61327.graph_c0进行同源比较及表位分析,推测芒果过敏原c61327.graph_c0与其他物种的同源过敏原产生交叉过敏反应。此前也有研究表明不同物种间的过敏原蛋白氨基酸序列高度同源,且过敏原结合表位具有相同的免疫活性(Song et al.,2008),如对芒果花粉过敏的人群,也对白桦树和木瓜的产生过敏反应(Renner et al. 2008)。今后应基于转录组测序,对本研究获得芒果的潜在过敏原进行体外表达和纯化等研究,并通过基因工程手技术减低过敏原基因的表达或培育低过敏的芒果品种。
4 结论
不同芒果过敏原基因具有组织表达特异性,大部分基因在花中高表达,说明对食用芒果后产生过敏反应的人群应避免触摸芒果花等组织。c61327.graph_c0与其他物种的同源过敏原产生交叉过敏反应。
参考文献:
谷冬梅,李会强. 2015. 由IgE介导的(速发型)芒果过敏及其过敏原的研究进展[J]. 中国免疫学杂志,(6):849-851. doi:10.3969/j.issn.1000-484X.2015.06.031. [Gu D M,Li H Q. 2015. The progress of mango immediate hypersensitive mediated by IgE and allergens[J]. Chinese Journal of Immunology,(6):849-851.]
武红霞,许文天,罗纯,姚全胜,王松标,马小卫,詹儒林. 2016. 芒果果实转录组数据组装及基因功能注释[J]. 热带作物学报,37 (11):2191-2198. doi:10.3969/j.issn.1000- 2561.2016.11.025. [Wu H X,Xu W T,Luo C,Yao Q S,Wang S B,Ma X W,Zhan R L. 2016. Transcriptome data assembly and gene function annotation of mango fruits[J]. Chinese Journal of Tropical Crops,37(11):2191-2198.]
吴序栎,张红云,刘志刚,王琳琳,邓志琼. 2010. 芒果过敏的研究进展[J]. 中国食品卫生杂志, 22(2):185-187. [Wu X L,Zhang H Y,Liu Z G,Wang L L,Deng Z Q. 2010. Progress on the study of mango allergy[J]. Chinese Journal of Food Hygiene,22(2):185-187.]
夏宏林,何颖,邹泽红,陶爱林. 2012. 苹果过敏原Mal d4 蛋白抗原表位预测及交叉反应分析[J]. 中国免疫学杂志,28(1):57-61. doi:10.3969/j.issn.1000-484X.2012.01.013. [Xia H L,He Y,Zou Z H,Tao A L. 2012. Prediction of antigenic epitopes and cross-reactivity of apple allergen Mal d4[J]. Chinese Journal of Immunology,28(1):57-61.]
閆慧清,黄小龙,马兆成. 2017. 食用芒果过敏的研究进展[J]. 食品科学,38(3):305-309. doi:10.7506/spkx1002-6630-201703048. [Yan H Q,Huang X L,Ma Z C. 2017. Progress in understanding hypersensitivity reaction after ingestion of mango fruits[J]. Food Science,38(3):305-309.]
Dautt-Castro M,Ochoa-Leyva A,Contreras-Vergara C A,Pacheco-Sanchez M A,Casas-Flores S,Sanchez-Flores A,Kuhn D N,Islas-Osuna M A. 2015. Mango(Mangifera indica L.) cv. Kent fruit mesocarp de novo transcriptome assembly identifies gene families important for ripening[J]. Front Plant Science,6:62-73. doi:10.3389/fpls.2015. 00062.
Diaz-Perales A,Collada C,Blanco C,Sanchez-Monge R,Carrillo T,Aragoncillo C,Salcedo G. 1999. Cross-reactions in the latex-fruit syndrome:A relevant role of chitinases but not of complex asparagine-linked glycans[J]. Journal of Allergy and Clinical Immunology,104(3Pt1):681-687. doi:10.1016/s0091-6749(99)70342-8.
Fasoli E,Righetti P G. 2013. The peel and pulp of mango fruit:A proteomic samba[J]. Biochimica Biophysica Acta,1834(12):2539-2545. doi:10.1016/j.bbapap.2013.09. 004.
Funes E,Milan J M,Lopez J D,Pagán J A,López J D,García F J,Negro J,Hernández J,Polo F,Rico P. 1999. Allergy to Anarcadiaceae. Identification of allergens[J]. Allergo-logy Immunology Clinical,14(2):82-89.
Gabriel M F,Uriel N,Teifoori F,Postigo I,Sunen E,Martinez J. 2017. The major Alternaria alternata allergen,Alt a 1:A reliable and specific marker of fungal contamination in citrus fruits[J]. International Journal of Food Microbio-logy,257:26-30. doi:10.1016/j.ijfoodmicro.2017.06.006.
Goldman D L,Li X,Tsirilakis K,Andrade C,Casadevall A,Vicencio A G. 2012. Increased chitinase expression and fungal-specific antibodies in the bronchoalveolar lavage fluid of asthmatic children[J]. Clinical and Experimental Allergy,42(4):523-530. doi:10.1111/j.1365-2222.2011. 03886.x.
Hoang V L,Innes D J,Shaw P N,Monteith G R,Gidley M J,Dietzgen R G. 2015. Sequence diversity and differential expression of major phenylpropanoid-flavonoid biosynthetic genes among three mango varieties[J]. BMC Genomics,16:561-572. doi:10.1186/s12864-015-1784-x.
Hu Y,Duan S,Zhang Y Z,Shantharaj D,Jones J B,Wang N. 2016. Temporal Transcription Profiling of Sweet Orange in Response to PthA4-Mediated Xanthomonas citri subsp. citri Infection[J]. Phytopathology,106(5):442-451. doi:10.1094/PHYTO-09-15-0201-R.
Kinder H,Scharf B,Steinhart H,Raschke A. 1999. Investigation of IgE-binding patterns and allergenicity of mango fruit allergens in different varieties[J]. Internet Symposium on Food Allergens,1(2):43-49.
Kozakov D,Beglov D,Bohnuud T,Mottarella S E,Xia B,Hall D R,Vajda S. 2013. How good is automated protein docking?[J]. Proteins,81(12):2159-2166. doi:10.1002/prot. 24403
Moreno A,Pineda F,Alcover J,Rodriguez D,Palacios R,Martinez-Naves E. 2016. Orthologous Allergens and Diagnostic Utility of Major Allergen Alt a 1[J]. Allergy Asthma Immunology Research,8(5):428-437. doi:10.4168/aair. 2016.8.5.428.
Noratto G D,Bertoldi M C,Krenek K,Talcott S T,Stringheta P C,Mertens-Talcott S U. 2010. Anticarcinogenic effects of polyphenolics from mango(Mangifera indica) varieties[J]. Journal of Agricultural and Food Chemistry,58(7):4104-4112. doi:10.1021/jf903161g.
Renner R,Hipler C,Treudler R,Harth W,Suss A,Simon J C. 2008. Identification of a 27 kDa protein in patients with anaphylactic reactions to mango[J]. Journal of Investigational Allergology and Clinical Immunology,18(6):476-481. doi:10.1089/jir.2007.0142.
Sharma A,Wai C M,Ming R,Yu Q Y. 2017. Diurnal cycling transcription factors of pineapple revealed by genome-wide annotation and global transcriptomic analysis[J]. Genome Biology Evolution,9(9):2170-2190. doi:10.1093/ gbe/evx161.
Song J,Zhang H,Liu Z,Ran P. 2008. Mango profilin:Clo-ning,expression,and cross-reactivity with birch pollen profilin Bet v 2[J]. Molecular Biology Report,35(2):231-237. doi:10.1007/s11033-007-9075-5.
Swellmeen L,Shahin R,Al-Hiari Y,Alamiri A,Shaheen O. 2017. Structure based drug design of Pim-1 kinase fo-llowed by pharmacophore guided synthesis of quinolone-based inhibitors[J]. Bioorganic and Medicinal Chemistry,25(17):4855-4875. doi:10.1016/j.bmc.2017.07.036.
Tsai W C,Wu T C,Chiang B L,Wen H W. 2017. Cloning,expression,and purification of recombinant major mango allergen Man i 1 in Escherichia coli[J]. Protein Expression and Purification,130:35-43. doi:10.1016/j.pep.2016. 06.009.
Tsujimura Y,Obata K,Mukai K,Shindou H, Yoshida M,Nishikado H,Kawano Y,Minegishi Y,Shimizu T,Karasuyama H. 2008. Basophils play a pivotal role in immunoglobulin-G-mediated but not immunoglobulin-E-me-diated systemic anaphylaxis[J]. Immunity,28(4):581-589. doi:10.1016/j.immuni.2008.02.008.
Tu M X,Wang X H,Feng T Y,Sun X M,Wang Y Q,Huang L,Gao M,Wang Y J,Wang X P. 2016. Expression of a grape(Vitis vinifera) bZIP transcription factor,VlbZIP36,in Arabidopsis thaliana confers tolerance of drought stress during seed germination and seedling establishment[J]. Plant Science,252:311-23. doi:10.1016/j.plantsci.2016. 08.011.
Wellhausen A,Schoning B,Petersen A,Vieths S. 1996. IgE binding to a new cross-reactive structure:A 35 kDa protein in birch pollen,exotic fruit and other plant foods[J]. Zeitschrift für Ern?hrungswiss,35(4):348-355. doi:10. 1007/BF01610553.
Wu H X,Jia H M,Ma X W,Wang S B,Yao Q S,Xu W T,Zhou Y G,Gao Z S,Zhan R L. 2014. Transcriptome and proteomic analysis of mango(Mangifera indica Linn) fruits[J]. Journal of Proteomics,105:19-30. doi:10.1016/j. jprot.2014.03.030.
Wu T C,Tsai T C,Huang C F,Chang F Y,Lin C C,Huang I F,Chu C H,Lau B H,Wu L,Peng H J,Tang R B. 2012. Prevalence of food allergy in Taiwan:A questionnaire-based survey[J]. International Medicine Journal,42(12):130-135. doi:10.1111/j.1445-5994.2012.02820.x.
Zhao F,Durner J,Winkler J B,Traidl-Hoffmann C,Strom T M,Ernst D,Frank U. 2017. Pollen of common ragweed(Ambrosia artemisiifolia L.):Illumina-based de novo sequencing and differential transcript expression upon elevated NO2/O3[J]. Environmental Pollution,224:503-514. doi:10.1016/j.envpol.2017.02.032.
(責任编辑 陈 燕)

