小麦BES1基因家族的比较基因组学分析
姜玉梅,姜如云,李俊畅,杨进威,牛吉山,李 磊
(1. 河南农业大学农学院,河南郑州 450046; 2. 河南农业大学国家小麦工程技术研究中心,河南郑州 450046)
BES1基因家族是植物特有的调节油菜素内酯(brassinosteroids,BRs)信号通路的重要转录因子,可通过调节BRs信号传导的代谢通路发挥作用[1-3]。研究表明,BRs是一种重要的植物内源激素,影响多种生物学进程,比如植物茎的伸长、叶片发育、花粉管生长、木质部分化、衰老、光形态发生、胁迫响应等[4],其在生长发育以及胁迫应答过程中的生理活性远高于其他植物激素[5]。Sahni等[6]研究发现,BES1主要通过激活下游基因的转录,调控BRs靶基因的表达,最终调控植物的生长发育和抗逆能力。Yang等[7]研究发现,在拟南芥中,BES1通过调节BRs的生物合成,抑制防御素和硫苷生物合成基因的表达,对植物防御反应起促进作用。BES1在BRs信号通路中上调表达,不仅可以调控靶基因的表达并促进茎的伸长[1],还可以抑制黄酮类化合物的生物合成,使植物能够及时从生长状态转换为响应紫外线胁迫的状态[8]。小麦(TriticumaestivumL.)是一种重要的粮食作物,其生长由于BRs的保护,可免受各种逆境胁迫的冲击,包括低温、高温、盐度、干旱、组织损伤、病原体入侵、昆虫攻击等[9-12]。截止目前,BES1基因家族已经在拟南芥[13]、油菜[14]、棉花[15]、玉米[16]、番茄[17]、茶树[18]等作物中得到鉴定,但对禾本科作物中BES1基因家族的鉴定还相对较少。因此,研究BES1基因家族成员在小麦中的功能就显得更为重要。
近年来,小麦以及其他禾本科作物的基因组序列陆续发布,为进一步研究BES1基因在小麦以及其他禾本科作物中的功能和进化关系奠定了基础。本研究对小麦等6种禾本科作物和十字花科模式植物拟南芥在系统发育、基因结构和共线性关系等方面进行了全基因组比较分析,同时进一步探讨了小麦BES1基因家族成员在小麦不同组织(器官)的表达模式,以期为进一步探索BES1基因家族成员的功能及其进化情况奠定基础。
1 材料与方法
1.1 BES1基因家族的鉴定
从植物参考基因组数据库Ensembl Plants(http://plants.ensembl.org/)下载小麦、大麦、水稻、玉米、高粱、谷子(6种常见的禾本科作物)以及拟南芥(十字花科模式植物)的基因组数据,包括基因组序列文件和蛋白序列文件。通过Pfam网站(http://pfam.xfam.org/)下载BES1蛋白保守结构域(PF05687)的隐马尔可夫模型,利用HMMER网站(https://www.ebi.ac.uk/Tools/hmmer/)进行序列比对,阈值设置为1×10-5,每条序列保留其对应的最长转录本序列作为最终结果,删除冗余蛋白序列,获得各个物种的BES1家族成员。对得到的BES1家族成员用SMART网站(http://smart.embl.de/index2.cgi)进行蛋白保守结构域的鉴定确认,对验证通过的小麦BES1家族成员利用Expasy网站(https://web.expasy.org/compute_pi/)对其氨基酸序列的分子质量、等电点等理化性质进行分析。
1.2 系统发育分析
利用MEGA 7.0软件[19]内置的CluatalW工具对小麦、大麦、水稻、玉米、高粱和谷子BES1基因家族成员的氨基酸序列进行比对分析。使用邻近法(neighbor-joining)构建系统发育树,bootstrap参数为1 000。得到的进化树利用iTOL网站(https://itol.embl.de/)进行美化。
1.3 基因结构分析
从SMART网站获得小麦BES1基因家族成员的蛋白保守结构域的位置信息。从基因组注释文件中提取小麦BES1基因家族成员的内含子、外显子、染色体位置等信息。利用TBtools软件[20]进行氨基酸序列保守结构域和对应核酸序列基因结构的可视化。
1.4 染色体定位和共线性分析
根据下载的小麦基因组数据库中的物理位置信息,利用R语言包Circos将所有BES1家族成员的基因定位到小麦染色体上。采用多重共线性扫描工具MCScan X对基因重复事件进行分析,用TBtools软件内置的Multiple Systeny Plot绘图工具进行物种间共线性分析。
1.5 小麦BES1基因家族的表达分析
从小麦基因表达谱网站Wheat Expression Browser (http://www.wheat-expression.com/)获取BES1家族成员在小麦不同组织(器官)中的表达量数据,基因表达水平以TPM(transcripts per million)为单位,利用TBtools软件绘制表达量热图。
2 结果与分析
2.1 小麦和6个代表性植物BES1基因家族成员的鉴定结果
利用从Pfam数据库中下载得到的隐马尔可夫模型,对小麦基因组数据进行序列比对,以e-value<1×10-5作为筛选标准,在小麦中共鉴定到20个BES1基因家族成员,按照小麦BES1基因家族成员在染色体上的位置信息对其进行命名(表1)。除TaBES1-U位于未知染色体外,其他家族成员分布在14条染色体上。小麦BES1基因家族成员编码蛋白氨基酸长度为178~686 aa,分子量为19.27~75.48 kDa,等电点为5.4~9.4。
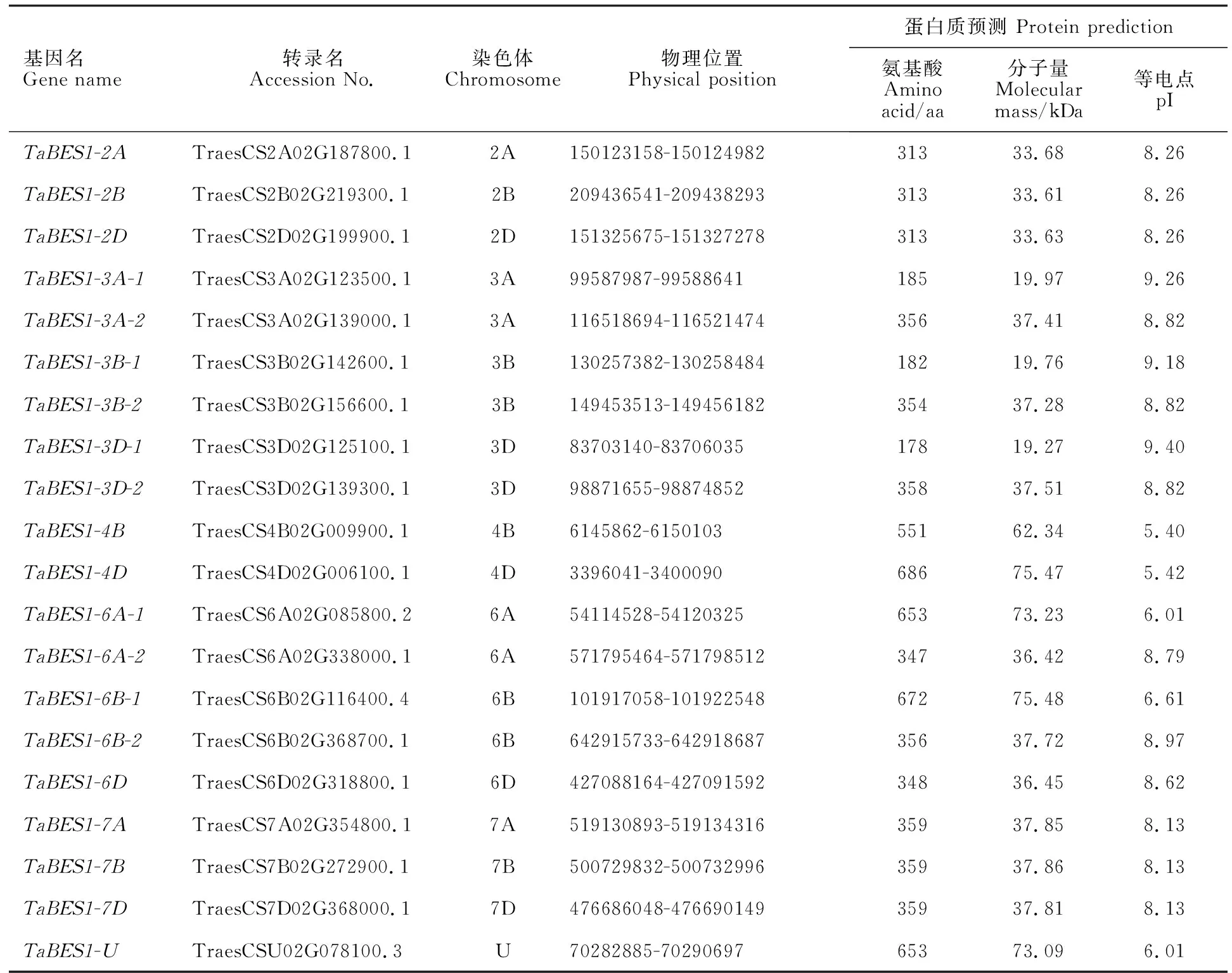
表1 小麦BES1基因家族成员Table 1 BES1 gene family in wheat
在水稻、玉米、高粱、谷子、大麦和拟南芥中分别鉴定到6、11、10、11、9和8个BES1基因家族成员。可以看出,水稻的BES1基因家族成员数最少,小麦的最多。
2.2 小麦和6个代表性植物BES1基因家族的系统发育分析
为了研究小麦、水稻、玉米、高粱、谷子、大麦和拟南芥中BES1基因的进化关系,利用这7个物种BES1家族基因的蛋白序列构建系统发育树。根据ClustalW比对结果,去掉进化距离较远的一个谷子基因KQL10972,对其他74个BES1基因家族成员利用MEGA 7.0软件构建系统发育树(图1),根据拟南芥BES1家族成员的分类,同时结合不同禾本科作物的进化关系,将74个BES1基因家族成员划分为Group A、Group B、Group C、Group D四大类,分别包含31、11、26和6个家族成员。小麦和6个代表性植物BES1基因家族成员在四个大类中的分布见表2。
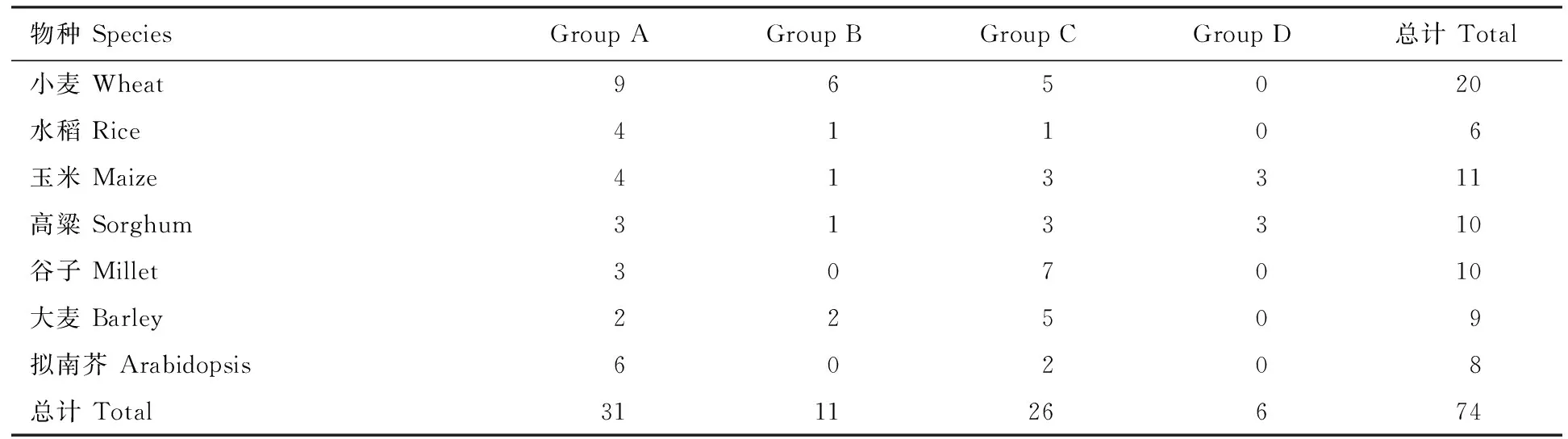
表2 BES1基因家族成员在系统发育树的分布情况Table 2 Distribution of BES1 gene family members in phylogenetic tree
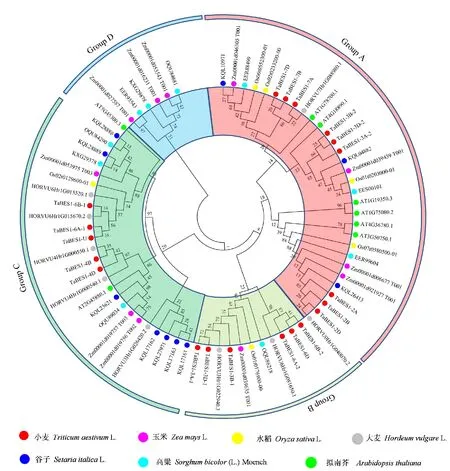
图1 小麦和6个代表性植物BES1的系统发育树Fig.1 Phylogenetic tree of BES1 in wheat and the six representative plants
2.3 BES1基因家族的基因结构
对小麦和6个代表性植物的BES1家族成员进行基因结构分析,结果(图2)表明,在小麦中,BES1基因家族成员外显子数目差异较大,大部分有2个外显子,最多有10个。整体来说,不同物种的外显子和内含子的结构与进化树分组有较好的对应关系,如Group A、Group B和Group D中的BES1基因一般含有2个外显子,而Group C中除两个大麦BES1基因外,其他BES1基因家族成员均含有较多的外显子。
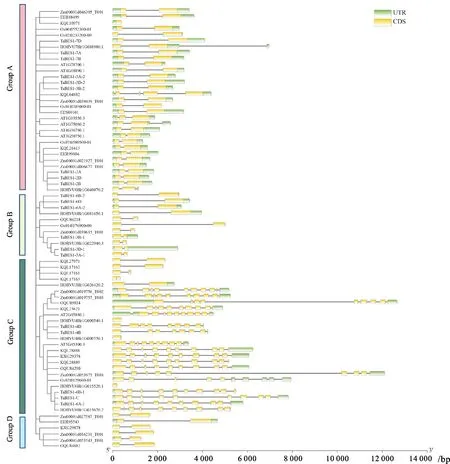
图2 小麦和6个代表性植物 BES1基因家族成员的基因结构分析Fig.2 Gene structure analysis of BES1 gene family members in wheat and the six representative plants
2.4 小麦和其他6个代表性植物的共线性分析
对小麦和其他6个代表性植物进行共线性分析,结果(图3)表明,小麦和拟南芥基因组中仅有3个TaBES1基因存在共线性关系,小麦和水稻、玉米、高粱、谷子、大麦的共线性基因对数目分别为25、16、15、10、11,说明小麦和水稻基因组拥有的共线性基因对数目最多。TaBES1-3A-1、TaBES1-3B-1和TaBES1-3D-1同时存在于小麦与拟南芥、水稻、玉米、高梁、谷子和大麦这6个物种的共线性基因对中,表明这3个基因可能在单子叶植物和双子叶植物中普遍存在,形成于物种分化之前,拥有更长的进化时间。
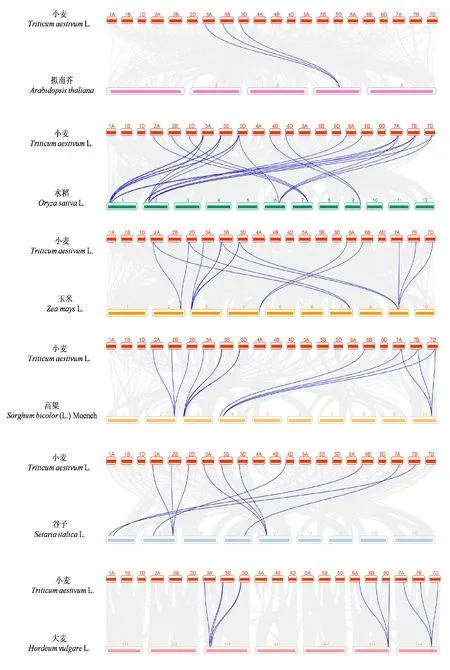
根据同源共线性分析结果,灰色线条代表两个物种间所有存在共线性的基因对,蓝色高亮线条代表小麦TaBES1基因与对应物种间的共线性基因对。According to the results of homologous synteny analysis,gray lines represent all synteny gene pairs between the two species,and blue lines represent synteny gene pairs between wheatTaBES1 genes and other species.图3 BES1基因在小麦和6个代表性植物间的共线性分析Fig.3 Collinearity analysis of BES1 genes between wheat and the six representative plants
2.5 TaBES1基因在不同组织中的表达模式
对20个TaBES1基因家族成员的表达模式进行分析,结果(图4)表明,TaBES1基因在不同组织中的表达模式差异明显。TaBES1-2A、TaBES1-2B、TaBES1-2D、TaBES1-3A-2、TaBES1-3B-2和TaBES1-3D-2这6个基因在穗部(特别是雄蕊中)表达量明显较高。TaBES1-7A、TaBES1-7B和TaBES1-7D这3个基因在根部表达量较高。有8个TaBES1基因(TaBES1-3A-1、TaBES1-3B-1、TaBES1-3D-1、TaBES1-6A-1、TaBES1-6A-2、TaBES1-6B-1、TaBES1-6B-2和TaBES1-6D)在各个组织(器官)表达量均很低或不表达。综上,小麦BES1基因在不同组织(器官)中发挥的功能可能存在差异,部分基因的表达呈现出组织特异性。
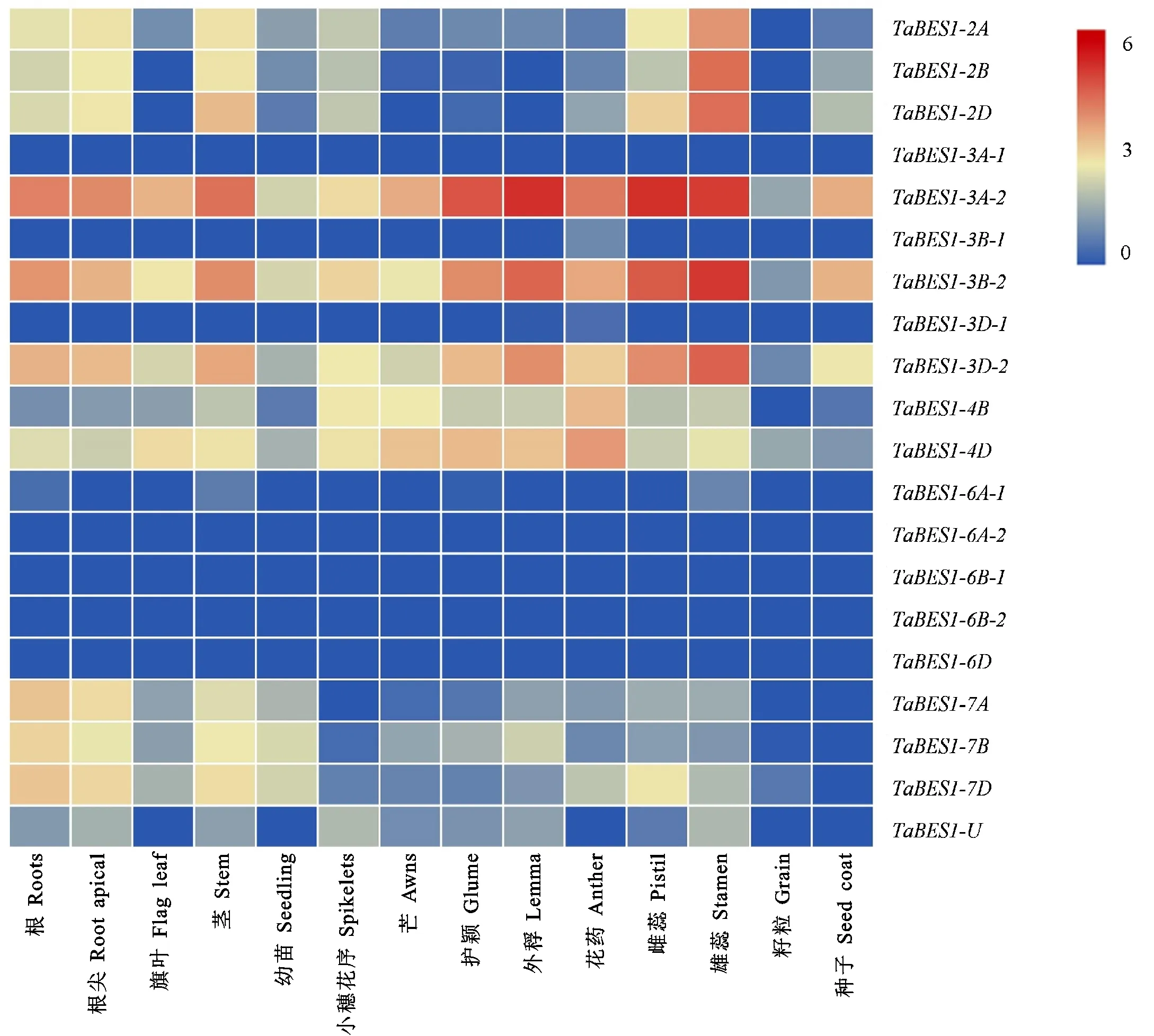
图4 小麦TaBES1基因在不同组织器官中的表达谱分析Fig.4 Expression patterns analysis of TaBES1 genes in different tissues of wheat
3 讨 论
小麦是典型的异源六倍体,其基因组在所有禾本科作物中最为庞大,约为水稻基因组的40倍[21]。本研究共鉴定到20个小麦BES1基因家族成员,在数量上超过了水稻、玉米、高粱、谷子、大麦等其他禾本科作物。普通小麦是具有AABBDD基因组的异源六倍体,这可能是小麦中BES1基因大量存在的一个关键原因,相比其他禾本科作物,小麦BES1基因经历了更长的进化时间。
在系统发育树中,相同亚家族中大部分BES1基因有着相似的基因结构,表明结构相似的BES1基因有着相似的进化历史和基因功能[22-23]。共线性分析显示,小麦BES1基因与拟南芥的共线性最低,但是与其他禾本科作物的共线性关系相对较高,说明禾本科作物之间亲缘关系更近,尤其是小麦与水稻之间的共线性关系最高。此外,在小麦和大麦的基因组共线性分析中,小麦第三、第六、第七染色体群中的基因分别对应大麦3H、6H、7H染色体中的基因,也有着良好的共线性关系,说明小麦和大麦基因组高度同源,这对研究小麦和大麦之间的进化关系有很大帮助。
BES1转录因子家族是植物体内一类重要的基因家族,也是BRs信号转导的核心成员,可以协同调控植物的生长发育和抗逆性[24]。在小麦中, 抑制BRs合成的代谢途径,会降低小麦的光合作用、籽粒产量以及对高光、高温的耐受性[25]。此外,BRs在对小麦抗旱性、磷酸盐稳态和谷物质量的调节中,也扮演着重要的角色[26]。在拟南芥中,BES1基因可以被EMS1-TPD1-SERK1/2介导的信号转导激活,从而控制拟南芥的绒毡层发育[13,27],影响植株育性。表达模式分析显示,TaBES1基因在小麦穗和根中的表达量较高,特别是TaBES1-3A-2、TaBES1-3B-2和TaBES1-3D-2在小麦雄蕊和雌蕊中的表达量较高,说明这三个基因在小麦穗发育过程中发挥着重要作用。

