转基因大豆‘ZH 10-6’数字PCR精准定量检测方法的建立
刘 双 赵 新 李瑞环 刘 娜 兰青阔 檀建新 王 永,*
(1.天津市农业科学院 生物技术研究所,天津 300381;2.河北农业大学 食品科技学院,河北 保定 071001)
自1996年国际上首次种植转基因作物至今已有24年的时间。转基因作物的发展突飞猛进,很多国家和地区均实施了标识管理制度,并设定了阈值。如欧盟实施强制性的转基因标识制度[1],我国目前对转基因产品也实施强制标识制度。目前,转基因检测的方法主要是基于核酸检测的聚合酶链式反应(polymerase chain reaction,PCR)技术[2]、核酸杂交技术[3]和基因芯片技术[4];基于蛋白质检测的Western Blot[5]和酶联免疫吸附测定[6]等。其中应用最广泛的是基于核酸检测的普通PCR方法和实时荧光PCR方法。我国已经研制了‘ZH 10-6’大豆的定性PCR检测方法,实时荧光定量PCR方法[7]已初步建立,但是其数字PCR定量方法尚未见报道。相对于转基因成分定性PCR检测方法,定量PCR检测方法不仅可以检测产品含有的转基因成分,而且可以确定转基因成分的含量,更加有助于转基因产品的监管。
数字PCR(digital polymerase chain reaction,dPCR)是在实时荧光PCR基础上发展起来的微量DNA分子定量检测新技术。dPCR是将PCR体系分配到足够小的反应单元中,实现每个反应单元只有单个模板分子进行PCR扩增,再采用泊松分布原理,根据阳性微滴与阴性微滴数的比例计算目标分子拷贝数,实现绝对定量[8-10]。该方法降低了标准曲线对测量结果产生影响等问题,降低了基体效应,实现了PCR扩增的样品分离,消除了本底信号的影响,提高了低拷贝DNA的扩增灵敏度[11]。相比实时荧光PCR,数字PCR具有更好的测量独立性,且无需任何校准物,具有更高的特异性、灵敏度、精确性和稳定性。
转基因耐除草剂大豆品种‘ZH 10-6’是由中国农业科学院作物科学研究所研发的转G2-EPSPS和GAT基因耐除草剂大豆新品种[12]。研发人采用农杆菌介导转化法,将整合有G2-EPSPS和GAT2个基因的独立表达载体DNA导入大豆受体中,通过多代筛选获得对草甘膦具有抗性的‘ZH 10-6’转化体,在我国具有重要产业化应用前景,已获得转基因安全证书。但是其数字PCR定量检测方法尚未见报道。本研究以‘ZH 10-6’特异性序列为靶标,通过对引物探针浓度的优化确定扩增体系,并对其特异性、灵敏度和准确度等进行测试,旨在建立该转基因大豆品种‘ZH 10-6’的微滴式数字PCR精准定量检测方法,以期为该转基因大豆品种的安全评价、行政监管和知识产权保护提供重要的技术支撑。
1 材料与方法
1.1 试验材料
转基因耐除草剂大豆品种‘ZH 10-6’;非转基因大豆品种‘ZH 10’;转基因玉米品种混合样(‘Bt 11’、‘Bt 176’、‘MON 810’、‘MON 863’、‘GA 21’、‘NK 603’、‘T 25’、‘TC 1507’、‘MON 89034’、‘MON 88017’、‘59122’、‘MIR 604’、‘3272’、‘MON 87460’、‘MIR 162’、‘DAS 40278-9’、‘双抗12-5’、‘IE09S034’、‘C0030.3.5’、‘C0010.3.7’、‘4114’、‘MON 87427’和‘5307’);转基因大豆品种混合样(‘GTS 40-3-2’、‘MON 89788’、‘CV 127’、‘A 5547-127’、‘A 2704-12’、‘305423’、‘356043’、‘MON 88302’、‘73496’、‘MON 87769’、‘MON 87705’、‘FG 72’、‘DAS 68416-4’和‘SHZD 32-1’);转基因油菜品种混合样(‘Ms 1’、‘Ms 8’、‘RF 1’、‘RF 2’、‘RF 3’、‘T 45’、‘oxy 235’、‘Topas 19/2’、‘MON 88302’和‘73496’);转基因水稻品种混合样(‘TT 51-1’、‘科丰6号’、‘M 12’、‘科丰8号’、‘科丰2号’、‘G6H1’和‘T1 C-19’);转基因棉花品种混合样(‘MON 531’、‘MON 1445’、‘MON 15985’、‘MON 88913’、‘GHB 614’、‘COT 102’)。以上样品均由本实验室收集保存。
1.2 方法
1.2.1基因组DNA提取
以转基因耐除草剂大豆品种‘ZH 10-6’新鲜叶片为材料,按照德国QIAGEN公司的DNeasy Plant Mini Kit提取试剂盒纯化获得基因组DNA,利用NanoDrop ND-1000核酸蛋白定量仪(Thermo Scientific公司,美国)测定DNA质量并将其稀释至统一浓度25 ng/μL,于-20 ℃保存备用。
1.2.2引物和探针设计
转基因耐除草剂大豆‘ZH 10-6’的外源基因插入位置为第17号染色体,插入位点为含有G2-EPSPS和GAT2个基因的完整表达框,以及只含G2-EPSPS基因的表达框,不同外源基因片段之间以大豆基因组DNA片段相连接(图1)。
根据转基因耐除草剂大豆‘ZH10-6’外源插入片段的2个5′端侧翼序列,采用Primer Express 3.0设计了多种引物探针组合,引物由上海生工有限公司合成,引物探针信息,见表1。
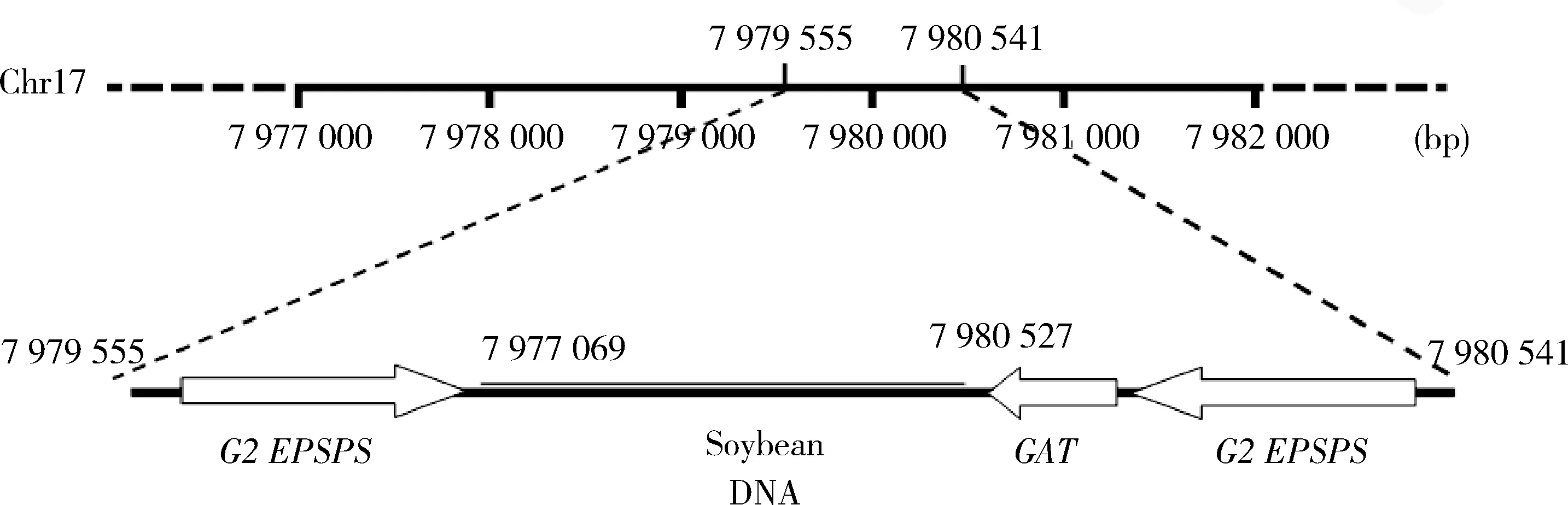
图1 转基因耐除草剂大豆‘ZH10-6’外源基因插入位点Fig.1 Insertion site of foreign gene in transgenic herbicide-tolerant soybean ‘ZH10-6’
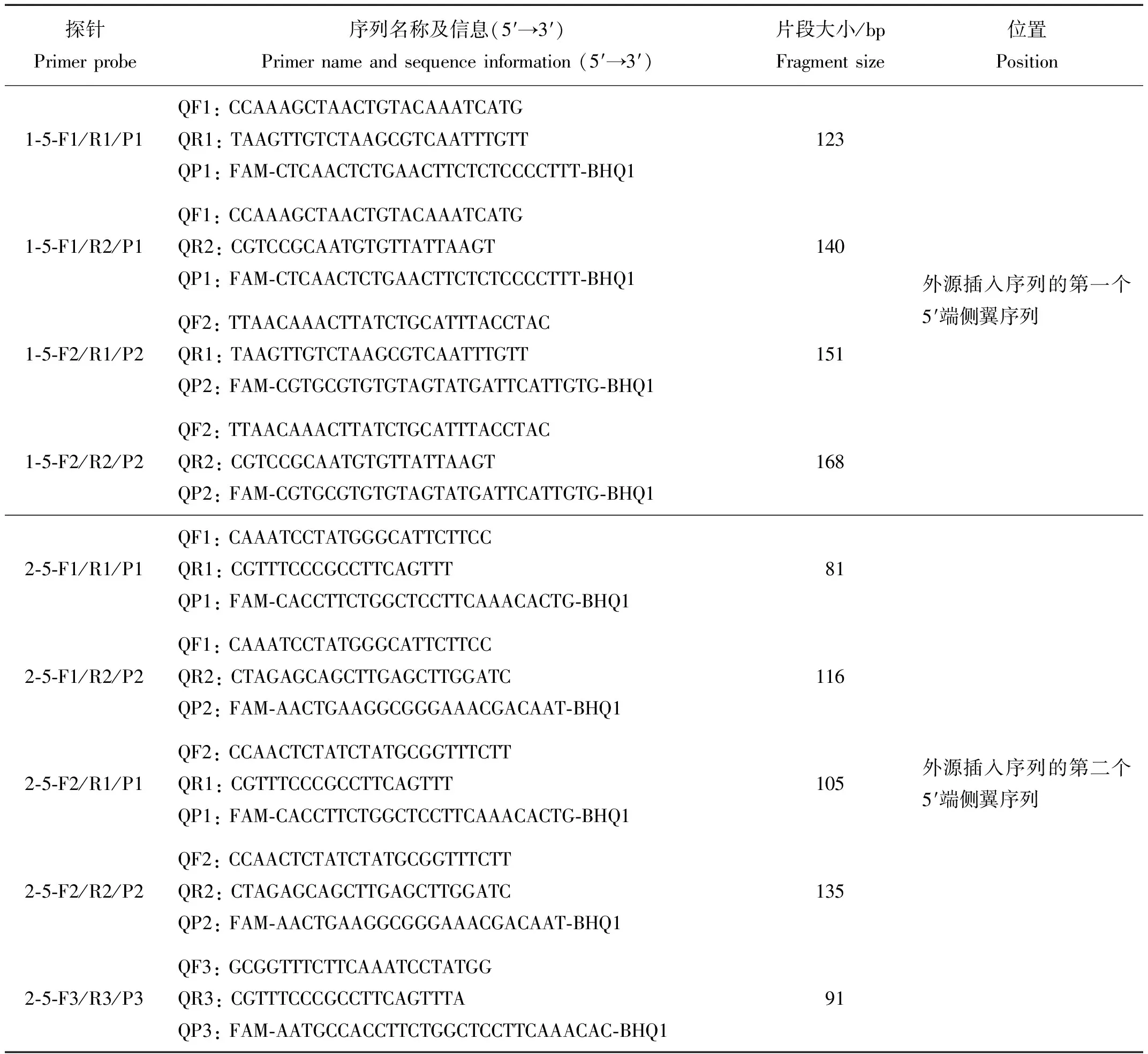
表1 微滴式数字PCR引物探针序列信息Table 1 Sequence information of droplet digital PCR primer probe
1.2.3反应体系和条件
PCR反应的总体积为20 μL,包括BIO-RAD ddPCR Supermix for Probes 10 μL,10 μmol/L上游引物1 μL,10 μmol/L下游引物1 μL,10 μmol/L探针1 μL,25 ng/μL DNA模板1 μL,用无菌ddH2O补充至20 μL。
PCR扩增反应条件:94 ℃预变性10 min,1个循环;94 ℃变性30 s,60 ℃退火1 min,40个循环;98 ℃酶失活10 min,1个循环。
1.2.4引物探针初筛
使用上述通用体系和程序,以转基因耐除草剂大豆‘ZH 10-6’基因组DNA为模板,采用合成的引物探针组合配制20 μL的PCR体系,将体系混匀后转移至微滴生成卡槽的样品列小室中,在oil列加入70 μL的微滴生成油,盖上胶垫放入微滴生成器BIO-RAD QX100 Droplet Generator(Bio-Rad公司,美国)中由仪器自主生成微滴,再将生成的微滴小心转移至96孔板中,175 ℃于BIO-RAD PX1 PCR Plate Sealer(Bio-Rad公司,美国)热封膜,最后置于Analytik jena Easy Cycler梯度PCR仪(Bio-Rad公司,美国)中进行PCR扩增,待PCR扩增结束后,将96孔板放入微滴分析仪BIO-RAD QX100 Droplet Reader(Bio-Rad公司,美国)中读取信号,并进行分析。筛选阳性和阴性微滴界限区分清晰且仅以转基因耐除草剂大豆‘ZH 10-6’基因组DNA为模板时,生成有阳性微滴的引物探针组合,每个反应设置2个平行。
1.2.5PCR反应体系和程序优化
根据BIO-RAD ddPCR Supermix for Probes推荐的引物探针体积,分别设置5个浓度梯度的引物探针(体系终浓度为0.3、0.4、0.5、0.6和0.7 μmol/L)对微滴式数字PCR体系进行优化,筛选出阳性与阴性微滴界限清楚,且荧光信号相对较高的引物探针组合作为本研究的最优反应体系。
分别设置6个梯度的退火温度(54、56、58、60、62 和64 ℃),对微滴式数字PCR程序进行优化,筛选出阳性微滴与阴性微滴界限清楚,且荧光信号相对较高的引物探针组合作为本方法的最优反应条件。
1.2.6特异性测试
以转基因玉米混合样,转基因大豆混合样,转基因油菜混合样,转基因水稻混合样,转基因棉花混合样,非转基因大豆样品的基因组DNA为模板,采用已优化的反应体系进行PCR扩增,对检测方法的特异性进行测试,每个反应设置2个平行。
1.2.7定量检测线性动态范围测定
将转基因耐除草剂大豆‘ZH 10-6’的基因组DNA(100%)用缓冲液稀释至50.000%、10.000%、2.000%、0.400%、0.080%和0.016% 共7个浓度梯度,采用优化后的体系进行PCR扩增以确定本方法的线性动力范围,每个反应3次重复。
1.2.8检出限和定量限验证
以定量检测线性动态范围测定中线性范围下限且RSD≤25%的基因组DNA浓度为模板,进行PCR扩增,每个反应设置10个平行,统计PCR反应结果确定本方法的定量限(limit of quantification, LOQ);采用可以稳定检测的基因组DNA浓度,设置10个重复进行PCR扩增,统计10个平行PCR反应的结果,确定本方法的检出限(limit of detection, LOD)。
1.2.9方法适用性测试
制备转基因耐除草剂大豆‘ZH 10-6’含量分别为100.00%、5.00%和1.00%的样品:以非转基因大豆‘ZH 10’和转基因耐除草剂大豆‘ZH 10-6’进行质量浓度配比混合,得到已知含量的定量测试样品。提取其基因组DNA作为模板,进行数字PCR扩增,通过计算得到测试样品的含量,对方法的定值适用性进行测试。
2 结果与分析
2.1 引物初筛
采用通用体系进行PCR扩增,初步筛选确定了阳性微滴与阴性微滴界限区分明显且荧光信号值相对较高的引物探针组合2-5-F3/R3/P3(图2),作为本研究的引物探针进行后续的体系优化和验证试验。
2.2 微滴式数字PCR体系优化
由表2、图3和图4可知,微滴式数字PCR体系中引物和探针的终浓度为0.5 μmol/L,退火温度56 ℃时,阳性微滴与阴性微滴界限清楚,且荧光信号值较高,确定扩增体系的引物终浓度为0.5 μmol/L,探针终浓度为0.5 μmol/L。
2.3 特异性测试
由图5可知,仅以转基因耐除草剂大豆‘ZH 10-6’的基因组DNA为模板时,生成有阳性微滴,且阳性微滴与阴性微滴界限清楚,而以其他转基因作物混样和非转基因大豆的基因组DNA为模板时,没有阳性微滴生成,表明本研究所筛选的引物探针特异性良好,可以用于后续测定。
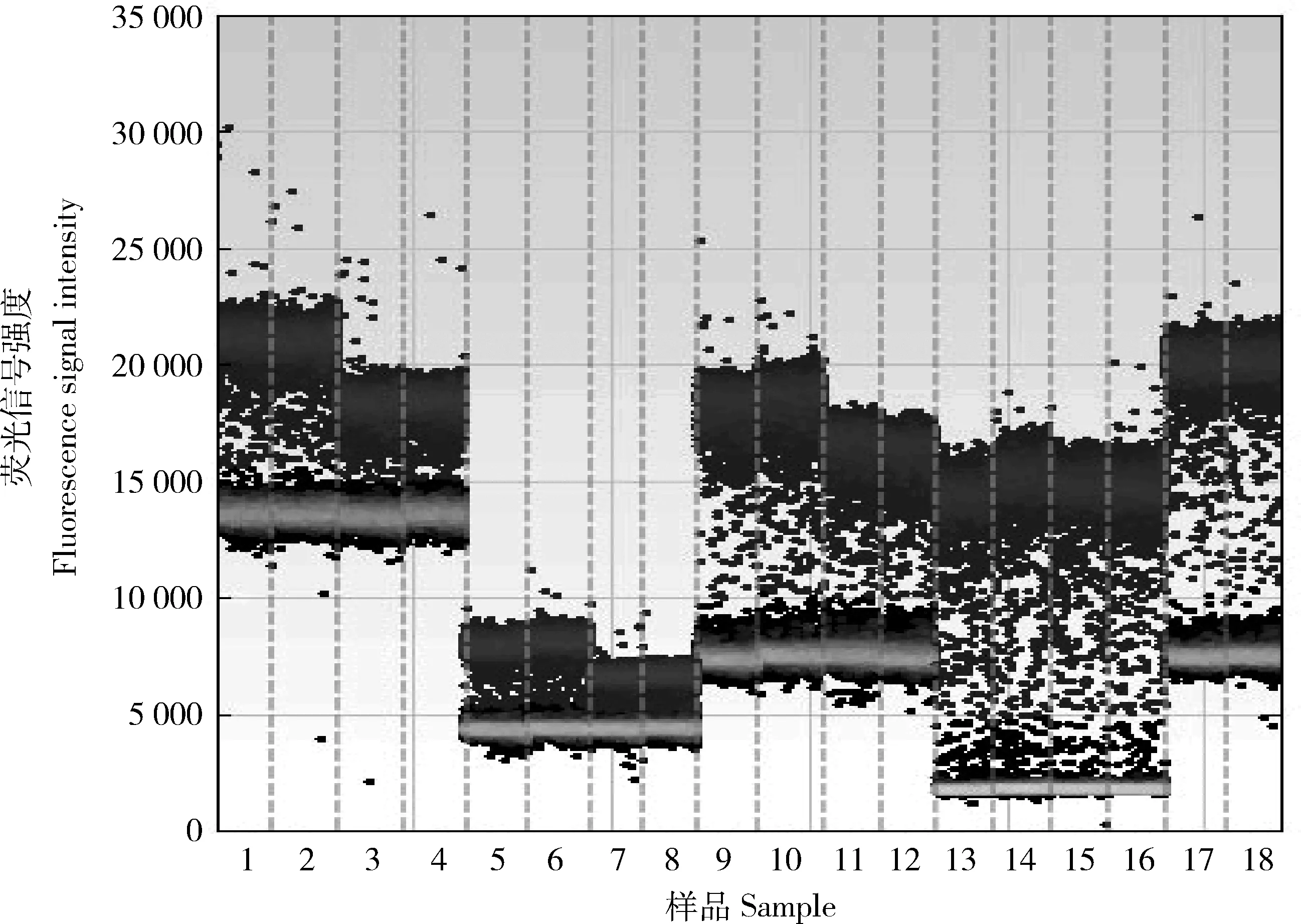
1和2,引物组合1-5-F1/R1/P1;3和4,引物组合1-5-F1/R2/P1;5和6,引物组合1-5-F2/R1/P2;7和8,引物组合1-5-F2/R2/P2;9和10,引物组合2-5-F1/R1/P1;11和12,引物组合2-5-F1/R2/P2;13和14,引物组合2-5-F2/R1/P1;15和16,引物组合2-5-F2/R2/P2;17和18,引物组合2-5-F3/R3/P3。1 and 2, primer combination 1-5-F1/R1/P1; 3 and 4, primer combination 1-5-F1/R2/P1; 5 and 6, primer combination 1-5-F2/R1/P2; 7 and 8, primer combination 1-5-F2/R2/P2; 9 and 10, primer combination 2-5-F1/R1/P1; 11 and12, primer combination 2-5-F1/R2/P2; 13 and 14, primer combination 2-5-F2/R1/P1; 15 and 16, primer combination 2-5-F2/R2/P2; 17 and 18, primer combination 2-5-F3/R3/P3.图2 微滴式数字PCR引物初步筛选结果Fig.2 Preliminary screening results of droplet digital PCR primers

表2 微滴式数字PCR体系优化拷贝数结果Table 2 Optimized copy number results of droplet digital PCR system
2.4 定量线性范围测定
由表3和图6可知,大豆内源基因Lectin和转基因耐除草剂大豆‘ZH 10-6’特异性序列标准曲线的相关系数R2均>0.99。在模板含量为66.0个拷贝时,大豆内源基因Lectin和转基因耐除草剂大豆‘ZH 10-6’特异性序列的RSD≤25.00%,初步确定本研究的定量检测下限为66.0个拷贝。
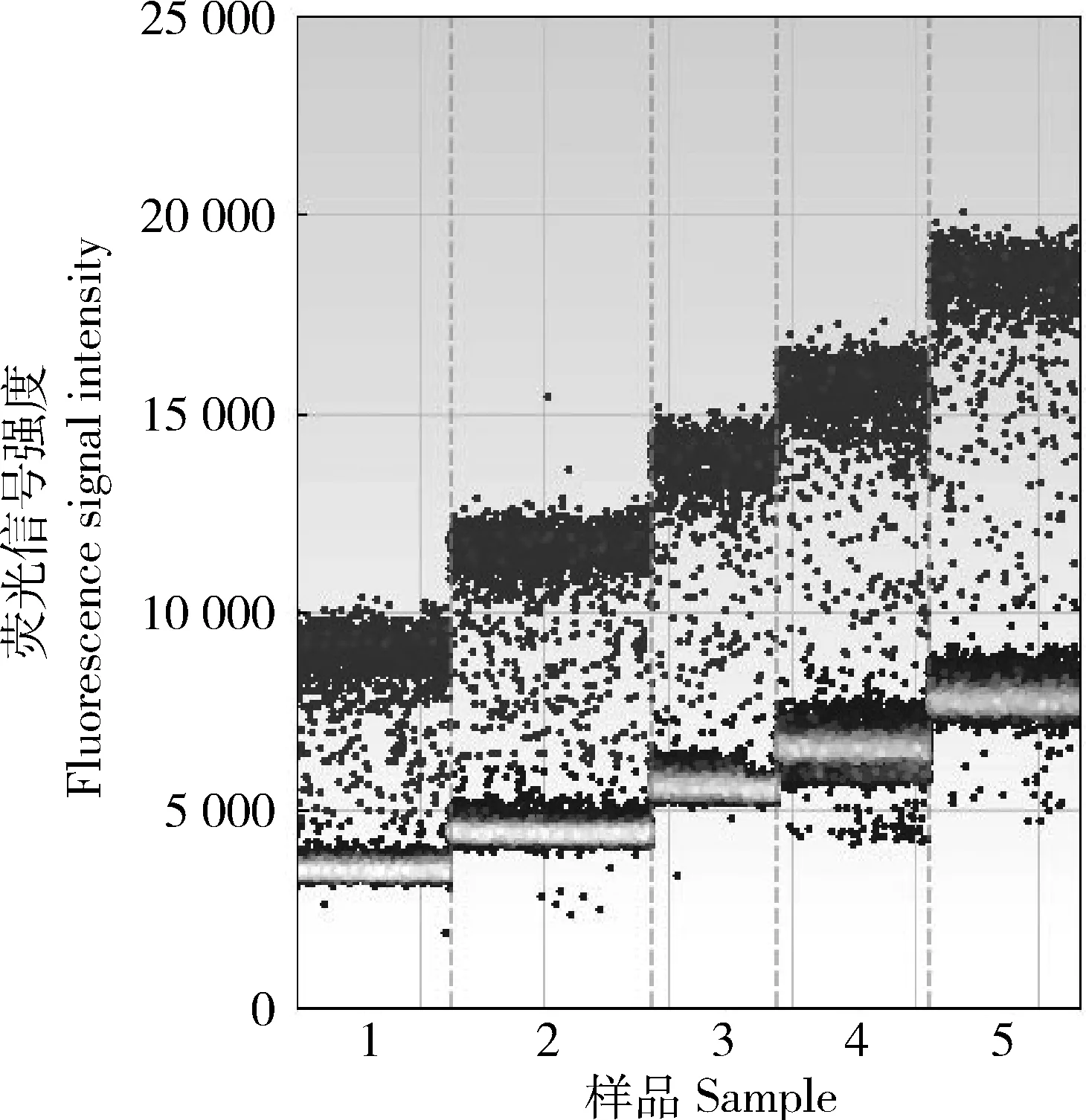
1,2,3, 4和5分别是引物浓度0.3,0.4,0.5,0.6和0.7 μmol/L。1, 2, 3, 4 and 5 indicate primer concentration 0.3, 0.4, 0.5, 0.6 and 0.7 μmol/L respectively.图3 微滴式数字PCR体系优化结果Fig.3 Optimization results of droplet digital PCR system
2.5 检出限和定量限验证
由表4可知,10次重复内源基因Lectin的RSD为19.44%,转基因大豆‘ZH 10-6’特异性序列的RSD为16.00%,内源基因和特异性序列扩增RSD均≤25.00%,故确定定量限为66.0个拷贝。
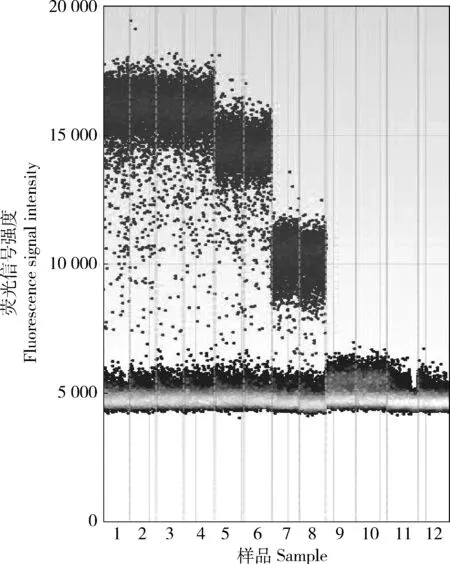
1和2,54 ℃;3和4,56 ℃;5和6,58 ℃;7和8,60 ℃;9和10,62 ℃;11和12,64 ℃。图4 微滴式数字PCR程序退火温度优化结果Fig.4 Optimization annealing temperature results of droplet digital PCR program

1和2,阳性对照;3和4,转基因耐除草剂大豆‘ZH 10-6’;5和6,非转基因大豆样品;7和8,转基因玉米混样;9和10,其他转基因大豆混样;11和12,转基因油菜混样;13和14,转基因水稻混样;15和16,转基因棉花混样;17和18,阴性对照。1 and 2, positive control; 3 and 4, genetically modified herbicide-tolerant soybean ‘ZH 10-6’; 5 and 6, non-transgenic soybean sample; 7 and 8, genetically modified corn mixed samples; 9 and 10, other genetically modified soybean mixed samples; 11 and 12, genetically modified rapeseed mixed samples; 13 and 14, genetically modified rice mixed sample; 15 and 16, genetically modified cotton mixed sample; 17 and 18, negative control.图5 微滴式数字PCR特异性检测结果Fig.5 Specific detection results of droplet digital PCR

表3 微滴式数字PCR定量线性范围测试结果Table 3 Test results of quantitative linear range of droplet digital PCR
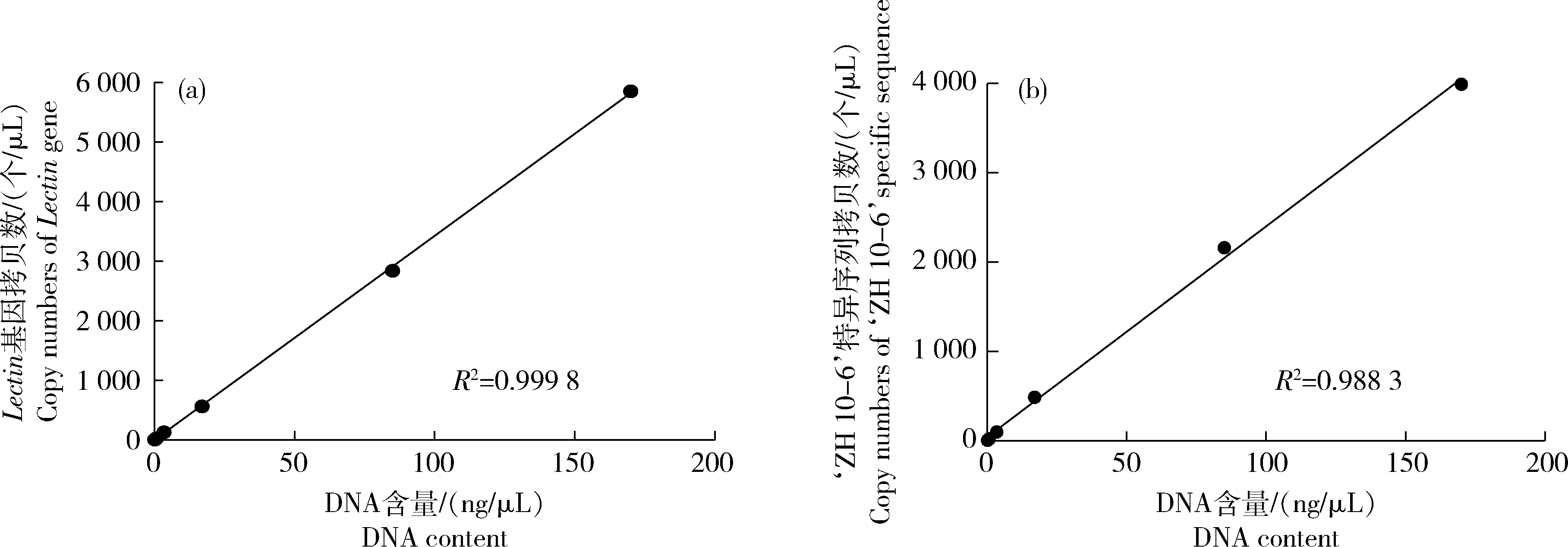
图6 大豆内源Lectin(a)和转基因耐除草剂大豆‘ZH10-6’特异性序列(b)基因定量线性范围PCR扩增结果Fig.6 The quantitative linear range PCR amplification results of soybean endogenous Lectin (a) and genetically modified herbicide-tolerant soybean‘ZH10-6’ specific sequence (b)
以含量为13.0个拷贝的DNA为模板扩增时,10次扩增均有阳性微滴生成;以含量为2.6个拷贝的DNA为模板扩增时,10个重复中有8次检测达到阳性微滴,2次未检测到阳性微滴,确定检出限为13.0个拷贝,见表4。
2.6 适用性测试结果
由表5和图7可知,模拟样品定值实际结果与预计结果偏差均在25%以内,表明本方法的定值适用性良好。
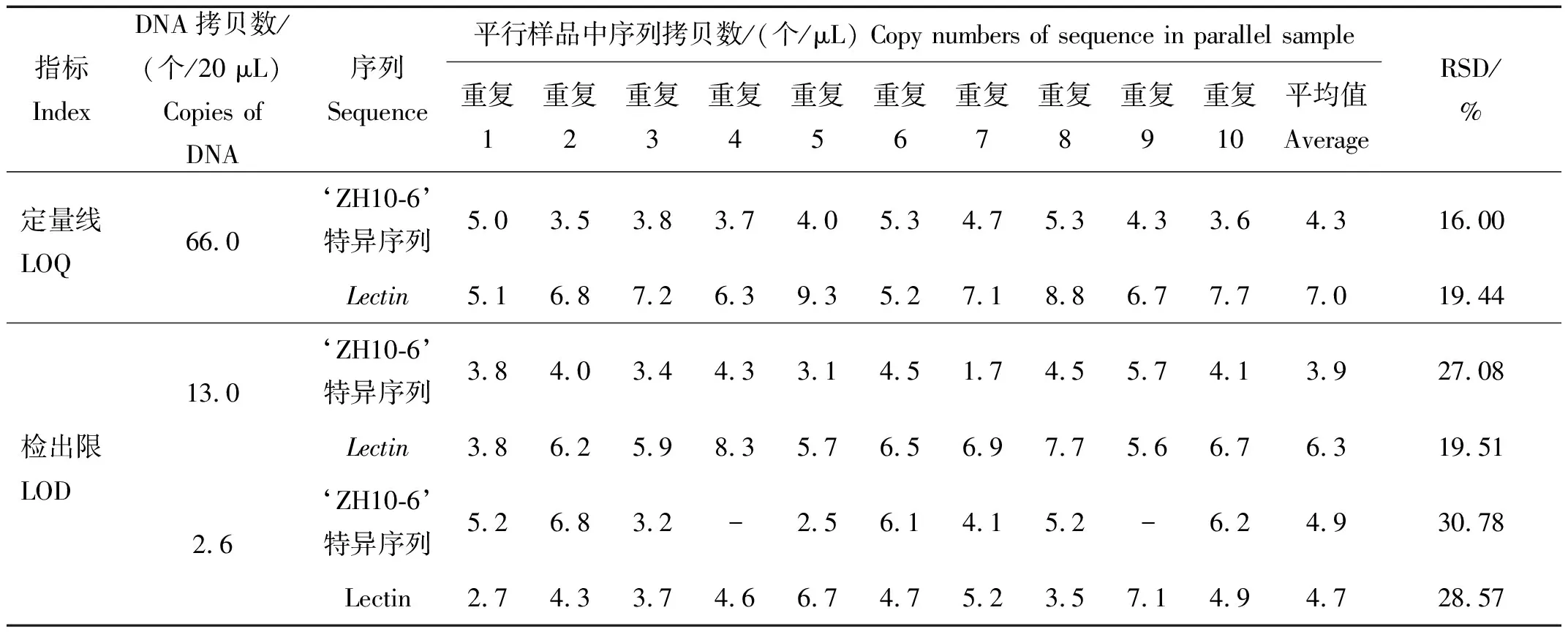
表4 微滴式数字PCR定量限(LOQ)和检出限(LOD)Table 4 Limit of quantification (LOQ) and limit of detection (LOD) of the droplet digital PCR

表5 数字PCR方法适用性测定Table 5 Applicability determination of digital PCR method
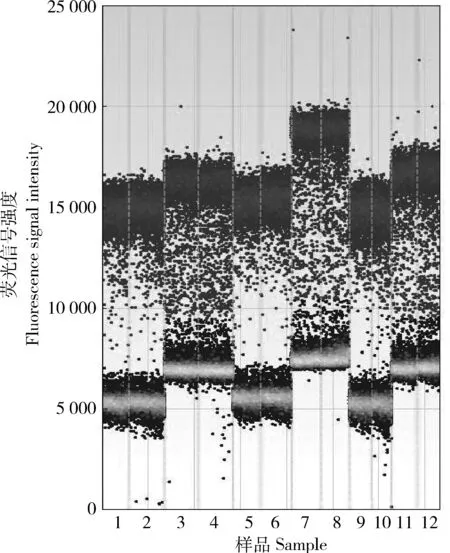
1和2,样品1内源基因;3和4,样品1外源基因;5和6,样品2内源基因;7和8,样品2外源基因;9和10,样品3内源基因;11和12,样品3外源基因。1 and 2, sample 1 endogenous gene; 3 and 4, sample 1 exogenous gene; 5 and 6, sample 2 endogenous gene; 7 and 8, sample 2 exogenous gene; 9 and 10, sample 3 endogenous gene; 11 and 12, sample 3 foreign gene.图7 数字PCR方法适用性测定结果Fig.7 Applicability test results of digital PCR method
3 讨 论
随着转基因技术的广泛应用,多数国家对转基因产品均强制规定转基因成分含量高于某一阈值时须进行标识,如何精准测定转基因成分的含量已经成为现今转基因产品检测技术研究的一个关键点。目前,用于分析基因拷贝数的方法主要有Southern Blot方法、实时荧光PCR定量方法和数字PCR定量方法。
Southern也被称为凝胶电泳杂交技术。该方法利用了硝酸纤维素膜或尼龙膜的特性,即可吸附DNA进行印迹实验。首先待测样品进行凝胶电泳,然后将电泳的DNA条带转印到膜上,后直接在膜上进行被测DNA和同位素标记的探针之间的杂交。最后,通过放射自显影设备来检测杂交结果,以检测DNA样品中包含的特定DNA序列。Southern印迹法在检测靶基因的拷贝数,鉴定转基因植物,开发分子标记和筛选DNA文库中起重要作用。但该项技术的操作步骤繁杂,要获得理想的结果,通常会花费较多的时间和精力。传统Southern印迹分析的探针通常用放射性同位素标记,但可能会对操作人员和环境造成放射性危害。传统的Southern Blot方法分析基因拷贝数操作复杂、耗时长、工作量大且准确性相对较差[13]。
实时荧光PCR相对定量检测方法是转基因检测中常用的方法之一。该方法具有特异性强、灵敏度高、可定量分析靶标序列含量等优点,相对于普通PCR来说,实时荧光PCR相对定量检测方法检出限更低。实时荧光PCR定量方法分析基因拷贝数较Southern Blot方法耗时短,但依赖于标准品,需要同时构建内源基因和转化体特异性序列的标准曲线,是一种依赖于构建标准曲线的相对定量分析方法[14-15],检测结果也存在一定的不确定性。
微滴式数字PCR精准定量检测方法是配置PCR体系后,通过微滴生成器将混匀后的体系生成上万个油包水微滴,再放入PCR仪中进行扩增,最后放入微滴读取仪中读取阳性微滴和阴性微滴的个数,从而来精准定量靶标序列。微滴式数字PCR精准定量方法[16]较前2种分析基因拷贝数的方法,操作更加简单,不需要标准物质,不依赖于标准曲线的构建,不仅可以更加准确的定量研究靶标序列,而且检测时间更短,结果由仪器判读也可避免操作人员的主观因素,获取数据更加方便,结果也更加稳定可靠,实现了真正意义上的绝对定量[17-18]。微滴式数字PCR精准定量检测方法的不足之处是比实时荧光PCR检测方法成本高。
本研究采用的微滴式数字PCR精准定量方法用于分析基因的拷贝数,可有效避免实时荧光定量PCR扩增中体系的扩增效率、标准曲线构建的准确性对检测结果带来的影响,对于拷贝数少的基因定量检测更加高效,应用前景广阔[19-20]。本研究建立的转基因耐除草剂大豆‘ZH 10-6’的数字PCR精准定量检测方法可以对转基因耐除草剂大豆‘ZH 10-6’进行精准定量,精确度和准确度均符合《农业农村部公告第323号-21-2020转基因植物及其产品成分检测数字PCR方法制定指南》[21]的相关要求,相较实时荧光定量PCR方法依赖于标准曲线的定量方法,更适合于转基因产品的分析[22]。
4 结 论
本研究基于转基因耐除草剂大豆‘ZH 10-6’的5’端边界序列,建立的转基因耐除草剂大豆‘ZH 10-6’数字PCR检测方法特异于‘ZH 10-6’品种检测,在相对标准偏差≤25.00%的情况下本方法的检出限(LOD)为13.0个拷贝、定量限(LOQ)为66.0个拷贝;PCR扩增反应模板含量与样品拷贝数之间线性R2≥0.998,DNA含量线性范围为0.08%~100.00%。本方法重复性好,精密度和准确度均符合相关要求,适用于对农产品和食品中转基因大豆‘ZH 10-6’特异性序列的精准定量分析。