橡胶树KT/HAK/KUP基因家族成员的鉴定与表达分析
肖小虎 林显祖 龙翔宇 秦云霞 阳江华 方永军






摘 要:为研究巴西橡胶树钾转蛋白(KT/HAK/KUP)基因家族在生长发育和逆境胁迫方面的生物学功能,从橡胶树基因组中鉴定得到31个KT/HAK/KUP基因家族成员,并从基因结构、染色体定位、系统进化和表达分析等方面进行全面系统的分析。研究结果发现,KT/HAK/KUP基因家族31个成员分布在3个Contig和15个染色体上,8号染色体上数目较多为8个成员,其中5个为串联重复;各成员编码的氨基酸残基数目在288~881个不等,蛋白分子量分布在31.82~98.96 kDa,其蛋白产物均定位于细胞质膜上;内含子数目为5~11个,进化上分为4个明显分支。在表达方面,部分成员呈现明显的组织特异性,在叶片发育过程中成员HbKUP5、HbKUP6、HbKUP9和HbKUP14表达发生明显变化,乙烯利刺激后HbKUP30和HbKUP31在胶乳中的表达明显上调,在抗寒品种‘93~114’中,HbKUP5和HbKUP9在低温处理后明显下调表达。本研究结果为深入解析KT/HAK/KUP基因家族的功能和培育高钾抗逆的橡胶树品种提供了理论基础。
关键词:巴西橡胶树;KT/HAK/KUP;基因家族;逆境胁迫;表达分析
中图分类号:S794.1 文献标识码:A
Identifcation and Expression of KT/HAK/KUP Genes in Hevea brasliensis
XIAO Xiaohu1, LIN Xianzu1,2, LONG Xiangyu1, QIN Yunxia1, YANG Jianghua1, FANG Yongjun1*
1. Key Laboratory of Biology and Genetic Resources of Rubber Tree, Ministry of Agriculture and Rural Affairs / Rubber Research Institute, Chinese Academy of Tropical Agricultural Sciences, Haikou, Hainan 571101, China; 2. College of Tropical Crops, Hainan University, Haikou, Hainan 570228, China
Abstract: In order to study the biological functions of KT/HAK/KUP gene family in the growth and development and stress of Hevea brasiliensis, 31 KT/HAK/KUP genes were identified, and the gene structure, chromosomal location, evolution and expression were analyzed. 31 members of KT/HAK/KUP gene family were distributed on 3 contig and 15 chromosomes. There were 8 members on chromosome 8, and 5 of which were tandem repeats. The number of amino acid residues ranged from 288 to 881, and the molecular weight of the proteins ranged from 31.82 to 98.96 kDa, the proteins were located on the cytoplasmic membrane, with 5 to 11 introns. All the mumbers could be divided into four distinct branches in evolution. The expression of HbKUP5, HbKUP6, HbKUP9 and HbKUP14 were significantly changed during leaf development. And the expression of HbKUP30 and HbKUP31 in latex were significantly up-regul¬ated under ethephon stimulation. In cold resistant cultivar ‘93-114’, the expression of HbKUP5 and HbKUP9 was significantly down-regulated under low temperature treatment. The results of this study would provide theoretical guidance for further study the functions of KT/HAK/KUP gene family and high potassium stress resistant rubber tree varieties breeding.
Keywords: Hevea brasiliensis; KT/HAK/KUP; gene family; stress; expression analysis
DOI: 10.3969/j.issn.1000-2561.2022.01.001
在植物生長发育的过程中,钾(K+)离子作为第二信使扮演着至关重要的角色。植物主要通过钾转运蛋白来实现对K+的吸收转运,KT/ HAK/KUP(K+ uptake permease/high-affinity K+/K+ transporter)是植物体内最大的K+转运蛋白基因家族,在植物养分代谢、生长调控和抵御胁迫等过程中发挥着重要的作用[1]。目前已经从水稻[2]、玉米[3]、番茄[4]、杨树[5]等植物中克隆得到多个KT/HAK/KUP基因家族成员。研究表明,KT/ HAK/KUP家族成员属于跨膜蛋白,主要定位于细胞的膜系统,包含10~15个跨膜域。在拟南芥中,钾饥饿(无K+)可以诱导AtHAK5的表达,在钾离子浓度小于50 μmol/L时athak5突变体种子萌发缓慢,根生长受抑制,钾离子吸收能力降低[6]。钾饥饿同样可以诱导水稻OsHAK5表达,将OsHAK5在烟草BY2细胞中表达,盐胁迫条件下细胞大量积累K+而非Na+,表明OsHAK5是对盐敏感的高亲和K+转运蛋白基因[7]。拟南芥AtKUP1-4在发育时期的茎尖、花序分生组织、叶基部等组织中表达量有不同程度的增加,推测这些基因参与拟南芥的生长发育过程[8]。另外,盐胁迫和激素刺激均能调控KUP基因的表达,表明KUP在维持细胞K+、Na+平衡和K+信号感知方面具有重要功能[9-10]。
天然橡胶是一种重要的工业原料和军事战略物资,巴西橡胶树是天然橡胶生产的主要来源。目前关于巴西橡胶树KT/HAK/KUP基因家族的研究尚未见报道,这限制了我们对巴西橡胶树钾离子运输和分配的全面了解。随着橡胶树基因组测序的完成和不断优化,使我们能够对整个橡胶树KT/HAK/KUP基因家族进行全面系统的分析。本研究从橡胶树转录组和基因组数据库中鉴定得到31个KT/HAK/KUP基因家族成员,并从基因结构、系统进化和表达模式等方面对这些家族成员进行全面系统的分析。研究结果将有助于全面了解KT/HAK/KUP基因家族成员在橡胶树钾离子转运和分配,以及生长发育和胁迫应答调控方面的重要功能。
1 材料与方法
1.1 材料
本研究不同组织转录组测序所用材料为巴西橡胶树(Hevea brasiliensis)‘热研7-33-97’,除了根来自于‘热研7-33-97’组培苗外,其他组织(包括胶乳、树皮、叶片、种子、雌花和雄花)均来自正常割胶橡胶树(开割两年以上),叶片不同发育时期的材料来自于一年生的‘热研7-33- 97’嫁接苗;乙烯利处理的材料为‘热研7-33-97’正常开割树(3天一刀,不涂乙烯利刺激),用1.5%乙烯利在0、3、12、24 h等4个不同时间点涂抹橡胶树割面。以上材料均来自海南省儋州市中国热带农业科学院院区实验基地。
1.2 方法
1.2.1 橡胶树KT/HAK/KUP家族成员的鉴定 橡胶树基因组数据(包括基因组fasta文件和gff3文件注释文件)来自本实验橡胶树基因组升级版本(未发表数据)。根据已发表文献,从NCBI下载拟南芥[11]、水稻[12]、杨树[5]和木薯[13]的KT/ HAK/KUP蛋白序列,利用HMMER软件构建KT/ HAK/KUP的HMM模型,然后利用该模型对橡胶树蛋白数据库进行检索,得到橡胶树KT/ HAK/ KUP家族成员,然后通过Pfam和CDD数据库,对鉴定出的橡胶树KT/HAK/KUP进行保守域验证。
1.2.2 基因结构和进化分析 根据橡胶树基因组GFF3文件包含的相关注释信息对橡胶树KT/ HAK/KUP家族成员的基因结构和染色体定位进行分析,利用TBtools[14]软件进行数据可视化作图。利用MEGA 6.0[15]软件构建系统进化树,采用Neighbor-Joining方法进行分子系统学分析,进行1000次bootstrap统计学检验,利用软件Figtree(http://tree.bio.ed.ac.uk/software/figtree/)对进化树进行调整修饰。
1.2.3 基因的表达模式分析 利用solexa转录组测序数据[16]对橡胶树KT/HAK/KUP基因在不同组织、不同叶片发育时期和乙烯利处理下的表达进行分析。首先对原始数据去除低质量序列,然后利用程序RSEM[17]进行表达分析,最后结合进化树信息,利用TBtools绘制热图。
2 结果与分析
2.1 橡胶树KT/HAK/KUP家族成员的鉴定
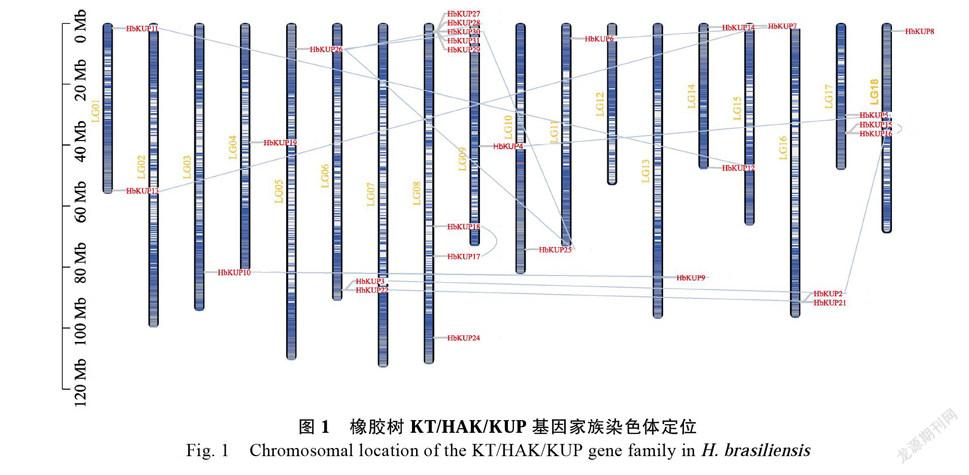
本研究利用已发表的拟南芥、水稻、杨树和木薯KT/HAK/KUP蛋白氨基酸序列构建HMM模型,对橡胶树蛋白数据进行检索,得到31个KT/HAK/ KUP基因家族成员同源序列。通过Pfam和CDD数据库分析发现,31个成员都含有KT/ HAK/KUP蛋白的所特有的保守结构域,命名为HbKUP1~HbKUP31(表1)。KT/HAK/KUP基因家族31个成员分布在3个Contig和15个染色体上,8号染色体上成员数目较多共8个,其中5个为串联重复;各基因成员编码的氨基酸残基数目在288~881个不等,蛋白分子量分布在31.82~ 98.96 kDa,大多编码碱性蛋白,等电点分布在5.04~ 9.3之间,其蛋白产物均定位于细胞质膜(表1)。
2.2 橡胶树KT/HAK/KUP家族成员的染色体定位
从31个橡胶树KT/HAK/KUP家族成员的染色体定位可以看出(图1,表1),8号染色体上成员数目最多有8个成员,17号染色体上有3个成员,1、6、14和16号染色体上均有2个成员,
3、4、5、9、10、11、13、15和18号染色体,以及3个Contig上面均有1个成员(表1)。其中8号染色体上5个成员成簇分布,6、16和17号染色体上有成员成对分布,这些成员可能是进化中通过基因复制产生的串联重复,而6和16号染色体上基因的复制可能发生6和16号染色复制之前,这从两个染色体间成员的同源性高于每个染色体上2个成员之间的同源性可以看出(蓝线连接表明有较高同源性)。从图中可以看出,串联复制和染色体复制事件是KT/HAK/KUP家族成员扩张的重要因素。
2.3 基因结构和进化分析
利用基因组序列及注释文件,对橡胶树KT/ HAK/KUP基因的外显子/内含子结构进行了分析。结果发现,各家族成员内含子数目在5~11之间,在相同进化分支中的各成員在内含子数目
和相对位置上更为相似,部分成员出现了较大内含子,如HbKUP2、HbKUP17和HbKUP18(图2B);在蛋白结构域方面,大部分家族成员都具有相似的motif,也有部分成员出现了motif的缺失,如HbKUP12和HbKUP19在蛋白的N端,HbKUP22的C端,以及HbKUP1、HbKUP18和HbKUP27中部有motif的缺失,这部分成员可能在进化过程中产生了功能分化(图2A)。
为了比较橡胶树和其他植物KT/HAK/KUP蛋白在进化上的相互关系,选取了杨树(Populus trichocarpa, Pt)、拟南芥(Arabidopsis thaliana, At)、水稻(Oryza sativa, Os)、木薯(Manihot esculenta, Me)和蓖麻(Ricicus communis, Rc),以及橡胶树(Hevea brasiliensis, Hb)KT/HAK/KUP蛋白氨基酸序列,利用软件MEGA 6.0采用Neighbor-Joining法,并进行1000次bootstrap统计学检验构建进化树(图3)。从图中可以看出,KT/HAK/KUP家族在进化上分为4个大的分支Ⅰ、Ⅱ、Ⅲ和Ⅳ,其中Ⅰ和Ⅱ又分为Ⅰ- a,Ⅰ-b和Ⅱ-a,Ⅱ-b。在每个分支中橡胶树和木薯聚在一起,这和二者的亲缘关系更近是一致的。在Ⅱ-b中HbKUP27和其他4个成员HbKUP28~ HbKUP31聚在一起,而这5个成员对应的基因成簇出现在8号染色体上,通过串联复制而来,其中HbKUP27在进化过程中出现基因结构缺失。从后续表达分析可以看出HbKUP27失去功能不表达,而HbKUP30和HbKUP31则主要在胶乳中起作用。
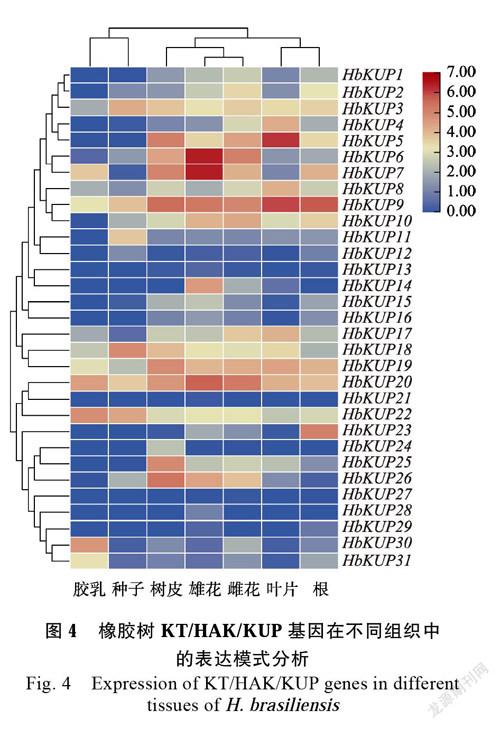
2.4 橡胶树KT/HAK/KUP基因家族成员的表达分析
利用本实验室已有转录组测序数据对31个橡胶树KT/HAK/KUP基因在不同组织、不同叶片发育时期和乙烯利处理后不同时间点的表达情况进行了分析。其中不同组织包括胶乳、树皮、叶片、根、种子、雌花和雄花。从图4可以看出,部分家族成员功能相对比较保守,在各组织中均有表达,如HbKUP3、HbKUP8、HbKUP9、HbKUP18、HbKUP19、HbKUP20和HbKUP22(图4);部分成员在胶乳中表达丰度较高,如HbKUP20、HbKUP22和HbKUP30;叶片中HbKUP5和HbKUP9表达丰度相对较高,而树皮中HbKUP5、HbKUP7、HbKUP9和HbKUP26表达丰度较高。另外,在根中HbKUP9的表达丰度也是最高的,说明HbKUP9在各组织钾离子运输中具有重要作用,而在进化过程中又进化出组织特异性表达的新成员。
KT/HAK/KUP基因在叶片发育过程中的表达情况如图5所示,在叶片发育过程中部分家族成员的表达发生明显变化,如HbKUP5和HbKUP9明显上调表达,在稳定态叶中表达丰度达到最高;HbKUP6呈明显下调表达,在库叶中表达丰度较高;而HbKUP14的表达呈先上升后下降的趋势,主要在变色期的叶中表达。以上基因的表达可能参与调节叶片细胞的渗透压,进而影响叶片的生长发育。
为分析乙烯刺激对胶乳中基因表达的影响,进行了乙烯利刺激实验。结果表明,在乙烯刺激前后,HbKUP20、HbKUP22和HbKUP30的表达丰度都相对较高,这和前面不同组织表达分析结果一致。HbKUP22、HbKUP30和HbKUP31受乙烯刺激影响,上调表达,HbKUP30和HbKUP31上升趋势最为明显(图6)。从前面基因的染色体定位分析可以看出,HbKUP30和HbKUP31是在8号染色体,通过串联复制进化而来,这2个基因可能是在橡胶树进化过程中产生的用来调控乳管渗透压的特异表达基因。在割胶过程中,乳管细胞将失去大量细胞质,需要补充大量水分和碳源,渗透压的调节显得尤为重要。另外,钾在植
物中还参与多种酶的活化,具有重要的生物学功能。由以上结果可以推测HbKUP30和HbKUP31在橡胶树乳管胶乳再生过程中具有重要作用。
低温寒害是制约我国橡胶种植面积的重要因素,已有文献表明KT/HAK/KUP在植物抗逆方面具有重要功能。因而本研究对KT/HAK/KUP基因在低温胁迫下的表达情况进行了分析。结果表明,HbKUP5和HbKUP9在叶片中表达相对较高,和前面基因在不同组织中的表达分析结果一致,但HbKUP5和HbKUP9在低温敏感品种‘热垦501’与抗寒品种‘93-114’叶片中的表达存在明显不同,在4℃低温处理1 h后HbKUP5和HbKUP9在‘热垦501’中表达呈上升趋势,而在‘93-114’中表达呈明显的下降趋势(图7),这可能和2种材料对低温的响应机制不同有关。由以上结果可以推测,HbKUP5和HbKUP9的表达和橡胶树的抗寒具有一定的相关性。
3 讨论
天然橡胶是一种重要的工业原料和军事战略
物质,巴西橡胶树是天然橡胶的主要来源。天然橡胶树的生成需要消耗大量的原料,钾作为植物生长所必需的三大主要元素之一,在调控植物生长发育和参与植物逆境胁迫应答等方面具有重要作用,钾离子对于橡胶树胶乳的再生也必然具有重要作用。本研究从橡胶树基因组中鉴定出31个KT/HAK/KUP家族基因,在进化上主要分为4个大的分支,从水稻[12]、杨树[5]、木薯[13]等不同植物的研究可以看出,KT/HAK/KUP家族成员的数目在进化过程中出现了较大幅度的扩增,主要通过染色片段复制和基因的串联复制实现。但通过分析可以发现,其他植物的串联重复大多是2~3个成员的串联重复[18-19],而在橡胶树中却出现了多达5个成员的串联重复。从基因的表达还可以看出,这5个串联重复成员中仅2个成员HbKUP30和HbKUP31主要在胶乳中表达,其他成员在各组织中均不表达或表达量很低,在乙烯利处理后HbKUP30和HbKUP31的表达都出现了明显的上升趋势,这和乙烯处理后胶乳产量呈上升趋势一致。从橡胶树的进化来看,橡胶树乳管是在进化过程中产生的一种特化组织,乳管中合成了大量的次生代谢产物天然橡胶烃,而这个过程需要大量的原料補充,这其中也包括钾离子,因而橡胶树在进化过程中进化出了适应这一功能的基因HbKUP30和HbKUP31,进而保障了乳管中天然橡胶生物合成的顺利进行。本研究从基因家族成员的鉴定、染色体定位、基因结构、系统进化和表达模式等方面对橡胶树KT/HAK/KUP各家族成员进行了系统的分析,从中筛选出橡胶树乳管和叶片等组织中主要表达的成员。研究结果为深入研究KT/HAK/KUP基因家族的功能和培育高钾抗逆的橡胶树品种提供了理论基础。
参考文献
[1] LI W H, XU G H, ALLI A, YU L. Plant KT/HAK/KUP K+ transporters: Function and regulation[J]. Seminars in Cell and Developmental Biology, 2018, 74: 133-141.
[2] YANG Z F, GAO Q S, SUN C S, LI W J, GU S L, XU C W. Molecular evolution and functional divergence of HAK potassium transporter gene family in rice (Oryza sativa L.)[J]. Journal of Genetics and Genomics, 2009, 36(3): 161-172.
[3] ZHANG Z B, ZHANG J W, CHEN Y J, LI R F, WANG H Z, WEI J H. Genome-wide analysis and identification of HAK potassium transporter gene family in maize (Zea mays L.)[J]. Molecular Biology Reports, 2012, 39(8): 8465-8473.
[4] HYUN T K, RIM Y, KIM E, KIM J S. Genome-wide and molecular evolution analyses of the KT/HAK/KUP family in tomato (Solanum lycopersicum L.)[J]. Genes & Genomics, 2014, 36(3): 365-374.
[5] HE C Y, CUI K, DUAN A G, ZENG Y F, ZHANG J G. Genome-wide and molecular evolution analysis of the Poplar KT/HAK/KUP potassium transporter gene family[J]. Ecology and Evolution, 2012, 2(8): 1996-2004.
[6] YOYNG J P, MARKUS G, JULIAN I S, MYEON H C. High-affinity k+ transport in arabidopsis: athak5 and akt1 are vital for seedling establishment and postgermination growth under low-potassium conditions[J]. Plant Physiology, 2010, 153 (2): 863-875.
[7] TOMOAKI H, MITSUO S, TOMOYUKI O, KOICHIRO T, PULLA K N, MAKI K, ATSUHIKO S, HIDEKI N. Rice sodium-insensitive potassium transporter, OsHAK5, confers increased salt tolerance in tobacco BY2 cells[J]. Journal of Bioscience and Bioengineering, 2011, 111(3): 346-356.
[8] LI Y, PENG L R, XIE C Y, SHI X Q, DONG C X, SHEN Q R, XU Y C. Genome-wide identification, characterization, and expression analyses of the KT/HAK/KUP potassium transporter gene family reveals their involvement in K+ deficient and abiotic stress responses in pear rootstock seedlings[J]. Plant Growth Regul, 2018, 85 (2): 187-198.
[9] HU J, MA Q, KUMAR T, DUAN., ZHANG J, YUAN H, WANG Q, KHAN S, WANG P, WANG S. ZxSKOR is important for salinity and drought tolerance of Zygophyllum xanthoxylum by maintaining K+ homeostasis[J]. Plant Growth Regulation, 2016, 80(2): 195-205.
[10] ADAMS E, SHIN R. Transport, signaling, and homeostasis of potassium and sodium in plants[J]. Journal of Integrative Plant Biology, 2014, 56(3): 231-249.
[11] MARKUS G, PASCAL M. Potassium transporters in plants-Involvement in K+ acquisition, redistribution and homeostasis[J]. FEBS Letters, 2007, 581(12): 2348-2356.
[12] MADHUR G, QIU X H, WANG L, XIE W B, ZHANG C J, XIONG L Z, LIAN X M, ZHANG Q F. KT/HAK/KUP potassium transporters gene family and their whole-life cycle expression profile in rice (Oryza sativa)[J]. Molecular Genetics & Genomics, 2008, 280(5): 437.
[13] OU W J, MAO X, HUANG C, TIE W W, YAN Y, DING Z H, WU C L, XIA Z Q, WANG W Q, ZHOU S Y, LI K M, HU W. Genome-wide identification and expression analysis of the KUP family under abiotic stress in cassava (Manihot esculenta Crantz)[J]. Frontiers in Physiology, 2018, 9: 17.
[14] CHEN C J, CHEN H, ZHANG Y, HANNAH R T, MARGARET H F, HE Y H, XIA R. TBtools: An integrative toolkit developed for interactive analyses of big biological data[J]. Molecular Plant, 2020, 13(8): 1194-1202.
[15] KOICHIRO T, GLEN S, DANIEL P, ALAN F, SUDHIR K. MEGA6: molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology & Evolution, 2013, 30(12): 2725- 2729.
[16] XIAO X H, YANG M, SUI J L, JIYAN QI, YONGJUN FANG, SONGNIAN HU, CHAORONG TANG. The calcium-dependent protein kinase (CDPK) and CDPK-related kinase gene families in Hevea brasiliensis-comparison with five other plant species in structure, evolution, and expression[J]. Febs Open Bio, 2017, 7(1): 4-24.
[17] LI B, DEWEY C N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome[J]. BMC Bioinformatics, 2011, 12(1): 323-323.
[18] ZHOU J, ZHOU H J, CHEN P, ZHANG L L, ZHUJ T, LI P F, YANG J, KE Y Z, ZHOU Y H, LI J A, DU H. Genome-wide survey and expression analysis of the KT/HAK/ KUP family in Brassica napus and its potential roles in the response to K+ deficiency[J]. international journal of molecular sciences, 2020, 21(24): 9487.
[19] YANG X, ZHANG J J, WU A M, WEI H L, FU X K, TIAN M M, MA L, LU J H, WANG H T, YU S X. Genome-wide identification and expression pattern analysis of the HAK/KUP/KT gene family of cotton in fiber development and under stresses[J]. Frontiers in Genetics, 2020, 11: 566469.

