多特征融合的乳腺癌组织病理学图像识别的方法
乔世昌,胡红萍,郝 岩,白艳萍
(中北大学 a.理学院; b.信息与通信工程学院, 太原 030051)
癌症是世界范围内致死率最高的疾病之一,其中乳腺癌(breast cancer)是全球女性死亡率最高的癌症之一[1]。乳腺癌组织病理学检查是乳腺癌诊断的“金标准”,因此对乳腺癌组织病理学图像的识别成为了医学图像处理领域的研究热点。常用的乳腺癌组织病理学图像识别算法包括基于传统的人工特征提取方法[2],以及基于深度学习的分类方法[3-4]。人工提取乳腺癌组织病理学图像特征的方法需要手动设计图像中感兴趣的区域,并提取其中的特征,再对提取的特征进行选择,在分类精度方面没有基于深度学习算法得到的分类精度高。但基于深度学习的方法对乳腺癌组织病理学图像进行分类需要大量的被标记的图像数据,而在实际中,大量被标记的乳腺癌组织病理学图像是很缺乏的。Araújo等[5]利用卷积神经网络(convolutional neural network,CNN),以及CNN与支持向量机(support vector machine,SVM)相结合的方法对Bioimaging 2015数据集进行二分类,其分类精度分别为80.6%和83.3%。Wang等[6]应用所提出的方法对Bioimaging 2015数据集进行四分类,得到了83.0%的分类精度。Spanhol等[7]提出了BreaKHis乳腺癌组织病理学图像数据集,并对该数据集进行肿瘤的良恶性分类,其分类的准确范围为80%~85%。
本文提取了乳腺癌组织病理学图像的纹理特征以及颜色特征并将其进行融合。首先对乳腺癌组织病理学图像进行染色分离处理,提取了染色分离后4个方向上的灰度共生矩阵特征和小波特征以及Tamura纹理特征,并根据颜色自动相关图提取原始图像的颜色特征,同时也提取染色分离前水平方向上的灰度共生矩阵特征作为纹理信息的补充,最后将提取到的特征进行融合,并输入到SVM中进行训练,从而实现乳腺癌组织病理学图像的分类,并得到83.33%的分类准确率。实验直接对原始尺寸大小的图像进行特征提取,不需进行裁剪、平移等扩充图像数据的步骤,可操作性较强。
1 研究方法
由于组织病理学图像自身的特点,实现乳腺癌组织病理学图像的自动分类是一项极具挑战性的工作。组织病理学图像中的细胞重叠现象、颜色分布不均匀以及图像之间细微的差异性等因素,为乳腺癌组织病理学图像特征的提取带来很大困难[8]。常见的图像特征包括图像的纹理特征、形状特征、颜色特征以及灰度分布等。本文通过灰度共生矩阵、小波变换以及Tamura特征的方法对乳腺癌组织病理学图像提取了纹理特征,并利用颜色自动相关图提取了其颜色特征,进而将提取到的特征进行融合,并输入到分类器中以实现乳腺癌的自动分类,图1为本文方法整体框架。

图1 本文方法框架
1.1 灰度共生矩阵
灰度共生矩阵(gray level co-occurrence matrix,GLCM)是一种常用的纹理统计分析方法[9],图像纹理特征的差异由像素值的排列分布来反映。通过空间特性来描述灰度图像的纹理特征,能反映出图像灰度关于相邻间隔、方向以及变化幅度上的空间信息。取大小为N×N的图像中任意一点(x,y)以及偏离于该点方向θ,距离d的另一点(x+a,y+b),其灰度值分别为p1、p2,并统计灰度值为p1、p2同时出现的概率p。概率p的计算如下式所示:
P(i,j,d,θ)={[(x,y),(x+a,y+b)]|f(x,y)=
p1,f(x+a.y+b)=p2}
(1)
其中,x,y=0,1,…,N-1。图像上所有在θ方向,距离为d,灰度值为p1、p2的像素点对出现的频率构成灰度共生矩阵,并通过计算得到共生矩阵的部分特征值,这些特征值分别代表图像的某些纹理特征。θ常取值为0°、45°、90°和135°,显然a=0,b=1时,θ=0°;a=-1,b=1时,θ=45°;a=1,b=0时,θ=90°;a=1,b=1时,θ=135°[10]。给出图像Ⅰ,如图2所示,则该图像以距离为1,90°方向上的灰度共生矩阵为
(2)

图2 图像矩阵Ⅰ
1.2 小波变换
小波变换的实质是将信号分解为一系列小波函数的叠加。由于小波变换在时域、空域以及频域同时具有良好的局部化性质,而图像的纹理描述是图像的局部区域特征,并且具有尺度性,当分辨率不同时,同一局部会表现出不同的纹理特征。应用小波变换将图像分解为它的多分辨率表示,且图像的多分辨率表示提供了图像纹理沿频率轴方向基于尺度的信息分布[11]。而图像自身的特点决定了在将小波变换应用到图像处理中时,需要使用二维小波变换。令f(x1,x2)∈L2(R2)表示一个二维信号,ψ(x1,x2)表示二维基本小波。则二维小波变换为:
(3)

本文应用小波变换对乳腺癌组织病理学图像进行多尺度分解,得到各尺度上的近似细节分量、水平细节分量、垂直细节分量以及对角细节分量。由于近似细节分量是原图像的低频分量,水平细节分量、垂直细节分量以及对角细节分量是原图像的高频分量,而图像的高频分量包含了其主要的纹理信息,因此取不同尺度上各子图高频分量的能量作为图像的纹理特征。
1.3 颜色自动相关图
颜色特征是彩色图像基本的视觉特征,与其他视觉特征相比,对图像本身的方向,尺寸以及视角的依赖性较小,而与图像中所包含的物体或场景相关。常用的提取颜色特征方法有颜色直方图、颜色矩等。而颜色直方图描述的是不同色彩在整幅图像中所占的比例,无法描述图像中的对象。颜色矩一般只有9个分量(3个颜色分量,每个分量上3个低阶矩),特征维数较少,难以完整描述图像的颜色信息。因此,由于颜色直方图以及颜色矩的局限性,本文选取颜色自动相关图(color auto-correlogram)来描述乳腺癌组织病理学图像的颜色特征。
颜色自动相关图是在颜色相关图的基础上得到的。颜色相关图不仅可以反映一幅图像中某种颜色的像素数量占整个图像的比例,同时还可以反映不同颜色对之间的空间相关性[12]。对图像I而言,令Ic(i)表示颜色为c(i)的所有像素,则颜色相关图可以表示为:
(4)
其中,|p1-p2|表示p1和p2的距离,Pr为求概率的运算。即颜色相关图可以看作是一张用颜色对
1.4 Tamura纹理特征
Tamura等[13]基于对纹理视觉感知的心理学研究提出了一种纹理特征的描述方法,并定义了6个用于描述纹理的特征量,分别为粗糙度、对比度、方向度、线性度、规则度和粗略度。这6个特征量的具体定义为:
1) 粗糙度
粗糙度反映了图像灰度变化的剧烈程度,纹理粒度越大则该纹理图像越粗糙。其定义为:
(5)
其中,M、N为图像尺寸大小,(i,j)为图像像素位置,Skbest=2k为整幅图像中各像素最优尺寸,k为参与计算的像素范围。
2) 对比度
对比度是对像素强度分布情况的统计,反映了灰度图像中最亮和最暗灰度的层级,其差异范围决定了对比度的大小。其定义为:
(6)
其中,α4=μ4/σ4为灰度统计量的峰态,μ4表示四阶矩均值,σ为图像灰度值的均方差。
3) 方向度
方向度反映了图像纹理沿某方向集中的强度。其定义为:
(7)
其中,p表示某个峰值,np为峰的数目,ωp表示围绕方向角的峰值与谷值的范围,r为与角度θ量化水平相关的归一化因子,φp为第p个峰值的位置,HD(φ)为相应方向上的直方图。
4) 线性度
线性度反映图像纹理是否具有线性结构。其定义为:
(8)
其中,Pd(i,j)表示距离为d的一组元素,方向编码分别为i、j时出现的次数,m为划分角度的数量。
5) 规则度
当图像中存在某一局部区域与整幅图像纹理特征不一致时,图像的规则度减弱。其具体定义为:
Freg=1-η(σcrs+σcon+σdir+σlin)
(9)
其中,σcrs、σcon、σdir、σlin分别为Fcrs、Fcon、Fdir、Flin的标准方差,η为标准化因子。
6) 粗略度
粗略度是粗糙度和对比度2个特征量的合成。其定义为:
Frgh=Fcrs+Fcon
(10)
2 多特征融合的乳腺癌图像识别
2.1 数据集
本文所研究的乳腺癌组织病理学图像数据来自Bioimaging 2015乳腺癌组织病理学图像分类挑战,所有图像均在相同的采集条件下数字化,放大倍数为200倍,像素大小为0.42 μm×0.42 μm(2 048×1 536像素)。图像经过苏木精与伊红(hematoxylin and eosin,H&E)染色,由于苏木精和伊红的特性,组织病理学图像中的蛋白质会被伊红染成粉红色,而苏木精则将细胞核染成蓝紫色。每幅图像均有四类标记,分别为正常组织(Normal)、良性病变(Benign)、原位癌(In Situ)以及浸润性癌(Invasive),其中正常组织与良性病变为非癌类,而原位癌与浸润性癌为癌类,如图3所示。
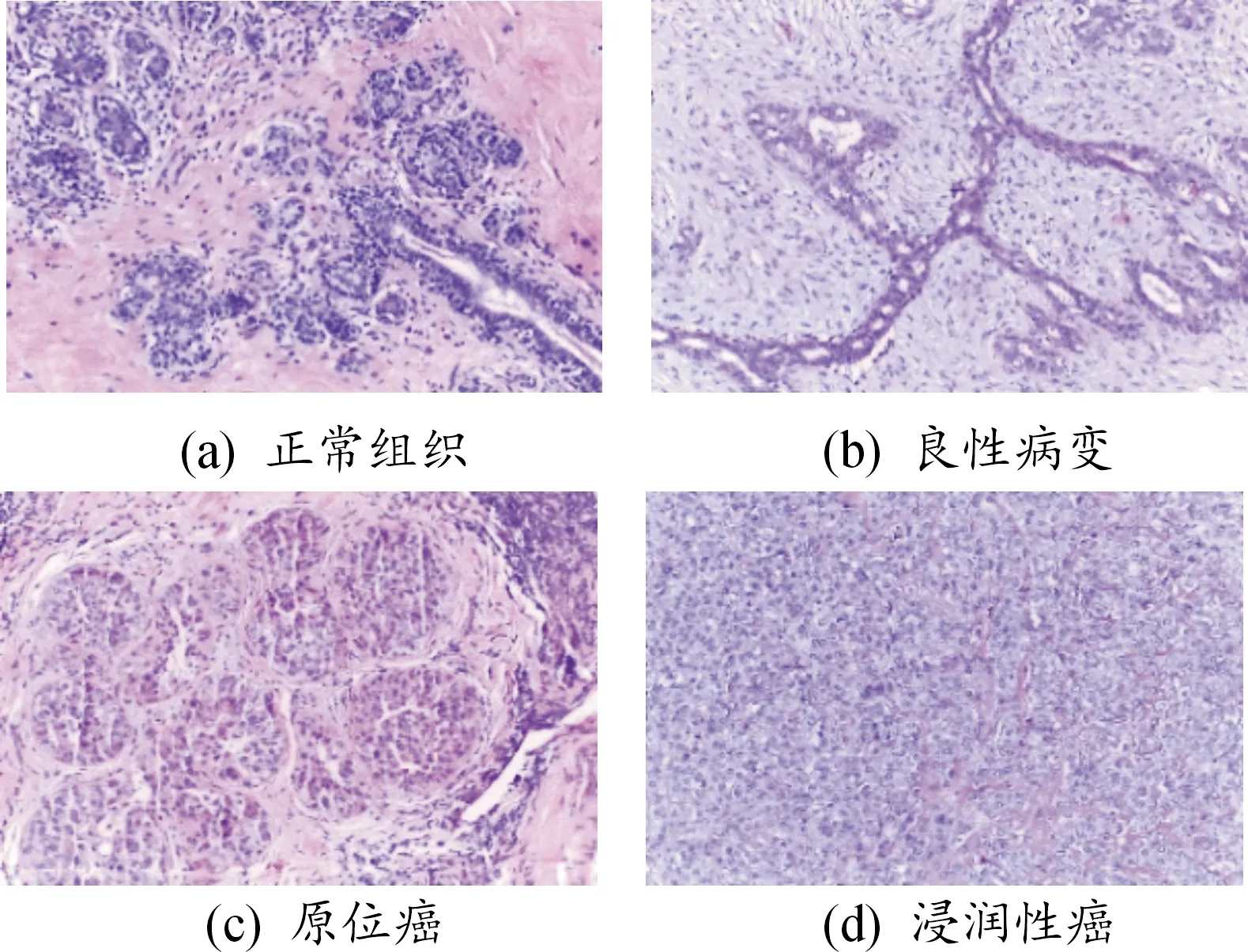
图3 Bioimaging 2015数据集中乳腺癌组织病理学图像示例
数据集中的类别标记由两名病理学专家负责,提供了图像内容的诊断,且专家之间存在意见分歧的病理图像被丢弃。该数据集由249幅图像的训练集和36幅图像的测试集组成(其中有16幅为模糊度增加的图像,称之为“扩展”测试集)。研究目标是利用机器学习的方法对该数据集进行客观的病理分类,将其分为癌类与非癌类。表1为该数据集各类数量分布。
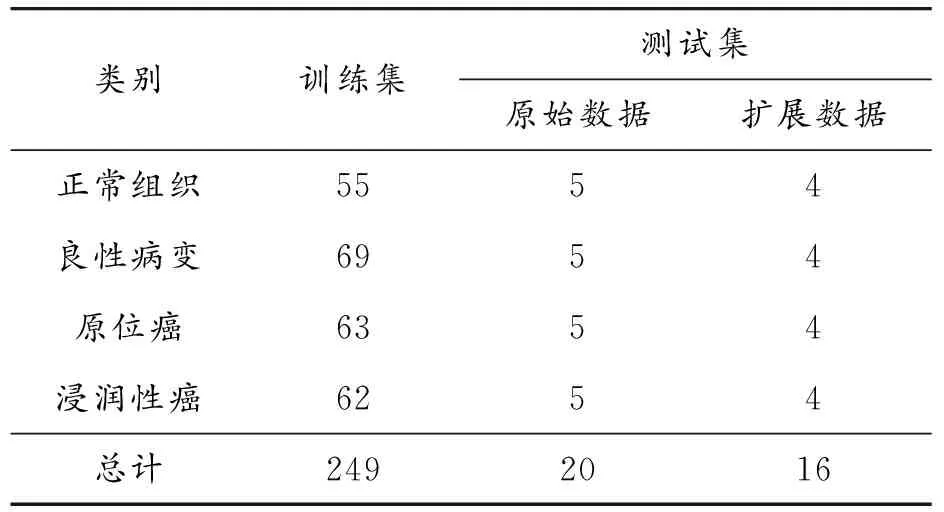
表1 Bioimaging 2015数据集各类组织病理学图像分布
2.2 预处理
组织病理学图像的染色分离对于病理学家和计算机辅助诊断都可能有帮助。用于自然图像的分离技术可能会导致组织病理学图像染色组织的结构特性发生改变,并产生不良的颜色失真。常用于H&E图像染色分离的方法是将RGB空间转换到光密度上实现的。由于染色分离是对每个染色剂密度图的估计,因此需考虑RGB颜色和每个像素的染色密度之间的关系:染色组织在一定的光谱中会根据其吸收染色剂的类型和数量使光衰减变弱。
令I∈Rm×n为透射光强度,其中m=3为RGB图像的通道数,n为图像像素总数,令I0为入射光强度,则光密度V可表示为[14]:
(11)
令V=WH,即给定观测矩阵V,目标是找到染色外观矩阵W和染色密度图矩阵H。即解决以下问题:
(12)
由于该问题是一个非凸优化问题,可能会收敛到局部最优而不是全局最优,并得出不理想的染色向量,因此Vahadane等[15]提出了一个稀疏非负矩阵分解(sparse non-negative matrix factorization,SNMF)框架,对式(12)添加稀疏约束:
(13)
其中,λ是稀疏正则化参数,j表示染色剂种类(j=1,2,…,r),对于H&E图像,r=2,并利用LARS-LASSO算法[16]对式(7)求解,得到W与H,从而实现H&E图像的染色分离。图4为采用该方法对Bioimaging 2015数据集中乳腺癌组织病理学图像的染色分离结果。
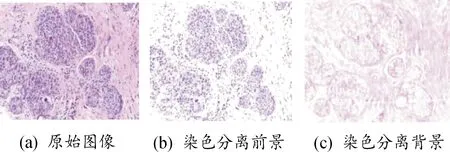
图4 乳腺癌组织病理学图像的染色分离结果
2.3 特征提取与分类
在图像识别中,原始图像具有大量的冗余信息,严重影响图像的分类精度。因此,选择合适的特征提取方法对图像识别至关重要,提取到有效信息的同时也降低了特征维度,避免了维数灾难问题。本文主要提取了乳腺癌组织病理学图像的纹理特征和颜色特征。具体来讲,不需要对该数据集中的图像进行裁剪、旋转、平移等扩充图像数据的操作,而是直接对乳腺癌组织病理学图像提取特征。在对图像进行染色分离之后,利用GLCM提取特征时,分别计算步长为1,角度为0°、45°、90°和135° 4个方向上的自相关、对比度、相异性、能量、熵、和方差、最大概率、方差、和平均值、和熵、聚类突出特征、聚类阴影特征、方差差异性、差异熵、反差分、逆差距,以及通过2种方法分别计算得出的相关性、同质性以及相关性的信息量度,构成一个88维的特征向量。在通过小波变换对乳腺癌组织病理学图像提取特征时,采用coif5小波基函数对图像进行3层分解,并提取每层高频分量的能量作为特征,共同构成一个9维的特征向量。另外,采取Tamura纹理特征方法提取了粗糙度、对比度、方向度、线性度、规则度和粗略度等6个纹理特征,并根据颜色自动相关图,对染色分离前的图像提取了128个分量作为颜色特征。本文采用基于径向基核函数支持向量机作为分类器对肿瘤的良恶性进行分类,其中令惩罚参数c为2,核函数参数g为1。
2.4 实验结果
本文将提取到的几种纹理特征以及颜色特征,分别输入到SVM分类器中进行乳腺癌图像分类。同时也比较了不变矩和Gabor特征等方法的分类效果。为验证图像预处理阶段中H&E图像染色分离对分类效果的影响,将上述各个特征分别进行了染色分离前后的分类效果比较。结果表明,对乳腺癌组织病理学图像染色分离后的分类精度比对原始图像直接进行特征提取再分类,在效果上有明显提升,如表2所示。另外,与其他特征相比,由不变矩提取到的特征分类精度较低,因此在特征融合中不考虑该方法。
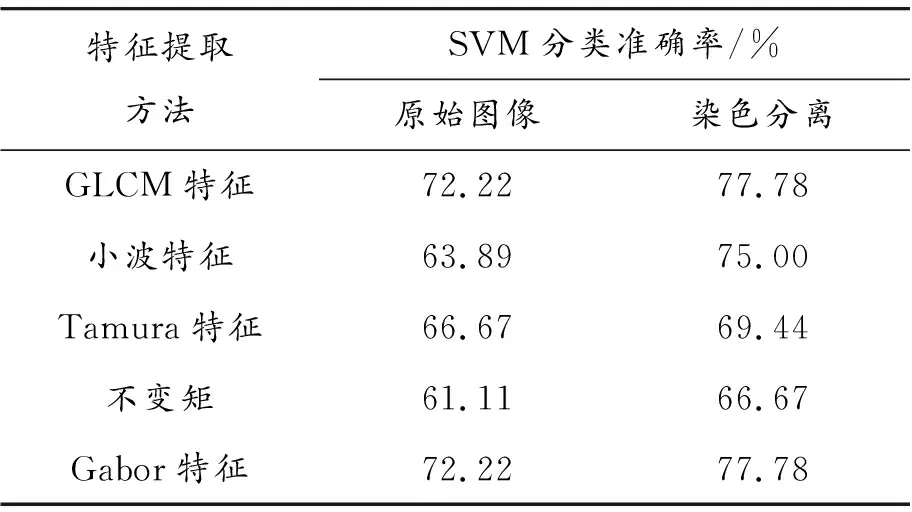
表2 基于SVM分类器的单一特征分类比较
在提取乳腺癌组织病理学图像的颜色特征时,将本文所采用的颜色自动相关图与颜色直方图以及颜色矩等的方法进行了比较,结果表明,所采用的颜色自动相关图方法对癌类与非癌类的识别效果最佳,如表3所示。

表3 基于SVM分类器的颜色特征分类比较
选取表2和表3中表现较好、维数较低的特征进行融合。以上特征提取方法分类效果最好的是GLCM特征和Gabor特征,但由于Gabor特征维数为7 680维,特征维数较大,与其他特征融合以后,分类效果不佳,因此,在特征融合时将这种特征排除在外,在GLCM特征上依次添加小波特征和颜色自动相关图以及Tamura纹理特征,分类准确率分别为77.78%、80.56%和80.56%。即将4个方法提取到的特征进行融合,分类精度得到了明显提高。另外,考虑到染色分离后的图像虽然突出了细胞核的分布、大小形态以及聚集密度等特点,但可能会减弱图像中细胞整体的分布轮廓。比如原位癌的病变部分主要发生在导管内,并未发生扩散,而浸润性癌中发生癌变的细胞,穿过导管壁并侵入到乳腺周围组织,这些分布信息可能在染色分离之后的图像中有所损失,因此,在将以上特征融合的基础上,考虑另外再提取原始图像步长为1,水平方向上的22维GLCM特征,以补充乳腺癌组织病理学图像纹理特征的信息。实验结果表明,将这5种特征融合之后,分类精度提高到83.33%,如表4所示,其中F1、F2、F3、F4、F5分别表示上述4方向上的88维GLCM特征、小波特征、颜色自动相关图、Tamura纹理特征以及水平方向上的22维GLCM特征。同时,在实验过程中,我们也对以上方法中的任意2种、3种以及4种特征进行了融合,但分类精度均未高于83.33%。
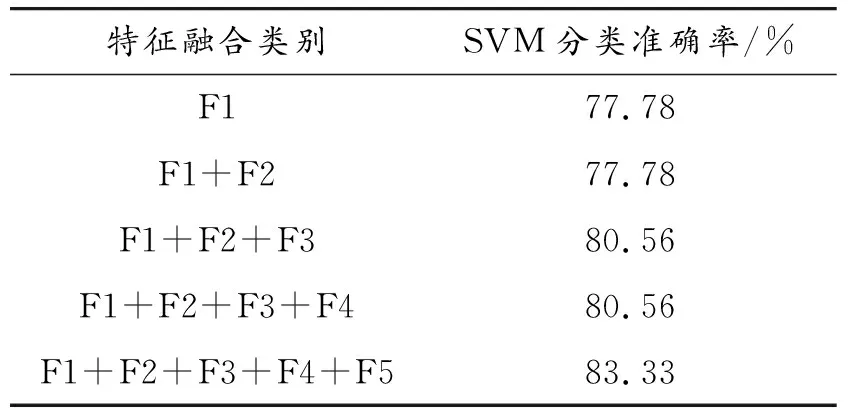
表4 基于SVM的多特征融合分类比较
另外,实验过程中,我们分别进行了5次独立实验,平均运行时间仅0.065 6 s,并将本文应用5种特征进行融合(F1+F2+F3+F4+F5)的方法与文献[5]中所采用的2种深度学习算法进行了比较,如表5所示。结果表明,文献[5]中最高的分类准确率为83.3%,但其运用的是深度学习算法,需要一系列的操作以扩充大量图像数据,且在训练中需要优化大量的参数,运行时间相对本文的方法较为缓慢,因此,本文提出的方法是一种快捷有效、易于实现且可行性强的方法。

表5 本文实验与相关文献分类结果对比
3 结论
通过对乳腺癌病理图像进行特征提取,减少了大量的冗余信息。由灰度共生矩阵、小波变换以及Tamura特征等方法提取了乳腺癌组织病理学图像染色分离后的纹理特征,同时根据颜色自动相关图提取了乳腺癌原始图像的颜色特征,并提取了图像染色分离前水平方向上的灰度共生矩阵特征作为纹理信息的补充,最后将提取到的特征进行融合,并通过SVM分类器进行分类。实验将Bioimaging 2015数据集分为癌类与非癌类,达到了83.33%的分类精度。实验表明:文中采用的方法是一种快捷有效、易于操作、正确率较高的乳腺癌组织病理学图像识别方法。在今后的工作中,将尝试进行乳腺癌组织病理学图像癌变部分的检测以及细胞核分割等方面的研究,进一步提高乳腺癌组织病理学图像的识别精度。

