橡胶树HbREF3基因的克隆及功能初步分析
录亿隆 刘星 张宇豪 刘开业 唐朝荣

摘 要:橡膠延伸因子(REFs)是橡胶粒子上的一种主要膜蛋白,在橡胶生物合成途径中具有延伸橡胶烃和稳定橡胶粒子等重要作用。对橡胶树不同组织的转录组测序发现,在胶乳中具有特异性的高表达,且其在HbREFs基因家族中表达水平也相对较高,仅次于表达丰度最高的。的开放阅读框为528 bp,编码175个氨基酸,对应的蛋白相对分子质量为19.62 kDa,理论等电点(pI)为5.28。系统进化树分析表明,HbREF3与橡胶树其他HbREFs虽均属于分组Ⅰ,但与HbREF1明显属于不同分支。HbREFs均含有REF保守结构域,但在保守motif的分布上,不同HbREFs蛋白含有保守motif数量不等,HbREF3比HbREF1多了一个motif。生信预测分析表明,该蛋白属于亲水性蛋白,无跨膜结构域,具有14个磷酸化位点。亚细胞定位分析发现HbREF3蛋白定位在内质网上,推测其可能参与橡胶粒子的形成和胶乳的再生。本研究结果为进一步阐明基因在橡胶树胶乳再生中的作用机制奠定了基础。
关键词:橡胶树;橡胶延伸因子HbREF3;生物信息学分析;亚细胞定位中图分类号:S794.1 文献标识码:A
Cloning and Primary Functional Analysis of Gene from
LU Yilong, LIU Xing, ZHANG Yuhao, LIU Kaiye, TANG Chaorong
College of Tropical Crops, Hainan University / Natural Rubber Cooperative Innovation Center of Hainan Province & Minstry of Education of PRC, Haikou, Hainan 570228, China
Natural rubber (NR) is an important strategic material related to national economy, livelihood and national security. It is of great significance to increase the production of natural rubber by focusing on the biosynthesis process of natural rubber. Rubber elongation factors (REFs) are mainly membrane proteins on rubber particles, which play an important role in extending rubber hydrocarbon and stable rubber particles during natural rubber biosynthesis process. It is an important approach for high-yield molecular marker-assisted selection breeding of rubber trees by identifying the key REFs gene that affects natural rubber yield. We found was highly and specifically expressed in latex, and its expression level was just less than that of which is the highest expression in latex in HbREFs gene family. The open reading frame of was 528 bp, encoding 175 amino acids. The molecular weight and theoretical isoelectric point of HbREF3 was 19.62 kDa and 5.28, respectively. Phylogenetic tree analysis showed that HbREF3 and HbREF1 belonged to different branches. All HbREFs proteins contain REF domain, and HbREF3 had one more motif than HbREF1. In silico studies showed that HbREF3 protein was a hydrophilic protein, without transmembrane domain, had 14 phosphorylation sites. The results indicated that HbREF3 may not be attached to the surface of rubber particles in a mosaic form, but directly or indirectly interacted with other proteins which embedded in the lipid monolayer membrane to form a complex attached on the surface of the rubber particles. The predicted localization result is different with HbREF1 which is embedded in the rubber particle monolayer membrane. The surface of rubber particles is composed of a monolayer of phospholipid membrane, the formation and development of rubber particles may be related to lipid synthesis. Subcellular localization analysis revealed that HbREF3 was localized in endoplasmic reticulum, we speculate that HbREF3 might be directly involved in rubber particle formation and latex regeneration by participating in lipid synthesis. Moreover, overexpression of REF/SRPPs family genes in nonrubber-producing plants (Arabidopsis and Capsicum) can significantly improve the drought resistance and accumulate a large number of lipid droplets. Therefore, we speculate that HbREF3 have a similar function to REF/SRPPs family proteins of nonrubber-producing plants in responding to drought in nature rubber harvesting process. We clarified the HbREF3 expression pattern and protein characteristics which would build a foundation for elucidating the molecular mechanism of in latex regeneration in rubber trees.
; rubber elongation factor HbREF3; bioinformatics analysis; subcellular localization
10.3969/j.issn.1000-2561.2022.03.007
天然橡胶(NR)因其具有较强的弹性、可塑性和良好的绝缘性,有效的散热性和低温下的延展性等独特性能,除了用于生产人们日常生活不可或缺的日用、医用等轻工业橡胶产品之外,主要作为战略物质用于生产军工和航空航天等国防器械,所以其经济价值高,应用前景广泛。近些年,虽然已有人工合成橡胶制品流向市场,但其性能不及天然橡胶,用途范围有限,所以天然橡胶的重要地位仍未被取代,尤其是在高精尖领域所用的橡胶制品仍以天然橡胶为原料,而且随着经济和科技的发展,人类在高精尖领域对天然橡胶的利用大大提高,需求量也不断增加。因此,研究天然橡胶的生物合成、提高天然橡胶产量具有重要意义。
橡胶树是大戟科橡胶树属植物,其胶乳的品质好、产胶量高,是全球天然橡胶的主要来源。橡胶树的胶乳来源于特化的乳管细胞,乳管细胞中有种特殊的细胞器负责胶乳主要成分橡胶烃的生物合成。这种特殊的细胞器被称为橡胶粒子,其形态主要呈球形和梨形,直径大小不一,据推测大橡胶粒子是由小橡胶粒子发育而来。橡胶粒子表面是附有多种膜蛋白的磷脂单层膜,它包裹着疏水性的聚异戊二烯,这些膜蛋白协同作用负责催化和调控天然橡胶分子链的合成与延伸。橡胶延伸因子(HbREFs)和小橡胶粒子蛋白(HbSRPPs)是橡胶树胶乳中丰度最高的蛋白,同属REF/SRPPs蛋白家族,它们与顺式异戊烯基转移酶(HbCPTs)和CPT结合蛋白(一种人Nogo B受体类似蛋白、也称HbCPTL)形成复合体定位在橡胶粒子上,是橡胶粒子膜蛋白的主要成分。
巴西橡胶树基因组显示,橡胶树中分别存在8个HbREFs和10个HbSRPPs。在非产胶植物中也有含REF结构域的蛋白,被称为REF/RPPs样蛋白,這些REF/SRPPs样蛋白是同源蛋白,均属于一个更大的植物胁迫相关蛋白家族。在橡胶树胶乳中,橡胶延伸因子占橡胶粒子膜蛋白的10%~60%,占胶乳总蛋白的6%~10%,在橡胶生物合成途径中具有十分重要的作用。在橡胶树胶乳中特异超高表达,是在胶乳中表达量最高的HbREFs家族基因,发现它的蛋白序列类似于羧基端被截断的HbSRPP1,推测其可能是由表达量较低的基因逐步进化而来。
本研究基于已发表的橡胶树基因组数据和转录组数据,克隆了在橡胶树胶乳中特异高表达的新的橡胶延伸因子基因(GeneID:110644926,与孙丽娜等报道的非同一个基因)。利用橡胶树、橡胶草、莴苣和拟南芥中REF/SRPPs的蛋白质序列构建了系统发育进化树,分析发现HbREF3可能是橡胶树物种中特异的一类橡胶粒子蛋白。并对其进行了蛋白质分子量、理论等电点、亲/疏水性、可能的磷酸化位点、跨膜结构域以及保守结构域等生物信息学分析。为进一步研究HbREF3蛋白在橡胶树中的作用,本课题组还预测了HbREF3蛋白的结构,并采用烟草瞬时表达系统分析其在细胞中的定位。本研究为深入阐明基因在橡胶烃生物合成中的作用机制提供了基础。
材料与方法
材料
1.1.1 植物材料 烟草使用的是本氏烟(种子为本实验室留存),在人工气候室(温度25℃、空气湿度60%,光照16 h/黑暗8 h)种植,选取生长约25 d的烟草叶片进行试验。
1.1.2 质粒及菌株 pCAMBIA1300-35S-EGFP购自淼灵质粒平台(http://www.miaolingbio.com/),内质网(ER)定位Marker和细胞核定位Marker的表达质粒为本实验室构建保存,pEASY-Blunt Zero克隆载体、大肠杆菌DH5α、根癌农杆菌GV3101购自北京全式金生物技术有限公司。
1.1.3 试剂 植物总RNA提取试剂盒(离心柱型Cat.RP55012)购自北京百泰克生物技术有限公司,逆转录试剂盒Revert Aid First Strand cDNA Synthesis kit(Cat.K1612)和限制性内切酶购自Thermo Fisher Scientific(中国),高保真DNA聚合酶试剂盒Prime STAR HS DNA Polymerase(Cat.R040A)购自TaKaRa公司,通用型DNA纯化回收试剂盒(Cat.DP204)购自天根生化科技(北京)有限公司,质粒提取试剂盒(Cat. C201-01)、同源重组试剂盒ClonExpress II One Step Cloning Kit(Cat.C112-01)购自诺维赞(南京)生物技术有限公司。
方法
1.2.1 橡胶树胶乳总RNA的提取和cDNA合成 胶乳总RNA的提取参照TANG等的方法,提取的总RNA使用InfiniteM200 Pro多功能酶标仪检测其浓度及质量,并使用2%的琼脂糖凝胶电泳对RNA的质量进一步确认。取1 μg RNA用Revert Aid First Strand cDNA Synthesis kit逆转录试剂盒进行逆转录,逆转录得到的cDNA置于–80℃备用。
1.2.2 基因的克隆及载体构建 利用本实验室发表的橡胶树基因组数据及注释文件查找基因的完整序列,运用Premier 5.0软件设计基因扩增引物HbREF3-F/HbREF3-R(表1)。PCR反应程序:95℃预变性5 min;95℃变性30 s,57℃退火30 s,72℃延伸50 s,32次循环后,72℃再延伸10 min,16℃待机。PCR产物经1%琼脂糖凝胶电泳检测后,将含有目的基因的条带用琼脂糖凝胶回收试剂盒纯化。将回收产物与pEASY-Blunt Zero克隆载体连接,转化DH5α感受态细胞后进行Sanger测序。使用限制性内切酶 Ⅰ和H Ⅰ对pCAMBIA1300-GFP载体进行双酶切,设计引物GFP-HbREF3-F/GFP-HbREF3-R(表1),扩增基因,通过同源重组构建pCAMBIA1300-载体。
1.2.3 生物信息学分析 使用在线网站(https:// ww.novopro.cn/tools/translate.html)将基因序列翻译为蛋白质序列,使用Prot Param (https://web.expasy.org/protparam/)在线软件预测HbREF3蛋白的理化性质。使用Prot Scale (https:// web.expasy.org/cgi-bin/protscale/protscale.pl)在线软件预测该蛋白亲/疏水性。使用TMHMM Server 2.0 (http://www.cbs.dtu.dk/services/TMHMM/)在线软件预测该蛋白是否有跨膜结构域。使用NetPhos(http://www.cbs.dtu.dk/services/Net)在线软件分析HbREF3蛋白中可能的磷酸化位点。使用SOPMA (https://npsa-prabi.ibcp.fr)和SWISS- MODEL (http://www.swissmodel.expasy.org)在线软件预测HbREF3蛋白的二级和三级结构。使用Pfam (https://pfam.xfam.org/)和MEME (https:// meme-suite.org/meme/)在线软件对HbREF3蛋白的结构域和保守序列进行分析。橡胶树、橡胶草、莴苣和拟南芥中REF/SRPPs家族氨基酸序列均參照已发表文章中提供的序列号从NCBI数据库下载,使用MEGA 7.0.26软件构建系统发育进化树。
1.2.4 HbREF3蛋白的亚细胞定位分析 将测序验证无误的表达载体pCAMBIA1300-利用化学转化法转化农杆菌GV3101,挑取的阳性单克隆用带筛选抗性的LB培养基摇至=0.4,然后在室温下5000 g离心10 min,收集菌弃上清,用SPARKES等的方法配置的侵染液重悬2次,调至=0.4,加入乙酰丁香酮至终浓度为100 μmol/L继续16℃、80 r/min避光摇2 h。用1 mL一次性无菌注射器注射生长约25 d的烟草叶片,避光处理30 h后,使用共聚焦显微镜观察荧光信号。
2 结果与分析
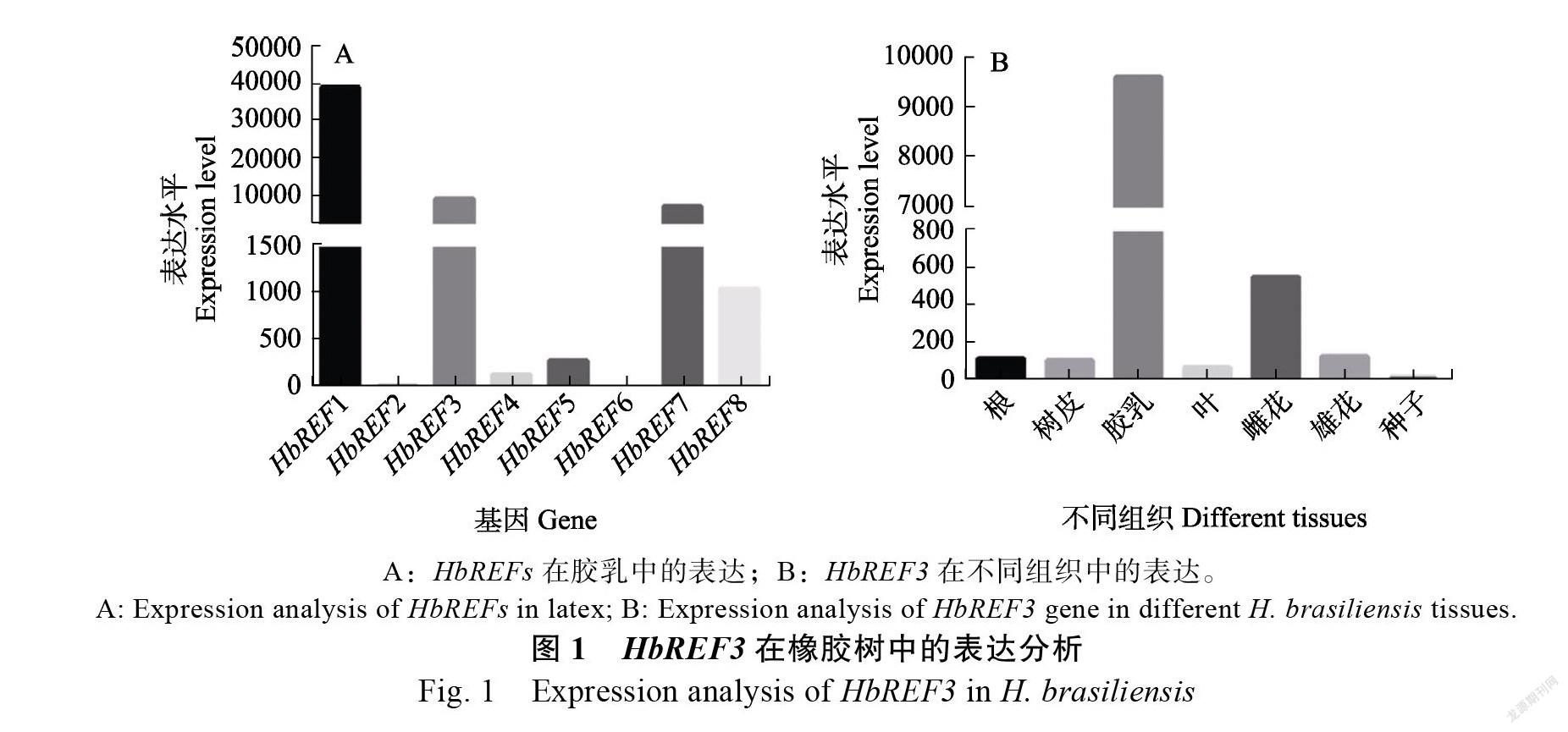
2.1 在橡胶树中的表达分析
利用实验室现有的橡胶树RNAseq数据,对在橡胶树中的表达进行可视化分析。数据表明,在HbREFs家族中除了基因之外,在橡胶树胶乳中的表达水平也比较高(图1A);而且在橡胶树不同组织中,在橡胶树胶乳中特异性表达(图1B)。
因此,本课题组对其在橡胶树胶乳中特异高表达的,且表达水平仅次于的进行进一步研究。
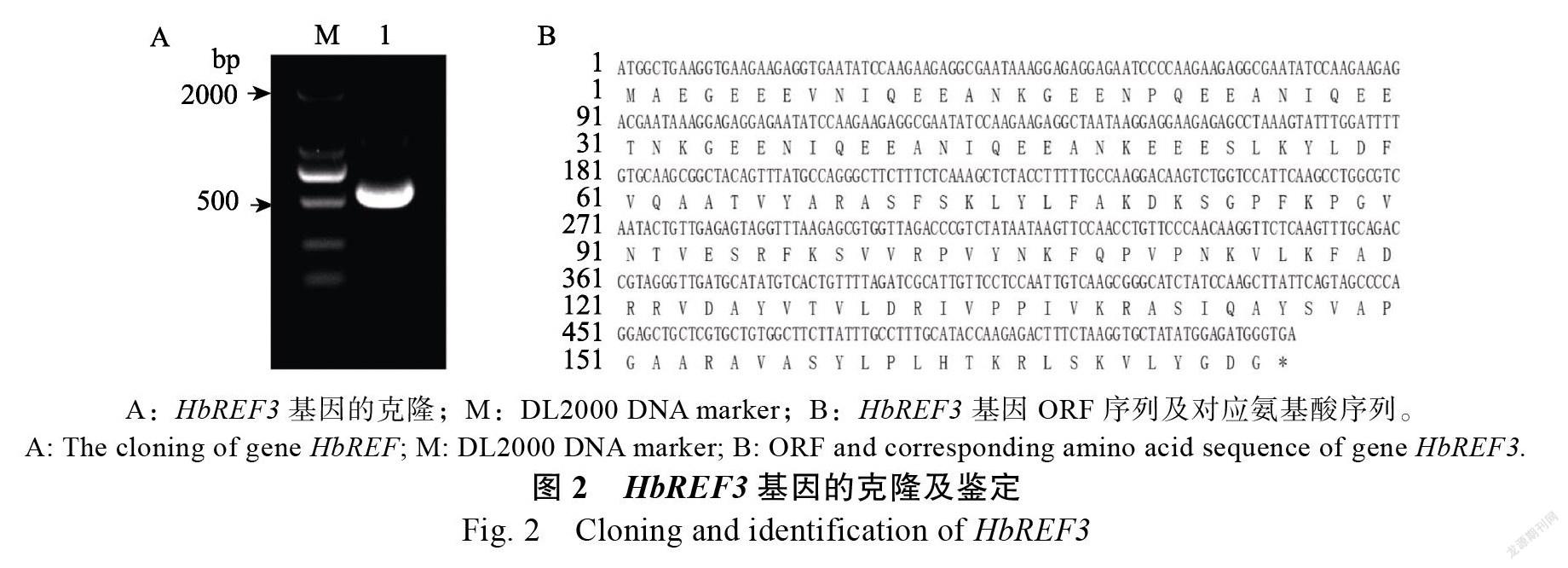
橡胶树基因的克隆与鉴定
本研究以橡胶树胶乳cDNA为模板,用引物HbREF3F/HbREF3R进行PCR扩增,得到约500 bp的片段(图2A)。通过Sanger测序分析发现扩增产物与基因组注释的基因开放阅读框序列完全一致,其全长528 bp,编码175个氨基酸(图2B)。
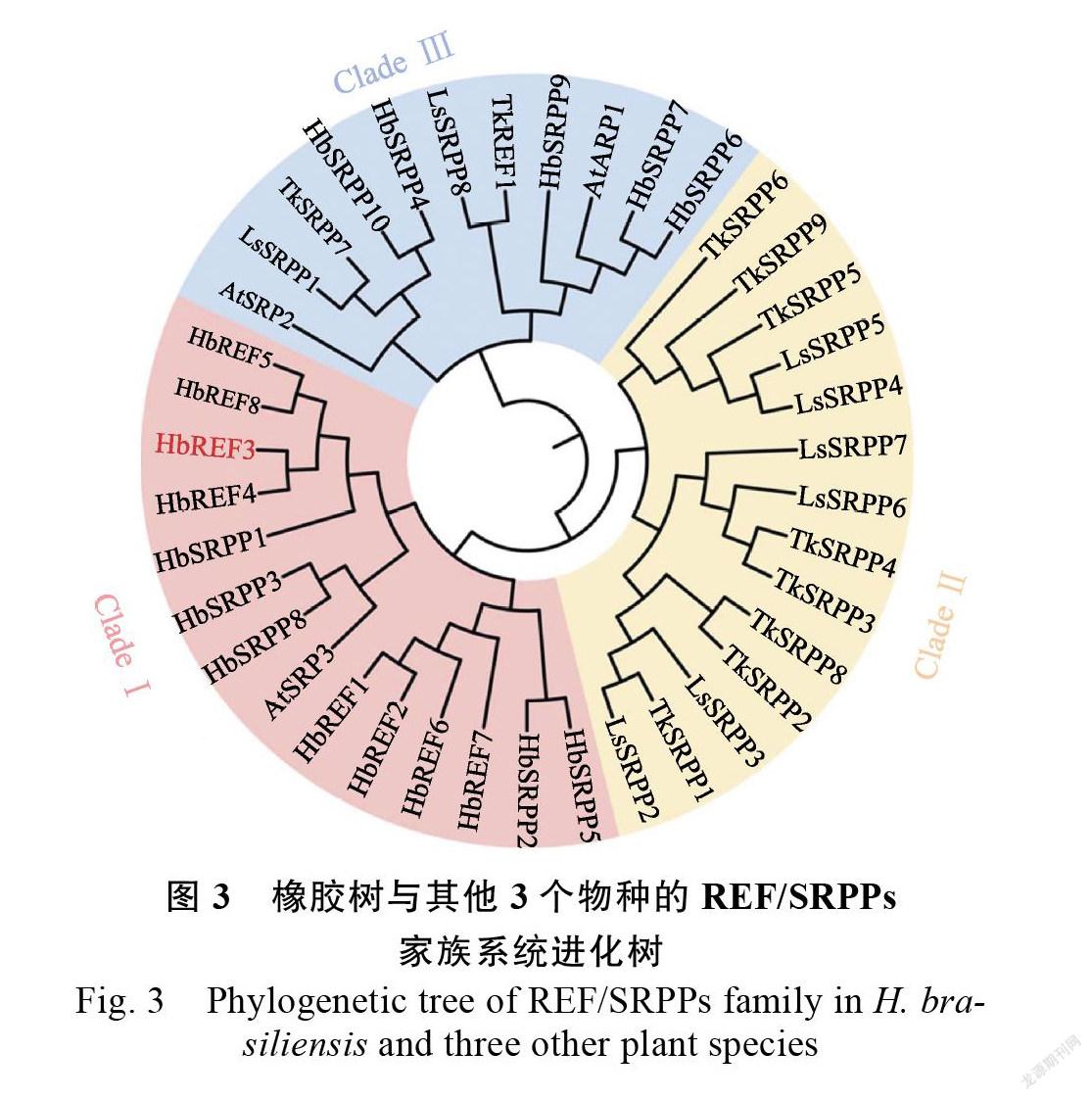
序列进化分析及蛋白保守结构域鉴定
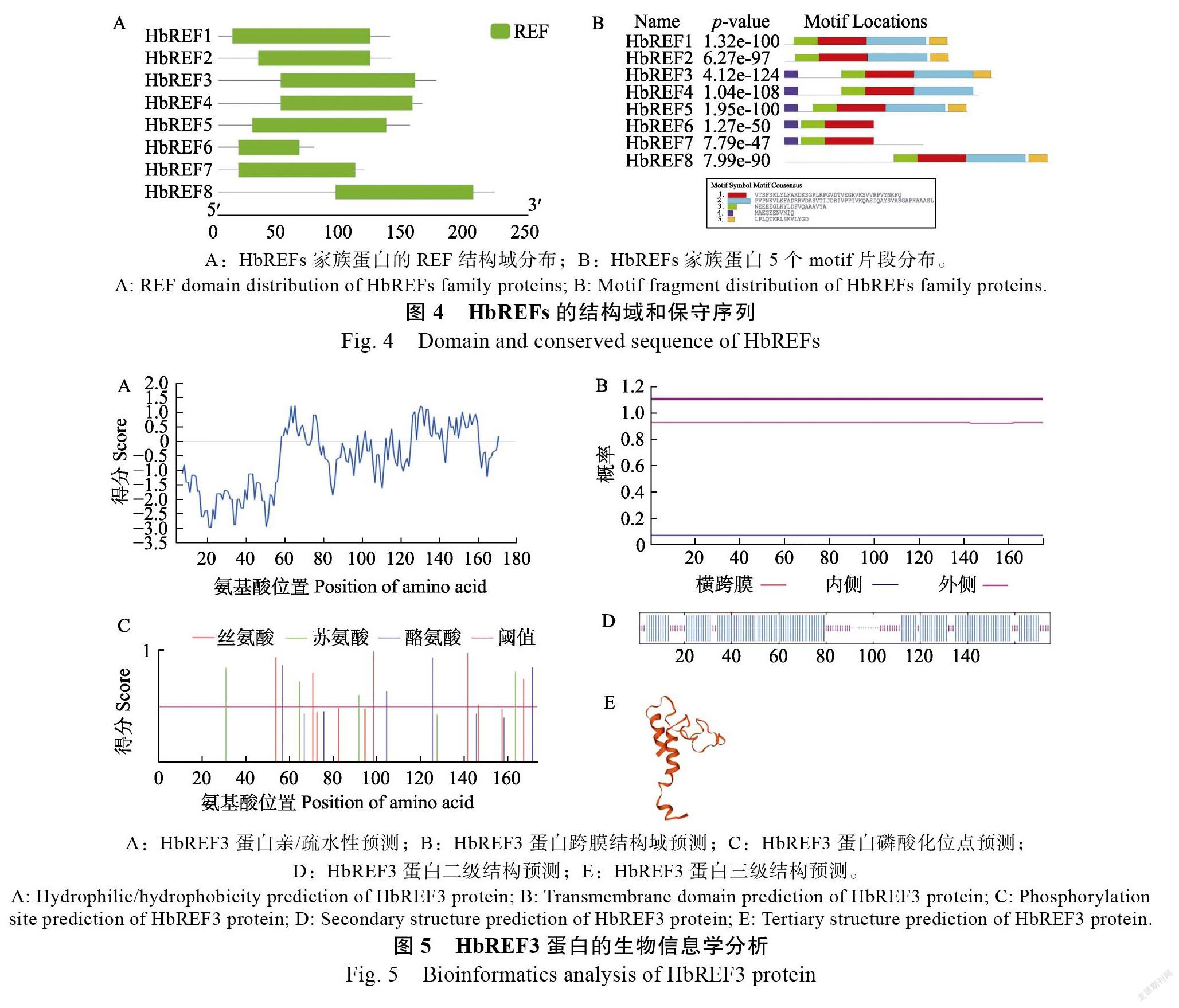
为了研究与其他REF/SRPPs家族基因的亲缘关系,根据已发表的橡胶树、橡胶草基因组文章,分别收集了18个橡胶树REF/SRPPs家族基因和10个橡胶草REF/SRPPs家族基因。此外,根据CHAKRABARTY报道的莴苣REF/SRPPs基因家族和KIM等报道的拟南芥REF/SRPPs基因家族,分别收集了8个莴苣REF/SRPPs家族基因和3个拟南芥REF/SRPPs家族基因。利用MEGA软件对上述39个REF/SRPPs家族基因的蛋白质序列构建了系统发育进化树。结果表明:HbREF3与橡胶树其他HbREFs家族蛋白都属于分组Ⅰ(Clade I),属于橡胶树特异的一类REFs基因,而橡胶草的TkSRPPs家族蛋白和大多数莴苣的LsSRPPs家族蛋白属于分组Ⅱ(Clade II),其余的分布在分组Ⅲ中(Clade III)(图3)。与在胶乳中特异高表达的虽然都属于分组Ⅰ,但明显属于不同分支(图3)。对橡胶树HbREFs基因家族氨基酸序列进行结构域和保守序列预测,发现它们均含有典型的REF结构域(图4A),但在保守motif的分布上,不同HbREFs蛋白含有保守motif数量不等(图4B)。因此推测不同基因之间可能存在功能上的分化。HbREF3有5个不同的motif片段,比HbREF1多一种motif片段(图4B),预示其功能与HbREF1可能有差异。
蛋白理化性質和生物信息学分析
对HbREF3蛋白序列及其理化性质进行预测分析发现,该蛋白相对分子质量为19.62 kDa,理论等电点(pI)为5.28,氨基酸组成中含量最高的是谷氨酸,占12.6%,不含半胱氨酸和色氨酸。对该蛋白的亲/疏水性和跨膜结构域的预测分析发现,HbREF3蛋白亲水区域分布明显多于疏水区域,属于亲水性蛋白(图5A),无跨膜结构域(图5B),推测该蛋白可能仅位于膜的外表面。磷酸化水平预测分析发现,该蛋白有14个磷酸化位点,分别是6个丝氨酸磷酸化位点、4个苏氨酸磷酸化位点和4个酪氨酸磷酸化位点,其中第99位氨基酸位点发生磷酸化修饰的可能性最高(图5C)。
HbREF3蛋白质二级结构预测分析发现,该蛋白的二级结构中α-螺旋占68%,无规则卷曲占25.14%,延伸链占6.86%(图5D)。通过SWISS-MOLD数据库进一步对HbREF3蛋白的三级结构进行预测,发现其与二级结构的预测结果一致(图5E)。
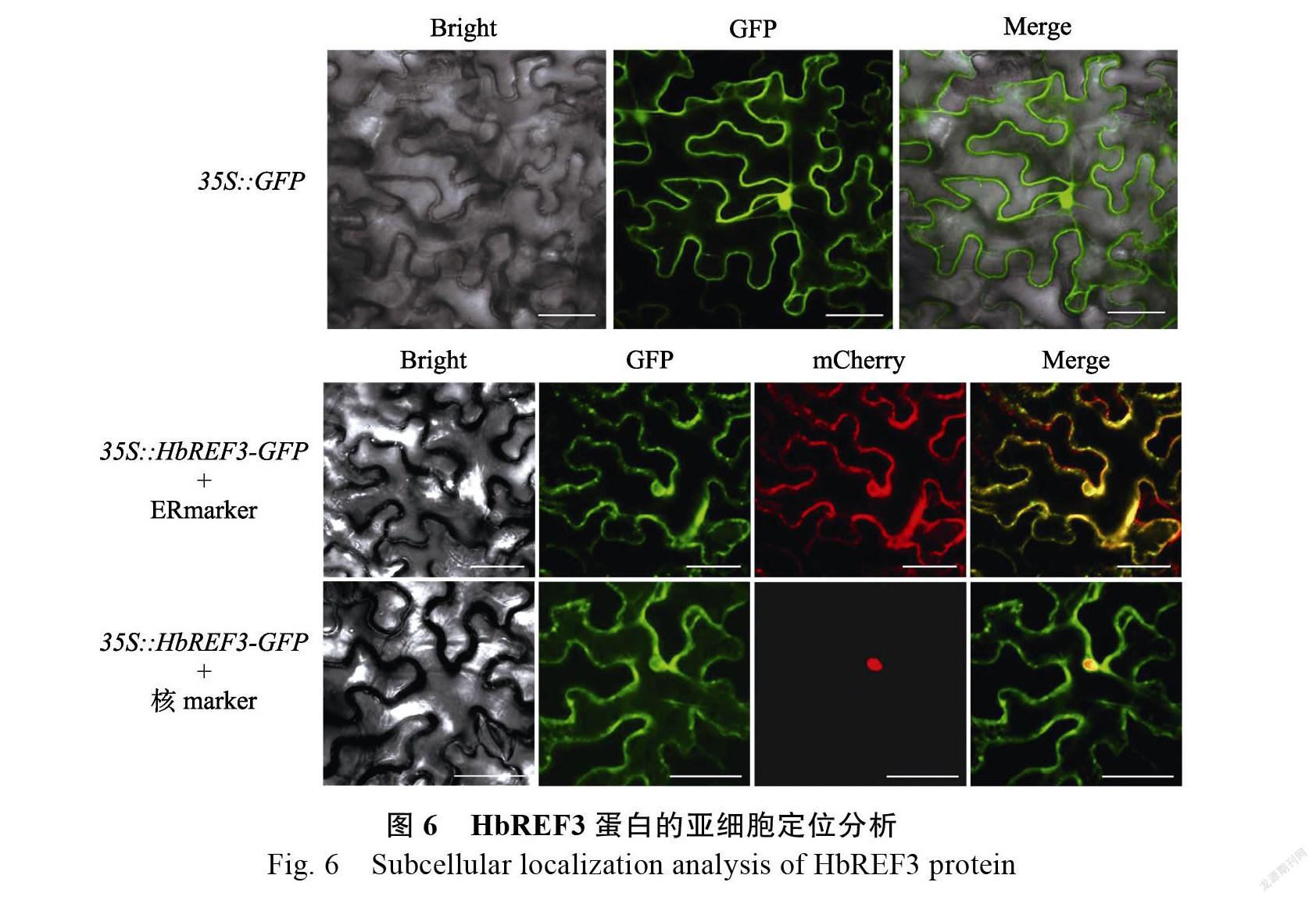
蛋白定位在内质网上
为进一步了解HbREF3蛋白在橡胶树中的功能,本课题组构建了pCAMBIA1300-H融合表达载体,采用农杆菌注射烟草叶片的瞬时表达方法,对HbREF3蛋白进行亚细胞定位(图6)。以35S::GFP作为对照,将HbREF3分别与内质网定位Marker和细胞核定位Marker共同转染,研究结果表明HbREF3蛋白定位于内质网上,且不定位于细胞核中。因此本课题组推测,HbREF3可能在橡胶粒子的形成和橡胶树胶乳的再生过程中起重要作用。
讨论
橡胶粒子作为聚合和贮存橡胶烃的特殊细胞器,对橡胶产量和胶乳的质量有很大影响;而橡胶延伸因子HbREFs是橡胶粒子中最丰富的蛋白质,在橡胶(顺式-1,4-聚异戊二烯)聚合延伸中起重要作用。前人研究表明基因在橡胶树胶乳中特异高表达,割胶和乙烯利处理均可以诱导其在胶乳中的上调表达,且基因在胶乳中的表达水平与胶乳产量呈正相关。本研究中的也是一个在橡胶树胶乳中特异性高表达的HbREFs家族基因,在橡胶树胶乳中的表达水平仅次于。编码175个氨基酸,预测该蛋白的相对分子质量为19.62 kDa、理论pI为5.28。系统发育进化树分析表明HbREF3虽然与基因表达水平在胶乳中特异最高表达的HbREF1属于一个大的分组,但它们在组Ⅰ中明显不属于一个分支,并且在motif片段分布上,橡胶树HbREFs家族中HbREF3有5个不同的motif片段,比HbREF1多一种motif片段,这也许会让它在蛋白质功能上与HbREF1有些许差异。因此,对HbREF3的研究或许会对分析HbREFs在产胶生物合成中的功能提供新的见解。
前人研究表明,天然橡胶被脂质单层膜包裹形成橡胶粒子,内部是疏水区,外部亲水区,而HbREFs与HbSRPPs、HbCPTLs和HbCPTs形成复合物附着在橡胶粒子表面的脂质单层膜上。在以往的HbREFs相关研究中,研究对象主要是HbREF1。本研究中,生信分析的预测结果显示HbREF3蛋白属于亲水性蛋白,无跨膜结构域,这说明HbREF3蛋白可能不是以镶嵌的形式附着在橡胶粒子表面,而是与镶嵌在膜上的其他蛋白直接或间接互作形成复合体附着在橡胶粒子表面的脂质单层膜上,这同HbREF1镶嵌在橡胶粒子膜中的报道明显不同。
橡胶粒子的形成和发育与天然橡胶的生物合成密切相关。研究发现,橡胶粒子表面是一层磷脂单层膜,橡胶粒子的形成和发育可能与脂质合成有关。YOKOTA等在酿酒酵母中过表达REF/SRPPs(、和)基因,发现这些蛋白质定位于内质网和脂滴中,它们的表达诱导了酵母中甾醇酯和三酰甘油的积累,并影响了脂滴的形态,表明REF/SRPPs影响脂质代谢。REF/SRPPs样蛋白在其他非产胶植物中也称胁迫相关蛋白,过表达的拟南芥植物表现出更旺盛的营养和生殖生长并显著增加了对干旱胁迫的耐受性。此外,发现萌发后的幼苗中有大量脂滴的积累。在拟南芥中过表达辣椒中的REF/SRPPs家族基因,促进转基因植株的根和茎生长以及抽薹提前,并且过表达也能增强拟南芥对干旱胁迫的耐受性。以上结果表明,REF/SRPPs蛋白可能在脂质的合成、组织的生长发育和响应干旱胁迫等方面发挥重要作用。本研究利用烟草叶片瞬时表达系统对HbREF3蛋白进行亚细胞定位发现,其定位在内质网膜上,推测HbREF3可能参与了脂质的合成,进而影响了橡胶粒子的形成和胶乳再生,并且该蛋白可能有类似于拟南芥中REF/SRPPs家族蛋白响应干旱胁迫的功能,在橡胶树中响应割胶胁迫,促进胶乳的再生。
综上,本研究对橡胶树基因进行克隆、生物信息学分析以及亚细胞定位,为进一步探究HbREF3蛋白在橡胶树中的功能奠定了基础。
参考文献
- XIANG Q, XIA K, DAI L, KANG G, LI Y, NIE Z, DUAN C, ZENG R. Proteome analysis of the large and the small rubber particles of using 2D-DIGE[J]. Plant Physiology and Biochemistry, 2012, 60: 207-213.
- CORNISH K, WOOD D F, WINDLE J J. Rubber particles from four different species, examined by trans-mission electron microscopy and electron-paramagnetic-resonance spin labeling, are found to consist of a homogeneous rubber core enclosed by a contiguous, monolayer biomembrane[J]. Planta, 1999, 210: 85-96.
- NAWAMAWAT K, SAKDAPIPANICH J T, HO C C, MA Y, SONG J, VANCSO J G. Surface nanostructure of natural rubber latex particles[J]. Colloids & Surfaces A (Physicochemical & Engineering Aspects), 2011, 390(1/3): 157-166.
- BERTHELOT K, LECOMTE S, ESTEVEZ Y, PERUCH F. REF (Hev b1) and SRPP (Hev b3): an overview on rubber particle proteins[J]. Biochimie, 2014, 106: 1-9.
- BERTHELOT K, LECOMTE S, ESTEVEZ Y, COULARY- SALIN B, PERUCH F. Homologous REF (Hevb1) and SRPP (Hevb3) present different auto-assembling [J]. Biochim et Biophysica Acta, 2014, 1844(2): 473-485.
- KO J H, CHOW K S, HAN K H. Transcriptome analysis reveals novel features of the molecular events occurring in the laticifers of (para rubber tree)[J]. Plant Molecular Biology, 2003, 53, 479–492.
- DENNIS M S, LIGHT D R. Rubber elongation factor from . Identification, characterization, and role in rubber biosynthesis[J]. Journal of Biological Chemistry, 1989, 264(31): 18608-18617.
- OH SK, KANG H, SHIN D H, J YANG, CHOW K S, YEANG H Y, WAGNER B, BREITENEDER H, HAN K H. Isolation, characterization, and functional analysis of a novel cDNA clone encoding a small rubber particle protein from [J]. Journal of Biological Chemistry, 1999, 274(24): 17132-17138.
- 孙丽娜. 橡胶树橡胶延伸因子的基因克隆与表达研究[D]. 海口: 海南大学, 2016.SUN L N. Studies on cloning and expression of gene from [D]. Haikou: Hainan University, 2016. (in Chinese)
- TANG C, QI J, LI H, ZHANG C, WANG Y. A convenient and efficient protocol for isolating high-quality RNA from latex of (para rubber tree)[J]. Journal of Biochemical Biophysical Methods, 2007, 70(5): 749-754.
- LIN T, XU X, RUAN J, LIU S, WU S, SHAO X, WANG X, GAN L, QIN B, YANG Y. Genome analysis of Rodin provides new insights into rubber biosynthesis[J]. National Science Review, 2018, 5(1): 78-87.
- CHAKRABARTY R, QU Y, RO D K. Silencing the lettuce homologs of small rubber particle protein does not influence natural rubber biosynthesis in lettuce ()[J]. Phytochemistry, 2015, 113: 121-129.
- PRIYA P, VENKATACHALAM P, THULASEEDHARAN A. Differential expression pattern of rubber elongation factor (REF) mRNA transcripts from high and low yielding clones of rubber tree ( Muell. Arg.)[J]. Plant Cell Reports, 2007, 26(10): 1833-1838.
- YAMASHITA S, YAMAGUCHI H, WAKI T, AOKI Y, MIZUNO M, YANBE F, ISHII T, FUNAKI A, TOZAWA Y, MIYAGI-INOUE Y. Identification and reconstitution of the rubber biosynthetic machinery on rubber particles from [J]. eLife, 2016, 5.
- KIM E Y, SEO Y S, LEE H, KIM W T. Constitutive expression of CaSRP1, a hot pepper small rubber particle protein homolog, resulted in fast growth and improved drought tolerance in transgenic plants[J]. Planta, 2010, 232(1): 71-83.

