环境DNA技术在湖泊生物资源调查中的应用进展
徐欣靖,皮杰,2,李德亮,刘新华,向建国,余建波*
(1.湖南农业大学动物科学技术学院,湖南 长沙 410128;2.湖南应用技术学院,湖南 常德 415100)
湖泊生态系统是地球上重要的生态系统之一,它既有蓄水、净化环境的作用,同时也具有保护水生生物多样性的作用。湖泊水生生物资源主要包括浮游生物、鱼类以及底栖生物等,它们共同组成湖泊的生物系统。进行湖泊水生生物多样性调查能够更好地了解湖泊物种组成,对生物多样性进行监测。但近年来,由于水利工程建设、水环境污染、栖息地被破坏、生物入侵等,引发湖泊生物资源面临物种灭绝、群体遗传多样性降低等问题,进而导致生物资源保护面临严峻挑战,因此对湖泊生物资源进行调查显得尤为重要。
水环境中生物种类组成复杂,有些种类个体微小、数量较少、不同生长阶段个体差异大,在进行调查时难以捕获、鉴定,这给湖泊资源调查工作带来诸多困难。在传统调查方法中,浮游生物是利用浮游生物网采集、浓缩、鉴定;鱼类是利用网捕、诱捕、声呐、实地调研;底栖生物则采用采泥器、底拖网等方法来调查。这些方法不仅费时费力、鉴别准确率低、操作烦琐,而且高度依赖鉴定人员专业知识水平。生物在自然水域环境中活动时,从黏液、皮肤、凋亡细胞等脱落的脱氧核糖核酸(DNA)片段散落于自然环境中(如:水、空气、土壤、冰)的DNA总和称为该自然环境中生物的环境DNA。环境DNA(eDNA)调查方法作为一种特异性强、灵敏度高、环境友好的资源调查技术,能够准确获取生物物种信息,对特定物种进行监测,可弥补传统调查方法的缺陷。eDNA技术是指从环境样品中直接提取DNA片段,并对其进行扩增、高通量测序,从而获得样品中多个物种信息的技术。目前eDNA技术在湖泊生物多样性检测、群落分布、资源评估方面都有相关的应用。现通过对eDNA技术的研究背景、在湖泊生物资源调查中的应用及对该技术的展望进行综述,旨在为后续开展的研究工作提供一定参考。
1 eDNA技术的研究背景
eDNA技术的初探可以追溯到20世纪末。1987年,Ogram等成功从海洋泥沼中提取出微生物DNA。20世纪末科学家通过采集鲸鱼粪便来鉴别种类,为eDNA技术在水域调查中的应用提供了灵感。2005年eDNA技术首次运用到水环境领域,通过特异性引物扩增出源自人及家畜的线粒体DNA,测试潜在的粪便污染。2008年Ficetoal等利用美国牛蛙基因特异性引物扩增其入侵水域eDNA,发现牛蛙入侵的量仅有1~2只/km,这是首次直接证明eDNA技术在水域环境检测中可行。2012年后,研究者逐渐尝试将eDNA技术应用于监测特定水域生物,这也使eDNA技术在水域环境物种从定性转变为相对定量,从单一物种的检测发展到多物种检测和特定物种检测,从仅为淡水检测延伸到海水检测,检测的目标种群也由低等过渡到高等,测序方式也从第一代的桑格测序向第三代高通量测序转变。自2015年后,由于eDNA研究的不断发展,逐渐成为生态环境研究领域的热点,研究领域也从水域eDNA向土壤eDNA、粪便eDNA等多元化发展;调查对象由鱼类转向浮游生物、底栖生物等;调查目的从物种调查向多样性、生物量丰度调查拓展。
2 eDNA技术在湖泊生物资源研究中的应用
目前,eDNA技术在湖泊生物资源研究中的应用主要分为物种监测、多样性监测和生物量评估3个方面,应用于浮游生物和底栖生物的研究主要集中在物种检测,在鱼类物种检测及多样性监测中应用相对成熟,见表1。
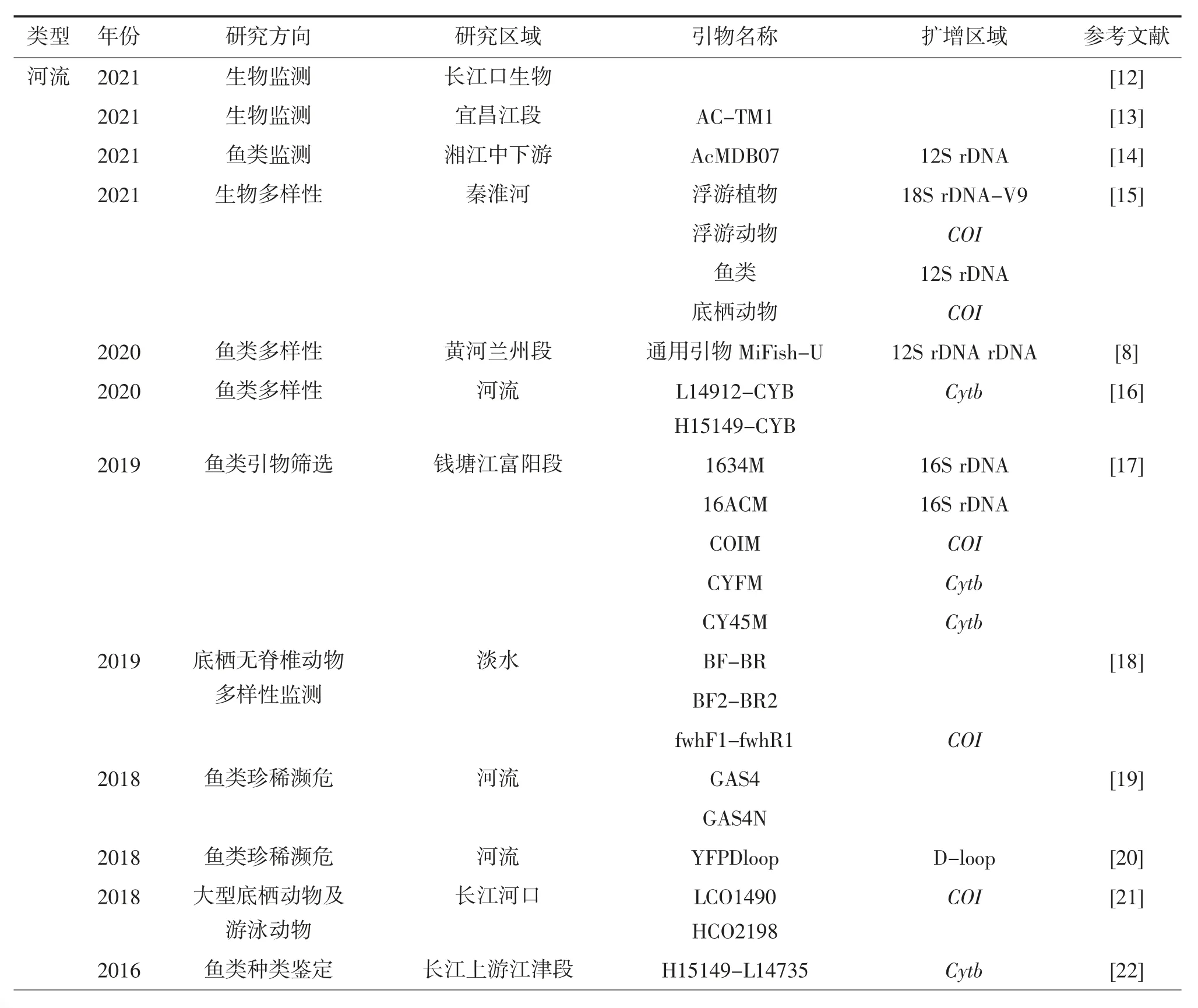
表1 国内eDNA技术在湖泊生物资源调查中的应用

续表
2.1 物种监测
eDNA技术现已经应用在湖泊物种调查、入侵种的监测、濒危种及稀有物种的保护中。生物入侵不仅对本地物种产生一定的威胁,而且还会对入侵地的生态系统功能产生损伤,造成生态系统退化、物种流失,因此有必要对入侵种进行监测。目前已报道的湖泊生物有浮游动植物、鲤、河鲈、克氏原螯虾、淡水珍珠贻贝等,其中,最早是2008年Ficetola等选用牛蛙基因上的小片段合成特异性引物,并且成功在湖泊中检测到该物种的存在,随后利用eDNA技术监测入侵种的工作逐渐展开。与国外相比,我国eDNA技术起步较晚,2005年有针对土壤eDNA的提取和纯化方法方面的研究报道;2014年王晓辉等针对海洋底泥中的eDNA的提取和纯化方法进行试验探究,为之后eDNA技术在水域物种监测中的成功应用奠定基础。目前eDNA技术应用于浮游生物及底栖生物调查的报道较少,有关于江豚、鲟等少数特定物种监测的应用报道。由于湖泊水域生态环境与生物的特异性,造成许多物种无法利用传统方法进行调查,尤其是濒危种与本地稀有种。传统方法调查速度慢、准确率低、调查结果无法及时获取,导致后续保护工作无法及时展开。eDNA技术的出现很好地弥补了传统方法的不足,并对调查物种与环境十分友好,通过采集水样对水体中含有的eDNA进行提取扩增数据分析及时获得调查水域的物种信息、分布、丰度。这项技术最初是在一些小型淡水湖泊中得到应用,而后逐渐向海陆不同生境不断扩展。近年来对濒危物种的研究主要集中在丹麦濒临灭绝的淡水欧洲气象泥鳅、哥伦比亚河上游的濒危大鳞大麻哈鱼、澳大利亚的濒危淡水鱼、切萨皮克湾的河鲱等,其中海洋生物褐背蝠鲼的发现是eDNA技术运用于海洋的一个成功案例。由于生态系统地理环境极其复杂,使用eDNA技术对濒危、稀有物种进行调查,降低了搜索难度,在一定程度上提高了检测效率和准确率。
2.2 多样性监测
由于人类过度捕捞、生境的变化和外来种的入侵,导致调查湖泊水域生物多样性组成发生变化,虽已采取相应措施,但成效并不显著。传统潜水、声呐、拖网操作难耗时长,因而eDNA技术的成功引入,为研究生物多样性的变化提供一个更加快速准确的方法。同时研究人员开始将该项技术运用于一些地形复杂、难以利用传统方法采样的区域中。
鱼类作为脊椎动物众多类群中种类最多的物种,是对湖泊水域生态调查的重要生物指标。2012年,Thomsen等在丹麦温带海洋生态系统中采用eDNA技术开展海水样品中鱼类多样性的研究,并将其与传统的浮潜、拖网和围网等调查方式进行对比,发现由于海洋环境地形的影响以及专业知识的限制,eDNA监测分析结果比传统鉴定方式更为客观和可靠。此后,研究者开始对eDNA技术在分析水域生物多样性的影响因素方面进行探索,并从海水水域研究不断向淡水湖泊、河流区域拓展,将其投入到实践中应用。2018年,Anaı¨s等基于的eDNA宏条形码开展了一次大规模的北极生物监测,共检测出181个物种;分析eDNA在水柱深度、潮汐池和季节方面的时空分布,提出,eDNA采样需要考虑时空维度方面因素才能准确评估群落结构的变化。这又一次证明eDNA技术在水生生物多样性调查应用中的可行性。
2.3 生物量评估
对目标生物资源的丰度调查是对水生生物资源监控的重要内容,通过对原始环境中eDNA浓度进行测定并比对,可探究该调查区域目标生物的生物量高低。近年来,不断有专家对各地的物种密度进行调查,发现物种的密度与其eDNA浓度之间存在线性关系,即可以通过某物种eDNA的含量来估算其生物量。常见验证方法是对人工养殖试验区的固定物种进行密度计算,而后对该区域eDNA浓度进行测定后绘制密度-浓度曲线。但是该方法适用范围较窄,仅限于水样中eDNA浓度与该物种数量的相关性较强的物种,对于带有坚硬外壳的虾、贝类,其eDNA浓度与生物量之间的关系并不明显。在通过eDNA浓度对生物量调查的同时,也可以对物种的遗传多样性进行相应分析,来判断物种的数量及结构差异,进而对物种的优良性状是否稳定遗传提供参考。
3 存在的问题及展望
3.1 采集水样后的处理与保存
由于生物释放到水体中的DNA会受到水环境的变化被降解,导致提取的DNA含量少,水样处理与保存不当会直接影响后验结果。2014年Matthew等针对eDNA研究现状的文献进行检索,并且使用SYBR-Green定量PCR方法对鲤eDNA降解的环境因素进行探究,发现eDNA降解的速度与生化需氧量、叶绿素和水环境总的eDNA浓度呈现负相关。另外,影响eDNA降解的非生物因素有紫外线辐射、温度、pH值、溶解氧、盐度和沉积物等,这也导致同批水样的质量有所不同。常见水样处理方式有2种,分别为采样过程中在线抽滤与冷藏保存采集的样品带回实验室抽滤;保存方式为-20、-80℃、无水乙醇浸泡等。目前已有针对延长DNA保存时间的相应研究,但关于如何避免污染、提升提取技术、选择更好的滤膜材料的研究相对较少。
3.2 eDNA扩增引物的选择
目前针对水生动物研究最常扩增的是、16SrDNA、RHO、等区域基因,但因生境、种类不同,同一种引物扩增出来的效果也不同。葛玉双等针对eDNA现有技术方法在水生生物多样性调查中的应用进行研究,得出目前有2种类型引物,一种是用于检测物种的特异性引物,另一种是用于生物多样性评估的通用引物。设计引物一般分为2步:一为获取目标物种的DNA序列,二为根据调查目的的不同设计引物。在未来针对特定物种进行引物开发,将对eDNA技术的推广提供很大帮助。
3.3 选择对比序列的基因库
目前常用的基因库有NCBI、BLOD和Mito chondrial Genome Database of Fish等大型开放数据库,这些数据库既包含淡水鱼种类,也包含了海水鱼种类。在对比采集淡水水体提取的eDNA时,可能由于操作或引物选择的问题会对比出海水生物种类。对淡水湖泊中鱼类种类鉴定最准确的方法,是根据当地鱼类志、其他文献记载以及采集当地现有已知鱼类样本,对样本DNA提取,建立本地鱼类数据库,再进行鱼类多样性调查,以获得准确的调查数据。
国内使用eDNA技术调查湖泊生物资源的研究相对国外较少,仅限于对长江、黄河、舟山近海等流域,以及洱海、太湖、鄱阳湖等部分湖区的水生生物进行了调查,研究对象涉及浮游动物、鱼类、底栖生物等。虽然自2019年以来国内针对湖泊水域生态eDNA的研究明显增多,但由于我国幅员辽阔、地形复杂,导致利用传统方法进行全面水生生物资源调查难度极大,新型eDNA方法能够很好地弥补传统方法缺陷,将来该项技术会应用于更多的湖泊调查中。
在各种实际应用中,对于特定水域利用eDNA技术进行物种调查的特有方法建立还有待提升。在生物量评估中,该方法具有广阔的发展前景,但目前生物量的评估仅能够从序列的OTU丰度情况来进行相应推断,虽然可以使用荧光定量PCR对特定基因进行定量,但针对物种的绝对丰度及生物量仅能预测,不能准确评估。现已有学者将生物体及基因库的大小纳入影响因子的分析中,如何将该技术在湖泊水生生物调查中进行更好的应用是未来研究的主要方向。

