猪FABP1和FABP2基因的分子特征及组织表达分析
熊讯 李永 阮涌 陈晨 宋林锦 石鹏飞 孙金魁 许厚强


摘要:【目的】明確脂肪酸结合蛋白(FABPs)对香苏杂交猪脂肪沉积的影响,为后续进一步探究猪脂肪细胞内FABP1和FABP2基因对脂肪酸代谢的调控作用打下基础。【方法】采集香苏杂交猪和柯乐猪的心脏、肝脏、脾脏、肺脏、肾脏、大肠、小肠、背最长肌及脂肪等组织样品提取总RNA,利用PCR克隆香苏杂交猪FABP1和FABP2基因编码区(CDS)序列,通过ProtParam、ProtScale、SOPMA、SWISS-MODEL、PSORTⅡPreadict及SignalP-5.0等在线软件进行生物信息学分析,并采用实时荧光定量PCR检测FABP1和FABP2基因在香苏杂交猪和柯乐猪各组织中的表达情况。【结果】香苏杂交猪FABP1和FABP2基因CDS序列分别为384和399 bp,分别编码127和132个氨基酸残基,对应的编码蛋白相对分子量为14107.23和15217.34 Da,均为亲水性蛋白;其二、三级结构均以β-折叠和无规则卷曲为主,无信号肽剪切位点,主要定位于细胞质。香苏杂交猪FABP1和FABP2氨基酸序列表现为与人类、牛、山羊的FABP1和FABP2氨基酸序列高度同源,对应的相似性均在80.0%以上,除斑马鱼外,与其他参考物种的相似性也在74.0%以上,故推测FABP1和FABP2基因序列的保守性较高; STRING预测结果显示,与这2个蛋白可能互作的蛋白有FABP5、FABP7、过氧化物酶体增殖物激活受体α(PPARα)、猪载脂蛋白A4(APOA4)、APOA1及脂酰辅酶A氧化酶1(ACOX1)等。FABP1和FABP2基因在香苏杂交猪和柯乐猪的各组织中均有表达,且以在肝脏、小肠和脂肪中的相对表达量较高,而背最长肌中的相对表达量最低;此外,FABP1和FABP2基因在香苏杂交猪肝脏和小肠中的相对表达量极显著高于其在柯乐猪对应组织中的相对表达量(P<0.01)。【结论】FABP1和FABP2基因在猪的各组织中均有表达,且以在甘油三酯和脂肪酸合成代谢的相关组织(肝脏、小肠和脂肪)中特异性高表达,可能对脂肪的合成代谢起重要调控作用。
关键词:猪;FABP1基因;FABP2基因;分子特征;脂肪酸代谢
中图分类号:S828.1 文献标志码: A 文章编号:2095-1191(2022)04-0935-11
Molecular characteristics and tissue expression analysis of FABP1 and FABP2 genes in pig
XIONG Xun1, LI Yong2, RUAN Yong1, CHEN Chen1, SONG Lin-jin1,
SHI Peng-fei1, SUN Jin-kui1, XU Hou-qiang*
(1College of Zoology,Guizhou University/Key Laboratory of Animal Genetics, Breeding and Reproduction in the Plateau Mountainous Region, Ministry of Education/Guizhou Key Laboratory of Animal Genetics, Breeding and Reproduction, Guiyang, Guizhou 550025, China; 2College of Life Sciences, Guizhou University, Guiyang, Guizhou 550025, China)
Abstract:【Objective】To clarify the effect of fatty acid binding proteins (FABPs) on fat deposition in Xiangsu hybrid pigs, so as to lay the foundation for further exploring the regulatory role of FABP1 and FABP2 genes on fatty acid metabolism in adipocytes of pigs. 【Method】Total RNA was extracted from the heart, liver, spleen, lung, kidney, large intestine, small intestine, longest back muscle and adipose tissue samples of Xiangsu hybrid pigs and Kole pigs, and the sequences of FABP1 and FABP2 gene coding region (CDS) of Xiangsu hybrid pigs were cloned by PCR, and were analyzed by ProtParam, ProtScale, SOPMA, SWISS-MODEL, PSORTⅡPreadict and SignalP-5.0 online software for bioinformatics analysis, and RT-qPCR was used to detect the expression of FABP1 and FABP2 genes in various tissues of Xiangsu hybrid pigs and Kole pigs. 【Result】CDS sequences of FABP1 and FABP2 genes of Xiangsu hybrid pigs were 384 and 399 bp, encoding 127 and 132 amino acid residues, respectively, corresponding to the relative molecular weights of 14107.23 and 15217.34 Da, which are both hydrophilic proteins; their secondary and tertiary structures were dominated by β-folding and irregular coiling, with no signal peptide shear sites,and localized in the cytoplasm. The amino acid sequences of both FABP1 and FABP2 of Xiangsu hybrid pigs showed high homology with those of human, bovine and goat, and the corresponding similarities were above 80.0%, and the similarities with other reference species, except zebrafish, were also above 74.0%, so the conserved sequences of FABP1 and FABP2 genes were presumed to be high; STRING prediction results showed that the proteins that might interact with these 2 proteins were FABP5, FABP7, peroxisome proliferator-activated receptor α (PPARα), porcine apolipoprotein A4 (APOA4), APOA1 and acyl-co A oxidase 1 (ACOX1), etc. FABP1 and FABP2 genes were expressed in all tissues of the Xiangsu hybrid pigs and Kole pigs, and were mainly expressed in various tissues, and the relative expression of FABP1 and FABP2 genes was higher in liver, small intestine and fat, while the relative expression in the longest dorsal muscle was the lowest; in addition, the relative expression of FABP1 and FABP2 genes in liver and small intestine in Xiangsu hybrid pigs was significantly higher than that in the corresponding tissues of the Kole pigs (P<0.01). 【Conclusion】FABP1 and FABP2 genes are expressed in various tissues of different pigs, with specific high expression in tissues related to triglyceride and fatty acid anabolism (liver, small intestine and adipose), which may play an important role in regulating lipid anabolism.
Key words: pig; FABP1 gene; FABP2 gene; molecular characterizatics; fatty acid metabolism
Foundation items: Guizhou Science and Technology Plan Project [QKHFQ〔2018〕4007(002)];Guizhou Agricultural Major Industrial Scientific Research Tackling Project (QJH-KY〔2019〕011)
0 引言
【研究意義】脂肪酸结合蛋白(Fatty acid-binding proteins,FABPs)是一族同源性细胞内小分子蛋白,广泛存在于脊椎动物和非脊椎动物细胞内,主要负责将细胞膜上的长链脂肪酸转运至磷脂和甘油三酯合成的位置(邹增丁等,2007;董飚等,2016;陈静等,2021)。FABPs一般由126~134个氨基酸组成,不同家族成员间的氨基酸序列相似性为22%~73%,但其三维结构高度保守(Zimmerman and Veerkamp,2002)。Zhang等(2020)研究表明,在18个物种中共发现12种FABPs基因,但未发现单一物种同时存在12种FABPs基因,且每个基因的结构各不相同,究其原因可能是通过串联复制从共同祖先进化而来。L-FABP/FABP1(肝型)和I-FABP/FABP2(肠型)基因是较早被发现的2个FABPs基因,对脂肪代谢调控起重要作用(陈璐,2013)。因此,开展FABP1和FABP2基因的分子特征及组织表达分析研究,对揭示FABP1和FABP2基因调控脂肪酸代谢的作用机制起重要意义。【前人研究进展】目前,有关FABP1和FABP2基因在家畜上的研究主要集中在鸡、鸭和羊上,二者对长链脂肪酸具有高度的结合力(Ockner et al.,1972),通过对长链脂肪酸摄取、细胞内转运、PPAR代谢通路等调控脂肪代谢(崔璐等,2016);同时能结合潜在的有毒脂肪酸,降低脂肪酸对细胞的毒性,而对细胞起保护作用(Wang et al.,2015)。FABP1和FABP2可与配体靶向结合而影响酶功能,改变膜结构及其流动性来调节酶活性,并通过激活核受体以调节基因表达(Jefferson et al.,1990;Lawrence et al.,2000;Gajda et al.,2013)。FABP1基因在鸭的各组织中均有表达,对脂肪代谢和多种脂肪代谢相关基因起调控作用(何俊,2013)。鸡FABP2基因表达能促进脂肪沉积,故可作为影响鸡脂肪性状的候选基因(Wang et al.,2006;俞英等,2011);FABP2基因在鸭小肠各段位均有表达,以十二指肠和空肠前半段的表达量较高,且干扰FABP2基因对脂类吸收相关基因有影响(陈芳等,2011);FABP2基因在贵州白山羊9个组织中均有表达,表达较高的组织包括肝脏和十二指肠(钱成,2016)。此外,Samulin等(2008)研究表明,FABP1基因在细胞增殖过程中发挥重要作用,同时与FABP2、FAPP3、FABP4、FAPP5、PPARA、PPARG1和PPRG2基因的共同表达可能对猪脂肪沉积的代谢调控过程有重要影响。脂肪又称甘油三酯(Triglyceride,TG),是由长链脂肪酸和甘油缩合而成。脂肪代谢是在各种酶的作用下经消化分解成甘油和脂肪酸等,并释放能量的过程(Hunter,2001)。脂肪酸作为一种信号分子,能调节细胞与脂质代谢,对生物体有重要的调控作用(Hotamisligil and Bernlohr,2015)。尽管所有FABPs家族成员都参与脂肪酸的代谢,但脂肪酸功能的多样性很有可能反映在不同组织中FABPS基因的表达差异上,且不同功能可能由组织特异性的蛋白质—蛋白质和蛋白质—膜相互作用驱动(Storch and Thumser,2010)。【本研究切入点】至今,有关FABP1和FABP2基因在家畜上的研究较少,尤其在猪各组织中的具体分子结构、功能及相互作用尚无系统研究报道。【拟解决的关键问题】通过生物信息学分析香苏杂交猪FABP1和FABP2基因的分子结构和功能,采用实时荧光定量PCR检测这2种基因在香苏杂交猪各组织中的表达差异,明确FABPs对香苏杂交猪脂肪沉积的影响,为后续进一步探究猪脂肪细胞内FABP1和FABP2基因对脂肪酸代谢的调控作用打下基础。
1 材料与方法
1. 1 试验材料
香苏杂交猪和柯乐猪均来自贵州大学种猪场,随机选取相同饲养条件下的成年猪各3头,依照GB/T 17236—2008《猪屠宰操作规程》进行屠宰,采集心脏、肝脏、脾脏、肺脏、肾脏、大肠、小肠、背最长肌及脂肪等组织样品用于总RNA提取。TRIzol® Reagent总RNA提取试剂盒、GenStar逆转录试剂盒(StarScript II Onestep RT-PCR Kit)购自西宝生物科技(上海)股份有限公司;荧光定量试剂TB Green® Premix Ex TaqTM II(Tli RNaseH Plus)、pMD19-T载体、大肠杆菌(Escherichi acoli)DH5α感受态细胞购自宝日医生物技术(北京)有限公司;DL2000 DNA Marker、胶回收试剂盒及卡那霉素(Kanamycin)购自生工生物工程(上海)股份有限公司。
1. 2 引物设计与合成
根据NCBI已发布的猪FABP1基因(NM_001004 046.2)、FABP2基因(NM_001031780.1)核苷酸序列,采用Primer Premier 5.0设计实时荧光定量PCR扩增特异性引物及编码区(CDS)扩增引物(表1),以GADPH为内参基因。所有引物均委托生工生物工程(上海)股份有限公司合成。
1. 3 组织总RNA提取及cDNA合成
采用TRIzol® Reagent总RNA提取试剂盒分别提取香苏杂交猪和柯乐猪的组织总RNA,以超微量分光光度计测定其浓度和纯度,合格的RNA置于-80 ℃冰箱中保存。采用StarScript II Onestep RT-PCR Kit反转录合成cDNA,-20 ℃保存备用。
1. 4 实时荧光定量PCR检测各组织表达差异
以cDNA为模板,采用实时荧光定量PCR检测FABP1、FABP2基因在香苏杂交猪和柯乐猪心脏、肝脏、脾脏、肺脏、肾脏、大肠、小肠、背最长肌及脂肪等组织中的表达情况。实时荧光定量PCR反应体系20.0 μL:2×TB Green Premix Ex Taq II(Tli RNaseH Plus) 10.0 μL,上、下游引物(10 μmol/L)各0.8 μL,50×ROX Reference Dye或50×ROX Reference Dye II 0.4 μL,cDNA模板2.0 μL,灭菌水6.0 μL。然后通过2-ΔΔCt法计算目的基因在不同组织中的相对表达量,并以SPSS 26.0进行单因素方差分析(One-way ANOVA)。
1. 5 目的基因CDS序列扩增及测序
以香苏杂交猪cDNA为模板,PCR扩增目的基因CDS序列。PCR反应体系20.0 μL:cDNA模板1.0 μL,上、下游引物各0.4 μL,2×Taq PCR StarMix 10.0 μL,ddH2O 8.2 μL。扩增程序:94 ℃预变性2 min;94 ℃ 30 s, 51 ℃ 30 s,72 ℃ 60 s,进行30个循环;72 ℃延伸7 min。PCR扩增产物以1.5%琼脂糖凝胶电泳进行检测。胶回收试剂盒回收纯化PCR扩增产物,然后与pMD19-T载体在16 ℃下连接12 h,再转化DH5α感受态细胞,涂板后37 ℃恒温培养12 h,经抗性筛选,挑选出单个菌落进行扩繁,菌液PCR验证呈阳性的菌液送至生工生物工程(上海)股份有限公司测序。
1. 6 FABP1和FABP2基因生物信息学分析
利用DNAStar-MegAlign进行FABP1和FABP2基因同源性分析,运用ProtParam(https://web.expasy. org/protparam/)和ProtScale(https://web.expasy.org/protscale/)分别进行编码蛋白理化性质及亲/疏水性分析,以SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html%5D)和SWISS-MODEL(https://swissmodel.expasy.org/interactive)预测蛋白的二、三級结构,利用PSORTⅡPreadict(https://www.genscript.com/psort.html)和SignalP-5.0(https://services.healthtech.dtu.dk)对FABP1和FABP2蛋白进行亚细胞定位及信号肽预测,采用STRING(https://string-db.org/)预测FABP1蛋白与FABP2蛋白间的相互作用,最后以MEGA 7.0进行FABP1和FABP2氨基酸同源性分析,并基于氨基酸序列相似性构建系统发育进化树。
2 结果与分析
2. 1 猪FABP1和FABP2基因CDS序列扩增结果
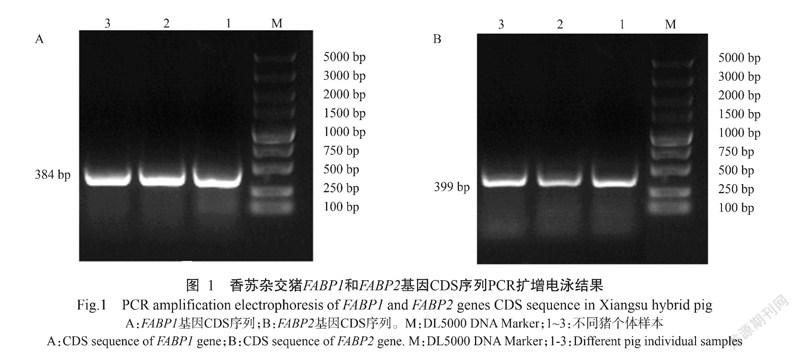
以香苏杂交猪cDNA为模板,PCR扩增FABP1和FABP2基因CDS序列,PCR扩增产物经1.5%琼脂糖凝胶电泳检测,结果在400 bp附近分别获得清晰单一的目的条带(图1),且扩增片段长度与预期结果相符。
2. 2 目的基因克隆载体构建及鉴定结果
将香苏杂交猪FABP1和FABP2基因CDS序列的胶回收产物与pMD19-T载体连接过夜,涂板转化DH5α感受态细胞后,挑选单个菌落进行菌液PCR验证,结果显示,香苏杂交猪FABP1和FABP2基因片段大小分别为384 bp(图2-A)和399 bp(图2-B),与预期结果一致。将阳性菌液测序结果与NCBI已发布的猪基因序列进行比对,结果显示,香苏杂交猪FABP1和FABP2基因序列与野猪的FABP1和FABP2基因序列完全吻合,不存在碱基或氨基酸突变。以上结果也表明,pMD19-T-FABP1和pMD19-T-FABP2克隆载体构建成功。
2. 3 香苏杂交猪FABP1和FABP2基因生物信息学分析结果
2. 3. 1 香苏杂交猪FABP1和FABP2基因编码蛋白理化性质 香苏杂交猪FABP1基因CDS序列长384 bp,共编码127个氨基酸残基。经ExPASy-ProtParam预测得知,FABP1蛋白分子式为C623H1021N163O197S5,原子数为2009,相对分子量为14107.23 Da,理论等电点(pI)为6.60;以赖氨酸含量最高,占11.8%,含量最低的分别是半胱氨酸、组氨酸和脯氨酸,各占0.8%;蛋白不稳定指数为10.51,属于稳定蛋白。由图3-A可看出,香苏杂交猪FABP1蛋白的第10位谷氨酰氨亲水性最强(-2.144),而第116位甘氨酸疏水性最强(1.322),且负值氨基酸数远多于正值氨基酸,故推测该蛋白为亲水性蛋白。
香苏杂交猪FABP2基因CDS序列长399 bp,共编码132个氨基酸残基,FABP2蛋白分子式为C680H1072N184O204S4,原子数为2144,相对分子量为15217.34 Da,理论等电点为6.61;以赖氨酸含量最高,占10.8%,含量最低的是组氨酸,仅占0.8%;蛋白不稳定指数为24.24,属于稳定蛋白。由图3-B可看出,香苏杂交猪FABP2蛋白的第14位天冬酰氨亲水性最强(-2.711),而第65位亮氨酸疏水性最强(1.967),负值氨基酸数远多于正值氨基酸,故推测该蛋白为亲水性蛋白。
2. 3. 2 香苏杂交猪FABP1和FABP2蛋白二、三级结构 SOMPA预测结果显示,香苏杂交猪FABP1蛋白二级结构由α-螺旋、β-折叠、β-转角和无规则卷曲组成,以β-折叠为主,占34.65%,其他3种结构依次为无规则卷曲(占31.50%)、α-螺旋(占22.83%)和β-转角(占11.02%);香苏杂交猪FABP2蛋白二级结构同样由4种结构组成,其中,β-折叠占40.15%,无规则卷曲占28.03%,α-螺旋占18.94%,β-转角占12.88%。SWISS-MODEL预测结果(图4)显示,香苏杂交猪FABP1和FABP2蛋白三级结构与其二级结构预测结构基本一致。
2. 3. 3 香苏杂交猪FABP1和FABP2蛋白信号肽及亚细胞定位 SignalP-5.0预测结果(图5)显示,香苏杂交猪FABP1和FABP2蛋白均无信号肽剪切位点,表明FABP1和FABP2蛋白可能不是分泌蛋白。通过PSORTⅡPreadict对香苏杂交猪FABP1和FABP2蛋白进行亚细胞定位预测,结果发现,FABP1蛋白定位于细胞质、细胞核和线粒体的可能性分别为56.5%、30.4%和13.0%,FABP2蛋白定位于细胞质、细胞核和线粒体的可能性分别为52.2%、26.1%和21.7%。
2. 3. 4 香苏杂交猪FABP1和FABP2蛋白的互作蛋白
使用STRING预测香苏杂交猪FABP1和FABP2蛋白的互作蛋白,结果(图6)显示与这2个蛋白可能互作的蛋白有FABP5、FABP7、过氧化物酶体增殖物激活受体α(PPARα)、猪载脂蛋白A4(APOA4)、APOA1及脂酰辅酶A氧化酶1(ACOX1)等。其中,FABP5和FABP7蛋白同属于FABPs家族,也是参与脂肪酸吸收、运输、储存及信号转导和基因转录的关键细胞内分子(Sharifi et al.,2013);PPARα通過对参与脂肪酸氧化基因的转录调节,在脂肪酸催化中发挥重要作用,因此其编码基因被视为影响猪脂肪特征的候选基因(Stachowiak et al.,2014);APOA4能促进脂质运输和新陈代谢,在小肠中合成、在胸腺中包装,然后分泌至肠道淋巴中并通过循环输送到脂肪和其他组织(Qu et al.,2021);ACOX1作为过氧化物β-氧化途径的第一个限速酶,受PPARα基因表达的调节,对脂肪酸氧化和沉积至关重要,尤其是在长链脂肪酸的脂质代谢过程中(Wu et al.,2018)。
2. 4 FABP1和FABP2氨基酸序列同源比对分析结果
使用MegAlign对不同物种的FABP1和FABP2氨基酸序列进行同源比对分析,结果显示,香苏杂交猪FABP1氨基酸序列与斑马鱼、人类、马、鸡、小鼠、大鼠、山羊、牛和绵羊的FABP1氨基酸序列相似性分别为62.2%、89.8%、78.7%、72.7%、82.8%、82.0%、83.6%、83.6%和82.8%(图7-A),除了与斑马鱼的相似性较低外,与其他几个物种均有较高的相似性;利用MEGA 7.0构建基于FABP1氨基酸序列相似性的系统发育进化树(图8-A),发现香苏杂交猪与人类的亲缘关系最近,其次是与牛的亲缘关系,与鸡和斑马鱼的亲缘关系相对较远。香苏杂交猪FABP2氨基酸序列与斑马鱼、人类、马、鸡、小鼠、大鼠、山羊、牛和绵羊的FABP2氨基酸序列相似性分别为67.7%、86.5%、76.7%、74.4%、79.7%、83.5%、88.7%、88.0%和88.7%(图7-B),与山羊和绵羊的相似性最高,与斑马鱼的相似性最低;基于FABP2氨基酸序列相似性构建的系统发育进化树(图8-B)显示,香苏杂交猪与牛的亲缘关系最近,与山羊和绵羊的亲缘关系较近,而与鸡和斑马鱼的亲缘关系相对较远。
2. 5 FABP1和FABP2基因在香苏杂交猪和柯乐猪不同组织中的表达情况
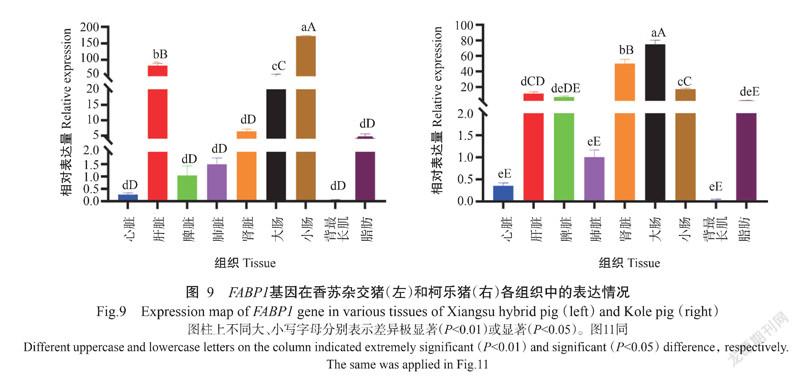
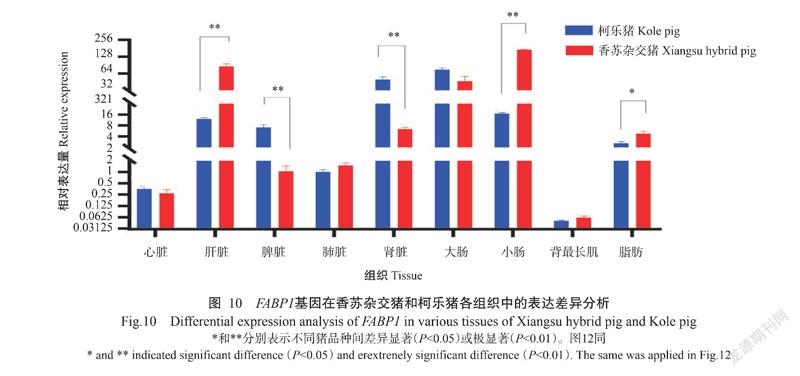
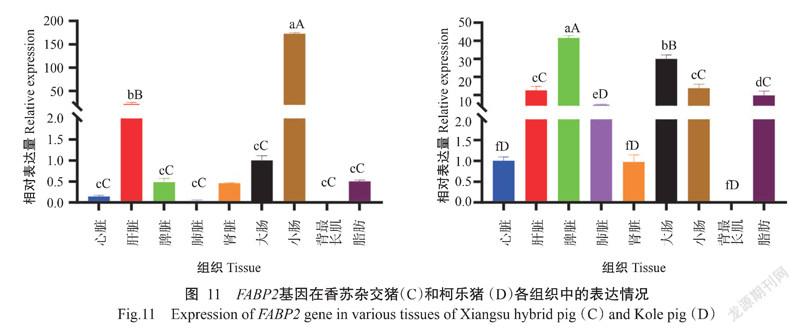
2. 5. 1 FABP1基因在香苏杂交猪和柯乐猪不同组织中的表达情况 FABP1基因在香苏杂交猪和柯乐猪不同组织中均有表达,FABP1基因在香苏杂交猪各组织中的相对表达量排序为小肠>肝脏>大肠>肾脏>脂肪>肺脏>脾脏>心脏>背最长肌(图9-A),在小肠、肝脏和大肠中的相对表达量极显著高于在其他组织中的相对表达量(P<0.01,下同),其他组织间的相对表达量无显著差异(P>0.05,下同)。FABP1基因在柯乐猪各组织中的相对表达量排序为大肠>肾脏>小肠>肝脏>脾脏>脂肪>肺脏>心脏>背最长肌(图9-B),在大肠中的相对表达量极显著高于在其他组织中的相对表达量,背最长肌中的相对表达最低。FABP1基因在香苏杂交猪肝脏和小肠中的相对表达量极显著高于其在柯乐猪对应组织中的相对表达量(图10),在脂肪中的相对表达量显著高于其在柯乐猪脂肪中的相对表达量(P<0.05,下同),在脾脏和肾脏中的相对表达量则极显著低于其在柯乐猪对应组织中的相对表达量。
2. 5. 2 FABP2基因在香苏杂交猪和柯乐猪不同组织中的表达情况 FABP2基因在香苏杂交猪和柯乐猪的不同组织中也均有表达,FABP2在香苏杂交猪各组织中的相对表达量排序为小肠>肝脏>大肠>脂肪>脾脏>肾脏>心脏>肺脏>背最长肌(图11-C),在肝脏和小肠中的相对表达量极显著高于在其他组织中的相对表达量,其他组织间的相对表达量差异不显著。FABP2基因在柯乐猪各组织中的相对表达量排序为脾脏>大肠>小肠>肝脏>脂肪>肺脏>心脏>肾脏>背最长肌(图11-D),以脾脏中的相对表达量最高,显著高于其他组织中的相对表达量,而背最长肌中的相对表达量最低。FABP2基因在香苏杂交猪肝脏和小肠中的相对表达量极显著高于其在柯乐猪对应组织中的相对表达量(图12),在脂肪中的相对表达量显著高于其在柯乐猪脂肪中的相对表达量,在脾脏和肾脏中的相对表达量也极显著低于其在柯乐猪对应组织中的相对表达量。
3 讨论
FABPs是由多个编码14~15 kD蛋白的基因组成,具有结合疏水生物分子如脂肪酸的能力(田鎏等, 2017),通过与PPARs进行功能性合作(Venkatachalam et al.,2017),而有助于脂质代谢相关基因的转录调控,对动物的脂肪沉积产生重要影响。近年来,有关FABP1和FABP2的研究报道主要集中在人类和斑马鱼上,且多与疾病、癌症及免疫等相关,而针对畜禽脂肪酸代谢调控的研究较少,具体调控机制也尚未明确(Chen et al.,2020;Tsai et al.,2020)。本研究成功克隆获得香苏杂交猪FABP1和FABP2基因CDS序列,与NCBI已发布的猪基因序列进行比对,发现与野猪的FABP1和FABP2基因序列完全吻合,不存在碱基或氨基酸突变,说明这2个基因在香苏杂交猪上稳定遗传。SignalP-5.0信号肽预测结果显示,香苏杂交猪FABP1和FABP2蛋白均无信号肽剪切位点,与Chen等(2020)在鱼类上的研究结果一致,表明FABP1和FABP2可能不是分泌蛋白。香苏杂交猪FABP1和FABP2氨基酸序列表现为与人类、牛、山羊的FABP1和FABP2氨基酸序列高度同源,对应的相似性均在80.0%以上,除斑马鱼外,与其他参考物种的相似性也在74.0%以上,由此推测香苏杂交猪FABP1和FABP2基因序列的保守性较高,不易发生突变,与Ordovas(2009)的研究结果一致。此外,香苏杂交猪FABP1、FABP2分别有56.5%和52.2%的可能位于细胞质,其二、三级结构主要由β-折叠和无规则卷曲构成。
FABP1和FABP2主要以小分子蛋白的形式存在于细胞内,共同参与细胞内脂肪酸的运输,但其具体功能有所不同。FABP1是肠细胞中高度丰富的细胞内蛋白,存在于整个肠道,且以十二指肠和空肠的表达水平较高(Agellon et al.,2002)。FABP1有2个FA结合位点,较其他FABPs具有更高的结合力,可直接通过扩散机制将脂肪酸转移至细胞膜上(Hsu and Storch,1996;Storch et al.,2002)。FABP2主要在小肠和肝脏中表达,与其他FABPs家族成员一样,仅拥有单一的结合位点,对不饱和脂肪酸具有较高的亲和力,通过碰撞机制将脂肪酸转移至细胞膜上(Hsu and Storch,1996;Storch et al.,2002)。本研究通过实时荧光定量PCR检测发现,FABP1、FABP2基因在香苏杂交猪和柯乐猪的各组织中均有不同程度的表达,可能与基因在组织中的特异性有关(Alves-Costa et al.,2008;Liu et al.,2008)。肝脏、小肠和脂肪是甘油三酯和脂肪酸合成代谢的主要部位,能改变肌间脂肪及肌内脂肪的含量(Jensen et al.,1990;Hunter,2001)。无论是香苏杂交猪还是柯乐猪,FABP1、FABP2基因均以在肝脏、小肠和脂肪中的相对表达量较高,以背最长肌中的相对表达量最低,表明FABP1和FABP2基因可能广泛参与各组织的表达调控。此外,FABP1和FABP2基因在香苏杂交猪肝脏和小肠中的相对表达量极显著高于其在柯乐猪对应组织中的相对表达量,在脂肪中的相对表达量则显著高于其在柯乐猪脂肪中的相对表达量,故推测这2个基因对香苏杂交猪的调控作用大于柯乐猪。综上所述,FABP1和FABP2基因在猪的肝脏、小肠和脂肪中呈特异性表达,可能与脂肪酸的合成代谢有关。该结论为揭示FABP1和FABP2基因对脂肪酸代谢的调控机制提供了基础理论依据。
4 结论
FABP1和FABP2基因在猪的各组织中均有表达,且以在甘油三酯和脂肪酸合成代谢的相关组织(肝脏、小肠和脂肪)中特异性高表达,可能对脂肪的合成代谢起重要调控作用。
参考文献:
陈芳,张昊,卢立志. 2011. 鸭I-FABP基因的克隆、组织表达模式及功能研究[J]. 畜牧兽医学报,47(5):1041-1048. [Chen F,Zhang H,Lu L Z. 2011. Molecular cloning,expression pattern and function of duck I-FABP gene[J]. Acta Veterinaria et Zootechnica Sinica,47(5):1041-1048.] doi:10.11843/j.issn.0366-6964.2016.05.023.
陳静,尤瑞国,刘慧敏,杨国庆. 2021. 柠檬醛对小鼠生长性能、肌内脂肪含量及脂肪酸代谢酶的影响[J]. 河南农业大学学报,55(4):721-726. [Chen J,You R G,Liu H M,Yang G Q. 2021. Effects of citral on growth performance,intramuscular fat content and fatty acid metaboli-zing enzymes in mice[J]. Journal of Henan Agricultural University,55(4):721-726.] doi:10.16445/j.cnki.1000-2340.20210531.001.
陈璐. 2013. 脂肪酸结合蛋白(FABPs)基因多态性对畜产品品质的影响[J]. 畜牧与饲料科学,34(10):82-84. [Chen L. 2013. Effects of fatty acid-binding proteins (FABPs) gene polymorphisms on animal products quality[J]. Animal Husbandry and Feed Science,34(10):82-84.] doi:10.3969/j.issn.1672-5190.2013.10.040.
崔璐,陈星,周颖,张淑君. 2016. FABPs家族基因的研究进展[J]. 畜牧与兽医,48(7):128-131. [Cui L,Chen X,Zhou Y,Zhang S J. 2016. Research progress on FABPs family genes[J]. Animal Husbandry & Veterinary Medicine,48(7):128-131.]
董飚,秦豪榮,王健,孙国波,纪荣超. 2016. 鸭A-FABP基因多态性在品种和世代间的比较分析[J]. 河南农业科学,45(8):131-135. [Dong B,Qin H R,Wang J,Sun G B,Ji R C. 2016. Comparative study of polymorphism in breeds and generations based on duck A-FABP gene[J]. Journal of Henan Agricultural Sciences,45(8):131-135.] doi:10.15933/j.cnki.1004-3268.2016.08.025.
何俊. 2013. 鸭L-FABP、A-FABP基因多态性与肌内脂肪含量的相关性及其对脂肪代谢相关基因表达的影响[D]. 杭州:浙江大学. [He J. 2013. Study on the associations of duck L-FABP and A-FABP gene polymorphisms with intramuscular fat contents and their influences on the expression of lipid-metabolism related genes[D]. Hangzhou:Zhejiang University.]
钱成. 2016. 贵州白山羊L-FABP和H-FABP基因表达、多态性与生长性状的遗传效应研究[D]. 贵阳:贵州大学. [Qian C. 2016. Association of expression,polymorphism and effects of L-FABP and H-FABP gene with growth traits in Guizhou white goat[D]. Guiyang:Guizhou University.]
田鎏,张昌军,刁红录. 2017. 脂肪酸结合蛋白研究进展[J]. 生命科学,29(2):164-169. [Tian L,Zhang C J,Diao H L. 2017. Advances in fatty acid binding proteins research[J]. Chinese Bulletin of Life Sciences,29(2):164-169.] doi:10.13376/j.cbls/2017021.
俞英,王栋,孙东晓,徐桂云,李俊英,张沅. 2011. 鸡L-FABP基因全长cDNA克隆表达及与杂种鸡脂肪沉积的关系[J]. 遗传,33(7):763-767. [Yu Y,Wang D,Sun D X,Xu G Y,Li J Y,Zhang Y. 2011. Whole cDNA sequence cloning and expression of chicken L-FABP gene and its relationship with lipid deposition of hybrid chickens[J]. Hereditas,33(7):763-767.] doi:10.3724/SP.J.1005.2011. 00763.
邹增丁,陈立祥,苏建明. 2007. 脂肪酸结合蛋白生物学特性及对脂肪代谢调控的研究进展[J]. 饲料工业,28(11):24-26. [Zou Z D,Chen L X,Su J M. 2007. Research progress in fatty acid-binding protein biology and regulation to fatty metabolism[J]. Feed Industry,28(11):24-26.] doi:10.3969/j.issn.1001-991X.2007.11.007.
Agellon L B,Toth M J,Thomson A B R. 2002. Intracellular lipid binding proteins of the small intestine[J]. Molecular and Cellular Biochemistry,239(1-2):79-82. doi:10. 1023/A:1020520521025.
Alves-Costa F A,Denovan-Wright E M,Thisse C,Thisse B,Wright J M. 2008. Spatio-temporal distribution of fatty acid-binding protein 6 (FABP6) gene transcripts in the developing and adult zebrafish (Danio rerio)[J]. The FEBS Journal,275(13):3325-3334. doi:10.1111/j.1742-4658. 2008.06480.x.
Chen X,Gao Y,Wu G,Gu J,Cai Y,Xu J,Cheng H. 2020. Molecular cloning,tissue expression,and transcriptional regulation of FABP1 and FABP2 in javelin goby(Syne-chogobius hasta)in response to starvation stress[J]. Comparative Biochemistry and Physiology. Part B:Biochemistry and Molecular Biology,250:110484. doi:10.1016/j.cbpb.2020.110484.
Gajda A M,Zhou Y X,Agellon L B,Fried S K,Kodukula S,Fortson W,Patel K,Storch J. 2013. Direct comparison of mice null for liver or intestinal fatty acid-binding proteins reveals highly divergent phenotypic responses to high fat feeding[J]. Journal of Biological Chemistry,288(42):30330-30344. doi:10.1074/jbc.M113.501676.
Hotamisligil G S,Bernlohr D A. 2015. Metabolic functions of FABPs—Mechanisms and therapeutic implications[J]. Nature Reviews Endocrinology,11(10):592-605. doi:10. 1038/nrendo.2015.122.
Hsu K. T,Storch J. 1996. Fatty acid transfer from liver and intestinal fatty acid-binding proteins to membranes occurs by different mechanisms[J]. Journal of Biological Chemi-stry,271(23):13317-13323. doi:10.1074/jbc.271.23.13317.
Hunter J E. 2001. Studies on effects of dietary fatty acids as related to their position on triglycerides[J]. Lipids,36(7):655-668. doi:10.1007/s11745-001-0770-0.
Jefferson J R,Powell D M,Rymaszewski Z,Kukowska-Latallo J,Lowe J B,Schroeder F. 1990. Altered membrane structure in transfected mouse L-cell fibroblasts expres-sing rat liver fatty acid-binding protein[J]. The Journal of Biological Chemistry,265(19):11062-11068. doi:10. 1016/S0021-9258(19)38557-6.
Jensen G L,Mascioli E A,Seidner D L,Istfan N W,Domnitch A M,Selleck K,Babayan V K,Blackburn G L,Bistrian B R. 1990. Parenteral infusion of long- and medium-chain triglycerides and reticuloendothelial system function in man[J]. Journal of Parenteral and Enteral Nutrition,14(5):467-471. doi:10.1177/0148607190014005467.
Lawrence J W,Kroll D J,Eacho P I. 2000. Ligand-dependent interaction of hepatic fatty acid-binding protein with the nucleus[J]. Journal of Lipid Research,41(9):1390-1401. doi:10.1016/S0022-2275(20)33451-9.
Liu R Z,Li X D,Godbout R. 2008. A novel fatty acid-bin-ding protein (FABP) gene resulting from tandem gene duplication in mammals:Transcription in rat retina and testis[J]. Genomics,92(6):436-445. doi:10.1016/j.ygeno. 2008.08.003.
Ockner R K,Manning J A,Poppenhausen R B,Ho W K L. 1972. A Binding Protein for fatty acids in cytosol of intestinal mucosa,liver,myocardium,and other tissues[J]. Science,177(4043):56-58. doi:10.1126/science.177.4043. 56.
Ordovas J M. 2009. Genetic influences on blood lipids and cardiovascular disease risk:Tools for primary prevention[J]. The American Journal of Clinical Nutrition,89(5):1509S-1517S. doi:10.3945/ajcn.2009.27113E.
Qu J,Fourman S,Fitzgerald M,Liu M,Nair S,Oses-Prieto J,Burlingame A,Morris J H,Davidson W S,Tso P,Bhargava A. 2021. Low-density lipoprotein receptor-related protein 1 (LRP1) is a novel receptor for apolipoprotein A4 (APOA4) in adipose tissue[J]. Scientific Reports:11:13289. doi:10.1038/s41598-021-92711-0.
Samulin J,Berget I,Lien S,Sundvold H. 2008. Differential gene expression of fatty acid binding proteins during porcine adipogenesis[J]. Comparative Biochemistry and Physio-logy. Part B:Biochemistry and Molecular Biology,151(2):147-152. doi:10.1016/j.cbpb.2008.06.010.
Sharifi K,Ebrahimi M,Kagawa Y,Islam A,Tuerxun T,Yasumoto Y,Hara T,Yamamoto Y,Miyazaki H,Tokuda N,Yoshikawa T,Owada Y. 2013. Differential expression and regulatory roles of FABP5 and FABP7 in oligodendrocyte lineage cells[J]. Cell and Tissue Research,354(3):683-695. doi:10.1007/s00441-013-1730-7.
Stachowiak M,Szydlowski M,Flisikowski K,Flisikowska T,Bartz M,Schnieke A,Switonski M. 2014. Polymorphism in 3' untranslated region of the pig PPARA gene influen-ces its transcript level and is associated with adipose tissue accumulation[J]. Journal of Animal Science,92(6):2363-2371. doi:10.2527/jas.2013-7509.
Storch J,Thumser A E. 2010. Tissue-specific functions in the fatty acid-binding protein family[J]. Journal of Biological Chemistry,285(43):32679-32683. doi:10.1074/jbc.R110.135210.
Storch J,Veerkamp J H,Hsu K T. 2002. Similar mechanisms of fatty acid transfer from human anal rodent fatty acid-binding proteins to membranes:Liver,intestine,heart muscle,and adipose tissue FABPs[J]. Molecular and Cellular Biochemistry,239(1-2):25-33. doi:10.1023/A:1020 546321508.
Tsai I T,Wu C C,Hung W C,Lee T L,Hsuan C F,Wei C T,Lu Y C,Yu T H,Chung F M,Lee Y J,Wang C P. 2020. FABP1 and FABP2 as markers of diabetic nephropathy[J]. Internationl Journal of Medical Sciences,17(15):2338-2345. doi:10.7150/ijms.49078.
Venkatachalam A B,Parmar M B,Wright J M. 2017. Evolution of the duplicated intracellular lipid-binding protein genes of teleost fishes[J]. Molecular Genetics and Geno-mics,292(4):699-727. doi:10.1007/s00438-017-1313-5.
Wang G Q,Bonkovsky H L,de Lemos A,Burczynski F J. 2015. Recent insights into the biological functions of liver fatty acid binding protein 1[J]. Journal of Lipid Research,56(12):2238-2247. doi:10.1194/jlr.R056705.
Wang Q,Li H,Li N,Leng L,Wang Y. 2006. Tissue expression and association with fatness traits of liver fatty acid-binding protein gene in chicken[J]. Poultry Science,85(11):1890-1895. doi:10.1093/ps/85.11.1890.
Wu X F,Liu Y,Gao C F,Chen X Z,Zhang X P,Li W Y. 2018. Novel alternative splicing variants of ACOX1 and their differential expression patterns in goats[J]. Archives Animal Breeding,61(1):59-70. doi:10.5194/aab-61-59-2018.
Zhang Y R,Zhang J M,Ren Y H,Lu R H,Yang L P,Nie G X. 2020. Tracing the evolution of fatty acid-binding proteins (FABPs) in organisms with a heterogeneous fat distribution[J]. FEBS Open Biology,10(5):861-872. doi:10.1002/2211-5463.12840.
Zimmerman A W,Veerkamp J H. 2002. New insights into the structure and function of fatty acid-binding proteins[J]. Cellular and Molecular Life Sciences,59(7):1096-1116. doi:10.1007/s00018-002-8490-y.
收稿日期:2021-12-20
基金項目:贵州省科技计划项目(黔科合服企〔2018〕4007(002)号);贵州省农业重大产业科学研究攻关项目(黔教合KY字〔2019〕011号)
通讯作者:许厚强(1957-),https://orcid.org/0000-0002-4696-5671,博士,教授,博士生导师,主要从事细胞分子生物学及动物遗传育种研究工作,E-mail:gzdxxhq@126.com
第一作者:熊讯(1997-),https://orcid.org/ 0000-0001-9653-1153,研究方向为动物遗传育种与繁殖,E-mail:445182432@qq.com

