链霉菌沉默基因簇激活在天然产物生物合成中的研究进展
马 正,施 越,章金垚,俞晓平
(中国计量大学 生命科学学院 浙江省生物计量及检验检疫技术重点实验室,浙江 杭州 31008)
链霉菌(Streptromyces)是在土壤中发现的丝状革兰氏阳性细菌[1],是放线菌中最大的一个种属,可产生多种抗病毒、抗真菌[2-4]等具有生物活性的次级代谢产物。自20世纪40年代首次报道链霉菌能产生抗生素以来,已从链霉菌中发现了大量的新型抗生素[5]。然而,经过几十年传统生化筛选分离,化合物发现的重复率不断提高。同时,众多广谱抗生素耐药性病原微生物不断涌现,因此,新型天然化合物的挖掘和创制显得尤为重要。
随着基因组测序方法、生物信息学技术的发展[6-8],目前包括antiSMASH[9]和PRISM[10]等在内的诸多分析工具已用于预测天然产物生物合成基因簇及其编码产物的化学结构[11]。大量链霉菌基因组测序分析表明:多数链霉菌基因组中8%~10%基因序列与次级代谢相关,一般都蕴含着几十个次级代谢产物生物合成基因簇,其数目远远超过目前从链霉菌中分离得到的化合物种类[12-13]。由此表明,链霉菌具有合成新天然化合物的巨大潜力[14]。然而,在常规实验室条件下,大多数天然产物(natural products,NP)的生物合成基因簇(biosynthetic gene cluster,BGC)不表达或低表达,这些处于“沉默”状态的生物合成基因簇,被称为沉默基因簇(silent biosynthetic gene cluster)或隐性基因簇(cryptic biosynthetic gene clusters,CBGCs)。目前,如何激活链霉菌中“沉默”基因簇的表达,已成为开发新天然化合物方面的研究热点。
本文将从调控基因的过表达/敲除、基因簇重构/异源表达、共培养、核糖体工程技术、基于报告基因的定向激活、clustered regularly interspaced short palindromic repeats/CRISPR-associated proteins 9(CRISPR/Cas9)编辑技术等方面综述一些激活链霉菌沉默生物合成基因簇的研究进展。
1 利用调控基因激活沉默基因簇
链霉菌细胞内存在复杂且庞大的调控系统,链霉菌次级代谢产物的生物合成受到包括途径特异性、多效性以及全局性调控基因在内的多层级严格调控[15]。因此,无论是通过正向调控基因的过表达还是负向调控基因的敲除,都是激活沉默基因簇表达最直观的方法之一。
1.1 过表达正调控基因
SARP(Streptomycesantibiotic regulatory proteins)普遍存在于链霉菌中并参与调控次级代谢产物的生物合成基因簇,该类调控基因的表达可激活沉默生物合成基因簇。Wu等人利用组成型启动子ermE*将12个SARP类调控基因在Streptomycestsukubaensis L20中过表达,发现tsuR1基因的过表达会产生新型蒽环类药物tsukubarubicin[16],它对枯草芽孢杆菌具有拮抗活性。
Krause等在变铅青链霉菌(Streptomyceslividans)中过表达始旋链霉菌(Streptomycespristinaespiralis)的SARP类调控基因papR2,导致S.lividans十一烷基灵菌红素(undecylprodigiosin,Red)的基因簇被激活。主要机理是由于过表达的papR2弥补了S.lividansRed的生物合成基因簇中SARP调节因子(RedD)的调节功能[17],从而实现Red的生成。除了SARP类调控基因外,LuxR家族调控基因也是常见的调控因子[18]。例如在链霉菌SN-593中过表达LuxR家族调控基因RevQ和RevU可成功激活苯甲醌类抗生素reveromycin A的合成[19]。
1.2 敲除负调控基因
有些调控因子发挥正向调控作用,但有些调控因子的表达反而导致基因簇的沉默,不利于次级代谢产物的生物合成。Li等人在S.coelicolorA3(2)发现一个I型PKS的基因簇(cpk),通过敲除途径特异性负调控基因scbR2(γ-丁内酯蛋白),使菌株丧失放线紫红素(Act)、Red、钙依赖性抗生素(CDA)生产的同时,成功激活出一种黄色色素coelimycin P1[20]。
AdpA(A因子依赖蛋白A)是一种多效性调节因子,通常能参与调节细胞周期分化和抗生素的合成[21]。Becerril等构建粘土链霉菌Streptomycesargillaceus的AdpA缺失突变株(Streptomycesargillaceu-ΔadpAa),分析发现该突变株发酵主产物米曲霉素(mithramycin)的产量明显下降的同时有两个新物质germicidin B/C被激活产生[22],这两个化合物已证实对孢子萌发具有协调作用[23]。同样,Xu等人报道圈卷产色链霉菌Streptomycesansochromogenes中oviedomycin的生物合成基因簇由于adpA基因的缺失而被激活[24]。
1.3 转录因子诱饵
转录因子诱饵(transcription factor decoy,TFD)是模拟阻遏蛋白可能与目标基因簇特异性结合一段DNA片段,通过构建质粒导入宿主增加TFD的拷贝数,“吸引”阻遏蛋白与之结合,从而“解锁”阻遏蛋白对基因簇的负向调控,实现目标基因簇的激活[25](图1)。TFD策略已广泛应用于哺乳动物细胞和临床研究[26-27],Wang等人首次尝试在链霉菌中采用TFD策略激活沉默基因簇[28],在S.lividans66中激活了放线菌素和十一烷基灵芝菌红素生物合成基因簇的表达。该策略也成功激活了Streptomycessp. F-5635中8个沉默基因簇中的3个沉默基因簇(编码butyrolactol A、oxazolepoxidomycin A)的表达(成功率为37.5%)[28]。
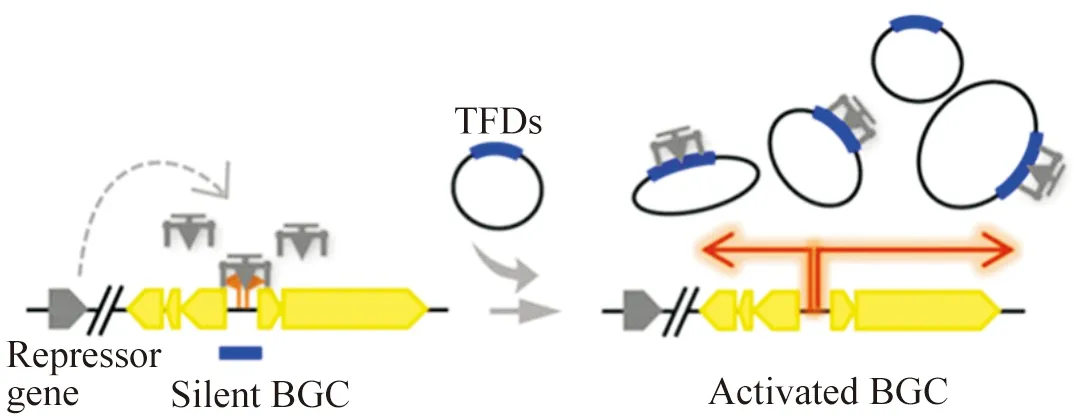
图1 转录因子诱导示意图[28]
2 基因簇重构与异源表达
基因簇处于“沉默”状态的原因是复杂和多方面的,往往是由于受到直接或间接性阻遏造成的,因此,去除或“绕过”阻遏因素是激活沉默基因簇的关键。异源表达作为一种将天然产物化学类型与其基因型联系起来的遗传操作工具,即将沉默基因簇在遗传代谢背景清楚的模式菌株中表达,可有效解除天然菌株中复杂调控网络的影响,从而激活沉默基因簇的表达[29]。选择合适的宿主是沉默基因簇异源表达成败的关键。选取宿主一般有一下三点原则:①宿主遗传背景清楚,易培养,生长周期短;②易于转化和遗传改造,有利于异源沉默基因簇的高效表达;③宿主能供给异源沉默基因簇编码次级代谢物合成所需的前体物质,尽可能少有竞争途径。
2.1 沉默基因簇异源表达及其宿主选择
目前,多个链霉菌已被开发成为异源基因簇表达的良好宿主,如S.coelicolor、S.lividans、S.albus等。比如,S.lividans的Act、Red和CDA基因簇在转录水平上基本处于“沉默”状态,且菌株遗传背景清楚,合成副产物少,因此,S.lividans可作为沉默基因簇异源表达的理想宿主[30]。例如,工程菌株S.lividansΔDYA11是通过删除其11个内源性生物合成基因簇(大小为228.5 kb)并插入另外两个噬菌体ΦC31attB整合位点构建而成[31]。将核苷类抗生素tunicamycin、兰硫肽deoxycinnamycin的生物合成基因簇在该菌株中进行了表达,结果发现S.lividansΔDYA11合成deoxycinnamycin和tunicamycin的产量是在未改造菌株S.lividnasTK24中表达后产量的4倍。
StreptomyccesalbusJ1074是SalI限制修饰系统存在缺陷的S.albusG衍生株,其存在7个rRNA操纵子且具有较小的基因组(6.8 Mbp),因此,S.albusJ1074在大多数沉默基因簇异源表达方面具有代谢背景相对“简洁”、生长速率快等优势[32],已被广泛用作已知基因簇或沉默基因簇异源表达的理想宿主[33-34]。最近Myronovskyi等人报道了对底盘菌株S.albusJ1074的改造,通过对15个内源基因簇(大小500 kb)的缺失和ΦC31attB整合位点的增加,获得衍生株S.albusDel14[35],并成功实现了来源Frankiaspp.沉默基因簇的异源表达,产生了新型化合物fralnimycin[36]。
2.2 基因簇重构和修饰
异源表达能“绕过”原宿主的调控网络来激活沉默基因簇,但通常表达产物的产量较低,分离提取困难。因此,需要提高异源沉默基因簇的转录强度而提升次级代谢产物产量。目前,通过生物合成基因簇的重构和修饰(包括DNA组装和启动子工程[37]),即利用组成型启动子或诱导型启动子替换原启动子(native promoter),增强基因转录水平,从而更有效的激活沉默基因簇在宿主中的异源表达[38-40](图2)。
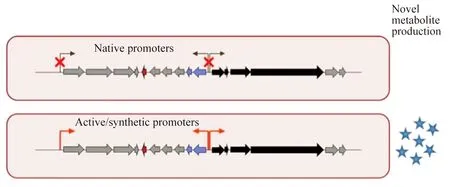
图2 利用启动子替换激活沉默基因簇示意图[40]
在宿主S.coelicolorM145中,Du等以neo(卡那霉素抗性基因)为报告基因,比较了3个组成型启动子ermEp*、SF14p、hrdBp和一个诱导型启动子tcp830p的转录活性,表明hrdBp表达活性最好。谷氏菌素(gougerotin)生物合成基因簇中GouA、GouM-B(一种多顺反子转录单位)和GouN的自身启动子被hrdBp取代,将重构的基因簇在S.coelicolorM146中进行异源表达后,谷氏菌素的产量较未修饰表达的产量增加了10倍以上[41]。
为实现Streptomycesorinoci壮观霉素生物合成基因簇在S.lividans中的表达,Shao等人对其基因簇进行重构[42],利用7种不同的强启动子和1种诱导型链霉菌启动子分别启动基因簇中8个基因的表达,借助TAR克隆技术(transformation-associated recombination,TAR)将重构的基因簇整合到S.lividans基因组上进行异源表达,最终壮观霉素的产量达100 g/L。
3 共培养
自然环境下,链霉菌与许多细菌长期共存且彼此相互作用[43]。因此,在这些细菌和链霉菌共培养(模拟链霉菌与其他菌株持续相互作用的环境)的过程中,会产生多种“诱导子”,从而极大地影响链霉菌的次级代谢,刺激产生新的次级代谢物。目前共培养激活沉默基因簇的机制主要可以分为四种情况:①空间和营养的竞争从而产生“抵制”;②菌株之间的物理接触;③菌株之间化学信号传递(如信号分子);④菌株间遗传信息的“水平转移”。
3.1 链霉菌和非链霉菌细菌共培养
链霉菌和非链霉菌的细菌共培养时,链霉菌会识别细菌表面包括受体蛋白在内的一些蛋白,并产生应答,从而激活新的代谢物来防御“入侵”。枯草芽孢杆菌(Bacillussubtilis)和大肠杆菌(Escherichiacoli)等模式菌株均可被用作链霉菌的共培养菌株。例如当StreptomycesMg1与B.subtilis3610共培养时,StreptomycesMg1会产生抑制甚至裂解B.subtilis3610的查尔霉素A(Chalcomycin A)[44]。活菌体或热灭活的枯草芽孢杆菌均能激活S.coelicolor和S.lividans中Red的产生[45-46]。细菌代谢产生的小分子物质也可作为信号分子激活链霉菌产生新的天然产物。例如,Perez等发现黄色粘球菌(Myxococcusxanthus)与S.coelicolor共培养时,黄色粘球菌会分泌裂解酶,从而激发S.coelicolor形成异常菌丝和产生Act以阻止黄色粘球菌的“破坏”[47]。
除上述提及的枯草芽孢杆菌,黄色粘球菌之外,另外一些病原菌、致病菌(例如耐甲氧西林金黄色葡萄球菌Staphylococcusaureus、铜绿假单胞菌Pseudomonasaeruginosa)和链霉菌共培养时,也能激活链霉菌合成granatomycin D、dihydrogranaticin B等物质[48]。
3.2 不同链霉菌共培养
在链霉菌-链霉菌共培养过程中,由可扩散底物如γ-丁内酯(γ-butyrolactone)和次级代谢物本身介导的种间相互作用被认为是激发次生代谢的主要因素[48]。特别是,GBL(如A因子、virginiae butanolides和IM-2)是广泛分布的信号分子,参与链霉菌间的信号传递[50]。由Streptomyces153产生的聚醚抗生素普罗莫霉素(promomycin),可作为离子载体,通过形成孔道增加K+离子通过细胞膜的外流,抑制细菌生长,而外流的K+离子同时也作为链霉菌种间通讯的信号分子,诱导共培养的Streptomyces574产生抗生素。此外包括盐霉素、莫能菌素和黑霉素在内的其它聚醚抗生素也可激活Streptomyces574抗生素的产生[51]。
除信号分子外,一种链霉菌的次级代谢物本身也可刺激另一种与之共培养的链霉菌次级代谢产物的合成[52]。Streptomycessp. SPB74、S.albusJ1074、S.viridochromogenesDSM40736和Streptomycessp.E14在内的4种链霉菌均可产生铁载体siderophores,在与S.coelicolorM145共培养时,可作为“诱导子”激活S.coelicolorM145产生出γ-actinorhodin、pridiginine(可作为免疫抑制剂抗癌药物)、celichelin、desferrioxamine B(可用于治疗铁中毒)等[53]。
4 核糖体工程
核糖体工程是一种旨在通过调节核糖体相关元件(核糖体蛋白/rRNA)来激活沉默基因并增加抗生素产量的方法,这种方法主要基于引入对攻击核糖体药物的抗性突变,如rpoB(编码ribonucleic acid polymerase,RNAP的β亚基)和rpsL(编码核糖体蛋白S12)基因的突变分别对利福平和链霉素产生抗性,影响菌株的转录和翻译水平,从而改变其代谢产物的合成能力(图3)。目前此方法已在菌株改良和新型抗生素发现等方面得到了较好的应用[54-56]。Shentu等分别利用利福平(Rifampicin,Rif)、链霉素(Streptomycin,Str)、庆大霉素(Gentamicin,Gen)、巴龙霉素(Paromomycin,Par)和红霉素(Erythromycin,Ery)等筛选获得淀粉酶产色链霉菌(Streptomycesdiastatochromogenes)1628的自发单一抗性突变株[57],其中的Str低耐受突变株SD143发生缺失突变,激活S.diastatochromogenes1628产生了多种次级代谢产物。
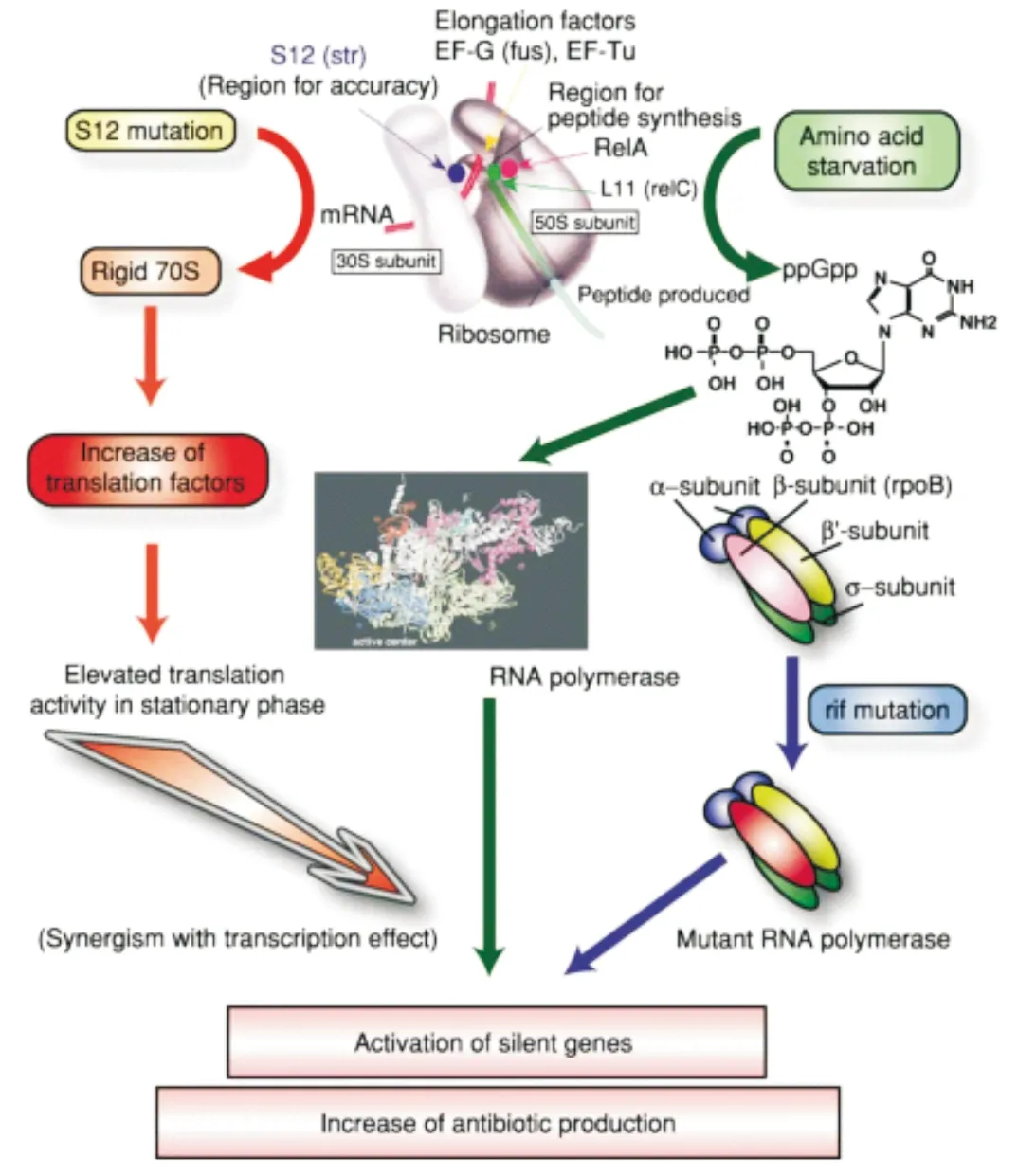
图3 核糖体工程原理示意图[59]
目前关于核糖体工程技术的机制研究较为深入,对于Rif抗性突变株,rpoB基因发生H437D或H437L有益突变有助于次级代谢产物的大幅提升甚至激活沉默基因簇;对于Str抗性突变株产生了正向效果,往往是rpsL基因发生K88E的有益突变,影响核糖体循环因子frr的表达水平从而提高翻译效率[58-59]。Zhang等人[60]利用一种即插即用(plug-and-play)质粒系统直接过表达含上述有益突变的外源rpsL和rpoB基因,并分别在链霉菌S.lividans、S.coelicolorA3(2)、Streptomycessp. FXJ6.204和Streptomycessp. FXJ8.102中进行表达,成功激活了多个沉默基因簇,其中Streptomycessp. FXJ8.102产生了两种新型芳香族聚酮类抗生素piloquinone和homopiloquinone。
5 依赖报告基因的沉默基因簇定向激活
上述利用共培养、核糖体工程技术等策略虽已成功应用于沉默基因簇的激活,但结果具有较高的随机性,通过不可预知的阻遏因素的解除,实现沉默基因簇的激活,但事先无法预判,无法精准实现目标沉默基因簇的激活。目前已有不少报道,以报告基因为指示(reporter gene-guided),结合随机突变筛选等手段,可实现目标沉默基因簇的定向激活[61-62]。
Guo等建立了一种高效的基于报告基因(neo)的核糖体工程突变株筛选策略(reporter-guided mutant selection,RGMS),成功激活了StreptomycesvenezuelaeISP5230中杰多霉素(jadomycin)基因簇的表达和Streptomycessp. PGA64中沉默基因簇pga的表达,分离得到了2个新的蒽醌类化合物gaudimycins D/E[63]。
Li等人在RGMS基础上,用邻苯二酚双加氧酶-卡那霉素抗性基因(XylE-neo)双报告基因体系取代了单报告基因neo[64]。通过表型(菌落颜色变黄)和卡那霉素抗性进行两轮筛选鉴定,成功实现Streptomyceslavendulae中链霉素F(streptothricin F)合成基因簇的激活。
6 激活策略的新工具:CRISPR/Cas9系统
链霉菌遗传操作涉及的基因/基因簇敲除,基因簇重构,启动子替换等在链霉菌遗传改造等方面已有许多成功的案例,但传统的遗传操作周期长,且较为繁琐[65]。近期,随着CRISPR/Cas9(Clustered regulatory interspaced short palindromic repeats/CRISPR-associated protein 9)技术的应运而生,为链霉菌遗传操作的简洁化和高效化提供了契机[66-69]。CRISPR/Cas9生物学的基本信息及其作用机制方面已有较为详细的研究,其作用原理是利用Cas9内切酶与引导RNA(gRNA)形成复合物,并被引导到与gRNA间隔区序列互补的Protspacer序列,特异性剪切双链断裂,然后再通过天然的非同源末端连接修复(NHEJ)或同源定向修复(HDR)机制修复双链,从而完成基因敲除等操作[70-71]。Pristinamycin I(PI)和Pristinamycin II是由Streptomycespristinaespiralis产生的两种组分,Meng等人[72]利用CRISPR/Cas9技术敲除Pristinamycin II生物合成基因簇和缺失阻遏调控基因papR3,构建了一株PI单组分产生菌,大幅增加PI生物效价。
与此同时,CRISPR/Cas9技术在激活链霉菌沉默基因簇方面也得到了较好地应用。利用CRISPR/Cas9技术在S.albusJ1074的NRPS基因簇和PKS-NRPS基因簇的核心基因上游区域插入组成型启动子permE*,从而激活6-epi-alteramides A/B的产生[73]。同样,基于CRISPR/Cas9技术用kasOp*启动子代替沉默基因簇核心基因的天然启动子,成功激活了S.albus、S.lividans、玫瑰孢链霉菌StreptomycesroseosporusNRRL15998等5株链霉菌中10个沉默基因簇的表达[74-75]。这些沉默基因簇涉及I、II和III型PKS、非核糖体多肽合成酶(NRPS)、PKS-NRPS杂合基因簇等。同时,CRISPR/Cas9技术在编辑基因组、优化底盘细胞性能、基因定点突变等方面也得到了广泛应用,显示出作为分子生物学工具的巨大潜力[76-79]。
7 结论与展望未来
从微生物次级代谢产物中分离天然活性化合物是开发新型药物的主要途径之一,但传统的筛选方法面临较大的瓶颈,如:分离纯化前无法预知活性物质结构是否具有新颖性,利用活性追踪分离得到的往往是已报道的物质[80],且耗时长。近年来,随着基因组测序技术和生物信息学的不断发展,激活沉默基因簇已成为新型药物创制的重要研究策略之一[81]。
在这篇综述中,我们总结了目前激活沉默基因簇的一些常用的方法。如过表达正调控基因、敲除负调控基因对调控网络进行修饰,或绕过原宿主的调控进行异源表达,以激活沉默基因簇的表达。随机诱变和核糖体工程技术等虽展现出一定的优势,但筛选过程工作量大、周期长,同时对沉默基因簇的激活具有一定的盲目性。为克服随机筛选的盲目性和繁琐性,以报告基因为导向,结合高通量筛选定向激活沉默基因簇也在逐渐被越来越多的学者采用。随着CRISPR/Cas9技术的出现和发展,在遗传改造链霉菌以及激活沉默基因簇研究方面逐渐体现出了优越性。相信随着生物信息学的发展、组学的深入研究,以及链霉菌遗传改造技术的进步,将会有更多的链霉菌沉默基因簇被挖掘和激活,创制更多的新天然化合物。

