基于生物信息学分析DDX5在头颈部鳞癌中的表达及意义
王文龙 刘郭琦 宋娇娇 于栋林 唐 娟 薛博元 黄玉梅
1 滨州医学院附属医院口腔颌面外科 山东 滨州 256603;2 滨州医学院附属医院口腔修复科 山东 滨州 256603
头颈部鳞状细胞癌(head and neck squamous cell carcinoma, HNSCC)是一种常见的头颈部肿瘤,约占头颈部肿瘤的90%。它常发生于口腔、口咽部和喉部,是一种病死率高达40%~50%的疾病[1-2]。世界范围内每年有超830 000人被诊断患有HNSCC,超过43 000人死于HNSCC[3]。HNSCC的治疗方法包括手术切除、放疗、化疗、免疫治疗、靶向治疗等,但这些方法没有显著提高其5年总体生存率[4]。多数患者往往在癌症晚期确诊,并且治疗效果及预后较差[5]。因此,明确HNSCC的发生发展机制,找出其特异性标志物及通路,对HNSCC的早期发现及靶向和免疫治疗具有举足轻重的作用。
已有研究阐述了circRNAs的多种功能,如circRNA可以作为miRNA的内源RNA(competing endogenous RNAs,CeRNA),与其他RNA竞争miRNA配对[6-7]。circRNA是前体mRNA(pre-mRNA)经过直接反向剪接,将其5′剪接供体和3′剪接受点共价连接形成的单链环状RNA,拥有比线性RNA更稳定的共价闭环结构,不受核糖核酸酶的影响,更耐降解。基于来源和结构,circRNA主要分为外显子circRNA、内含子circRNA和外显子-内含子circRNA[8]。circRNA还可调节细胞核内的转录以及与蛋白质因子结合[9]。circRNA已被证明与人类多种疾病有关,包括癌症、神经退行性疾病和心血管疾病等[10-14]。
DEAD-box解旋酶5(DEAD-box helicases 5,DDX5)是一种通过ATP水解产生的能量催化双链RNA解旋的酶,因其保守序列Walker B 基序中含有Asp-Glu-Ala-Asp(D-E-A-D)而得名[15]。DDX5最早被发现于猿猴病毒40T抗原的免疫交叉反应过程中[16]。DDX5参与调节各种细胞RNA代谢发挥了重要作用,共同激活许多转录因子[17-19]。DDX5在多种癌症中的异常表达影响了癌细胞发生发展,并且与预后、生存以及肿瘤分期等有显著相关性[20-21]。
1 材料与方法
1.1 线上数据库分析 通过线上数据库Timer(http://timer.cistrome.org/)以及UALCAN (http://ualcan.path.uab.edu/index.html)预测DDX5人类多种恶性肿瘤,特别是头颈部鳞癌,与邻近正常组织的表达情况以及意义。
1.2 材料 于滨州医学院口腔颌面外科术中切取标本时分别留取大小约为3 mm×3 mm×3 mm的头颈部鳞癌及对应癌旁组织各5份,组织体积与RNAstore(样本保存液)按照体积比1∶10保存,然后将所有样本储存于液氮中。本研究的实验程序和方案均经滨州医学院附属医院医学伦理委员会批准。纳入标准为:由我院病理科术后常规病理确诊为HNSCC;为首次手术且术前未经放疗和(或)化疗;患者知情同意。
1.3 RNA提取 使用Trizol试剂(Invitrogen/Thermo Fisher Scientific公司,美国)从HNSCC和癌旁组织样本中提取总RNA。使用NanoDrop ND-1000系统(Thermo Fisher Scientific公司,美国)测量RNA表达水平。RNA完整性采用标准变性琼脂糖凝胶电泳方法评估。
1.4 微阵列分析 利用5对癌组织和癌旁组织进行微阵列分析,以便通过比较HNSCC和邻近对照样本来鉴定差异表达的circRNAs和mRNAs。根据Arraystar的标准程序进行样品制备和微阵列杂交。用RNaseR(Eicentre;Illumina,Inc.,美国)消化总RNA以去除线性RNA并丰富circRNA。然后,利用随机引发法(Arraystar Super RNA标记试剂盒,Arraystar公司,美国)扩增富集的circRNA,并转录成荧光cRNA。将标记的cRNA杂交到Arraystar Human circRNA Array V2(Arraystar公司,美国)上。清洗载玻片后,使用安捷伦G2505C扫描仪(安捷伦科技有限公司,美国)扫描阵列。采用安捷伦特征提取软件(11.0.1.1版)用于分析采集的阵列图像。mRNA使用Invitrogen SuperScript的ds-cDNA合成试剂盒,利用Oligo(dT)反转引物(100 pmol)将5 μg总RNA反转录为双链cDNA(ds-cDNA)。按照美国NimbleGen Systems公司的基因表达分析方案对ds-cDNA进行标记。取4 μg标记的ds-cDNA,使用NimbleGen杂交体系在42°C条件下使荧光标记的ds-cDNA与芯片杂交16~20 h,然后使用相应洗脱液洗涤,最后使用Axon GenePix 4000B 微阵列扫描仪对芯片进行扫描。随后的数据处理使用R.中的limma软件包进行两组之间表达的circRNA和mRNAs通过火山图过滤进行鉴定。HNSCC和相邻对照组之间差异表达的circRNA和mRNAs通过折叠变化筛选。
1.5 功能富集分析 基因本体论(gene ontology,GO)项目提供了一个受控词汇表来描述任何生物体中的基因和基因产物属性(http://www.geneontology.org/)。GO分类涵盖生物过程、细胞成分和分子功能。P值表示差异表达基因中GO术语富集的意义。京都基因和基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG,http://www.kegg.jp)路径分析用于收集路径簇,涵盖差异基因表达谱中的分子相互作用和反应网络。P值表示与特定基因相关的通路的重要性。
1.6 OncomiR数据中关键miRNA数据的下载 OncomiR(http://www.oncomir.org/oncomir/)基于癌症基因组图谱(the cancer genome atlas,TCGA)数据库对30种癌症类型的肿瘤发展和进展中的失调miRNA进行预测。本研究将与HNSCC发生发展相关的miRNA纳入分析(参数设置依次为Search by Cancer Type:Tumor Development;Select a significance threshold,P<0.05;Select a cancer type,HNSCC)。同时将差异倍数值大小前5位的circRNA与靶向结合位点的miRNA取交集,共同纳入本次研究。用线上工具(http://bioinformatics.psb.ugent.be/webtools/Venn/)画韦恩图,可视化miRNA交集结果。
1.7 建立ceRNA网络图 应用线上数据库Targetscan预测上述miRNA的靶 mRNA,将预测的结果与芯片所检测到的差异mRNA取交集,构建mRNA-miRNA关系对。保留上述预测circRNA-miRNA和mRNA-miRNA中共有的miRNA、mRNA和共同作用的circRNA。采用Cytoscape软件构建circRNA-miRNA-mRNA调控网络。
1.8 TCGA数据库中头颈部鳞状细胞癌的数据分析 本研究通过TCGA数据库下载了502例癌组织HNSCC患者临床资料并纳入研究,比较了DDX5的表达在癌组织中与患者的性别、TNM分期、临床分期和肿瘤解剖部位的关系。
1.9 RNA 提取及qRT-PCR RNA提取采用的是通用型 RNA 提取试剂盒(艾科瑞生物工程有限公司,中国)。根据制作商的说明,从样本中提取 RNA 样本,并储存于-80℃,存储时间不超过半年。实验步骤均于超净台中无酶条件下进行,使用 Evo M-MLV 反转录试剂盒Ⅱ(艾科瑞生物工程有限公司,中国) 对提取的 RNA 进行逆转录获得 cDNA,储存于在-20℃。 使用 SYBR Green Pro Taq HS qPCR 试剂盒(艾科瑞生物工程有限公司,中国)进行 qPCR。每组样品设3个独立副孔。PCR 条件为:95℃下30 s,95℃下5 s,60℃下30 s,共 40个循环。每组实验重复两次以上减少误差。对于定量分析,使用2-△△Cq 方法计算 mRNA 的相对表达水平。DDX5引物:正向 5′-GCCATTCCTAGAGAGAGGCGA-3′;反向 5′-CCTTAGGAGCACCACCGTAGA-3′。β-actin:正向 5′-TGGCACCCAGCACAATGAA-3′;反向 5′-CTAAGTCATAGTCCGCCTAGAAGCA-3′。
1.10 免疫组化 进行组织层面相关蛋白表达情况的分析,石蜡切片均由滨州医学院附属医院病理科完成,切片厚度在 3~5 μm,利用通用 SP 试剂盒(中杉金桥,中国)进行实验。切片于65℃烤片,二甲苯脱蜡 15 min×3次,梯度酒精水化 10 min×6 次,EDTA 抗原修复,过氧化氢酶、山羊血清依次室温孵育 10 min,一抗 37℃恒温箱中孵育 1 h,生物素标记山羊抗小鼠/兔 IgG 室温孵育 15 min,辣根酶标记链霉卵白素工作液孵育 10 min,DAB 显色,显色过程于显微镜下进行,显色时间约15~30 s,苏木素复染 1 min,盐酸酒精分化 2~3 次,冲水反蓝,中性树胶封片,最终由两名经验丰富的病理科医师进行判读结果。
1.11 统计学方法 采用GraphPad Prism8软件进行统计分析。qRT-PCR 数据分析采用两个独立样本t检验进行。CircRNA和mRNA的显著差异表达水平采用FDR筛选进行比较分析。P<0.05为差异有统计学意义。
2 结果
2.1 circRNA、mRNA微阵列分析 本研究对5对HNSCC组织进行circRNA、mRNA分析,共检测到287个circRNAs,其中有146个上调circRNAs和141个下调circRNAs (P<0.05,差异倍数>1.5)。共有1 053个失调mRNA,其中有377个mRNAs高表达和676个mRNAs低表达(log2差异倍数>1.3,P<0.05)。将显著上调和下调的mRNA表达绘制在火山图上和散点图(图1A、B),直接展示了两个对照组之间被检测的差异基因。聚类分析表明,circRNA在两组间存在差异表达(图1C)。由此可见,在HNSCC组织中,相较于配对的癌旁组织,mRNA、circRNA的表达谱是有差异的。

A. mRNA火山图;B. mRNA热图;C. circRNA热图。
2.2 GO和KEGG富集分析 GO分析可以分为分子功能(molecular function,MF)、生物过程(biological process,BP)和细胞组分(cellular component,CC)三个部分。本研究应用GO分析推断差异的mRNA可能存在的分子功能。1 053个差异mRNA主要涉及的CC主要富集在“细胞外细胞器”“细胞外膜结合细胞器”“胞外囊泡外小体”“细胞外周”“质膜”“膜的组成部分”等。主要涉及的MF主要富集在“外源性肽抗原的抗原加工和呈递”“肽抗原结合”“核糖体的结构成分”“核糖体的结构成分”以及“抗原结合”等。主要涉及的BP主要富集在 “外源性肽抗原的抗原加工和呈递”“肽抗原的抗原处理和呈递”“系统过程”“G蛋白偶联受体信号通路”“抗原处理和呈递”以及“感觉知觉中化学刺激的检测”等条目(图2)。本研究采用KEGG富集通路分析差异mRNA所参与的功能,基于差异表达的mRNA进行通路分析以推断参与的通路。差异的mRNAs主要富集在“抗原处理和呈递”“移植物抗宿主病”“自然杀伤细胞介导的细胞毒性”(图2)。

A~C分别为上调的mRNA在细胞组分、分子功能、生物过程中的前10个显著性富集GO项;D~F分别为下调的mRNA在细胞组分、分子功能、生物过程中的前10个显著性富集GO项。
2.3 与差异表达的circRNA结合的miRNA 本研究通过对差异表达的circRNA进行阈值筛选,以P<0.01, FDR<0.1为阈值,选取了差异倍数值大小在前5位的circRNA(包括hsa_circRNA_102485、hsa_circRNA_014280、hsa_circRNA_402089、hsa_circRNA_036186、hsa_circRNA_404474)。然后,利用Arraystar自制的miRNA靶点预测软件预测circRNA-miRNA关系对,选出前15个miRNA。接着,利用在线工具OncomiR筛选 HNSCC中肿瘤形成这一方面发现,与肿瘤形成、发展有关的miRNA共有371个。本研究通过与15个差异表达的miRNA取交集发现共有hsa-miR-103a-2-5p、hsa-miR-342-3p、hsa-miR-505-5p、hsa-miR-377-3p 4种miRNA(图3)。

图3 与差异表达的circRNA结合的miRNA筛选韦恩图
2.4 miRNA-mRNA预测 本研究利用数据库Targetscan对上述4种的差异miRNA预测其靶向的mRNA发现共有7 315个靶向的mRNA,再与本研究芯片中已筛选出的差异mRNA取交集发现共有75个的差异mRNA(图4),构建了miRNA-mRNA关系对。
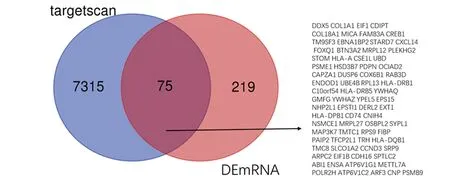
图4 miRNA-mRNA预测筛选韦恩图
2.5 构建ceRNA调控网络 本研究通过OncomiR、Targetscan数据库与芯片预测取交集,分别得到circRNA-miRNA以及miRNA-mRNA关系对,利用Cytoscape软件构建及可视化1个由2个circRNA、4个miRNA和75个mRNA组成的ceRNA网络图(图5)。
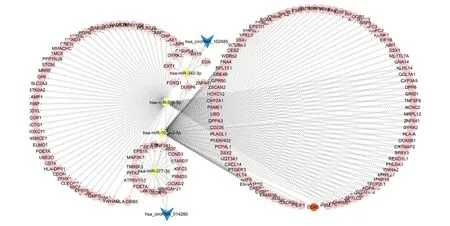
粉色圆点代表mRNA,黄色方块代表miRNA,蓝色箭头代表circRNA。
2.6 DDX5在泛癌中的表达 本研究前期通过对Timer数据库分析发现,与邻近正常组织比较,多种广泛类型人类肿瘤的DDX5 mRNA表达水平差异有统计学意义,在膀胱尿路上皮癌、乳腺浸润癌、胆管癌、结肠癌、食管癌、头颈部鳞癌、肾嫌色细胞癌、肝细胞癌、肺腺癌、肺鳞癌、皮肤黑色素瘤、胃癌、甲状腺癌、子宫内膜癌中有显著表达(图6A)。值得注意的是, DDX5在HNSCC中的表达上调,P<0.05。UALCAN数据库预测显示,与邻近正常组织比较,HNSCC中的DDX5高表达,P<0.05(图6B)。同时,本研究在上述ceRNA网络图中发现了DDX5的上游miRNA-103a-2-5p,circRNA-102485(图6C)。
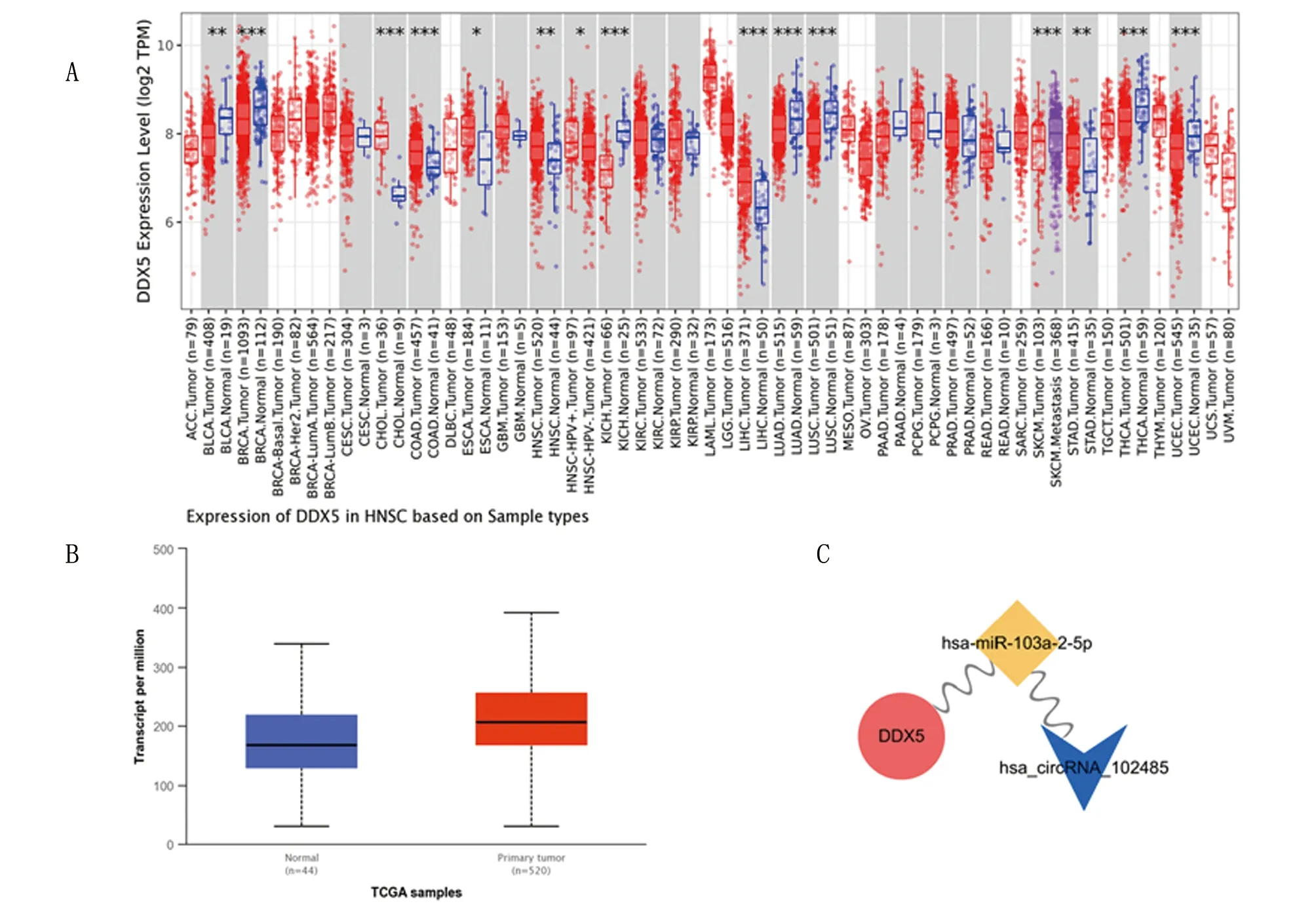
A. 研究所有TCGA肿瘤中DDX5基因在肿瘤和邻近正常组织之间的差异表达情况,*P<0.5,**P<0.01,***P<0.001;B. 预测与邻近正常组织比较,HNSCC中的DDX5高表达;C. 利用Cytoscape可视化miRNA-103a-2-5p-circRNA-102485-DDX5轴。
2.7 HNSCC中DDX5表达的临床分子特征 本研究对基于TCGA数据库下载的512例HNSCC样本进行分析,通过基础R包将FPKM(fregments per kilobase per million)格式的RNAseq数据转换成TPM(transcripts per million reads)格式,并进行log2转化,通过中位数区分出DDX5高表达(n=251)和DDX5低表达(n=251)。本研究发现,DDX5的表达与患者的临床分期(P<0.05)、性别(P<0.05)和肿瘤解剖细分(P<0.05)显著相关,但是与TNM分期无相关性(表1)。
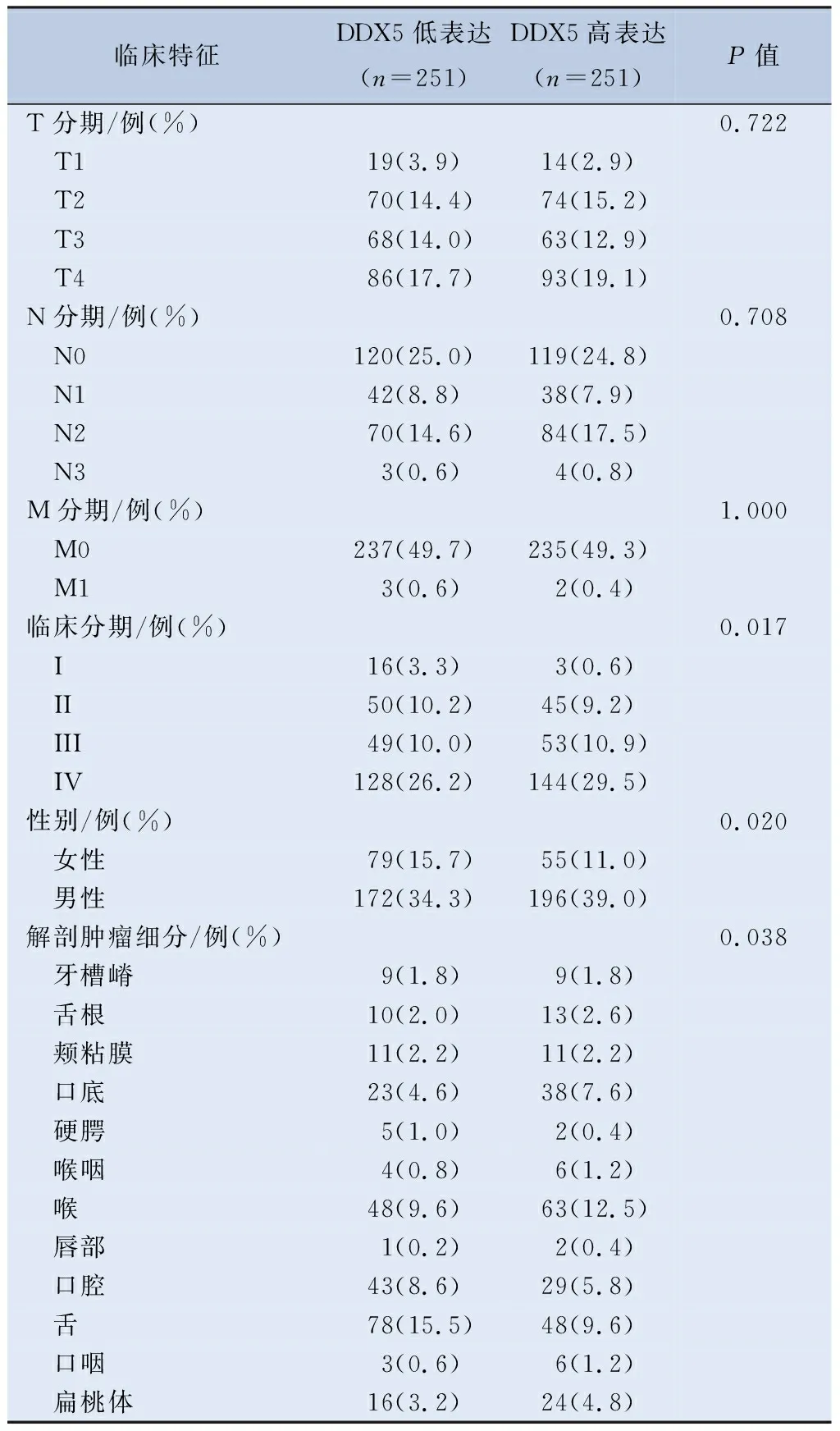
表1 HNSCC中DDX5表达的临床分子特征
2.8 DDX5在HNSCC中差异表达的验证 DDX5在组织中的阳性染色主要位于细胞质,在细胞核中也有少量表达。与癌旁组织比较,DDX5在HNSCC组织中阳性率相对较高(图7A)。本研究还收集用于芯片分析的组织在内的12对HNSCC样本的RNA,采用qRT-PCR验证生物信息学分析结果。结果显示,与癌旁组织比较,DDX5在HNSCC的表达增加,P<0.05(图7B)。这与本研究中芯片分析和免疫组化结果是一致的。

A. 免疫组化法检测HNSCC组织中的DDX5的表达(×100,×400),黑箭头表示癌旁组织,红色箭头表示癌组织;B. 癌及癌旁组织qRT-PCR结果,*P<0.05。
3 讨论
在过去10年里,HNSCC最显著的治疗进展是免疫疗法的应用。免疫疗法基于以下前提,即癌症需要免疫监视、癌细胞表型和/或肿瘤微环境的改变才能逃避免疫系统并在临床上表现出来。事实上,免疫系统被认为是HNSCC的发展、建立和传播的关键[22-23]。目前已有研究证明DDX5在HNSCC发生发展中的重要作用,但通过DDX5寻找上游靶基因以及发现DDX5及其上游基因构成的信号通路在HNSCC发生发展中的作用的研究还未有进展。因此,研究寻找HNSCC标志物DDX5及其相关基因通路有助于深入了解该疾病,并及时进行各种免疫疗法和靶向治疗。
环状RNA(circRNAs)是真核生物中共价闭合的内源性生物分子,具有组织特异性和细胞特异性表达模式,其生物发生受特定顺式作用元件和反式作用因子调控。许多circRNA通过作为microRNA或蛋白质抑制物,或调节蛋白质功能,或通过自身翻译发挥重要的生物学功能[24]。尽管这些转录的环状性质使它们的检测、定量和功能表征具有挑战性,但高通量RNA测序和circRNA特异性计算工具的进展推动了鉴定circRNAs 方法的发展[25]。
本研究通过基因芯片筛选来鉴定circRNA、mRNA在样本癌及癌旁组织中的表达谱,并通过ceRNA分析来评估circRNA-miRNA-mRNA的相互作用。基因芯片筛选结果显示,HNSCC组织以及癌旁组织共检测到287个circRNAs,发现有146个上调circRNAs和141个下调circRNAs。另外,发现共有1 053个失调mRNA,其中有377个mRNAs高表达和676个mRNAs低表达。以差异倍数值大小在前五位选出hsa_circRNA_102485、hsa_circRNA_014280、hsa_circRNA_402089、hsa_circRNA_036186、hsa_circRNA_404474。接着对miRNA进行重叠,筛选出hsa-miR-103a-2-5p、hsa-miR-342-3p、hsa-miR-505-5p、hsa-miR-377-3p。再对差异miRNA预测其靶向的mRNA,发现共有75个共同靶向的差异mRNA。本研究还构建了ceRNA网络图,通过先前Timer以及UALCAN数据库预测DDX5 mRNA在HNSCC的表达是显著上调的。总之,本研究发现了circRNA-102485-miRNA-103a-2-5p-DDX5轴。目前尚未有研究报道circRNA-102485、has-miR-103a-5p在肿瘤中的作用,尚需对其功能进行进一步研究。
DDX5作为一种ATP依赖性RNA螺旋酶,参与广泛的生物过程,包括转录、翻译、前体信使RNA加工或选择性剪接,从而影响细胞增殖、转移、分化以及凋亡等,由此认为,其与恶性肿瘤的发生发展密切相关[17, 21]。本研究先前发现,DDX5解螺旋酶在多种肿瘤中异常表达,且可作为乳腺癌、非小细胞肺癌、肝细胞癌、食管癌等的治疗靶点[8, 17, 19, 26-27]。本研究借助芯片技术以及多种线上数据库,利用Targetscan、OncomiR、Timer和UALCAN数据进行挖掘,发现DDX5在HNSCC中是高表达且有意义,故在ceRNA网络图中发现了circRNA-102485-miRNA-103a-2-5p-DDX5轴。在骨肉瘤中,Circ-XPR1通过分泌miR-214-5p产生的吸附作用可以调节DDX5的表达,从而对骨肉瘤细胞的增殖产生影响,为治疗相关靶点提供了基础[28]。本研究发现,与癌旁组织比较,HNSCC中的DDX5 mRNA水平和蛋白水平都明显上调。免疫组化发现,与癌旁组织比较,DDX5主要集中于癌巢,并且在qRT-PCR中DDX5在HNSCC的表达也相对上调,这也与本研究的芯片预测是一致的。DDX5作为一种共转录激活因子,与信号转导和转录激活因子-3形成DDX5/STAT3轴,以控制肿瘤促进因子的转录,包括成纤维细胞生长因子、血管内皮生长因子、转化生长因子-β、基质金属肽酶-2、缺氧诱导因子、白细胞介素-6等,从而影响肿瘤生长、集落形成、血管生成、癌细胞迁移[29]。DDX5在HNSCC中的高转录水平与临床分期以及肿瘤的解剖部位有密切关系,因此,DDX5在HNSCC中可作为潜在的诊断生物标志。与此同时,Xu等[30]发现DDX5可以作为生物治疗肺癌的一个靶点,miR-431抑制了DDX5的表达,同时明显抑制了肺癌细胞的增殖、侵袭和迁移。DDX5在胃癌中的表达是显著上调的,且与胃癌病理分期有密切关系[21]。DDX5作为组织特异性致癌分子的一种常见上游调节因子,为肠道疾病的治疗提供了极好的靶点[31]。
综上所述,本研究通过构建DDX5-miRNA-circRNA网络调控图,揭示了DDX5在HNSCC中的表达是明显上调的。DDX5在HNSCC中展示了其所参与的复杂的生理过程,具有成为HNSCC诊断的潜在生物标记物的潜力,但仍需要进一步的临床以及基础实验来验证。本研究通过采用芯片技术以及线上数据库的结合为HNSCC的研究机制和靶向治疗提供了新的潜在靶点。

