绿豆Alfin1-like基因家族的鉴定与干旱胁迫下的表达分析
周旭旭, 刘金洋, 陈 新, 薛晨晨, 陈景斌, 林 云, 闫 强, 吴然然, 朱月林, 袁星星
(1.南京农业大学园艺学院,江苏南京210095;2.江苏农业科学院经济作物研究所/江苏省高效园艺作物遗传改良重点实验室,江苏南京210014)
Alfin1-like蛋白家族是PHD蛋白家族中的1个亚家族,其N端有1个Alfin结构域,C端有1个保守的PHD-finger结构域。Alfin1-like基因只存在于植物中,其在进化上也是非常保守的,具有4个内含子和5个外显子[1]。Alfin1-like蛋白与植物的耐盐性有很大关系。第一个Alfin1蛋白是从苜蓿中分离出来的[2],过表达Alfin1基因可以增强植物的耐盐性[3]。在其他作物中,Alfin1-like蛋白在应对盐胁迫、干旱胁迫等逆境中都有很重要的调节作用,比如过表达大豆中GmPHD2(1种编码Alfin1的基因)的拟南芥表现出耐盐性[4]。在鹰嘴豆的干旱胁迫分析中,发现1个Alfin1-like转录因子在干旱胁迫下,上调表达, 该转录因子可能与抗旱相关[5]。
绿豆是亚洲重要的豆类作物,也是中国重要的小杂粮作物。绿豆富含蛋白质、维生素、矿物质和碳水化合物,是一种优质的膳食原料[6]。本课题组前期以苏绿1号为试验材料完成了全基因组测序和盐胁迫下的转录组测序,在获得的基因组及转录组数据中均能检测到Alfin1-like基因家族的10个成员。因此,本研究首先在全基因组水平上,通过比较基因组学分析对VrAL(绿豆Alfin1-like)基因进行鉴定,并对其家族成员启动子及基因复制进行分析,研究绿豆中该基因家族的复制特征。其次,用含有20% PEG6000(聚乙二醇6000)的Hoagland营养液处理绿豆幼苗,采用qRT-PCR探究VrAL基因家族对PEG模拟干旱的响应特点。旨在探索VrAL基因家族成员在干旱胁迫下基因表达的差异性。
1 材料与方法
1.1 材料的处理与获得
试验材料为绿豆[Vignaradiata(L.)Wilczek]苏绿1号,在江苏省农业科学院经济作物研究所温室内以蛭石为基质进行穴盘育苗,每盘培育50株幼苗,共2个穴盘,用1/2浓度的Hoagland营养液[7]进行底面吸肥,每2 d更换1次营养液。
播种后10 d,第1对真叶充分展开后,参照曹志敏等[8]的方法,在1个穴盘的营养液中添加20%的PEG6000模拟干旱胁迫;对照组穴盘的营养液中不添加PEG6000。参照谢敏等[9]的方法,在绿豆幼苗被胁迫0 h、12 h、24 h、36 h时分别在处理组和对照组随机取样5株,取叶片和根部组织各0.1 g保存于液氮中,为qRT-PCR分析备用。
1.2 全基因组VrAL家族成员鉴定
基因家族成员鉴定遵循以下步骤:①使用7条拟南芥AL蛋白序列和绿豆蛋白质数据库进行本地BLAST搜索,E值设置为1×10-10,获得绿豆AL家族候选序列,拟南芥和绿豆蛋白质数据从NCBI网站(https://www.ncbi.nlm.nih.gov/)下载[10]。②将AL家族候选序列在NCBI网站(https://www.ncbi.nlm.nih.gov/)上进行Pfam检测,去除不含Alfin1-like结构域的序列。③ 将获得的AL家族序列与本课题组之前完成的苏绿1号全基因组测序数据进行比较以确认所获得序列的准确性。④将10条VrAL家族蛋白质序列上传到在线网站ExPASy(https://web.expasy.org/protparam/)分析VrAL家族蛋白质的理化性质[11],用在线网站Cell-PLoc(http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2/)进行亚细胞定位预测[12]。
1.3 VrAL基因结构及进化分析
使用TBtools工具[13]进行基因结构分析并进行可视化操作。利用在线网站(Phytozome doe.gov)获得水稻、大豆、玉米的基因数据,从中挑选出用拟南芥AL基因家族注释的基因,并获得其氨基酸序列。再用Clustal W对拟南芥、水稻、大豆、玉米、绿豆AL家族的蛋白质序列进行序列比对。最后,在MEGA 7.0中用邻接(Neighbor-joining)法构建系统进化树,Bootstrap设置为1 000次,其他设置选择默认参数。
1.4 VrAL基因家族的染色体定位、启动子及基因复制分析
利用TBtools工具从绿豆基因组中VrAL基因家族5′端上游各选2 000 bp,使用PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)在线网站对基因家族成员进行启动子分析[14]。从预测结果筛选出与植物激素响应及逆境响应相关的元件,并用TBtools工具将其可视化。为了进一步分析基因重复事件,本研究拟分析非同义替换率(Ka)和同义替换率(Ks),估算各旁系之间的Ka/Ks比值,以检测该基因家族在进化中受到的选择压力。
1.5 VrAL家族蛋白质的多序列比对及互作蛋白质预测
利用DNAman对绿豆10个AL家族蛋白质进行多序列比对[9],并将绿豆10条AL蛋白的氨基酸序列上传至在线网站string(https://www.string-db.org/)进行互作蛋白质预测,Network type设置为full STRING,Required score设置为medium confidence(0.400)。
1.6 PEG模拟干旱胁迫下VrAL家族的qRT-PCR分析
利用植物RNA提取试剂盒[DP419,天根生化科技(北京)有限公司产品]来提取总RNA。使用植物反转录试剂盒(TSK302M,南京擎科生物科技有限公司产品)进行反转录。用内参基因检验反转录合成的cDNA第一条链的完整性。使用NCBI Primer designing tool(https://www.ncbi.nlm.nih.gov/tools/primer-blast/)在线设计VrAL基因家族的qRT-PCR特异性引物,并用TBtools-primer check检查引物的特异性。引物信息见表1。以反转录的 cDNA 为模板,使用荧光定量试剂盒(TSE201,南京擎科生物科技有限公司产品)进行qRT-PCR分析。每个样品进行5次生物学重复和3次技术重复。利用VrAL基因的循环数阈值(Ct)减去内参基因的Ct,得到△Ct后,用 2-△Ct法计算相对表达量[15],使用SPSS统计软件对基因表达水平进行 Duncan’s 检验,P<0.05表示差异显著,P<0.01表示差异极显著。

表1 VrAL基因家族及内参基因荧光定量引物
2 结果和分析
2.1 VrAL蛋白家族成员的生化特性
经过BLAST比对和挑选,绿豆全基因组中共鉴定到10个AL基因。根据它们在染色体上的分布,依次命名为VrAL1~VrAL10。绿豆VrAL基因的编码区(Coding sequence,CDS)位于717(VrAL8)~765 bp(VrAL3),编码的氨基酸序列长度为239~255 aa,相对分子质量为27 000~28 800,理论等电点为4.91(VrAL2)~6.24(VrAL9)。亲水性预测结果表明,VrAL蛋白均为亲水性蛋白质(表2)。VrAL蛋白亚细胞定位预测结果表明,VrAL蛋白均定位在细胞核。
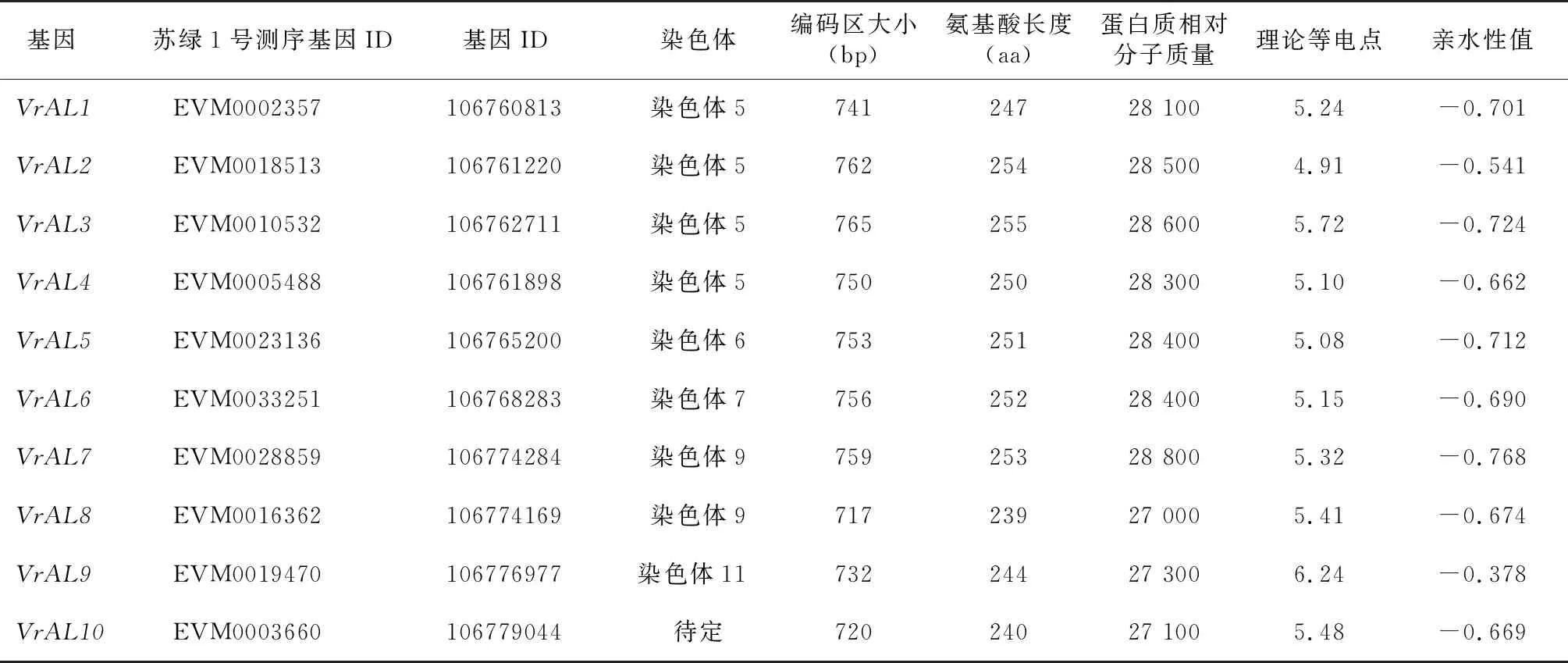
表2 绿豆AL基因家族成员及其编码蛋白质的生化特性
2.2 VrAL保守结构域、基因结构及进化树分析
从进化树可以看出,10个VrAL基因分出3个分支,VrAL1、VrAL6、VrAL7、VrAL3基因属于第一个分支,VrAL10、VrAL8、VrAL5、VrAL4、VrAL9属于第二个分支,VrAL2单独在最后一个分支上;从进化树上可以看出,VrAL基因家族的进化过程中存在基因复制事件,VrAL基因家族共存在5个基因对,包括8个基因(VrAL1/VrAL6、VrAL1/VrAL7、VrAL5/VrAL8、VrAL5/VrAL10、VrAL4/VrAL9)(图1a)。10个VrAL蛋白都含有Alfin1-like蛋白结构域(pfam12165)和PHD蛋白结构域(pfam00628)(图1b)。基因结构分析结果表明,10个VrAL基因比较保守,都含有4个内含子和5个外显子(图1c),这与拟南芥中AL基因结构相同[16]。
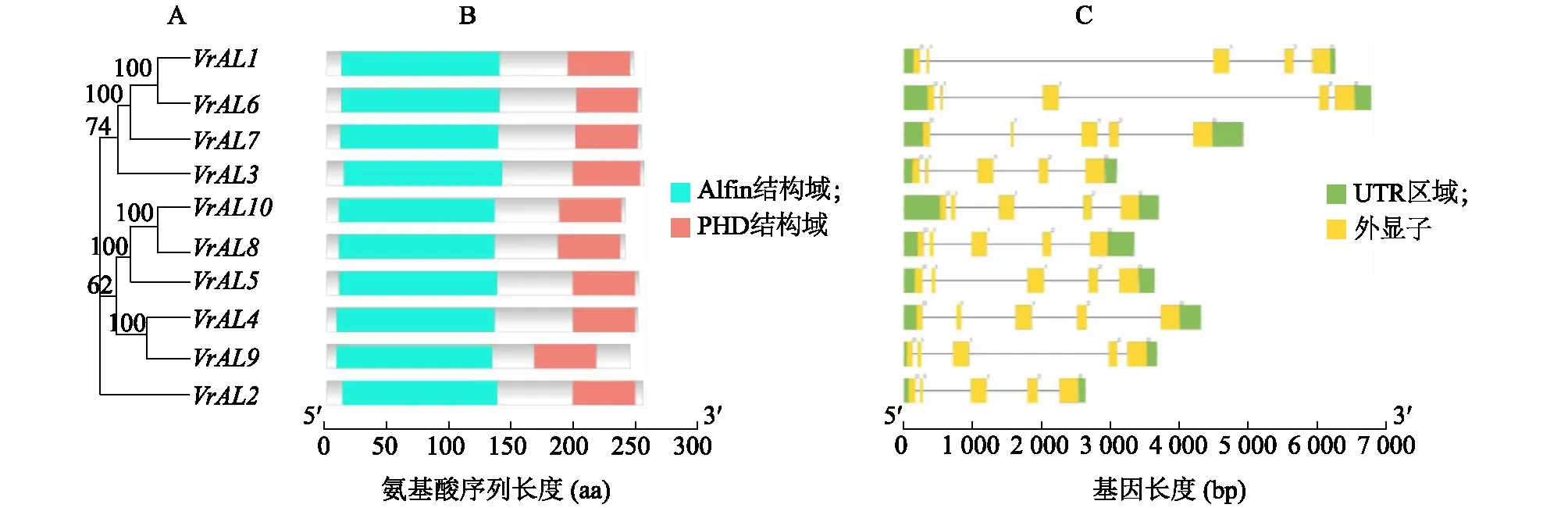
图1 VrAL基因家族系统进化树(A)、对应的Pfam结构(B)及基因结构(C)
利用拟南芥(7个)、大豆(16个)、玉米(17个)、水稻(8个)及绿豆(10个)AL蛋白全长序列构建系统进化树(图2)。根据进化关系,这58个AL蛋白可分为3类,VrAL在3类中都有分布。
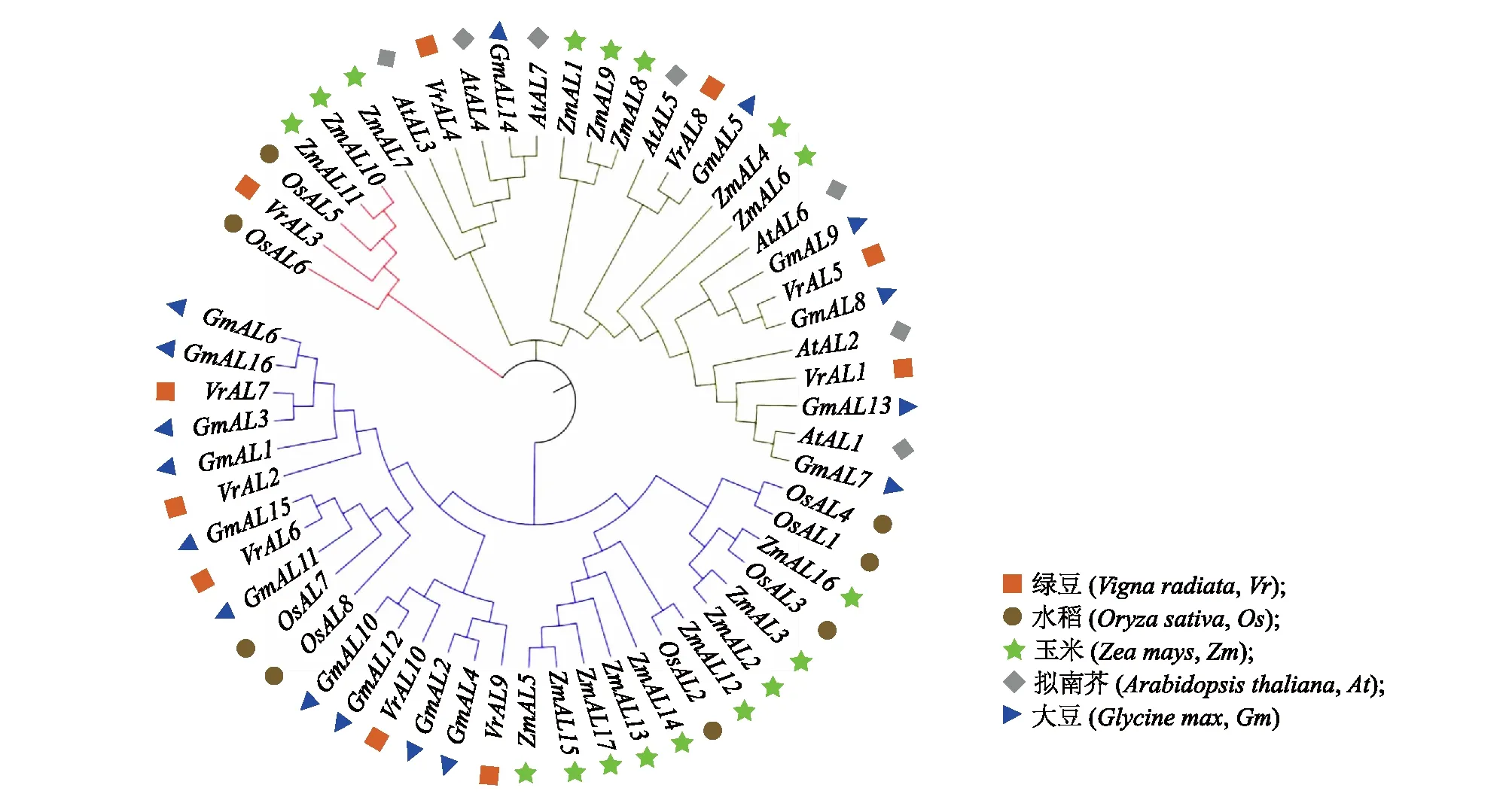
图2 绿豆、拟南芥、大豆、玉米及水稻AL基因家族系统进化树
2.3 VrAL基因的顺式作用元件、染色体定位及复制分析
从PlantCARE在线网站中预测VrAL基因家族成员上游2 000 bp序列,这些序列除了具有光响应、逆境响应和无氧响应元件外还具有多种植物激素(茉莉酸甲酯、生长素、水杨酸、脱落酸等)响应元件(图3)。其中VrAL1、VrAL2、VrAL4、VrAL6、VrAL8的启动子区域存在脱落酸响应元件,VrAL2、VrAL3、VrAL4、VrAL6、VrAL9的启动子区域存在茉莉酸甲酯响应元件,VrAL2、VrAL4、VrAL6、VrAL9的启动子区域存在干旱胁迫响应元件,VrAL4、VrAL6、VrAL7、VrAL8的启动子区域存在逆境响应元件,VrAL3、VrAL7、VrAL9的启动子区域存在SA响应元件,VrAL2、VrAL6、VrAL7的启动子区域存在生长素响应元件。AL基因家族在其他物种中被报道主要与植物的生长发育以及抗逆境胁迫有关,这与VrAL基因家族的启动子作用元件的分析结果也是一致的。
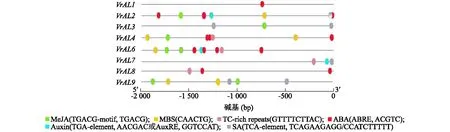
MeJA:茉莉酸甲酯响应元件;MBS:干旱胁迫响应元件;TC:逆境响应元件;ABA:脱落酸响应元件;Auxin:生长素响应元件;SA:水杨酸响应元件。
9个VrAL基因定位在5条染色体上,VrAL1、VrAL2、VrAL3、VrAL4定位在第5条染色体上,VrAL5定位在第6条染色体上,VrAL6定位在第7条染色体上,VrAL7、VrAL8定位在第9条染色体上,VrAL9定位在第11条染色体上,VrAL10暂时不确定定位在几号染色体上(表2)。绿豆种内的AL基因家族共线性分析结果表明,10个VrAL基因存在5个基因对,分别是VrAL1/VrAL6、VrAL1/VrAL7、VrAL5/VrAL8、VrAL5/VrAL10、VrAL4/VrAL9(图4)。Ka/Ks的计算结果显示,除了VrAL4/VrAL9由于序列差异过大无法计算Ka/Ks外,其他同源基因对VrAL1/VrAL6、VrAL1/VrAL7、VrAL5/VrAL8、VrAL5/VrAL10的Ka/Ks值都小于0.20(表3)。上述结果表明,重复的VrAL基因在进化过程中受到纯化选择[17]。片段复制是VrAL基因家族进化的主要方式,在VrAL基因家族进化中起重要作用,这与大豆AL家族中的研究结果一致[4]。
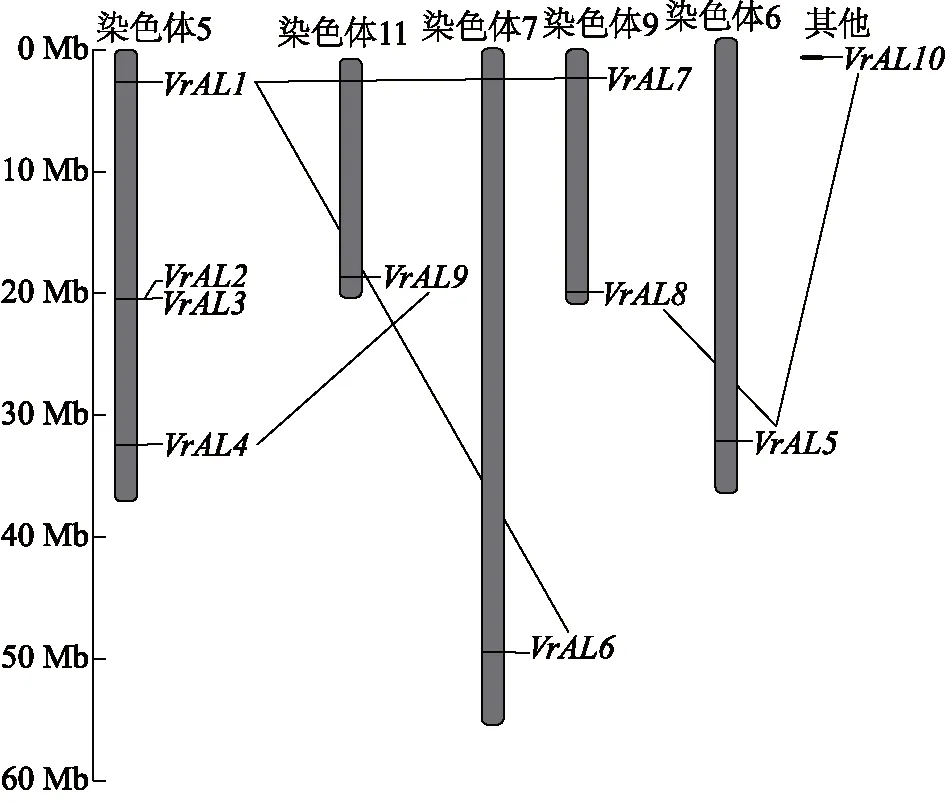
片段复制的基因用直线连接。
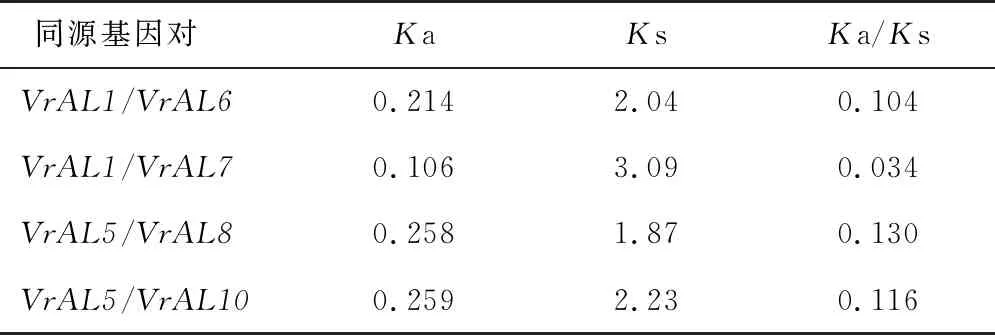
表3 VrAL基因家族同源基因对的Ka/Ks
2.4 VrAL家族蛋白质的多序列比对及其互作蛋白预测
10个VrAL蛋白的多序列比对结果如图5所示,其中N端含有1个长约130个氨基酸的Alfin结构域(pfam12165),C端含有1个长约50个氨基酸的PHD结构域(pfam00628)。其中加粗实线框为PHD结构域中的酪氨酸,10个VrAL蛋白中只有VrAL2在该位点不是酪氨酸。利用蛋白质互作预测在线网站String预测绿豆中与AL蛋白互作的蛋白质,其功能注释主要有 E3 泛素蛋白连接酶DRIP2及其亚体、组蛋白赖氨酸N-甲基转移酶ASHR3、ATP合成相关的酶、普通的应激A蛋白、DEAD-box ATP依赖性RNA解旋酶5等。

实线框为Alfin结构域(pfam12165);虚线框为PHD结构域(pfam00628);加粗实线框为酪氨酸。
2.5 VrAL基因家族在PEG模拟干旱条件下的qRT-PCR分析
如图6所示,在20%PEG培养条件下,绿豆根中有4个VrAL的相对表达量比对照组显著或极显著增加,分别是VrAL1(图6A)、VrAL5(图6B)、VrAL7(图6C)、VrAL9(图6D)。VrAL1在用20%PEG培养12 h后相对表达量开始升高, 24 h时相对表达量达到所选取时间段的最高值,约为同时间对照组的7倍,36 h时相对表达量下降,大约是同时间对照组的2倍。VrAL5和VrAL7在处理组中的相对表达量一直处于增长的趋势,在36 h时的相对表达量约是对照组的3倍。VrAL9在用20%PEG培育12 h时,相对表达量达到所选时间段中的最高值,是同时间段对照组的3倍,之后开始下降。VrAL2、VrAL3、VrAL4、VrAL6、VrAL8和VrAL10在根部处理组中的表达和对照组无明显差异。
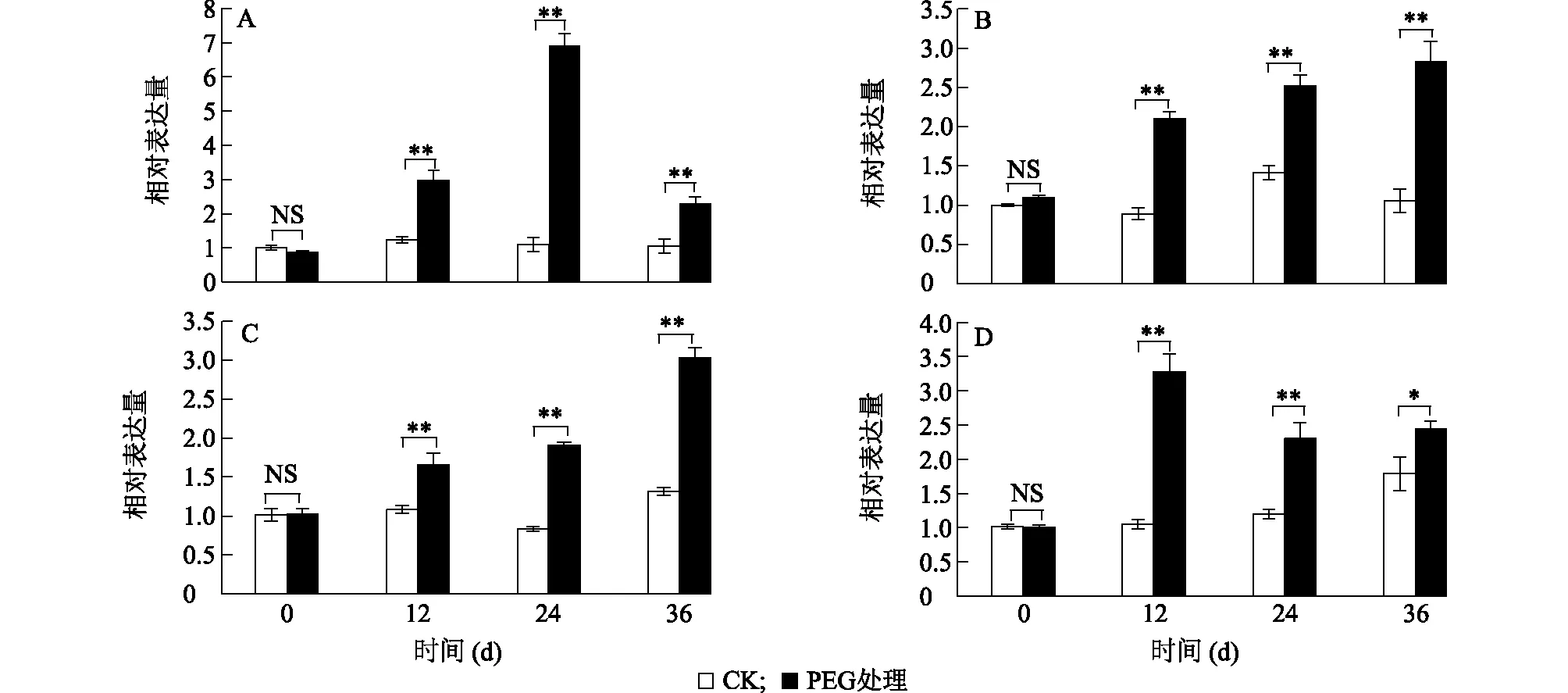
A、B、C、D分别表示根部4个基因VrAL1、VrAL5、VrAL7、VrAL9的qRT-PCR分析。*表示用t测验在0.05水平差异显著,**表示用t测验在0.01水平差异显著,NS表示两组间无显著差异(P>0.05)。
如图7所示,在绿豆叶片中有3个VrAL在用20%PEG培育后上调表达,分别是VrAL8(图7A)、VrAL9(图7B)和VrAL10(图7C)。VrAL8和VrAL9都是在用20%PEG培养12 h后相对表达量达到最高值,分别约是对照组的2.2倍和4.2倍,之后相对表达量开始下降。VrAL10在用20%PEG培养12 h后相对表达量开始上升,到36 h达到最高值,约是对照组的3倍。其他7个基因的相对表达量和对照组无明显差异。
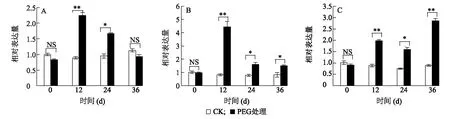
A、B、C分别表示叶片3个基因VrAL8、VrAL9和VrAL10的qRT-PCR分析。*表示用t测验在0.05水平差异显著,**表示用t测验在0.01水平差异显著,NS表示两组间无显著差异(P>0.05)。
3 讨论
3.1 生物信息学预测VrAL基因家族功能
本研究从绿豆基因组中鉴定出10个AL成员,这些成员的结构非常相似。与其他物种(拟南芥、大豆、玉米、水稻)的AL基因结构也非常相似,暗示AL基因家族在植物界是非常保守的。AL蛋白家族通常作为一种转录因子。VrAL蛋白的亚细胞定位预测结果显示其全部定位在细胞核,预测VrAL蛋白是作为一种转录因子发挥作用。顺式作用元件分析结果显示,大多数VrAL启动子序列中含有逆境和激素响应元件,预测VrAL在绿豆抗非生物胁迫中发挥重要作用。
从植物进化的关系上来看,植物中都存在AL基因,从低等植物莱茵衣藻(Chlamydomonasreinhardtii)到高等植物拟南芥(Arabidopsisthaliana)、苜蓿(MedicagosativaL.)、水稻(OryzasativaL.)、大豆[Glycinemax(L.) Merr.]、玉米(ZeamaysL.)、毛果杨(PopulustrichocarpaTorr. & Gray)、葡萄(VitisviniferaL.)、榆钱菠菜(AtriplexhortensisL.)和大白菜(BrassicarapaL.)中都有报道[4,18-20]。通过多序列比对和系统发育树分析发现,VrAL家族中有3组同源基因,VrAL1、VrAL6、VrAL7属于一组,VrAL5、VrAL8、VrAL10属于一组,VrAL4和VrAL9属于一组。这3组基因在进化过程中存在片段复制现象。通过蛋白质互作预测也发现与VrAL家族蛋白质互作的蛋白质主要有E3泛素蛋白连接酶DRIP2及其亚体、组蛋白赖氨酸N-甲基转移酶ASHR3、ATP合成相关的酶、普通的应激A蛋白、DEAD-box ATP依赖性RNA解旋酶5等,表明VrAL家族基因在调控绿豆生长发育过程中起着不可忽视的作用。
拟南芥AL蛋白具有与活性组蛋白标记H3K4me3/2和顺式作用元件GNGGTG/GTGGNG结合的能力,表明AL家族作为一种转录因子可能主要通过结合DNA来抑制其他基因的转录来起作用。AL蛋白与H3K4me3/2结合能力的有无通常与AL蛋白C末端PHD结构中的酪氨酸密切相关。拟南芥中的7个AL蛋白中只有AL3的PHD结构中不含有酪氨酸,组蛋白肽下拉试验结果也证明AL3确实不能与H3K4me3/2结合[21]。在VrAL蛋白的多序列比对中发现,VrAL2蛋白的PHD结构域不含酪氨酸,预测VrAL2蛋白可能没有与H3K4me3/2结合的能力。
3.2 VrAL基因参与干旱胁迫的响应
AL基因家族是PHD基因家族中的一个亚家族,但是只存在于植物的PHD家族中。AL转录因子家族,在植物的生长发育以及抵抗逆境胁迫中起重要作用。苜蓿中Alfin1蛋白可以调节MsPRP2基因的表达,该基因编码一种富含脯氨酸的细胞壁蛋白质,并积极调节植物的耐盐性和抗旱性[3,22-23]。在拟南芥中,AtAL5蛋白可与8个目标基因(SHMT7、bZIP50、CAX1、TAC1、FAO、OFE、NAC075和WRKY11)的启动子结合并抑制其转录,其中5个基因(TAC1、SHMT7、FAO、OFE和CAX1)的敲除突变体均表现出对盐、干旱和冷冻胁迫的耐受性增强[24],AtAL6控制多个下游基因的表达,包括ETC1、NPC4、SQD2和PS2,以促进缺磷条件下的根毛伸长[25]。在榆树中发现AhAL1结合GRF7、DREB1C和几个与ABA相关的编码基因的启动子区域,并下调其表达;在AhAL1转基因植物中DREB2A、DREB1A和3个ABF基因的表达量增加[26]。在对耐旱和干旱胁迫敏感的水稻的转录组比较中也发现,部分AL基因响应干旱胁迫[27]。在对鹰嘴豆的干旱胁迫分析中也发现1个对干旱响应表达上调非常明显的AL转录因子[5]。但是绿豆中的AL家族还没有相关的研究报道。在本试验中,PEG模拟的干旱胁迫下,绿豆根部和叶片中VrAL1、VrAL5、VrAL7、VrAL8、VrAL9和VrAL10上调表达。推测VrAL1、VrAL5、VrAL7、VrAL8、VrAL9、VrAL10的上调表达和绿豆抵抗干旱胁迫有关,具体的调控网络还有待进一步的研究。

