CRISPR/Cas生物传感器检测食源性病原体的研究进展
黄璐琦,孙旭飞,苑 宁,范少华,张 伟,4,5*
(1 河北农业大学食品科技学院 河北保定 071000 2 河北农业大学理工学院 河北沧州 061100 3 河北软件职业技术学院 河北保定 071000 4 河北农业大学生命科学学院 河北保定 071000 5 河北省人畜共患病原微生物分析与防控重点实验室 河北保定 071000)
食源性疾病作为全球范围内的重大公共卫生问题之一,关系着人们的身体健康和生命安全。目前,引起食源性疾病的病原体包括食源性致病菌、病毒、生物毒素、药物残留、重金属、非法添加物等[1]。据WHO 报道,全球每年约有6 亿人(几乎每10 人中就有1 人) 因食用受污染的食品而患病,导致约42 万人的死亡,造成3 300 万健康生命年损失。各国每年用来治疗食源性疾病的费用约高达150 亿美元[2]。其中,食源性致病菌和病毒是造成食源性疾病的重要原因[3-4]。常见的食源性致病菌有沙门氏菌、单核细胞增生李斯特氏菌、金黄色葡萄球菌、大肠杆菌等。常见的食源性病毒有诺如病毒、轮状病毒、冠状病毒、猪瘟病毒等[5-6]。生物传感器因具有响应时间快、成本低、不需要任何预富集步骤、可移植性和易于使用的优点,被广泛应用于食源性病原体的检测[7]。成簇的规律间隔的短回文重复序列及其相关蛋白(Clustered regularly interspaced short palindromic repeats/CRISPR associated proteins,CRISPR/Cas) 系统是于1987年由Ishino 等在大肠埃希菌中首次发现的[8]。它是由细菌进化出的一种具有免疫记忆的获得性免疫防御系统,用于抵制外源核酸及噬菌体的入侵[9]。CRISPR/Cas 系统具有较强的核酸识别特性,近年来逐渐被用作生物传感系统研究的识别元件。本文介绍并总结CRISPR/Cas 系统及其在食源性病原体检测中的应用。
1 CRISPR/cas 系统
近年来,由于成簇的规律间隔的短回文重复序列CRISPR 及其相关蛋白Cas 系统可以作为一种高效的基因组编辑工具,因此引起研究人员的关注。2019年12月,CRISPR 基因编辑技术入选了Nature 近10年中最具影响力的五项重大科学事件之一,并作为“变革生物技术”斩获了2020年诺贝尔奖[10]。CRISPR/Cas 系统通过Cas 效应蛋白-向导RNA(guide RNA,gRNA)双链体进行高特异性识别活性,使Cas 效应蛋白对靶DNA 产生切割活性[11],引起双链断裂(Double-stranded DNA break,DSB),进而激活细胞内源通路修复DSB 缺口,实现对靶位点的编辑[12]。有研究表明,CRISPR/Cas 系统分布于47%已测序细菌和90%已测序古细菌的基因组中,目前已知有2 大类6 个亚型[13-14]。
1.1 作用机制
完整的CRISPR 基因座包含一系列CRISPR阵列、CRISPR 相关Cas 蛋白和由不同间隔区(26~72 bp)分隔的短的、高度保守的重复序列(21~48 bp)[15-16]组成。当噬菌体或质粒等外源DNA 入侵宿主后,细菌通过CRISPR 序列与Cas 蛋白的共同作用来防御噬菌体的入侵,其作用机制包括“捕获-表达-干扰”3 个阶段的免疫反应。
在捕获阶段,首次入侵的噬菌体会被保守的适应性蛋白Cas1 和Cas2 识别加工成短片段,通过获取入侵噬菌体的核酸序列,在一些相关效应因子或一些效应蛋白的参与下将其整合到细菌的CRISPR 序列中,形成间隔子序列。在表达阶段,CRISPR 阵列被转录为前体crRNA(pre-crRNA),然后被加工为成熟的crRNA(crispr RNA)。1 类系统中,Cas6 作为一种核糖核酸内切酶可以切割pre-crRNA 中重复序列的单个磷酸二酯键使其成为成熟的crRNA[17]。2 类系统的机制较为复杂多样,有些需要RNase III 对pre-crRNA 进行加工[18],且这一加工过程需要反式激活tracrRNA (transactivating crRNA)的参与[19];有些可以独立转录出成熟的crRNA。在某些V 型以及所有VI 型系统中并未发现tracrRNA 的存在,Cas12a 和Cas13a可分别通过不同的机制直接加工pre-crRNA 形成成熟的crRNA[20]。在干扰阶段,效应蛋白复合物或单个的效应蛋白与gRNA 结合形成核糖核蛋白复合体,通过特异性识别前间隔序列毗邻基序(Protospacer adjacent motif,PAM),使gRNA 与入侵靶标碱基互补,Cas 核酸酶特异性切割外源核酸序列,从而达到防御的目的。作用原理图见图1。
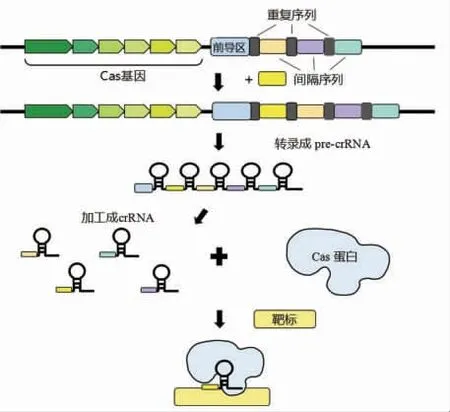
图1 CRISPR-Cas 系统结构部件机理示意图Fig.1 Diagrammatic illustration of mechanism of structural components of CRISPR-Cas system
1.2 分类
随着研究人员对CRISPR/Cas 系统的深入研究,CRISPR 系统中的各种Cas 核酸酶相继被发现。2015年,Makarova 等[21]根据CRISPR/Cas 系统中Cas 复合体的组成与Cas 蛋白结构特征,将CRISPR/Cas 系统分为2 类-5 型-18 亚类,这是首次对CRISPR/Cas 系统进行较为明确的划分,已得到普遍认可。在此基础上,研究人员于2016年又发现一种新型的CRISPR/Cas 系统,进一步对CRISPR/Cas 家族进行补充与完善。目前,根据CRISPR/Cas 系统参与干扰阶段的Cas 蛋白数量分为两大类,6 个亚型,每种亚型都有标志性的Cas 核酸酶[22]。第1 类包括I、III 和IV 型,这种类型需多种Cas 蛋白共同参与干扰机制。第2 类包括II 和V 和VI 型,这种类型只需一个具有多个结构域,分子质量较大的蛋白参与干扰阶段[23]。由于2 类系统简单、高效,因此常用于生物工程和CRISPR 诊断。目前研究最广泛的是Cas9 系统、Cas12 系统和Cas13 系统。不同类型Cas 系统分类见表1。
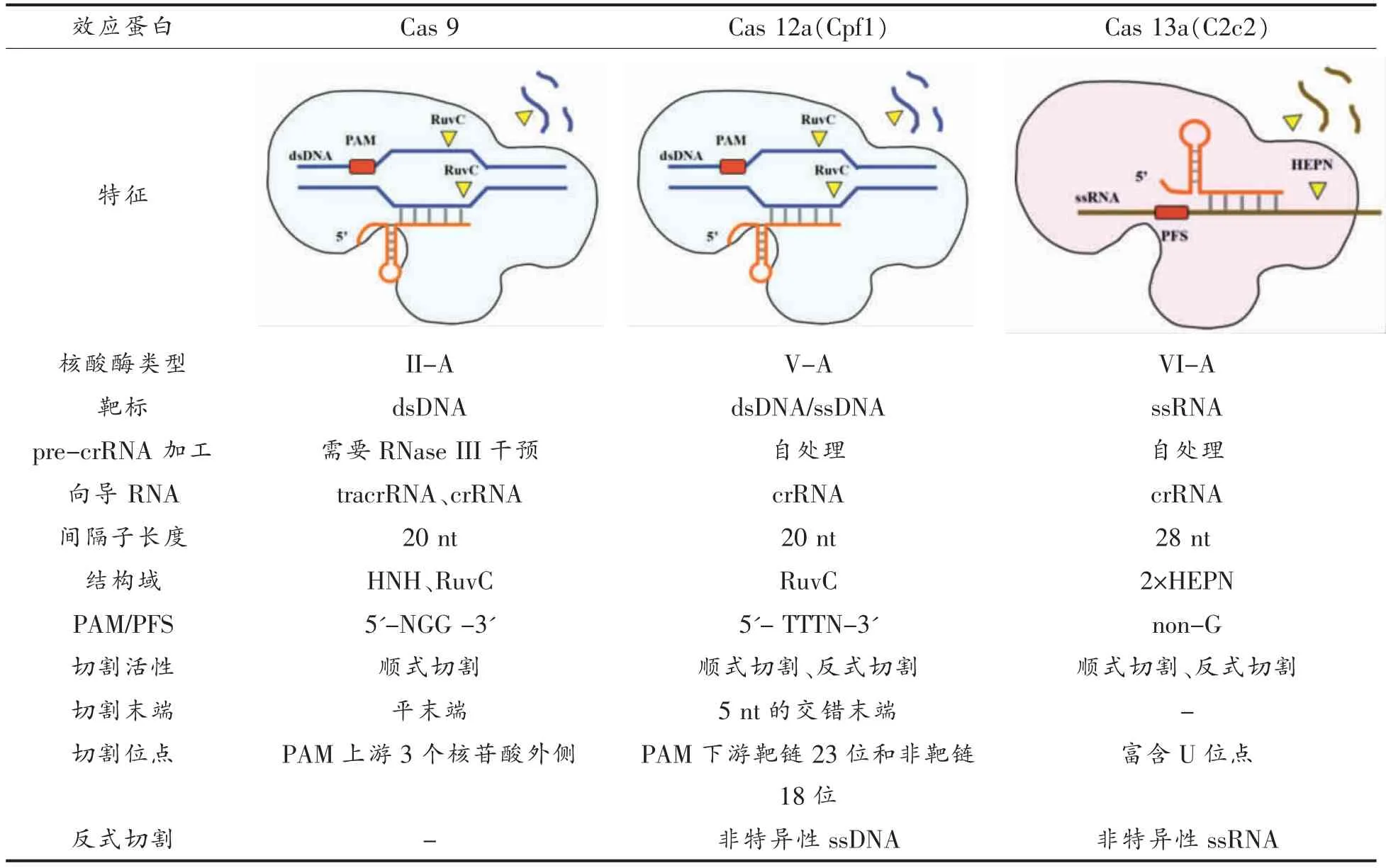
表1 CRISPR-2 类效应核酸酶[24,34,61]Table 1 Class 2 CRISPR-associated effector nucleases[24,34,61]
1.2.1 Cas9 系统 Cas9 是一种由双RNA 引导的具有DNA 核酸内切酶活性的效应蛋白,可以通过组氨酸-天冬氨酸-组氨酸样结构域 (His-Asn-His,HNH)和原核同源重组蛋白Ruv C 样结构域(Cross-over junction endodeoxyribonuclease Ruv C)诱导特定双链DNA(dsDNA)断裂[22,25]。2012年,Wiedenheft 等[26]发现从嗜热链球菌中纯化出的Cas9 可通过crRNA 靶向切割基因,这一发现极大地推动了研究人员对于Cas9 系统的研究。Cas9 系统是II 型CRISPR/Cas 系统中最具有标志性的家族,由crRNA、tracrRNA 和Cas9 蛋白3 个重要部分构成。Charpentier 和Doudna 团队[25]将tracrRNA和crRNA 的组合命名为单一的向导RNA(gRNA)。在基因编辑中,crRNA 和tracrRNA 还可以人工融合产生嵌合的单链导向RNA(sgRNA)。gRNA 与Cas9 的结合会产生活性核糖核蛋白复合物,对靶序列的识别、切割起着至关重要的作用[27]。
Cas9 系统的激活首先需对特异性目标进行识别,由tracrRNA 和crRNA 形成的gRNA 识别靶标PAM。PAM 最早由Mojica 等[28]于2009年提出,他们发现在原间隔子靶序列的一侧存在2~5 nt 的保守序列,这段序列可使CRISPR/Cas 系统区分出自己的基因组DNA 和入侵的核酸序列[29],在病毒入侵时加速识别[30],促使dsDNA 链分离,RNA-靶DNA 杂交形成R 环[31]。PAM 一般存在于间隔子靶序列下游3 nt 处,Cas9 蛋白的PI 结构域可识别5'-NGG-3' 的PAM[30]。在CRISPR/Cas9 系统中,对PAM 的识别会启动dsDNA 解链,使crRNA 链侵入并与靶碱基配对。靶标DNA 的识别严格依赖于DNA 靶位点下游同源PAM 的存在,同时也需PAM 旁边的种子序列与gRNA 的特异性互补结合[32]。种子序列即crRNA 与靶DNA 互补的前8~10 个碱基[25]。有研究表明,种子序列的存在能够进一步促进crRNA-靶DNA 的杂交,种子序列的错配还会导致酶切割活性的丧失[33]。在识别PAM 区和种子序列后,crRNA5' 末端的20 个核苷酸通过碱基互补对目标靶序列进行捕获,形成一个R 环结构[34]。一旦crRNA-DNA 杂交R-环开始形成,Cas9 就会启动效应蛋白的酶切特性,在位于HNH和RuvC 结构域的PAM 序列上游3 个碱基处以二价阳离子依赖的方式分别催化靶DNA 链和非靶DNA 链的切割,产生平的断裂DSB 位点[35-37]。
随着对Cas9 研究的深入,有研究发现了Cas9n(Cas9 nickase)[38],它是Cas9 变种中的一种。这种类型的Cas9 是由在RuvC 或HNH 域中引入D10A 或H840A 沉默突变产生的[39]。该酶可在靶标的相应链上产生切口。而当两个域都发生突变并同时失活,会形成缺乏核酸酶的另一变种dCas9(dead Cas9),失活的dCas9 核酸酶只保留了与DNA 的结合能力,可特异性地结合到靶DNA 序列上,但不发生切割[40]。dCas9 还与相应的转录激活或者抑制域结合,利用CRISPR/dCas9 系统,实现对靶标基因转录的激活或者抑制,实现基因位点的可视化成像等[41]。
1.2.2 Cas12 系统 与CRISPR/Cas9 类似,V 型Cas12 蛋白也是CRISPR 家族2 类成员之一,这种蛋白可在gRNA 的作用下特异性结合互补的靶单链DNA(ssDNA)和dsDNA[42],激发非特异切割活性。到目前为止,已发现Cas12 的多种亚型,有VA,Ve-B,Ve-C,Ve-U,其中对Cas12a (Cpf1)和Cas12b(C2c1)[43]的研究较多。
Cas12a 系统由crRNA 与Cas12 蛋白两部分组成。与CRISPR/Cas9 相比,CRISPR/Cas12a 构成分子质量较小,约有1 200 至1 500 个氨基酸[44]。Cas12a 效应蛋白具有一个特征的RuvC 核酸酶结构域,同时具有DNase 和RNase 活性[45]。与Cas9不同,Cas12a 系统中crRNA 的形成不需要tracr-RNA 或RNase III 的参与。pre-crRNA 由Cas12a自身的RNase 结构域加工,使Cas12 在CRISPR重复序列形成的发夹结构上游4 nt 处剪切precrRNA,产生中间crRNA 分子,在体内进一步加工成约43 nt 的成熟crRNA[46]。Cas12a 与成熟cr RNA 形成Cas12a-crRNA 核糖核蛋白二元复合体,发生构象变化,表现出对dsDNA 的切割活性。在crRNA 的引导下,Cas12a 识别位于靶标上富含T 的PAM 序列与5~8 nt 的种子序列[47]。当crRNA特异性识别靶标链形成RNA-DNA 结合体后,Cas12a 通过RuvC 结构域中的核酸酶位点,在PAM 序列的远端(3'方向)18 nt 与非靶标链PAM位点的远端(5'方向)23 nt 处对靶标链进行切割,从而形成5 nt 突出的黏性末端。除了高特异性的dsDNA 切割活性,Cas12a 还显示出与crRNA/目标DNA 形成三元复合物后持续的Cas12 裂解活性,可以无选择地发挥DNase 活性[46,48],通过RuvC 结构域裂解非特异的ssDNA,即反式切割活性[49]。
2015年,Shmakov 等[50]鉴定出一个新的CRISPR 系统蛋白Cas12b (又称为 C2c1)。与Cas12a 不同,CRISPR/Cas12b 系统是由tracrRNA和crRNA 组成的gRNA 为导向的Cas12b 蛋白核酸酶,可以识别5'-(A)TTN 的PAM 序列,切割双链DNA 后产生7 nt 突出的黏性末端[51-52]。由于Cas12a 仅带有用于DNA 切割的RuvC 结构域,因此还可以被修饰成核酸酶失活的Cas12a(dead Cas,dCas12a),用于基因编辑等领域。
1.2.3 Cas13 系统 2016年,研究人员在沙氏纤毛菌 (Leptotrichia shahii) 中发现一种新型2 类VI-A 型蛋白Cas13a(又称C2c2)[53]。同年,该研究团队在进行沙氏纤毛菌C2c2(Lsh Cas13a)的体外切割试验时发现,在靶RNA 存在的情况下,Cas13a-crRNA 复合物不仅可以切割靶RNA,还可以非特异性地切割侧枝ssRNA。迄今为止,CRISPR/Cas13 是目前CRISPR/Cas 家族中除第1类III 型外唯一靶向RNA,且具有非特异性切割活性的CRISPR/Cas 系统。已发现的Cas13 蛋白有4 种亚型[54],即VI-A(Cas13a/C2c2)、VI-B(Cas13b)、VI-C(Cas13c)和VI-D(Cas13d)。Cas13a(C2c2)亚型是第1 个广为人知且具有典型特征的Cas13蛋白。
Cas13a 系统是由crRNA 与Cas13 蛋白两部分构成。crRNA 是由pre-crRNA 在Cas13 的单独作用下形成的,不需要traRNA 的参与。Cas13a 蛋白是由1 个识别瓣叶(Recognition lobe,REC)和1个核酸酶瓣叶(Nuclease lobe,NUC)组成。其中,组成REC 叶片的Helical-1 结构域负责切割precrRNA 形成成熟的crRNA,组成NUC 叶片的2 个高等真核生物和原核生物核苷酸结合域(Higher eukaryotes and prokaryotes nucleotide-binding,HEPN)负责诱导Cas13a 发生协同构象变化,使无酶切活性状态的Cas13a-crRNA 复合物表现出RNase 活性,特异性地对靶向RNA 进行顺式切割[55-56]。Cas13a 蛋白一旦被靶RNA 活化,也会展现出针对ssRNA 的“附带切割”能力,裂解附近的其它ssRNA[57-58]。一直以来,由于不涉及dsDNA 解链的关系,靶标不包含PAM 序列。然而,当成熟的crRNA 与Cas13 蛋白结合形成Cas13-crRNA 复合物后,该复合物会在靶RNA 链3'端识别与PAM 类似的前间隔序列侧翼位点 (Protospacerflanking site,PFS),激活Cas13 蛋白在crRNA 互补位置外的侧翼序列附近切割靶RNA。据Abudayyeh 等[53]发现,Cas13a 复合物更偏向于在富U区进行非特异性切割。
为了扩展CRISPR/Cas13a 系统在RNA 研究上的应用,研究人员对Cas13a 蛋白的HEPN 结构域进行突变失活,可以产生无RNA 核酸酶活性的Cas13a(dead Cas13a,dCas13a)。dCas13a 能够特异性识别和结合靶向RNA 而不进行切割,通过与各种不同功能性蛋白进行融合,可实现RNA 从编辑到修改再到可视化的各种操作[39]。2017年,Smargon 等[59]还发现另一种同样具有靶向和编辑RNA能力的蛋白Cas13b。与Cas13a 不同的是,Cas13b发挥作用需要靶RNA 的两端均存在PFS 结构,这增加了该系统对ssRNA 切割的限制条件[60]。
2 CRISPR-传感器在食源性病原体检测中的应用
近年来,基于CRISPR/Cas 的技术已广泛应用于基因组编辑、基因转录调控、表观遗传工程和许多其它领域。CRISPR/Cas 系统之间的多样性和Cas 蛋白的特异性,使CRISPR/Cas 系统被用作生物传感器和生物传感系统开发中的识别元件。生物传感器虽具有灵敏、灵活、低成本和快速等优点,但也存在一些缺点,如特异性差。CRISPR/Cas系统具有可编程、高特异性的诱导切割活性,这为识别目标提供了机会,可满足生物传感器理想条件下对目标的检测。将CRISPR/Cas 系统与生物传感器结合,可利用其独一无二的识别与切割活性,满足对目标高特异、高灵敏的检测。目前有研究人员对基于CRISPR/Cas 的生物传感系统进行研究和开发[61]。特别是CRISPR/Cas 技术对核酸识别的特异性与gRNA 设计的便捷性,使得该系统在核酸检测方面展现出巨大的发展潜力[62]。
2.1 基于Cas9 系统检测食源性病原体
基于CRISPR/Cas9 系统的检测大多是利用其对靶标的识别与切割作用。Sun 等[63]开发一种由Cas9 触发的两步等温扩增方法,用于大肠杆菌O157:H7 的高灵敏检测,如图2a 所示。利用Cas9(H840A) 对特异链的切割来取代一些切刻内切酶。将两个Cas9(H840A):sgRNA 复合物识别并切割靶DNA 的一条链缺口,触发新链在缺口处延伸并置换原始DNA 链,进行SDA 反应。SDA 的产物进一步RCA 反应以产生长ssDNA 与荧光探针杂交。使荧光探针离开携带有猝灭基团的UiO66,导致荧光产生。通过荧光强度来定量检测目标DNA。该方法能够检测浓度为40 CFU/mL 的大肠杆菌O157:H7。Wang 等[64]开发一种简单巧妙的Cas9n 的扩增反应(Cas9nAR)。利用Cas9n 的特异性识别能力获得ssDNA,然后进行多次引发、延伸、切刻和置换反应,并用SYBR Green I 染料实时监测Cas9nAR 的进程,用强大的荧光信号反映dsDNA 扩增产物的量。该方法将鼠伤寒沙门氏菌作为靶标,通过高效的指数扩增机制,在60 min显示2 copy/反应的检测限。除了以荧光作为检测信号外,其它可视化手段在CRISPR/Cas9 的基础上也显示出良好的应用效果。Wang 等[65]以Cas9n触发的等温DNA 扩增技术为基础,以侧向流动试纸为检测平台,利用特异性sgRNAs 和引物,建立一种同时对鼠伤寒沙门氏菌和大肠杆菌的检测方法,如图2b 所示。分别用地高辛/生物素和FITC/生物素标记对基因组DNA 扩增子进行双标记,使其直接显示在一个简单的横向流动条上。该方法利用试纸平台的扩展性,实现多重目标的检测,具有较高的特异性和敏感性。对细菌的检测限为100 CFU/mL,整个分析从样品制备到读出结果可在3 h 内完成。Moon 等[66]针对新型冠状病毒报道了一种基于CRISPR/dCas9 系统的比色法,如图2c 所示。该方法设计了能够与新型冠状病毒靶RNA 杂交的PAM-mer 序列,并在其3' 端设置生物素,以允许其与链霉亲和素-辣根过氧化物酶结合,在微孔板上通过氧化四甲基联苯(TMB)胺来诱导颜色变化,成功实现了病毒的可视化检测。
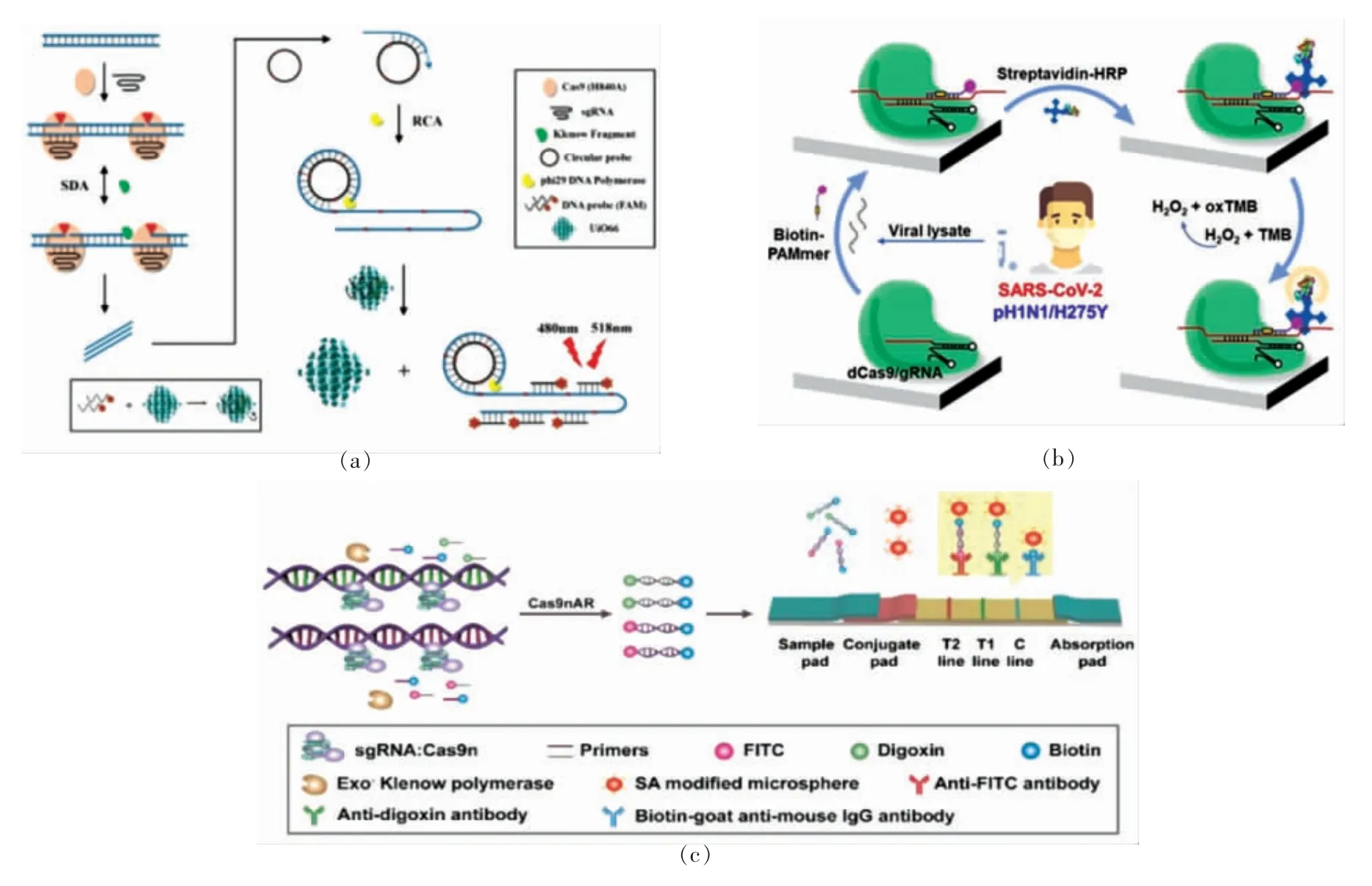
图2 基于CRISPR-Cas9 系统的生物传感分析[63, 65-66]Fig.2 Biosensing assays based on CRISPR-Cas9 system[63,65-66]
尽管CRISPR/Cas9 系统已被应用于基因编辑与检测,还需要2 个连续碱基为G 的PAM 序列对靶标进行识别。这类位点约占基因组的9.9%[67],在很大程度上限制了CRISPR/Cas9 的应用范围。此外,CRISPR/Cas9 系统在使用时可能产生严重的脱靶效应。哈佛大学实验室发现Cas9 的脱靶效率最高可达84%[68],这也是限制其应用的一大难题[69]。
2.2 基于Cas12 系统检测食源性病原体
基于CRISPR/Cas12 的生物传感器,利用Cas-crRNA 对靶序列的识别以及对非特异性ss-DNA(用作报告子)的切割作用,使读出信号发生变化。目前,CRISPR/Cas12 系统与荧光技术相结合被用于核酸检测,显示出高灵敏度、强特异性、快速的优势,具有良好的应用前景。
Li 等[70]利用Cas12a 的反式切割活性,与扩增技术结合,建立了CRISPR/Cas12a 的荧光检测平台,称为“Cas12aFDet”。该检测平台可降低被气溶胶污染的风险,将Cas 体系预先加在试管盖中,在扩增反应后,密封的反应管通过离心将PCR/RAA反应与Cas12a 介导的切割反应相结合,利用Cas12a 的裂解活性对报告探针进行切割,根据荧光强度在15 min 内即可完成对单增李斯特氏菌的检测。Zhang 等[71]发现CRISPR/Cas12a 系统还可以识别新的PAM 序列(UUUN),在此基础上,通过设计gRNA 即可实现对副溶血性弧菌的检测。该方法同样为避免气溶胶污染将Cas 体系与扩增体系预先加在同一试管中,在自制的紫外装置中可达到1.02×102拷贝/μL 的检测限。如图3a 所示,Wang 等[72]同样将PCR 与Cas12 体系集成在特殊的单个毛细管中,建立“CRISPCR”方法来检测大肠杆菌。这种单个毛细管不仅可以解决CRISPR/Cas 核酸酶的不耐热问题,还使反应在不到10 min内获得稳定的荧光信号。除与PCR 方法结合外,近年来快速发展的等温扩增方式也被应用于与CRISPR 系统相结合。Liu 等[73]将RPA 等温扩增技术与CRISPR/Cas12a 相结合,建立“RPA-Cas12a-FS”检测平台用于检测大肠杆菌、单核细胞增生李斯特菌、金黄色葡萄球菌和副溶血弧菌。该系统可在37 ℃下45 min 内特异性检测低至10 拷贝的目标基因水平。Mukama 等[74]也将CRISPR/Cas12 和LAMP 技术结合,开发一种超灵敏的特异性侧向流动生物传感器(LFB),用于铜绿假单胞菌的检测,如图3b 所示。该方法在纯样品和复杂样品中均显示单拷贝的灵敏性和高特异性。LFB 检测试纸使用简单,检测快速,便携廉价。Li 等[75]在建立“Cas12aFDet”后,同年,进一步利用Cas12a 的侧裂解诱导信号变化的特性,将其引入电化学传感平台,如图3c 所示。通过RAA 扩增,用修饰在电极上的MB-ssDNA 代替荧光信号标记的信号探针,对目标引入前、后的MB 信号进行监测,检测限低至0.68 amol/L。
针对病毒的检测,He 等[76]通过编程crRNA,使CRISPR/Cas12a 能够在识别靶标DNA 链后,切割荧光探针,达到对非洲猪瘟病毒的检测。在没有核酸扩增的情况下,2 h 内可获得1 pmol/L 的检测极限,延长反应时间还可使检测限达fmol/L 水平。彭小唤[77]应用改良的Cas12a 检测非洲猪瘟病毒,可在1 h 内将检测限提高至amol/L 水平。针对2019年爆发的新型冠状病毒,Guo 等[78]构建了基于RT-RAA 的CRISPR 系统SARS-CoV-2 的检测平台,检出限为1×104拷贝,显示出良好的特异性。如图3d 所示,Chen 等[79]报道了一种基于LAMP 和CRISPR/Cas12a 技术的无污染目视检测SARS-CoV-2 的方法,通过智能手机和便携式3D打印仪器,产生的荧光不需要任何专用仪器就可用肉眼看到,整个扩增和检测过程在40 min 内完成,达到20 拷贝的高灵敏检测。Ali 等[80]还将RTLAMP、CRISPR-Cas12 组成的“iSCAN”平台与侧流层析技术结合,显示出与荧光检测相同的结果,实现了对新型冠状病毒快速灵敏检测。
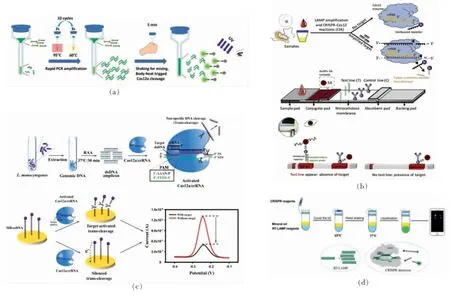
图3 基于CRISPR-Cas12 系统的生物传感分析[72,74-75,79]Fig.3 Biosensing assays based on CRISPR-Cas12 system[72,74-75,79]
基于CRISPR/Cas12a 的生物传感器与其它检测方法相比,虽具有灵敏度更高,特异性更强的优点,但仍有一定的局限性,如不同的扩增技术决定检测时间的长短;DNA 扩增产物转移过程中有气溶胶污染的风险。尽管有些研究将扩增过程与CRISPR 体系集成于单试管内进行,研究人员仍需考虑扩增体系和Cas12a 切割体系的相容性对试验结果的影响。
2.3 基于Cas13 系统检测食源性病原体
相比于Cas12 对靶标DNA 的非特异性切割活性,Cas13 同样具有出色的表现。由于DNA 在微生物细胞中长期存在,且无论其生理条件如何,靶向DNA 的方法很难指示细菌活性,因此以DNA 为检测目标的核酸分析方法无法区分死菌和活菌,会使假阳性率较高。相比之下,RNA 很容易在死细菌中快速消化,可被用来快速指示细菌的生存状态[81]。在食品中致病菌对人体的危害关键在于其保持一定的生长活性,从而产生的致病性。对食品中活的致病菌的检测至关重要。以RNA 为目的靶标的Cas13 在食源性致病菌上的检测方面具有不可估量的发展前景。
Zhou 等[82]建立了“CCB-D”的细菌传感策略,用于金黄色葡萄球菌的检测。如图4a 所示,将nuc基因作为分子标记,由PCR 扩增的产物转录成单链RNA (ssRNA),并借助crRNA 的可编程性和Cas13a 附带的RNase 活性,使FAM 荧光基团与BHQ1 荧光基团分离,产生显著增强的荧光。用该方法成功检测到低至1 CFU/mL 的金黄色葡萄球菌。通过可靠性、敏感性、特异性和简单性的验证,证实Cas13 在检测平台具有高灵敏度、高特异性的优势。而葛以跃等[83]将CRISPR-Cas13a 系统与RAA 技术相结合,建立一种副溶血性弧菌快速检测方法(RAA-Cas13a)。将靶核酸进行RAA 扩增和T7-RNA 聚合酶转录后,在crRNA 的引导下激活Cas13a 酶的附带切割活性,切割荧光探针从而释放荧光信号。该方法避免了传统RAA 荧光检测使用的合成相对复杂的RAA 荧光探针,降低了检测难度与成本。整个检测反应需1 h 左右,与实时PCR 具有相同的检测灵敏度,为10 拷贝/μL。苏璇等[84]同样用该方法建立了CRISPR/Cas13a 辅助RAA 方法检测金黄色葡萄球菌,达到10 CFU/mL的灵敏检测。Shen 等[85]基于核酸的变构探针和CRISPR/Cas13a 组合,提出一种“APC-Cas”的检测系统。用变构探针的适体区域结合靶标,通过扩增和转录过程获得能与Cas13 蛋白结合的ssDNA,进而产生强大的荧光信号,可选择性、敏感地定量检测牛奶等各类样品中的肠炎沙门氏菌。不同于大多需要转录步骤的CRISPR 检测手段,Zhang等[81]建立了一种可直接靶向检测致病菌RNA、无需逆转录、PCR 扩增和核酸标记的检测平台。如图4b 所示,利用发光RNA 适体(Broccoli)-CRISPR/Cas13a 系统来分析活蜡样芽胞杆菌。这种适体不同于其它基于CRISPR/Cas 的分析中使用的化学合成和修饰的RNA 探针,它可与DFHBI-1T 染料结合,产生强荧光信号。Broccoli-DFHBI-1T 复合物在CRISPR/Cas13a 识别到细菌RNA 后被Cas13激发的切割活性剪切,其结构被破坏,无法维持原有的结合状态,使DFHBI-1T 染料暴露,导致荧光淬灭,以此监测病原菌RNA 的存在。该方法成功地定量分析活菌,检测低至10 CFU 的蜡样芽孢杆菌,并精确定量105个CFU 总细菌中的活菌,从而更准确地估计蜡样芽胞杆菌破坏食物的能力。曾红棱等[86]同样利用该方法成功实现食品中单增李斯特菌的检测。
对于病毒的检测,Fozouni 等[87]报告了一种无扩增的CRISPR-Cas13a 分析方法,如图4c 所示,通过组合多个crRNA 来增加Cas13a 活性,该方法可从鼻拭子中直接检测SARS-CoV-2 的RNA,通过手机显微镜读取。在30 min 内可达到100 拷贝/μL 的灵敏度,可在5 min 内准确检测出1 组阳性临床样本中预先提取的RNA。

图4 基于CRISPR-Cas13 系统的生物传感分析[81-82,87]Fig.4 Biosensing assays based on CRISPR-Cas13 system[81-82,87]
目前,利用Cas13 的侧裂解活性在食源性病原体检测方面已有较大的进展,这一切割活性比基于Cas9 的检测更具应用价值。就Cas12 与Cas13 而言,有研究人员对基于Cas13 与Cas12 检测病原体的方法进行比较,发现Cas13 活性比Cas12 强,可产生更多的信号。在灵敏度方面,Cas13a 远优于Cas12a,使用Cas13a,反式切割信号可被Cs6 效应核酸酶进一步放大,从而达到极高的灵敏度[80]。在应用Cas13 的检测平台时,需使用RNA 荧光报告基团,其易受环境中RNA 酶降解,可能出现假阳性结果。同样也涉及气溶胶污染而导致的问题,需在转移操作步骤中加入石蜡油或矿物油等来严格控制污染的发生。此外,检测致病菌时,大多需进行转录过程,使预处理时间变长,可能在一定程度上无法满足快速检测的需求。
3 结语
自CRISPR 问世以来,作为基因编辑工具的Cas 系统被广泛用于各个领域,特别是在基因编辑、基因转录调控、基因分型等方面显现出独一无二的优势。基于CRISPR/Cas 系统的生物传感器,在检测肿瘤、癌症标记物、病毒等方向有了更加深入的研究与应用。在病原体的检测中,与传统检测方法相比,该方法操作简单,特异性强,灵敏度高,检测快速,使用试剂少,在温和条件下进行,可满足目前对即时检测的需求。在过去的几年里,基于CRISPR 的生物传感技术在核酸检测方面显示出很大的潜力,然而,仍处于起步阶段,存在一定的局限性,有待改善与提高,如在实际操作中容易发生气溶胶污染,以及Cas 蛋白对靶序列的识别高度依赖PAM 区等。未来,研究人员可以进一步挖掘其它的新型Cas 蛋白,配合适当的扩增方法以更好解决目前存在的不足,应用新技术、新平台,建立更为简单、高效的病原体检测方法。总之,将CRISPR/Cas 系统与生物传感器技术结合作为检测手段,为食源性病原体的检测提供了新思路、新方法。该技术还可用于控制食品从生产、加工、运输、贮存、口岸通关到销售和消费各个环节的生物性风险检测,提高检测方法的准确性和灵敏度,更大程度地保证食品质量安全。

