基于高光谱技术的土壤蛋白酶活性估测
王亚新,杨 莎,乔星星,王 超,冯美臣,肖璐洁,宋晓彦,张美俊,杨武德
(山西农业大学 农学院,山西 太谷 030801)
土壤酶主要由微生物、植物根系和土壤动物分泌。土壤酶是土壤中所有生物生化过程可以持续进行的动力,其活性反映了土壤生物化学过程的方向与强度,与土壤中各种有机、无机物质的转化密切相关[1]。土壤中各种含蛋白质物质(如几丁质、叶绿素、尿素等)在蛋白酶的催化下转化为无机态氮,供植物吸收利用,这个过程蛋白酶具有很强的专一性[2],是促进土壤氮循环的重要组分[3-4],可以作为一种氮矿化的指示剂[5]。殷陶刚等[6]研究指出,土壤水分降低会显著降低蛋白酶活性,而我国水资源分布不均,水分已成为制约我国农业发展的主要因素[7],因此,研究不同水分条件下土壤蛋白酶活性具有重要意义。目前土壤蛋白酶活性的测量方法大多为荷夫曼法、洛美科法及加勒斯江法[8],这些方法耗时较长,过程繁杂,在试验过程中可能造成较大误差,而且有研究认为这些不同方法的有效性在很大程度上仍是未知的[9]。
高光谱技术在估测土壤氮含量方面已有了较多研究[10-12],偏最小二乘(PLS)作为线性建模方法被广泛应用且取得了不错的估测效果[13-14]。随着高光谱技术的不断发展,机器学习、支持向量机等非线性模型也被用于土壤氮含量的估测,同样得到了较好的预测结果[15-17]。土壤光谱在实际测量中,容易受到仪器噪声和测量环境的影响,从而影响建模效果[18],而陶培峰等[19]通过研究证明,光谱预处理可以提高模型预测的精度。
土壤蛋白酶活性与土壤氮密切相关,而高光谱可以较为准确地估测土壤氮含量。此外,孙倩倩[20]通过研究证明,高光谱技术可以对植物超氧化物歧化酶和过氧化物酶活性进行有效预测;杨晨波[21]利用高光谱技术有效估测了土壤脲酶活性。那么高光谱技术是否可以对土壤蛋白酶活性进行准确估测呢?为了探究这个问题,本研究以冬小麦水分胁迫试验采集的土壤为研究对象,测定蛋白酶活性和土壤光谱反射率,对原始光谱数据进行不同的预处理,并对高光谱数据及土壤蛋白酶活性进行相关性分析,构建偏最小二乘(PLS)线性模型和支持向量机(SVM)非线性模型对土壤蛋白酶活性进行估测,旨在探究高光谱技术估测土壤酶活性的可行性。
1 材料和方法
1.1 试验材料
供试小麦品种为长6878和长4738。
1.2 试验设计
试验于2019年10月至2020年7月在山西农业大学农学院实验站进行,共设21个小区,各小区面积均为3 m×3 m,行距为20 cm,施用尿素227.85 kg/hm2、钾肥(硫酸钾)655.5 kg/hm2、磷肥(过磷酸钙)115.5 kg/hm2,作为底肥使用,不追肥。试验采取完全随机设计,根据田间持水量的百分比控制水分,设置2个水分胁迫时期:拔节期至抽穗期、开花期至灌浆期,3个水分胁迫程度:轻度胁迫(田间持水量的65%±5%)、中度胁迫(田间持水量的55%±5%)、重度胁迫(田间持水量的45%±5%),以不进行水分胁迫(田间持水量的75%~80%)为对照。在开花期至灌浆期不进行水分胁迫,在拔节期至抽穗期分别进行重度胁迫(T1)、中度胁迫(T2)、轻度胁迫(T3);在拔节期至抽穗期不进行水分胁迫,在开花期至灌浆期分别进行重度胁迫(T4)、中度胁迫(T5)、轻度胁迫(T6)。试验设计如表1所示。

表1 试验设计Tab.1 Test design
1.3 土壤样品采集
在冬小麦返青、抽穗、灌浆3个关键生育时期采集0~10、10~20、20~40、40~60 cm这4个土层的土壤。将相同处理的土壤充分混合成1份土壤样品,再将土壤样本去除动植物残体以及其他杂质后放在室内阴凉通风处进行风干,风干后过2 mm筛,采用加勒斯江法测量土壤蛋白酶活性。剔除异常值及缺失值之后,共获得土壤样品128个。
1.4 光谱数据采集
采用美国ASD公司生产的Field Spec Pro FR型便携式光谱仪采集光谱数据,该仪器波段范围为350~2 500 nm,其中,350~1 000 nm光谱分辨率为3 nm,光谱采样间隔为1.4 nm;在1 000~2 500 nm,光谱分辨率为10 nm,光谱采样间隔为2 nm。土壤光谱测定在暗室内进行,光纤探头视场角25°,探头垂直向下,距样品表面0.10 m,光源为1 000 W卤化灯,光源垂直高度0.5 m,入射角度45°。将处理好的样品放置到干净的黑色托盘内(高1.5 cm,直径9.5 cm),保持土壤厚度1 cm左右并保持土壤表面平整,每个样本平均选择5个光谱测定点,每光谱采集点获取10条光谱曲线,每土壤样本共计采集50个,平均后得到该样本的最终反射光谱。
1.5 光谱数据处理
由于在光谱边缘有较大的噪声影响,故删除光谱区域 350~399、2 451~2 500 nm,采用 400~2 450 nm波段范围内的光谱数据[22]。对原始光谱反射率数据进行不同的变换处理,采取的光谱预处理方法为 Savitzky-Golay Smoothing(SG)[23]、一阶导数(FD)、二阶导数(SD)[24]、归一化(Nor)[25]、去趋势化(Det)[26]、变量标准化(SNV)[27]和多元散射校正(MSC)[28]这7种变换。
1.6 模型构建方法
1.6.1 偏最小二乘(PLS) 通过将自变量和因变量的高维数据空间投影到相应的低维空间,分别得到自变量和因变量的相互正交的特征向量,再建立自变量和因变量的特征向量间的一元线性回归关系,不仅可以克服共线性问题和去除对回归无益噪声的影响,其在选取特征向量时也突出和强调自变量对因变量的解释和预测作用[29]。
1.6.2 支持向量机(SVM) SVM是一种基于机器学习理论的非线性方法,能够对线性和非线性关系进行建模。其可以消除波长变量共线性,解决因土壤各组分的吸收波段相互重叠干扰土壤有机质含量估算精度的问题,避免模型过度拟合[30]。
1.7 模型评价方法
本研究使用决定系数(R2)、标准偏差(RMSE)和相对预测偏差(RPD)来作为模型的评价指标。R2越大,RMSE越小,表示模型预测效果越好。一般认为,当RPD<1.4时,模型难以对样本进行有效预测;当1.4≤RPD<2.0时,模型可以对样本进行粗略地预测;RPD≥2,表明模型具有较好的预测精度和稳健度[31-33]。
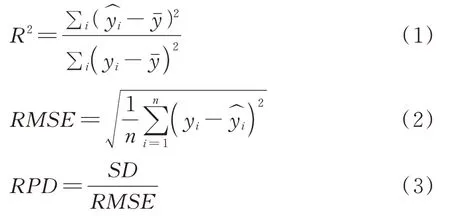
式中,n为样本数为土壤酶活性预测值,yi为土壤酶活性实测值为所有样本的土壤酶活性平均实测值;SD为土壤酶活性实测值的标准差。
1.8 数据处理
试验采用View Spec Pro 6.0对土壤光谱数据进行提取,采用The Unscrambler X 10.4对光谱数据进行预处理,采用Excel 2019以及SPSS 20.0进行数据整理与分析,采用MATLAB 2010进行模型的建立,采用Origin 2021制图。
2 结果与分析
2.1 土壤酶活性的描述性统计分析
将土壤样本按蛋白酶活性的实测值排序并按照3∶1的比例将数据分为建模集(n=96)和验证集(n=32),对数据集进行描述性统计分析(表2)。

表2 蛋白酶活性的描述性统计分析Tab.2 Descriptive statistical analysis of protease activity
本试验中蛋白酶总数据集的范围在0.14~3.29 mg/g,平均值为1.47 mg/g,偏度系数接近0,峰度系数在-1,本试验数据较为符合正态分布。建模集数据、验证集数据及全部数据的平均值、标准差和变异系数都较为一致,说明建模集和验证集的划分较为合理。
2.2 土壤蛋白酶的光谱反射率变化分析
按照四分位法将土壤蛋白酶活性由小到大排列并分成4等份,每份包含25%的蛋白酶样本数据,将每部分内的蛋白酶数据及相对应的光谱数据进行平均,得到不同蛋白酶活性水平下的光谱反射率,如图1所示。
由图1可知,蛋白酶活性在一定范围内土壤光谱反射率随着蛋白酶活性水平的增高而降低,蛋白酶活性超过一定范围后土壤光谱反射率随着蛋白酶活性水平的增加而升高。在不同土壤蛋白酶活性水平下的土壤光谱反射率曲线走势一致,全波段范围内蛋白酶光谱曲线整体呈上升趋势。在可见光(400~800 nm)波段范围内反射率迅速上升;在800~1 900 nm波段范围内反射率平缓上升;在1 400 nm附近有一个较小的吸收谷,在1 900 nm波段附近出现较大的吸收谷,在2 200 nm波段以后波峰和波谷交替出现。

图1 不同蛋白酶活性水平下的土壤光谱反射率变化曲线Fig.1 Variation curve of soil spectral reflectance under different protease activity levels
2.3 光谱数据与土壤蛋白酶的相关性分析
将蛋白酶活性与经过不同预处理的土壤光谱反射率进行相关性分析,结果如图2所示。由图2可知,原始光谱反射率与土壤蛋白酶活性呈正相关,在400~2 450 nm波段范围内,相关系数都在0.4附近且波动较小,经过SG预处理的光谱反射率和蛋白酶的相关性与原始光谱下的蛋白酶相关性趋势接近。Nor预处理的光谱反射率与蛋白酶的相关性在350~1 500 nm波段呈负相关,在1 500~2 500 nm波段呈正相关,其相关系数在-0.40~0.43。FD预处理和SD预处理的光谱反射率与蛋白酶活性在全波段呈正负相关,其相关性达到0.68。在Det、SNV和MSC预处理下的光谱反射率与蛋白酶都呈正负相关,但在500~1 300 nm波段Det预处理下为正相关,SNV和MSC预处理下为负相关,且在SNV和MSC预处理下相关系数达到最大,为0.99。与原始光谱数据相比,经过不同预处理(FD、SD、Det、SNV、MSC)的光谱数据与土壤蛋白酶的相关性得到了显著提高。
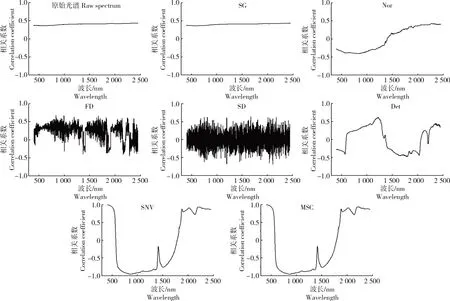
图2 不同预处理下土壤光谱与土壤酶活性的相关性分析Fig.2 Correlation analysis between soil spectrum and soil protease activity under different pretreatments
2.4 基于近红外光谱的土壤蛋白酶估测模型效果分析
光谱数据经7种预处理方法变换后,与土壤蛋白酶活性值之间建立PLS线性模型和SVM非线性模型,所建模型表现如图3所示。从图3可以看出,除基于MSC预处理所建的模型外,基于R、SG、FD、SD、Nor、Det、SNV所建立的PLS线性模型表现都优于SVM非线性模型。除MSC外,PLS线性的模型决定系数Rc2均在0.80以上,预测决定系数Rv2均在0.75以上,RMSEc均在0.31以下,RMSEv均在0.40以下,RPDc和RPDv均在2.0以上;基于MSC所建立的模型其R2也在0.6以上,RPD在1.6以上,可以粗略地估测蛋白酶活性,基于原始光谱和其他预处理所建的模型可以较为准确地估测蛋白酶活性。经过7种处理后所建立的土壤蛋白酶近红外SVM模型除基于原始光谱和SG预处理所建的模型外,基于其他6种预处理所建立的模型Rc2均在 0.70以上,Rv2均在 0.55以上,RMSEc均在0.43以下,RMSEv均在0.53以下,RPDc均在1.90以上,RPDv均在1.50以上,可以对蛋白酶活性进行有效估测。PLS线性模型和SVM非线性模型都基于导数变换预处理得到最佳的估测效果,R2在0.95以上,RMSE在0.20以下,RPD在4.0以上,基于FD预处理下的PLS线性模型估测效果最好,其模型 表 现 为 :Rc2=0.99、RMSEc=0.08、RPDc=10.51;Rv2=0.96、RMSEv=0.17、RPDv=4.68。

图3 基于不同预处理方法的PLS和SVM建模效果Fig.3 Modeling results of PLS and SVM based on different pretreatment methods
3 结论与讨论
在本研究中,一定酶活性水平下土壤光谱反射率随土壤蛋白酶活性的增加而降低,超出一定水平后,土壤光谱反射率随着土壤蛋白酶活性的增加而升高,但其反射率变化并不明显。根据张盼盼等[34]的研究,土壤蛋白酶活性增加的同时土壤有机碳含量也呈增加趋势,因此,在土壤蛋白酶活性超过一定水平后,可能由于土壤有机碳含量的影响使得土壤蛋白酶光谱反射率变化较小。
光谱在测量过程中容易受到测量仪器和测量环境的影响,前人通过研究发现,光谱预处理可以有效去除各种噪声影响[35]。本研究中,土壤蛋白酶光谱数据经不同预处理之后,土壤蛋白酶活性与土壤光谱的相关性得到了显著提高。不同预处理方式对土壤蛋白酶与土壤光谱之间相关性的影响不同,SNV与MSC预处理显著提高了短波近红外与中波近红外波段范围内光谱与土壤蛋白酶的相关性,Det预处理提高了短波近红外波段土壤光谱与土壤蛋白酶的正相关性,主要是由于预处理方法工作原理的不同,对光谱与蛋白酶活性相关性的影响不同。导数变换明显放大不同波段土壤蛋白酶的光谱反射率变化,使得更多的光谱信息显现出来,基于导数预处理所构建的PLS和SVM模型对于蛋白酶的估测效果显著优于其他预处理所建模型。这一结果与崔霞等[36]所提出的“导数变换处理具有去除部分线性或接近线性的背景值干扰的功能,有助于降低低频噪声对光谱的影响和突显细微信息差异的变化,能够较好的提高模型估算效果”的观点相同。对比不同预处理下的模型效果,多数光谱预处理都提高了模型的估测效果,大部分模型都是稳健和可靠的,可以用来快速估测土壤蛋白酶活性,证明光谱预处理能够去除噪声影响,有效提高模型的估测效果。在整体上,PLS模型的估测效果优于SVM模型的估测效果,PLS具有多元线性回归、主成分分析、典型相关分析3种分析方法的优点,在处理小容量样本数据方面具有独特的优势,能更有效地提取和利用光谱中的重要信息。但是CHANG等[37]也提出同一种模型方法在不同的试验中所达到的效果不同,并没有一种适用于所有试验的模型方法。因此,在不同的试验中,对于模型方法的选取可能需要从多方面考虑。
本研究以128个土壤样品为研究对象,测定其土壤蛋白酶活性和土壤光谱信息,对原始光谱反射率数据进行不同的变换处理,再分别与土壤蛋白酶活性进行相关分析,构建偏最小二乘(PLS)线性估测模型和支持向量机(SVM)非线性估测模型。研究结果表明,不同的光谱预处理方法显著提高了土壤光谱数据与土壤蛋白酶的相关性,其相关系数最高达到0.99。对比分析基于不同预处理下所建立的土壤蛋白酶PLS线性估测模型和SVM非线性估测模型的表现,导数变换处理下模型效果最好,大部分模型精度和准确性都较高,可以对土壤蛋白酶进行估测;基于一阶导数预处理所构建的PLS线性模型得到土壤蛋白酶活性的最佳预测效果(Rc2=0.99、RMSEc=0.08、RPDc=10.51;Rv2=0.96、RMSEv=0.17、RPDv=4.68)。

