侵染花叶青木的病毒种类鉴定及其基因序列分析
梁海文,兰平秀,柳勤海,谭冠林,陈小姣,赵雁,李凡
侵染花叶青木的病毒种类鉴定及其基因序列分析
梁海文1,2,兰平秀1,柳勤海1,谭冠林3,陈小姣1,赵雁2,李凡1
1云南农业大学植物保护学院,昆明 650201;2云南农业大学园林园艺学院,昆明 650201;3云南农业大学现代教育技术中心,昆明 650201
【目的】探明采自云南和海南表现为黄化、皱缩等表型的花叶青木病毒种类。【方法】首先,利用转录组测序技术对来自云南省昆明市的花叶青木样品进行检测;然后,根据转录组测序结果,采用RT-PCR对采自云南省昆明市和海南省海口市的73份花叶青木样品开展转录组测序结果中发现的病毒种类检测验证,并扩增获得相关病毒基因片段,对这些病毒的基因序列进行比对分析。【结果】转录组测序结果发现,送测的花叶青木样品中含有蚕豆萎蔫病毒2号(broad bean wilt virus 2,BBWV2)、菜豆黄花叶病毒(bean yellow mosaic virus,BYMV)、黄瓜花叶病毒(cucumber mosaic virus,CMV)、豌豆耳突花叶病毒1号(pea enation mosaic virus 1,PEMV-1)和豌豆耳突花叶病毒2号(pea enation mosaic virus 2,PEMV-2)5种病毒的侵染。但随后对来自云南和海南的73份花叶青木样品的RT-PCR验证中,只检测到BBWV2、BYMV、CMV和PEMV-2 4种病毒。其中,云南和海南的样品中均能检测到BBWV2,总检出率为11.0%;而BYMV、CMV和PEMV-2仅在云南的样品中被检测到,检出率分别为5.5%、1.4%和1.4%。从两地采集的花叶青木样品中既未发现PEMV-1的侵染,也未发现BBWV2、BYMV、CMV和PEMV-2的复合侵染。为了进一步分析BBWV2、BYMV、CMV和PEMV-2花叶青木分离物与来自其他寄主植物相关病毒分离物的分子变异,选取检测到上述4种病毒的样品,分别对BBWV2、BYMV和CMV 3种病毒的及PEMV-2的序列进行扩增,并对扩增获得的BBWV2 490 nt的small(GenBank登录号:OQ137565、OQ137566)、BYMV 499 nt的核心区域(GenBank登录号:OQ137568)、CMV 880 nt的(GenBank登录号:OQ137567)和PEMV-2 800 nt的核心区域序列(GenBank登录号:OQ137569)进行分析。序列比对分析结果表明,BBWV2云南与海南花叶青木分离物间存在85.9%的核苷酸序列一致性,二者与GenBank中其他BBWV2分离物分别存在79.8%—94.5%和80.2%—92.0%的核苷酸序列一致性;BYMV、CMV和PEMV-2云南花叶青木分离物与GenBank中相应病毒的核苷酸序列一致性分别为79.8%—99.0%、71.5%—99.4%和84.9%—98.0%。利用以上病毒相应基因核苷酸序列构建系统发育树,发现BBWV2云南和海南花叶青木分离物分别属于I组中的a亚组和b亚组;BYMV云南花叶青木分离物与伊朗(GenBank登录号:MN241051,MN241060)和伊拉克(GenBank登录号:JQ026005)蚕豆分离物具有较近的亲缘关系;CMV云南花叶青木分离物属于I组中的IB亚组,但与IB亚组的其他成员具有较远的亲缘关系;PEMV-2云南花叶青木分离物与云南豌豆分离物亲缘关系最近并聚为一组,但与其他分离物存在较远的亲缘关系。【结论】首次报道了花叶青木被病毒感染,同时也是BBWV2、BYMV、CMV和PEMV-2在花叶青木乃至桃叶珊瑚属植物上的首次报道,BBWV2、BYMV、CMV和PEMV-2花叶青木分离物与来自其他植物的相关病毒分离物存在较大的分子变异。
花叶青木;病毒鉴定;蚕豆萎蔫病毒2号;菜豆黄花叶病毒;黄瓜花叶病毒;豌豆耳突花叶病毒2号
0 引言
【研究意义】花叶青木(var.)为丝缨花科(Garryaceae)桃叶珊瑚属()常绿灌木,其叶片革质光亮且具有金黄色斑点,是青木()最常见的栽培变种,在国内外园林绿化中广泛应用。2021年,笔者发现昆明市园林绿化中的花叶青木叶片大多具有黄化斑点等表型,有的甚至整个植株叶片都布满黄色斑纹。这些花叶青木叶片的黄色斑纹表型与其原种青木叶片的表型差别很大,同时还观察到花叶青木“色彩斑斓”的叶片表型与植物病毒侵染引起的症状较为相似,而花叶青木主要通过扦插方式进行繁殖,极易导致病毒等病原物通过繁殖材料传播扩散。目前尚不清楚昆明市园林绿化中这些表现为黄化斑点的表型是花叶青木固有特性,还是因病毒感染所致。为此,通过对这些表现为黄化斑点等症状的花叶青木样品开展病毒种类鉴定及其分子变异分析,以期为花叶青木叶片表型与病毒侵染的相关性研究,以及彩叶观赏植物的品种选育等提供理论依据,同时也将为观赏植物病毒病的防控工作提供参考。【前人研究进展】多年生的园林观赏植物很容易遭受各种病原物的侵害,对其观赏价值等造成严重影响,但有些病毒的侵染对于观赏植物几乎没有危害,反而使其叶片和花朵的色彩变得更加丰富,进而增加其观赏价值和经济价值[1]。当今,彩叶植物的观赏价值远高于同源的绿叶观赏植物,在园林绿化中使用频率高,经济价值也随之提升。因此在观赏植物育种过程中,人们有意识地选择具有花叶或杂色花性状的品种并广泛引种栽培。有研究表明观赏植物丰富表型的产生与病毒侵染相关,如段永嘉等[2]在香石竹、唐菖蒲、紫罗兰及虞美人的杂色花品种上分别检测出香石竹斑驳病毒(carnation mottle virus,CarMV)、菜豆黄花叶病毒(bean yellow mosaic virus,BYMV)、芜箐花叶病毒(turnip mosaic virus,TuMV),探明了这4种植物的花瓣杂色均为病毒侵染所致。吴康承[3]从具有花叶症状的彩叶扶桑上分离得到的番茄花叶病毒(tomato mosaic virus,ToMV),将其接种普通朱槿后可产生全株花叶的朱槿。因此,有些植物病毒的侵染与观赏植物的彩色叶片或花瓣有关,有的病毒在提高观赏植物的观赏价值方面是有益的。截至目前,与花叶青木同源的青木上仅见日本学者报道过花椰菜花叶病毒科()杆状DNA病毒属()的桃叶珊瑚环斑病毒(aucuba ringspot virus,AuRV)[4]和伴生豇豆病毒科()线虫传多面体病毒属()的苏铁坏死矮化病毒(cycas necrotic stunt virus,CNSV)[5]两种病毒的侵染,尚未见有关花叶青木病毒方面的报道。深度测序(deep sequencing)也称下一代测序(next-generation sequencing,NGS)和高通量测序(high-throughput sequencing,HTS),是一种可在短时间内以较低成本对几十万到几百万条DNA分子进行序列测定的便捷测序技术,目前已在植物病毒鉴定和检测方面广泛应用,可鉴定已知病毒、发现新病毒及分析病毒间的进化关系等[6]。目前宏基因组测序(metagenome sequencing)、转录组测序(RNA sequencing,RNA-seq)和小RNA测序(small RNA sequencing,sRNA-seq)是深度测序技术中主要的病毒检测鉴定方法,其中转录组测序在植物病毒研究中应用最为广泛。Lan等通过去除rRNA的转录组测序,分别在表现为坏死、褪绿症状的山药样品中发现柘橙病毒属()的一种新病毒——山药褪绿坏死病毒(yam chlorotic necrosis virus,YCNV)[7],在诺丽上发现马铃薯Y病毒属()的一种新病毒——诺丽花叶病毒(noni mosaic virus,NoMV)[8];李梁利用转录组测序技术发现在表现为皱缩症状的藜上存在蚕豆萎蔫病毒2号(broad bean wilt virus 2,BBWV2)的侵染[9]。转录组测序也被用于分析病毒侵染后对观赏植物叶色变化的影响,如王炜等利用转录组测序探明滇山茶花叶呈色可能与病毒侵染后诱导了多个代谢途径的基因沉默有关[10]。【本研究切入点】彩叶植物的叶片呈色机理受多方面的影响,有的可能为品种本身遗传特性决定,有的或是被病毒感染所致。然而,病毒感染对园林植物观赏性方面的影响迄今鲜有报道,也尚未见有关花叶青木被病毒感染的报道。【拟解决的关键问题】利用转录组测序结合RT-PCR技术,对采自云南和海南表现为黄化、皱缩和褪绿斑等症状的花叶青木样品进行病毒种类检测鉴定,并对其中发现的病毒进行分子变异和系统发育分析。
1 材料与方法
1.1 样品采集
2021年8月,从云南省昆明市部分住宅小区景观花园中采集表现为黄化(图1-a)和皱缩、褪绿斑(图1-b)的花叶青木叶片样品58份,挑选具有典型症状的7份样品等量混合后于液氮中磨碎,送上海元莘生物医药科技有限公司进行转录组测序(RNA-seq),其余样品(包括转录组测序剩余的7份)于-20 ℃冰箱保存备用。
2022年2月,从海南省海口市采集表现为不规则黄斑和黄化(图1-c)的花叶青木样品15份,-20 ℃冰箱保存备用。
1.2 材料与试剂
DH5菌株为本实验室保存,CTAB为国产分析纯,pMD19-T Vector和Reverse Trancsriptase M-MLV(RNase H-)购自宝生物工程(大连)有限公司,DNA Polymerase和DNA纯化回收试剂盒(离心柱型)购自天根生化科技(北京)有限公司。
1.3 转录组测序
转录组测序样品的总核酸采用Invitrogen公司的Trizol试剂进行提取后,运用NanoDrop 2000(Wilmington,DE,USA)分光光度计对核酸质量进行检测,将OD260/280在1.6—1.8的核酸利用TruSeq RNA Sample Prep Kit除去rRNA,使用Illumina HiSeq X-ten系统进行测序。所得序列经CLC Genomic Workbench 9.5(QIAGEN)分析后,组装成200 bp以上的contigs,利用NCBI中的Blastn和Blastx进行比对分析并获取与病毒相关的contigs。数据分析由美国农业部农业研究服务署(USDA-ARS)国家种质资源实验室的李如慧博士帮助完成。
1.4 引物设计
根据转录组测序获取的contigs设计检测BYMV、PEMV-1和PEMV-2的特异引物,BBWV2和CMV的检测引物参考文献[11],引物详细信息见表1。

表1 花叶青木病毒检测引物
1.5 总核酸提取、RT-PCR及PCR产物克隆
采用Li等[12]的CTAB法提取样品总核酸。以总核酸为模板,按照Reverse Transcriptase M-MLV(RNase H-)试剂盒说明书进行反转录,获取病毒的cDNA。使用DNA Polymerase试剂盒进行PCR扩增,10 μL反应体系:10×Buffer(Mg2+plus)1 μL、dNTP mix(2.5 mmol·L-1)0.8 μL、上下游引物各0.2 μL、DNA Polymerase 0.2 μL、ddH2O 6.6 μL、cDNA 1 μL,轻弹管壁混匀后进行以下反应:94 ℃预变性2 min,94 ℃变性30 s,57 ℃退火30 s,72 ℃延伸若干30 s,扩增35次循环,72 ℃延伸10 min后,取全部PCR产物用1%琼脂糖凝胶于0.5×TBE缓冲液中电泳,35 min后于凝胶成像系统中观察扩增结果。
紫光灯下切下与引物设计相符的扩增片段,经SanPrep柱式胶回收试剂盒纯化后,连接至pMD19-T Vector,10 h后转化至大肠杆菌DH5,在含Amp的LB固体培养基上培养12 h后,挑取单菌落扩繁10 h并进行PCR验证,挑选阳性克隆送至生工生物工程(上海)股份有限公司进行测序。
1.6 序列分析
利用NCBI网站中的Nucleotide BLAST(https:// blast.ncbi.nlm.nih.gov/Blast.cgi)对测序结果进行比对分析,从GenBank数据库下载BBWV2、BYMV、CMV和PEMV-2的序列,利用Clustal Omega(https://www. ebi.ac.uk/Tools/msa/clustalo/)进行多重比对并截取与本研究获取序列一致的区域,删去相互间序列变异小于1%的分离物,利用MEGA7.0软件中的最大似然法(maximum likelihood,ML)分别构建BBWV2、BYMV、CMV和PEMV-2等病毒相关基因的系统发育树,其中构建系统发育树所使用的最适替代模型分别为TN93(Tamura-Nei)、T92(Tamura 3- parameter)、HKY(Hasegawa-Kishino-Yano)和K2(Kimura 2-parameter)。
2 结果
2.1 花叶青木样品转录组测序
通过对7份花叶青木样品混合物的转录组测序,共获得67 563 492的reads(读长),去除reads中的接头序列及由于接头自连等原因导致没有插入片段的reads,组装后获得长度大于200 bp的187 602个contigs(重叠群)。Blastn分析显示,长度532—5 297 bp的几个contigs分别与BBWV2、BYMV、CMV、PEMV-1和PEMV-2等病毒拥有94%—99%最高的核苷酸序列一致性(表2),暗示花叶青木样品中可能存在以上5种病毒的侵染。
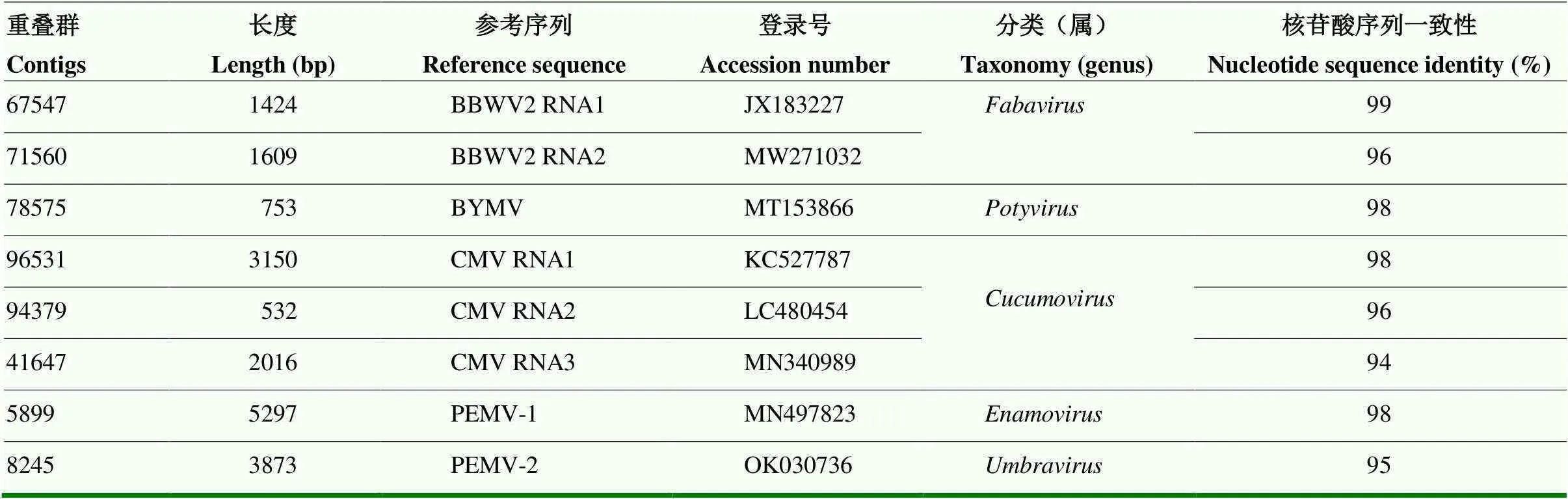
表2 利用转录组测序从花叶青木混合样中获取的病毒相关数据统计
2.2 花叶青木单一样品中转录组测序相关病毒的RT-PCR验证
为验证转录组测序获得的BBWV2、BYMV、CMV、PEMV-1和PEMV-2这5种病毒在花叶青木样品中的真实性,利用上述5种病毒的特异性检测引物(表1),对送转录组测序的7份采自昆明的花叶青木单一样品进行相关病毒的RT-PCR检测,其中4份样品能分别扩增到约490、499、880和800 nt与引物设计相符的病毒基因片段(图2)。将扩增产物进行克隆和测序,Blastn分析显示这些基因片段分别与GenBank中的BBWV2(OP722593)、BYMV(JQ026005)、CMV(MT786689)和PEMV-2(MN399704)拥有95%、99%、99%和96%的核苷酸序列一致性,证明花叶青木中存在BBWV2、BYMV、CMV和PEMV-2这4种病毒的感染,7份花叶青木单一样品均未检测到PEMV-1的感染。
2.3 云南和海南花叶青木样品中BBWV2、BYMV、CMV、PEMV-1和PEMV-2的检测结果
利用BBWV2、BYMV、CMV、PEMV-1和PEMV-2的特异检测引物,对采自云南(58份,含送转录组测的7份)以及海南(15份)花叶青木样品进行上述5种病毒的RT-PCR检测,共有14份样品检测到BBWV2、BYMV、CMV和PEMV-2,病毒总检出率为19.2%(表3)。其中检出BBWV2的样品数为8份,检出率最高,为11.0%;其次为BYMV,样品检出数为4份,检出率为5.5%;CMV和PEMV-2的样品检出率均较低,分别仅有1份样品检测到阳性条带,检出率为1.4%。BBWV2在云南和海南的样品中均被检测到,而BYMV、CMV和PEMV-2仅在云南的样品中被检测到。73份花叶青木样品中未发现PEMV-1的侵染,也未发现BBWV2、BYMV、CMV和PEMV-2等病毒之间的复合侵染。
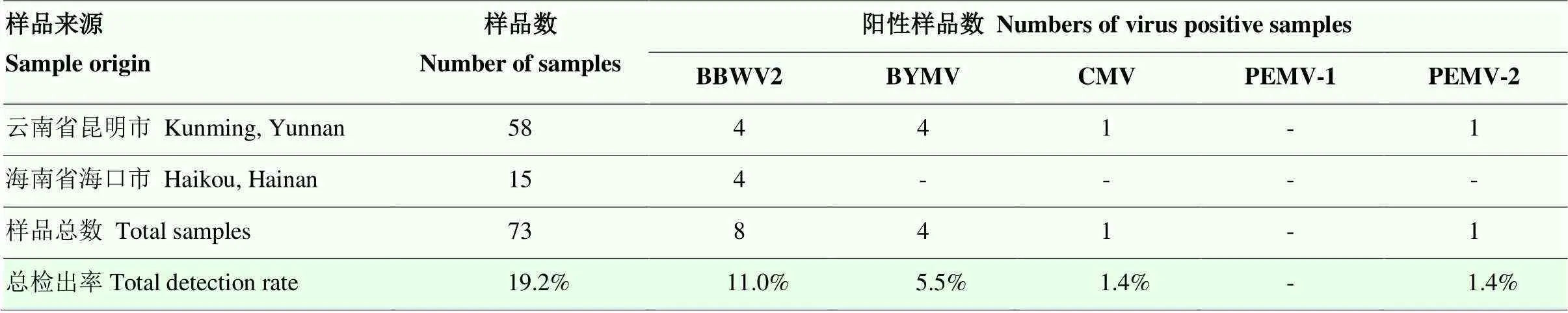
表3 云南和海南73份花叶青木样品BBWV2、BYMV、CMV、PEMV-1和PEMV-2的RT-PCR检测结果
-:未检出病毒no virus was detected
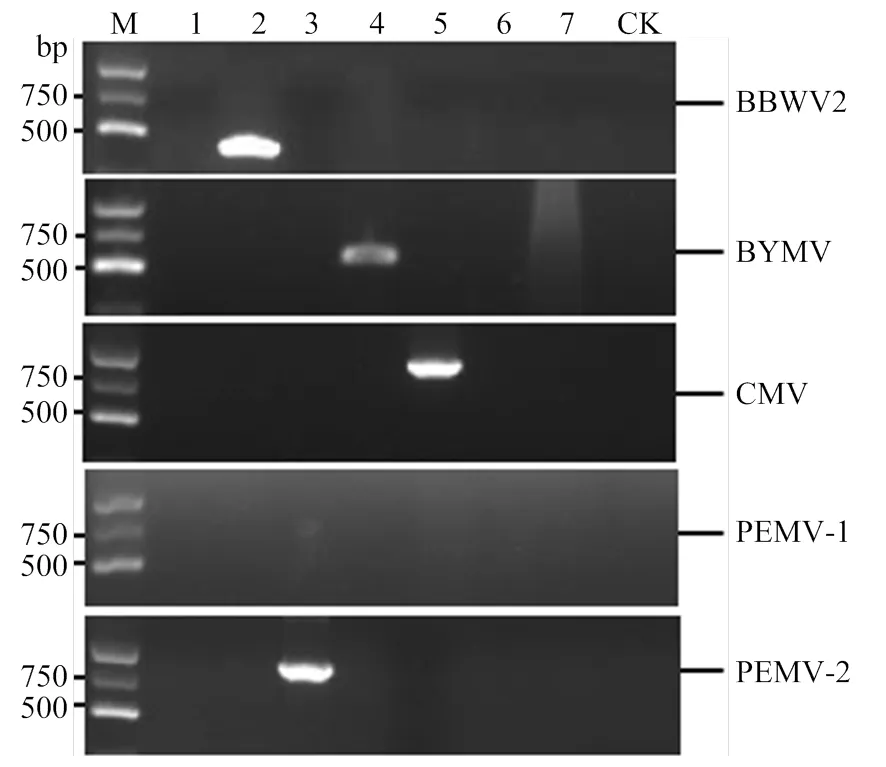
1—7:花叶青木样品A. japonica var. variegata sample;M:Marker;CK:阴性对照Negative control
2.4 BBWV2花叶青木分离物small cp的遗传变异及系统发育分析
结合RT-PCR检测结果,从云南和海南的BBWV2阳性花叶青木样品中各选择一份样品进行BBWV2扩增产物的测序,序列分析显示获取的490 nt片段为BBWV2的small序列。序列比对结果显示BBWV2花叶青木云南分离物(BBWV2-YNHYQM,GenBank登录号:OQ137565)与海南分离物(BBWV2-HNHYQM,GenBank 登录号:OQ137566)间具有85.9%的核苷酸序列一致性,且这两个BBWV2花叶青木分离物与GenBank中的其他BBWV2分离物分别具有79.8%—94.5%和80.2%—92.0%的核苷酸序列一致性。
从GenBank中获取BBWV2不同分离物的small基因序列,去除相互间序列差异小于1%的分离物,对来自中国、日本、韩国、英国、德国等不同国家和地区的番茄、辣椒、白术、山药、菊三七、车前草、本氏烟、蚕豆等不同寄主植物22个BBWV2分离物构建系统发育树,结果显示这些BBWV2分离物可分为I、II两组,I组又可分为a、b两个亚组,其中本文获得的两个BBWV2花叶青木分离物YNHYQM和HNHYQM分别分布于I组中的a亚组和b亚组,分别与来自德国的本氏烟分离物(OK058514)和来自云南的辣椒分离物(MW271032)具有较近的亲缘关系(图3)。
2.5 BYMV花叶青木分离物cp核心区的遗传变异及系统发育分析
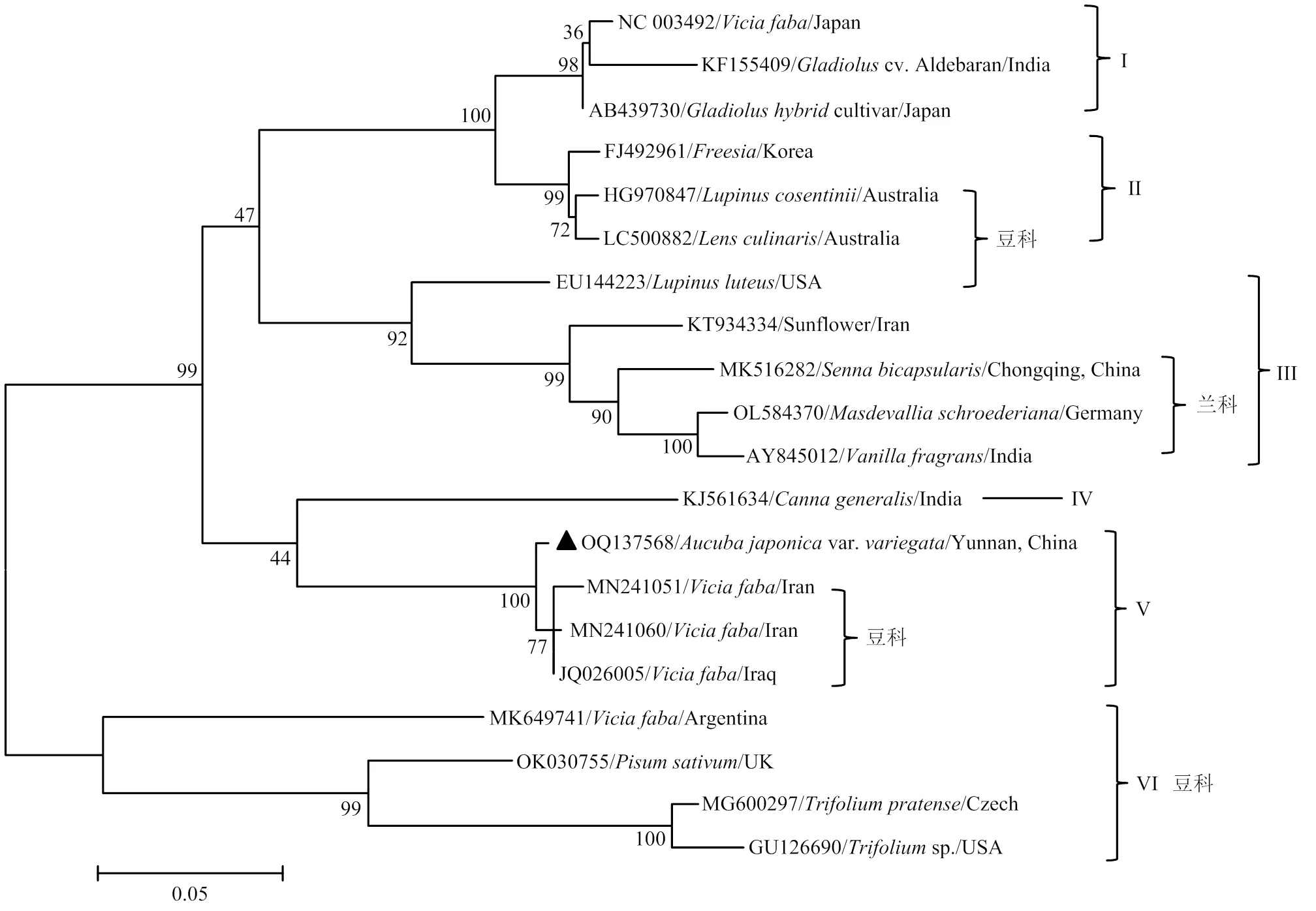
根据RT-PCR检测结果,对唯一一个含有BYMV感染的花叶青木样品进行病毒扩增序列分析,显示获取的499 nt片段为BYMV核心区(BYMV-YNHYQM,GenBank登录号:OQ137568)。从GenBank下载BYMV其他分离物相应区域序列进行多重比对,发现BYMV花叶青木分离物YNHYQM与其他分离物在核心区域具有79.8%—99.0%的核苷酸序列一致性,分子变异较大。去除相互间序列差异小于1%的分离物,利用来自中国、日本、韩国、印度、英国、美国、阿根廷、伊朗、伊拉克等不同国家和地区豆科、兰科等不同作物上的20个BYMV分离物构建系统发育树,显示这些BYMV分离物可分为6个组,其中YNHYQM分离物与来自伊朗(MN241051,MN241060)和伊拉克(JQ026005)的蚕豆分离物具有较近的亲缘关系,聚集在一起归为V组(图4)。各组中,来自豆科、兰科等相近寄主的分离物分别显示了相对较近的亲缘关系。
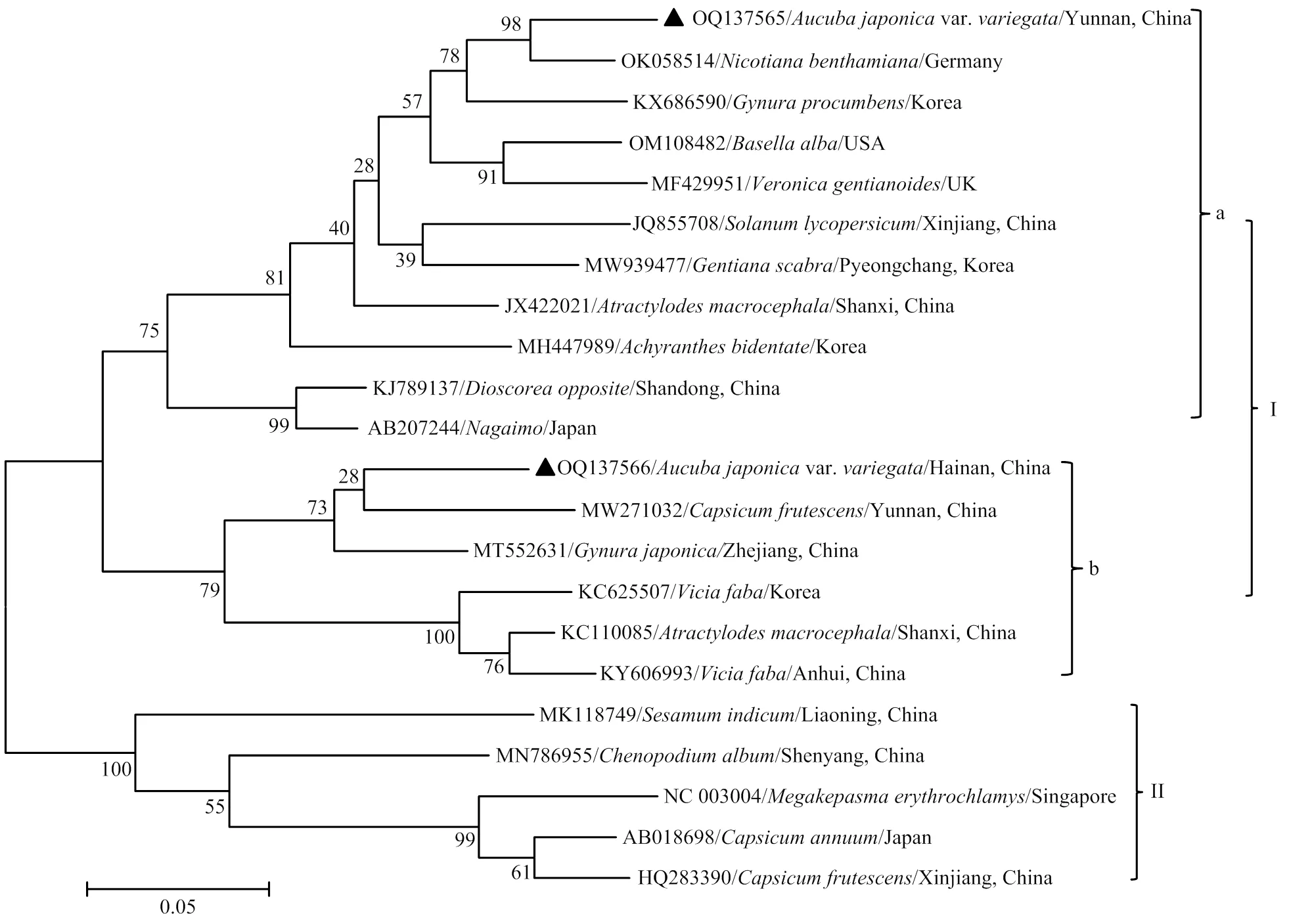
分离物GenBank登录号/寄主植物/地理来源GenBank accession number of different isolates/host plant/geographical origin;p:本研究获得的分离物The isolate obtained in this study;置信值:分支上的数值,用来评估该分支的可信度,置信值一般>70%时为可信The value on the branch is used to evaluate the credibility of the branch. The confidence value is generally greater than 70%;距离标尺:序列间差异数值的单位长度,发育树的比例尺The unit length of the value of the difference between sequences, the scale of the evolutionary tree。下同The same as below
2.6 CMV花叶青木分离物cp的遗传变异及系统发育分析
根据RT-PCR检测结果,对本文获得的唯一一个CMV花叶青木分离物880 nt序列进行分析,所获得的为CMV的(CMV-YNHYQM,GenBank登录号:OQ137567)。从GenBank下载CMV其他分离物相应区域序列进行多重比对,CMV花叶青木分离物YNHYQM显示了与其他分离物有71.5%—99.4%的核苷酸序列一致性。去除相互间序列差异小于1%的分离物,利用分布于IA、IB和II组的26个CMV分离物构建发育树,YNHYQM分离物属于IB亚组,但单独成一簇,与IB亚组的其他成员显示了较远的亲缘关系(图5)。
2.7 PEMV-2花叶青木分离物RdRp核心区的遗传变异及系统发育分析
根据RT-PCR检测结果,对获得的唯一一个PEMV-2花叶青木分离物800 nt的序列进行分析,显示为的核心区域(PEMV-2-YNHYQM,GenBank登录号:OQ137569)。从GenBank下载PEMV-2其他分离物相应区域进行多重序列比对,显示PEMV-2花叶青木分离物YNHYQM与其他PEMV-2分离物拥有84.9%—98.0%的核苷酸序列一致性。去除相互间序列差异小于1%的PEMV-2分离物构建系统发育树,发现这19个PEMV-2分离物可分为3个组,大多数的分离物聚集在I组;而II组又可分为a亚组和b亚组,其中德国豌豆分离物(MW961148)单独形成II组的a亚组,PEMV-2云南花叶青木分离物与云南的4个豌豆分离物聚集在II组的b亚组,亲缘关系较近;美国豌豆分离物(JF723436)与其他分离物亲缘关系较远,单独形成亚组III(图6)。
豆科 Leguminosae;兰科 Orchidaceae
3 讨论
3.1 云南和海南花叶青木的病毒种类
花叶青木是常见的园林植物,国内外报道过花叶青木上有炭疽病、白粉病、根腐病、褐斑病等病害的发生[13-17],但尚未见病毒病的相关报道。本研究利用转录组测序从采自昆明的疑似病毒病症状花叶青木样品中发现可能有BBWV2、BYMV、CMV、PEMV-1和PEMV-2感染。随后采用RT-PCR从供试的73份花叶青木样品中只检测到BBWV2、BYMV、CMV和PEMV-2,表明花叶青木中存在上述4种病毒的侵染。但4种病毒中只有BBWV2同时在云南和海南的样品中被检测到,而BYMV、CMV和PEMV-2仅在云南的样品中被检测到。自然界中,PEMV-1与PEMV-2通常复合侵染引起豌豆耳突花叶病,但植株中也存在仅检测到PEMV-2而检测不到PEMV-1的现象[18-19]。本研究转录组测序结果显示获得了PEMV-1的近全长基因组序列,但利用RT-PCR技术在73份花叶青木样品中均未检测到PEMV-1,原因有待进一步探究。此前日本科学家曾报道在与花叶青木同源的青木上发现有AuRV和CNSV侵染[4-5]。为此,本研究也利用这两种病毒的特异引物对供试的73份花叶青木样品进行RT-PCR检测,但未扩增到相关病毒的目标条带,因此供试的花叶青木样品中暂未发现含有AuRV和CNSV这两种病毒。
3.2 4种病毒花叶青木分离物与其他分离物的系统发育关系
BBWV2在全世界范围内均有发生和分布,在44个科186属328种植物上曾有报道[20-22]。本研究中BBWV2是唯一在云南与海南花叶青木样品上均检测到的病毒,但来自这两个地区的BBWV2阳性花叶青木样品上观察到的症状表现不一致,云南的阳性样品表现为边缘不清晰的淡黄色斑块,而海南的阳性样品嫩叶有边缘明显的黄色斑点,老叶黄化。两个BBWV2花叶青木分离物在small区域具有85.9%的核苷酸序列一致性,系统发育上分属于不同亚组,亲缘关系较远。尚不清楚这些差异是否是造成云南和海南两地花叶青木表型差异的主要原因。另外,本实验室也在花叶青木BBWV2阳性样品中检测到1种新病毒(结果未发表),因此云南和海南两地BBWV2阳性样品叶片症状不一的原因尚无法确定。BYMV是蚕豆和豌豆上发生最普遍和最严重的病毒之一,可侵染12个科的114种植物[23-24],本研究中获得BYMV花叶青木分离物与伊朗(MN241051和MN241060)和伊拉克(JQ026005)蚕豆分离物的亲缘关系较近,但更翔实的数据还有待BYMV花叶青木分离物全基因组序列的获取及进一步分析。CMV被称为全球第四大植物病毒,可侵染100多个科1 000多种植物并造成危害和严重经济损失[25-26]。根据症状特点、寄主范围、致病性及核苷酸序列同源性的差异,CMV可分为I和II两个亚群[27],亚群I进一步分为IA和IB两个亚组,亚群II和IA亚组全球分布,而IB亚组主要在亚洲发生[28]。本研究在花叶青木上发现的CMV分离物属于IB亚组,与IB亚组的印度和乌干达分离物最近,但与国内其他CMV分离物不在同一组,暗示CMV花叶青木分离物可能是从国外传入。另外,PEMV-2花叶青木分离物在系统发育中与本实验室获得的4个PEMV-2云南豌豆分离物(数据未发表)单独聚成一组,与其他国家PEMV-2豌豆分离物的亲缘关系较远,推测本研究获得的PEMV-2花叶青木分离物与云南豌豆中PEMV-2分离物可能有着相同的起源。不过,本研究中花叶青木4种病毒的来源还需进行更多深入的探究。
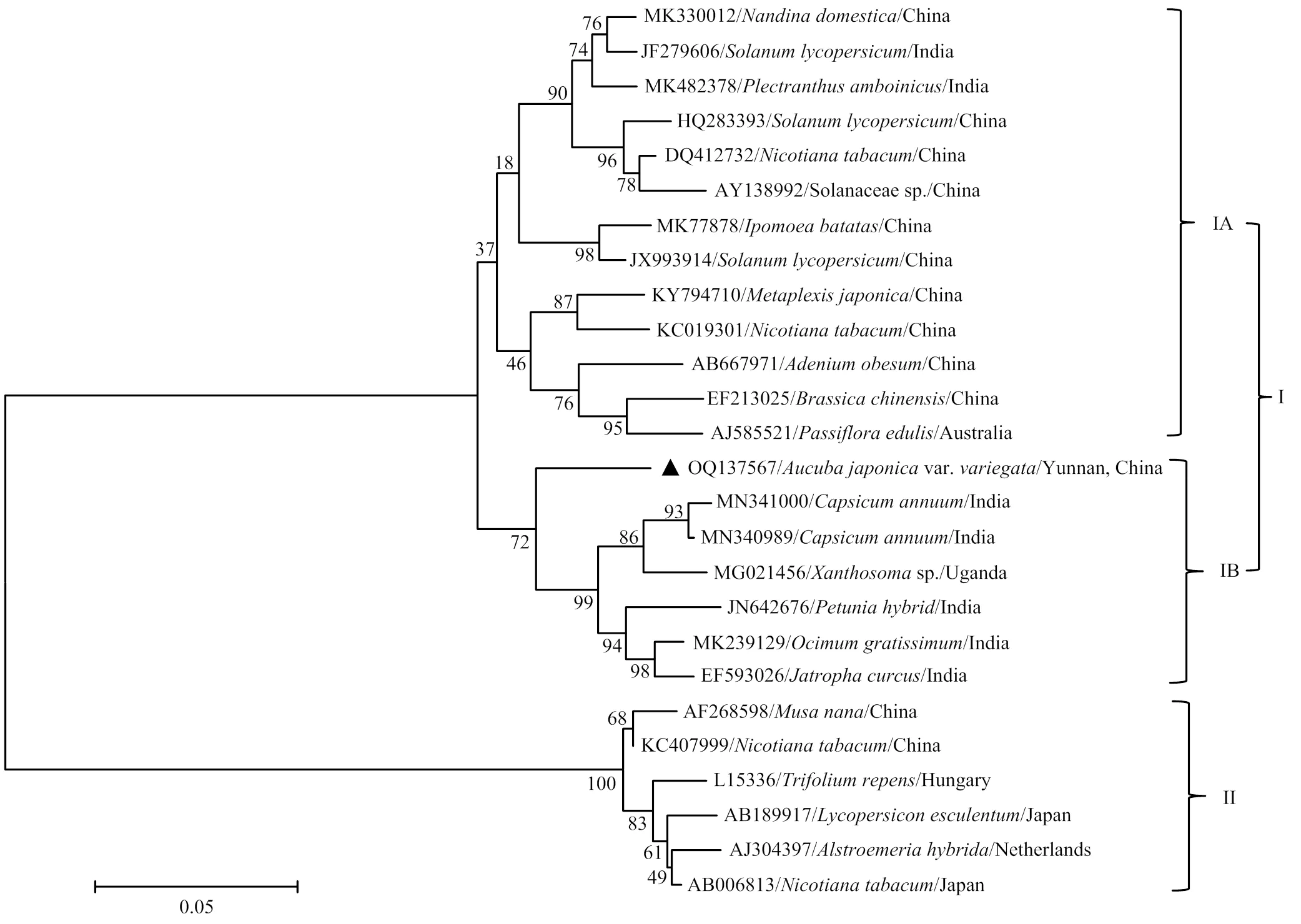
图5 基于CMV不同分离物cp构建的系统发育树
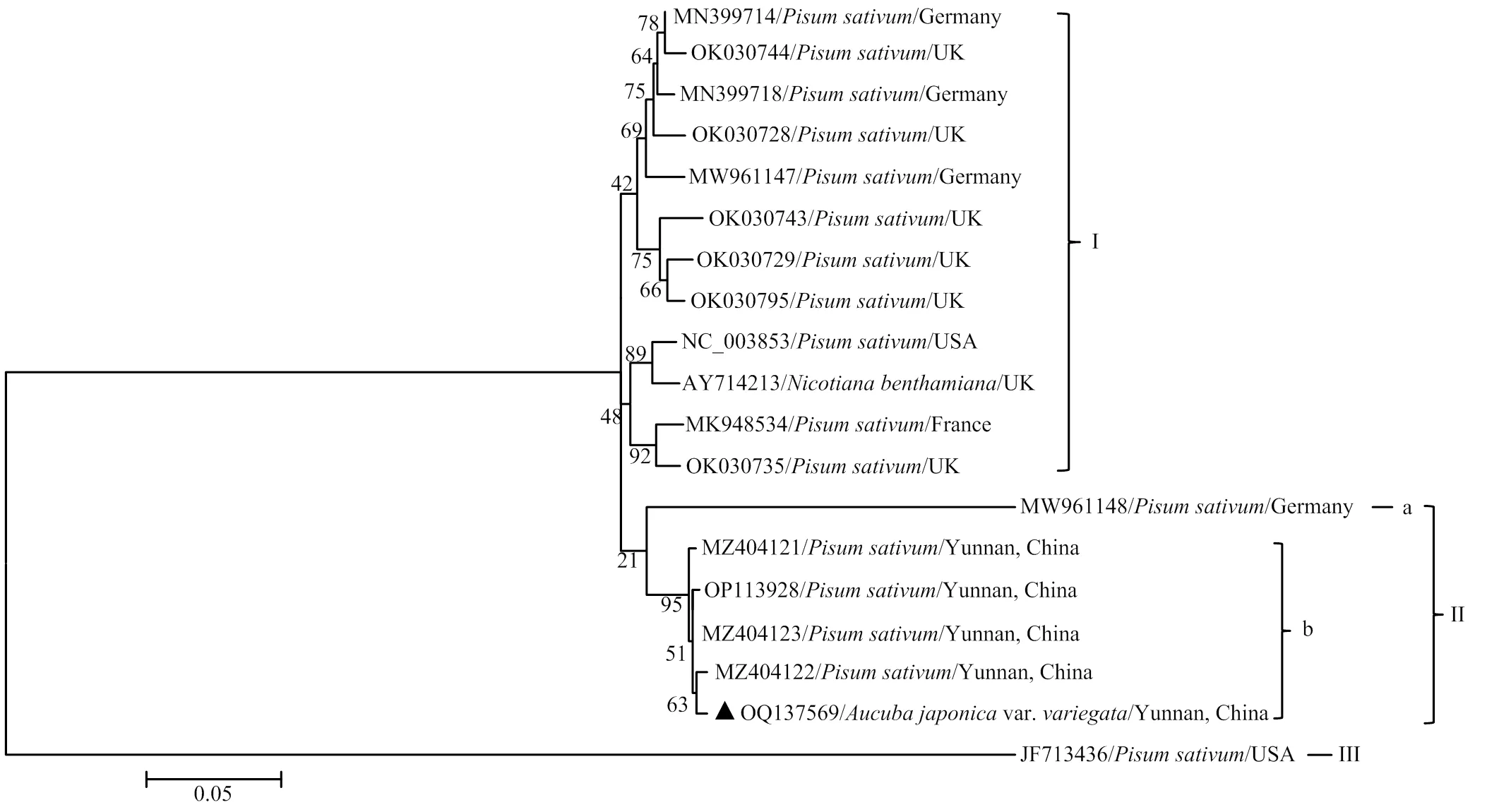
图6 基于PEMV-2不同分离物RdRp核心区构建的系统发育树
3.3 花叶青木叶片表型与病毒种类的相关性
在花叶青木上发现的4种病毒中,海南的样品只检测到BBWV2一种病毒,而云南的样品BBWV2、BYMV、CMV和PEMV-2这4种病毒均被检测到,可看出云南和海南的花叶青木样品上的病毒种类及数量差异较大,云南的病毒种类多样性更丰富。暂不清楚造成上述差异的原因,推测可能是两地栽培的花叶青木种质来源不同或是其周围植物及其携带的病毒种类各异、气候条件差异等,导致两地花叶青木上的病毒种类不一,致使两地的花叶青木叶片斑纹也不尽相同。本研究虽未在花叶青木样品中检测到AuRV和CNSV,但根据日本学者报道,青木受这两种病毒侵染后均产生黄色环斑,这与本研究供试的花叶青木样品表现的不规则黄色斑点表型相似,因此花叶青木叶片黄色斑纹表型的产生很有可能受病毒侵染影响,且病毒种类不一样,在花叶青木上造成的表型可能相同,也可能不相同。除了本文中所报道的BBWV2、BYMV、CMV和PEMV-2这4种病毒外,本实验室还在花叶青木上发现了多种新病毒(结果另文报道)。由于目前较难找到不含病毒的青木植株,且尚未分离出单一病毒分离物在花叶青木上进行回接试验并验证柯赫式法则,因此暂不能断定花叶青木上叶片斑纹的变化是否与病毒侵染有关;若与病毒侵染有关,则病毒种类与花叶青木上叶片斑纹的相关性及其形成机制等都值得深入研究。
4 结论
采用高通量测序并结合RT-PCR技术,从来自云南和海南的73份花叶青木样品中检测到BBWV2、BYMV、CMV和PEMV-2 4种病毒,证明花叶青木中存在上述4种病毒的侵染,这是BBWV2、BYMV、CMV和PEMV-2在花叶青木乃至桃叶珊瑚属植物上的首次报道。研究结果可为病毒侵染与花叶青木叶片表型相关性研究提供理论依据。
[1] 侯元凯. 彩叶植物研究进展. 世界林业研究, 2010, 23(6): 24-28.
HOU Y K. Study advances in color-leaf plants. World Forestry Research, 2010, 23(6): 24-28. (in Chinese)
[2] 段永嘉, 蔡红. 花卉杂色花的研究. 植物病理学报, 1998, 28(1): 85-89.
DUAN Y J, CAI H. Studies on the variegated flowers of flowering plants. Acta Phytopathologica Sinica, 1998, 28(1): 85-89. (in Chinese)
[3] 吴康承. 病毒造就的缤纷植物. 生命世界, 2010(7): 52-55.
WU K C. Colorful plants created by viruses. Life World, 2010(7): 52-55. (in Chinese)
[4] UKE A, PINILI M S, NATSUAKI K T, GEERING A D W. Complete genome sequence of aucuba ringspot virus. Archives of Virology, 2021, 166(4): 1227-1230.
[5] KUSUNOKI M, YAMASHITA S, CHANG M U, DOI Y, YORA K. Isolation of new, cycas necrotic stunt virus from,and. Japan Journal Phytopathology, 1979, 45: 571-572.
[6] MATSUMURA E E, COLETTA-FILHO H D, NOURI S, FALK B W,
NERVA L, OLIVEIRA T S, DORTA S O, MACHADO M A. Deep sequencing analysis of RNAs from citrus plants grown in a citrus sudden death-affected area reveals diverse known and putative novel viruses. Viruses, 2017, 9(4): 92.
[7] LAN P X, MENG Y, SHEN P, LI R H, MA Y, TAN S T, CHEN H R, CAO M J, LI F. Complete genome sequence of yam chlorotic necrosis virus, a novel macluravirus infecting yam. Archives of Virology, 2018, 163(8): 2275-2278.
[8] LAN P X, HE P, ZHANG Y K, ZHANG S, ZHANG Z B, CHEN X J, TAN S T , LUO H M, CAO M J, LI F. Molecular characterization of a novel potyvirus infecting noni. Archives of Virology, 2019, 164(12): 3099-3102.
[9] 李梁. 卫矛与藜上病毒的检测与鉴定[D]. 沈阳: 沈阳大学, 2020.
LI L. Detection and identification of viruses on theMaxin andL. plants[D]. Shenyang: Shenyang University, 2020. (in Chinese)
[10] 王炜, 郑伟, 徐晓丹, 陈菁, 王谈笑. 基于转录组测序的滇山茶花叶呈色机理分析. 西北植物学报, 2017, 37(9): 1720-1727.
WANG W, ZHENG W, XU X D, CHEN J, WANG T X. Coloring mechanism analysis of mosaic leaves inLindl. based on sequencing of transcriptome. Acta Botanica Boreali- Occidentalia Sinica, 2017, 37(9): 1720-1727. (in Chinese)
[11] 刘勇, 李凡, 李月月, 张松柏, 高希武, 谢艳, 燕飞, 张安盛, 戴良英, 程兆榜, 等. 侵染我国主要蔬菜作物的病毒种类、分布与发生趋势. 中国农业科学, 2019, 52(2): 239-261. doi: 10.3864/j.issn. 0578-1752.2019.02.005.
LIU Y, LI F, LI Y Y, ZHANG S B, GAO X W, XIE Y, YAN F, ZHANG A S, DAI L Y, CHENG Z B,. Identification, distribution and occurrence of viruses in the main vegetables of China. Scientia Agricultura Sinica, 2019, 52(2): 239-261. doi: 10.3864/j.issn.0578- 1752.2019.02.005. (in Chinese)
[12] LI R, MOCK R, HUANG Q, ABAD J, HARTUNG J, KINARD G. A reliable and inexpensive method of nucleic acid extraction for the PCR-based detection of diverse plant pathogens. Journal of Virological Methods, 2008, 154: 48-55.
[13] 李翰文, 刘欢欢, 张敏, 蔡琴, 刘丹, 李沛利. 成都市洒金珊瑚炭疽病病原鉴定及潜在侵染源初探. 植物保护, 2021, 47(4): 38-45.
LI H W, LIU H H, ZHANG M, CAI Q, LIU D, LI P L. Identification of the pathogen causing anthracnose and preliminary investigation of the potential infection source ofin Chengdu. Plant Protection, 2021, 47(4): 38-45. (in Chinese)
[14] LI P L, LIU D, GONG G S, Chen S R, Yang X X. First report ofcausing anthracnose onin Sichuan province of China. Plant Disease, 2016, 100(5): 1019.
[15] CHO S E, ZHAO T, LEE S H, LEE S Y, SHIN H D. First report of powdery mildew caused byonin Korea. Plant Disease, 2017, 101(6): 1033.
[16] 冯福娟. 洒金东瀛珊瑚菌核性根腐病初步研究. 浙江林业科技, 2006, 26(3): 47-49, 58.
FENG F J. A preliminary study on the root rot ofvar.D’Om-Brain. Journal of Zhejiang Forestry Science and Technology, 2006, 26(3): 47-49, 58. (in Chinese)
[17] 吕传红, 杨银虎. 洒金珊瑚特性及其培育技术. 现代园艺, 2015(7): 43-44.
LÜ C H, YANG Y H. Characteristics and cultivation techniques ofvar.. Contemporary Horticulture, 2015(7): 43-44. (in Chinese)
[18] GAAFA Y Z A, HERZ K, HARTRICK J, FLETCHER J, BLOUIN A G, MACDIARMID R, ZIEBELL H. Investigating the pea virome in Germany—old friends and new players in the field(s). Frontiers in Microbiology, 2020, 11: 583242.
[19] FOWKES A R, MCGREIG S, PUFAL H, DUFFY S, HOWARD B, ADAMS I P, MACARTHUR R, WEEKES R, FOX A. Integrating high throughput sequencing into survey design reveals turnip yellows virus and soybean dwarf virus in pea () in the United Kingdom. Viruses, 2021, 13(12): 2530.
[20] EDWARDSON J R, CHRISTIC R G. Fabaviruses//CRC handbook of viruses infecting legumes. Boca Raton: CRC Press, 1991: 263-274.
[21] 周雪平, 余永杰, 刘勇, 李德葆. 侵染豌豆和蚕豆的蚕豆萎蔫病毒研究. 浙江农业大学学报, 1995, 21(3): 221-226.
ZHOU X P, YU Y J, LIU Y, LI D B. Studies on two isolates of broad bean wilt virus fromand. Journal of Zhejiang Agricultural University, 1995, 21(3): 221-226. (in Chinese)
[22] 青玲, 吴建祥, 戚益军, 周雪平, 李德葆. 蚕豆萎蔫病毒单克隆抗体制备及检测应用. 微生物学报, 2000, 40(2): 166-173.
QING L, WU J X, QI Y J, ZHOU X P, LI D B. Preparation of monoclonal antibodies to broad bean wilt virus and application in virus detection. Acta Microbiologica Sinica, 2000, 40(2): 166-173. (in Chinese)
[23] SOFY A R, HMED A, ALNAGGAR A, DAWOUD R A, ELSHAARAWY R, SOFY M R. Mitigating effects of bean yellow mosaic virus infection in faba bean using new carboxymethyl chitosan-titania nanobiocomposites.International Journal of Biological Macromolecules, 2020, 163: 1261-1275.
[24] 涂丽琴, 吴淑华, 干射香, 崔晓艳, 赵文浩, 程兆榜, 陈新, 周益军, 季英华, 朱月林. 江苏省蚕豆上菜豆黄花叶病毒的分子鉴定. 江苏农业学报, 2019, 35(4): 804-810.
TU L Q, WU S H, GAN S X, CUI X Y, ZHAO W H, CHENG Z B, CHEN X, ZHOU Y J, JI Y H, ZHU Y L. Molecular identification of bean yellow mosaic virus infectingfrom Jiangsu province. Jiangsu Journal of Agricultural Sciences, 2019, 35(4): 804-810. (in Chinese)
[25] SCHOLTHOF K G, ADKINS S, CZOSNEK H, PALUKAITIS P, JACQUOT E, HOHN T, HOHN B, SAUNDERS K, CANDRESSE T, AHLQUIST P, HEMENWAY C, FOSTER G D. Top 10 plant viruses in molecular plant pathology. Molecular Plant Pathology, 2011, 12(9): 938-954.
[26] 宋嘉伟, 侯盼盼, 费莉玢, 龙奎, 刘颖, 高诗琪, 苏秀, 马良进. 黄瓜花叶病毒浙江白术分离物全基因组序列测定与分析. 植物病理学报, 2020, 50(2): 251-254.
SONG J W, HOU P P, FEI L B, LONG K, LIU Y, GAO S Q, SU X, MA L J. Complete genomic sequence analysis of cucumber mosaic virus isolated fromKoidz. in Zhejiang. Acta Phytopathologica Sinica, 2020, 50(2): 251-254. (in Chinese)
[27] JACQUEMOND M. Cucumber mosaic virus. Advances in Virus Research, 2012, 84: 439-504.
[28] 瞿青云, 沈文涛, 庹德财, 言普, 陈萍, 周鹏. 黄瓜花叶病毒香蕉分离物基因组全长克隆及侵染性克隆构建. 分子植物育种, https://kns.cnki.net/kcms/detail//46.1068.S.20221214.1136.002. html.
QU Q Y, SHEN W T, TUO D C, YAN P, CHEN P, ZHOU P. The genome full-length cloning and invasive cloning construction of cucumber mosaic virus banana isolate. Molecular Plant Breeding, https://kns.cnki.net/kcms/detail//46.1068.S.20221214.1136.002.html. (in Chinese)
Viruses Identification and Their Gene Sequences Analysis Infectingvar.
LIANG HaiWen1,2, LAN PingXiu1, LIU QinHai1, TAN GuanLin3, CHEN XiaoJiao1, ZHAO Yan2, LI Fan1
1College of Plant Protection, Yunnan Agricultural University, Kunming 650201;2College of Horticulture and Landscape, Yunnan Agricultural University, Kunming 650201;3Modern Education Technology Center, Yunnan Agricultural University, Kunming 650201
【Objective】The objective of this study is to identify the viruses infectingvar.plants with symptoms of yellowing and shrinking which collected from Yunnan and Hainan provinces.【Method】Firstly, RNA sequencing was used todetect the viruses infectingvar.plants from Kunming, Yunnan province. Then, based on the RNA sequencing results, RT-PCR was used to detect these viruses in 73var.field samplescollected in Kunming of Yunnan province and Haikou of Hainan province, and the related virus gene fragments were amplified, sequenced and analyzed.【Result】RNA sequencing results showed that 5 viruses were detected from the pooled diseased plants, such as broad bean wilt virus 2 (BBWV2), bean yellow mosaic virus (BYMV), cucumber mosaic virus (CMV), pea enation mosaic virus 1 (PEMV-1), and pea enation mosaic virus 2 (PEMV-2). However, only BBWV2, BYMV, CMV and PEMV-2 were detected in the 73var.samplesfrom Yunnan and Hainan. Among them, BBWV2 was detected in thevar.samples both from Yunnan and Hainan, with total detection rate of 11.0%, while BYMV, CMV and PEMV-2 were only detected in the Yunnan samples with detection rate of 5.5%, 1.4% and 1.4%, respectively. However, neither PEMV-1 nor the co-infections among BBWV2, BYMV, CMV and PEMV-2 was detected in these 73var.samples from Yunnan or Hainan. In order to further analyze the molecular variation of BBWV2, BYMV, CMV and PEMV-2 aucuba isolates with the related virus isolates from other host plants, the 490 nt of smallof BBWV2 (accession number OQ137565, OQ137566), 499 nt of the coreregion of BYMV (accession number OQ137568), 880 nt of theof CMV (accession number OQ137567), and 800 nt of the coreof PEMV-2 (accession number OQ137569) were amplified from thevar.samples infected with the above 4 viruses, and then the amplified gene sequences were analyzed, respectively. Sequence comparison analysis results showed that BBWV2 Yunnan aucuba isolate shared 85.9% nt identity with Hainan aucuba isolate, the BBWV2 Yunnan and Hainan aucuba isolates shared 79.8%-94.5% and 80.2%-92.0% nt identity with other BBWV2 isolates retrieved from GenBank, respectively. BYMV, CMV, and PEMV-2 aucuba isolates shared 79.8%-99.0%, 71.5%-99.4% and 84.9%-98.0% nt identity with other corresponding viruses in GenBank, respectively. Phylogenetic trees based on the gene nucleotide sequences of the above viruses revealed that the BBWV2 Yunnan and Hainan aucuba isolates were grouped into subgroup a and subgroup b in BBWV2 group I, respectively, and the BYMV Yunnan aucuba isolate showed a closest relationship with Iranian (accession number MN241051, MN241060) and Iraqi (accession number JQ026005) broad bean isolates. Moreover, the CMV Yunnan aucuba isolate was clustered into CMV IB subgroup of group I, but showed a distant relationship with other CMV isolates in the same subgroup. While PEMV-2 Yunnan aucuba isolate showed a closest relationship with Yunnan pea isolates and clustered into the same group, and had a distant relationship with other PEMV-2 isolates.【Conclusion】To our knowledge, this is the first record of virus infectingvar., and also the first report of BBWV2, BYMV, CMV and PEMV-2 infectingvar.as well as the plants in genus. Significant molecular variation was detected among the BBWV2, BYMV, CMV and PEMV-2 aucuba isolates with the related virus isolates from other plants worldwide.
var.; virus identification; broad bean wilt virus 2; bean yellow mosaic virus; cucumber mosaic virus; pea enation mosaic virus 2

10.3864/j.issn.0578-1752.2023.10.007
2023-02-01;
2023-03-14
国家重点研发计划(2022YFD1400700)、云南省专家工作站(202005AF150040)
梁海文,E-mail:13627546002@163.com。兰平秀,E-mail:lanpx22@126.com。梁海文和兰平秀为同等贡献作者。通信作者赵雁,E-mail:744234036@qq.com。通信作者李凡,E-mail:fanli@ynau.edu.cn
(责任编辑 岳梅)

