豌豆蚜特异性扩张分支气味受体基因的克隆及表达分析
田莹 陈瑞朋 黄天宇 陈国华 王冰 王桂荣

摘要
豌豆蚜Acyrthosiphon pisum氣味受体家族存在明显扩张现象,推测在豌豆蚜寄主适应过程中起着重要作用。本研究旨在克隆豌豆蚜特异性扩张分支上的气味受体基因,明确这些气味受体基因在不同组织中的表达水平。通过PCR技术克隆特异性扩张分支上气味受体基因的全长序列,采用实时荧光定量RTqPCR技术测定这些基因在触角及足中的表达水平。克隆获得豌豆蚜特异性扩张分支中4个气味受体基因全长序列(GenBank 登录号:OL739156-OL739159),分别编码374~376个氨基酸,序列相似性较高,为68.09%~83.11%,均具有7个跨膜结构域。荧光定量RTqPCR结果显示ApisOR6、ApisOR8、ApisOR13和ApisOR14基因在豌豆蚜成虫触角及足中均有表达,其中ApisOR8、ApisOR13和ApisOR14基因在触角中偏好表达,ApisOR13基因在触角中的表达量最高。本研究为解析豌豆蚜特异性扩张气味受体的功能提供了分子基础。
关键词
豌豆蚜; 气味受体; 选择压; 基因克隆; 表达分析
中图分类号:
S 433.3
文献标识码: A
DOI: 10.16688/j.zwbh.2022068
Molecular cloning and expression analysis of specific expansive odorant receptors clade in Acyrthosiphon pisum (Hemiptera: Aphididae)
TIAN Ying1,2, CHEN Ruipeng2, HUANG Tianyu2, CHEN Guohua1*, WANG Bing2*, WANG Guirong2
(1. College of Plant Protection, Yunnan Agricultural University, Kunming 650201, China; 2. State Key
Laboratory for Biology of Plant Diseases and Insect Pests, Institute of Plant Protection, Chinese
Academy of Agricultural Sciences, Beijing 100193, China)
Abstract
Gene family expansions of odorant receptors (OR) were found in the pea aphid, Acyrthosiphon pisum, and might play a vital role in the host plant adaptation. This study aims to clone odorant receptor genes of a specific expansive OR clade, and to assay their expression levels in different adult tissues. PCR was used to clone the fulllength cDNA sequences of four OR genes. Realtime quantitative PCR (RTqPCR) was used to assess the expression patterns of specific expansive OR genes in antennae and legs. The fulllength cDNA sequences of four OR genes (GenBank accession no.: OL739156-OL739159) of the specific expansive OR clade in A.pisum were cloned, encoding 374-376 amino acids, having the high sequence similarities from 68.09 % to 83.11 % and all containing seven transmembrane domains. The RTqPCR results showed that the four OR genes were expressed in both antennae and legs of A.pisum adult, among which, ApisOR8, ApisOR13, and ApisOR14 bias to express in the antennae, and the ApisOR13 has the highest expression level in the antennae. This study provides a basis for revealing the molecule mechanism of specific expansion ORs in A.pisum.
Key words
Acyrthosiphon pisum; odorant receptor; selection pressure; gene cloning; expression level analysis
昆虫的嗅觉在寄主定位、产卵场所选择、配偶选择和危害逃避等方面起着至关重要的作用[1]。昆虫能够通过灵敏的嗅觉系统识别数千种不同化学信号以感知其周围环境[2]。触角是昆虫主要的嗅觉感受器官,其表面分布着多种类型的感器,气味分子通过感器表面的极孔进入到淋巴液中,与气味结合蛋白(odorant binding protein, OBP)结合后被运输至感觉神经元树突膜上,激活神经元树突膜上表达的气味受体(odorant receptor, OR)后,将化学信号转变为电信号,在昆虫中枢神经系统中加工整合,引起昆虫的一系列行为反应[27]。
气味受体是一种疏水性膜蛋白,一般具有7个跨膜区域,具有300~500个氨基酸。昆虫的气味受体结构与脊椎动物G蛋白偶联受体相反,其N端位于细胞内,C端位于细胞外[4, 8]。对于昆虫OR的研究可追溯至1999年,第一个昆虫OR在黑腹果蝇Drosophila melanogaster中被鉴定出来[9],随着测序技术和生物信息学的快速发展,许多种昆虫的基因组被公布,例如冈比亚按蚊Anopheles gambiae[10]、意大利蜜蜂Apis mellifera[11]、埃及伊蚊Aedes aegypti[12]、赤拟谷盗Tribolium castaneum Herbst[13]、豌豆蚜Acyrthosiphon pisum[14]等,并且越来越多的昆虫的OR基因得以分析鉴定。基因组鉴定的OR多样性与昆虫生态学作用密切相关,包括昆虫遗传多态性和种间隔离,昆虫与植物之间复杂的化学通讯,以及昆虫的生态位分化及适应性进化等[1523]。因此,OR被作为昆虫进化生物学研究的重要模型,对昆虫OR的进化研究有助于揭示化学生态学机制。
豌豆蚜Acyrthosiphon pisum是第一个全基因组测序的半变态发育的模式生物[24]。目前已有染色体级别的豌豆蚜基因组可供研究者使用[25]。随着豌豆蚜基因组测序质量的不断提高,对于豌豆蚜气味受体的注释分析也在更迭变化。Smadja等率先在第一版豌豆蚜基因组中鉴定了79个ORs基因,与其他蚜虫ORs基因相比,基因数量显著增多[14]。随后,Robertson等[26]根据较高质量的豌豆蚜和大豆蚜Aphis glycines基因组和转录组对ORs基因进一步注释,完善并更正了Smadja等[14]的注释结果,所注释的OR数量增加至87个,以上系统发育分析结果表明了豌豆蚜气味受体家族正在经历快速扩张的现象。
基因家族的扩张和收缩与导致遗传变化的进化压力密切相关。正选择基因在适应性进化中十分重要[27],正选择作为基因进化的主要驱动力推动着物种有利突变的遗传,也有助于揭示物种复杂的适应性表型的内在遗传机制[2835]。气味受体在嗅觉感受过程中扮演着重要角色,处于正选择状态下的气味受体基因很可能与昆虫种群分化或者生态适应性改变有关,基因新拷贝在适应性进化驱动下获得新的功能[3637]。处于正选择状态下的豌豆蚜气味受体扩张分支的存在原因及意义同样值得探究。Smadja等使用PAML软件对豌豆蚜所有扩张的气味受体分支进化压和正选择位点进行预测,结果发现豌豆蚜气味受体的两个扩张分支Clade A和Clade B处于正选择状态,除去假基因以外,扩张分支Clade A中有17个OR基因,Clade B中有8个OR基因,它们明显经历了相对较新的基因复制[14]。这些基因的功能尚未明确,有待于进一步的研究。
本研究以Smadja等[14]分析的豌豆蚜气味受体扩张现象作为研究背景,对OR扩张分支Clade B中的气味受体基因的表达模式进行初步探究。我们将处于正选择状态的Clade B分支定名为OR6分支,采用PCR技术克隆了ApisOR6、ApisOR8、ApisOR13和ApisOR14这4个全长基因,通过荧光定量RTqPCR技术对克隆的4个基因在触角及足中的表达谱进行分析,以期为进一步研究豌豆蚜气味受体产生特异性扩张分支的原因及机制奠定必要的基础。
1 材料与方法
1.1 供试虫源及处理方法
本試验虫源为中国农业科学院植物保护研究所昆虫功能基因组饲养的豌豆蚜种群。使用土培蚕豆幼苗在人工气候箱内饲养豌豆蚜,饲养条件:温度 (21±2)℃,相对湿度 (70±5)%,光周期L∥D=16∥8 h。在此条件下豌豆蚜稳定地进行孤雌生殖。
豌豆蚜组织样品的准备:用镊子将豌豆蚜成虫触角和足取下,立即放入液氮中暂存,立即提取组织总RNA或者转入-80℃冰箱中保存备用。以相同方式收集不同批次的豌豆蚜组织样品3组,每组触角和足分别取自300头和200头豌豆蚜成虫。
1.2 总RNA提取与cDNA合成
使用微量总RNA提取试剂盒(天漠生物)按照说明书步骤提取豌豆蚜成虫触角和足2个部位的总RNA。利用NanoDrop 2000检测总RNA的浓度,1.2%琼脂糖凝胶电泳检测质量。使用Thermo反转录试剂盒按照说明书合成用于基因克隆及qPCR的cDNA,放入-20℃冰箱中保存备用。
1.3 引物设计
基于豌豆蚜基因组及生物信息学分析,选择豌豆蚜特异性扩张OR6分支上具有全长cDNA序列的4个气味受体ApisOR6, ApisOR8, ApisOR13和ApisOR14进行基因克隆和表达量分析。利用Primer Premier 5.0和Beacon Designer 8分别设计基因克隆引物和实时荧光定量引物,引物由生工生物工程(上海)股份有限公司合成,引物序列见表1。
1.4 基因克隆
以豌豆蚜的触角cDNA为模板,利用特异性引物(表1)克隆ApisOR6、ApisOR8、ApisOR13和ApisOR14基因全长序列。50 μL反应体系:2 × PrimeSTAR Mix(含DNA Polymerase, Buffer, dNTP Mixture)25 μL,正向和反向引物(10 μmol/L)各2 μL,cDNA模板 1 μL,ddH2O 20 μL。反应条件为:94℃预变性15 s;98℃变性10 s,50℃/55℃/56℃退火10 s,72℃延伸1 min,35个循环;72℃延伸2 min。PCR扩增产物经1.2%琼脂糖凝胶电泳检测,利用胶回收试剂盒(全式金生物技术有限公司,北京)回收目的条带。将回收后的PCR产物连接到pEASYBlunt克隆载体(全式金生物技术有限公司,北京)上,再转入Trans1T1感受态细胞(全式金生物技术有限公司,北京),涂布到含有IPTG和Xgal的LB固体培养基(氨苄青霉素 50 μg/mL)上,37℃培养过夜。经蓝白斑筛选,挑取单个阳性克隆在含有50 μg/mL氨苄青霉素的LB液体培养基中以200 r/min, 37℃,过夜培养,菌液送往北京六合华大基因科技有限公司进行测序。
1.5 序列分析和系统发育树构建
测序结果由DNAMAN软件完成拼接;利用EXPASY蛋白质组学网站(https:∥www.expasy.org)的翻译工具Translate tool (https:∥web.expasy.org/translate)将核酸序列翻译成氨基酸序列;使用TMHMM2.0网站 (http:∥www.cbs.dtμ.dk/services/TMHMM)预测跨膜结构域;使用Clustal X 2.0軟件进行氨基酸序列比对[38]。
使用RaxML 8.0构建豌豆蚜特异性扩张分支气味受体的进化树,氨基酸替换模型为JonesTaylorThornton(JTT),步长检验(Bootstrap)为1 000。建树用的豌豆蚜、大豆蚜以及棉蚜Aphis gossypii的气味受体序列来源于Huang等[39]的数据。
1.6 实时荧光定量RTqPCR
以豌豆蚜成虫触角和足cDNA为模板,以ApisNADH和ApisSDHB基因为内参[40],利用特异性引物(表1)通过RTqPCR技术检测ApisOR6、ApisOR8、ApisOR13和ApisOR14的表达水平。反应体系15 μL:
上、下游引物各0.6 μL,cDNA 0.75 μL,ddH2O 5.25 μL,50×Passive Reference Dye 0.3 μL,2×PerfectTM Green qPCR SuperMix 7.5 μL。反应条件为:94℃预变性2 min;95℃变性5 s,58℃退火34 s,72℃延伸10 s,40个循环。所有样品均使用独立的RNA进行3次重复,每个基因的标准曲线包含至少5个浓度值,以评估PCR的扩增效率,保证单个基因的扩增效率在90%至110%之间。
1.7 数据分析
对OR6分支4个气味受体在不同组织中的相对表达量进行数据分析。基因的相对表达量计算采用2-ΔΔCt法,计算公式为ΔCt=Ct目标基因-Ct内参基因;ΔΔCt=ΔCt样品-ΔCt参照物。利用SPSS软件对不同组织间的表达水平进行独立样本t测验,对4个基因相对表达量差异进行单因素方差分析,利用GraphPad软件作图。
2 结果与分析
2.1 豌豆蚜OR6分支4个气味受体基因克隆、序列分析与系统发育分析
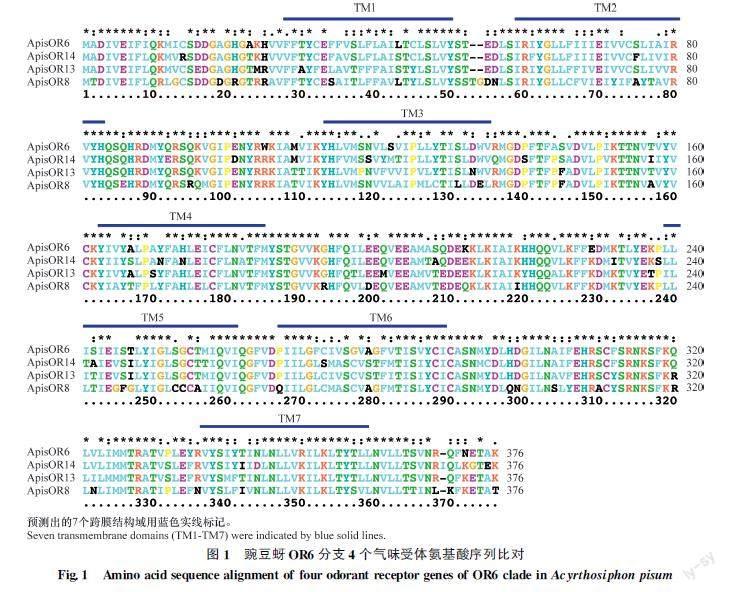
基于Smadja等[14]分析的豌豆蚜气味受体扩张分支Clade B (我们命名为OR6分支)中的气味受体基因序列,我们选择具有全长开放阅读框的ApisOR6、ApisOR8、ApisOR13和ApisOR14进行基因克隆,获得这4个基因的cDNA全长序列。4个气味受体基因的开放阅读框全长分别为1 122、1 128、1 122 bp和1 125 bp,编码374、376、374个和375个氨基酸。通过序列比对发现这4个气味受体基因的序列相似度较高,核苷酸序列一致性为82.76%~92.69%,氨基酸序列相似性为68.09%~83.11%。其中ApisOR6和ApisOR13的序列一致性最高,核苷酸序列一致性为92.69%,氨基酸序列一致性为83.11%。利用TMHMM2.0网站预测蛋白二级结构,发现4个气味受体均具有7个跨膜结构域(图1)。
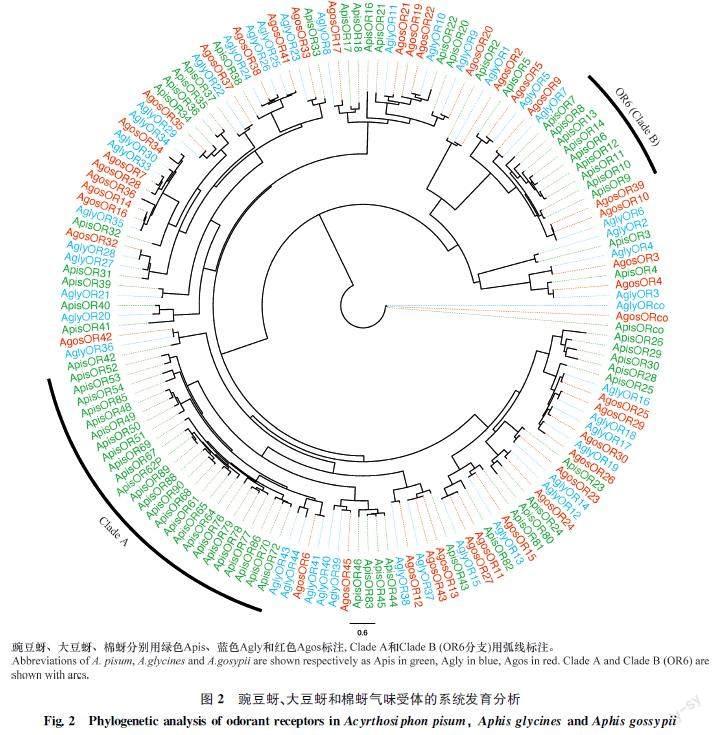
选择豌豆蚜、大豆蚜和棉蚜中已注释的OR构建系统发育树(图2),结果显示豌豆蚜OR中具有2个气味受体扩张分支Clade A和Clade B (OR6分支),这与Smadja等[14]的分析结果相对应。在OR6分支上的气味受体中ApisOR6和ApisOR14距离较近,ApisOR8和ApisOR13与ApisOR6关系较远。
2.2 4个气味受体基因在豌豆蚜成虫不同组织中的表达模式
利用实时荧光定量RTqPCR方法检测豌豆蚜OR6分支ApisOR6,ApisOR8,ApisOR13和ApisOR14基因在豌豆蚜成虫触角和足中的转录水平,结果显示4个ORs基因在豌豆蚜成虫触角及足中均有表达,其中ApisOR8、ApisOR13和ApisOR14这3个基因在触角中的表达量均極显著高于足中的表达量(P < 0.001),而ApisOR6在触角及足中的相对表达量没有显著差异(P>0.05)(图3)。对不同基因在触角中的表达水平进行进一步分析,结果显示ApisOR6、ApisOR8、ApisOR13和ApisOR14基因在触角中的相对表达量有显著差异,其中ApisOR13的表达量最高,ApisOR8的表达量最低(图4)。
3 结论与讨论
本研究克隆得到了豌豆蚜OR6特异性扩张分支上4个气味受体ApisOR6、ApisOR8、ApisOR13和ApisOR14的全长序列。对跨膜区域的预测结果显示,4个气味受体均具有7个跨膜结构域,符合昆虫气味受体典型的二级结构特征。序列分析结果显示,豌豆蚜OR6分支上这4个气味受体的核苷酸及氨基酸序列的一致性较高,表明这些基因可能存在基因复制事件。根据已报道的研究结果,我们推测这些受体在进化过程中可能具有不同的功能,是因为它们氨基酸序列具有一定的分化,进而导致嗅觉受体功能的改变[41]。系统发育进化树的分析结果表明,ApisOR8、ApisOR13、ApisOR6和ApisOR14 4个气味受体聚集在一支,其中ApisOR6和ApisOR14距离更近,表明它们的序列相似性更高,可能功能更接近;我们推测ApisOR14、ApisOR13和ApisOR6可能是由 ApisOR8 分化出来的,ApisOR13和ApisOR8可能与ApisOR6存在功能差异,但这一推论需要通过进一步的功能验证。
实时荧光定量PCR结果显示,ApisOR6、ApisOR8、ApisOR13和ApisOR14这4个气味受体基因在豌豆蚜成虫的触角及足中均有表达,在组织表达模式上存在一定差异。基因的表达水平较高往往表示该基因在维持正常生理状态中发挥着重要作用[39, 4244],ApisOR8、ApisOR13和ApisOR14基因在触角中偏好表达,表明这3个气味受体在豌豆蚜的嗅觉识别过程中起到一定的作用。研究发现,在嗅觉识别过程中发挥重要作用的气味受体其功能与其表达模式有密切联系[45-46],ApisOR13基因在触角中的表达量最高,推测ApisOR13在豌豆蚜正常嗅觉识别中可能发挥着相对重要的作用。OR6分支基因表达模式的分析为豌豆蚜基因组中气味受体扩张分支基因功能的研究奠定了一定的分子基础。
豌豆蚜基因组的发表为揭示该物种在寄主选择、适应性进化和生态物种形成等方面提供了研究基础[14, 2426, 4749]。对豌豆蚜气味受体家族的分析结果显示有两个特异性扩张分支受到了正选择压力的影响,其进化机制及形成原因尚不清楚,推测可能与豌豆蚜对寄主植物等生存环境识别的进化特点有关,即蚜虫对新寄主的适应可能来自于正选择驱动的化学感受基因的适应性进化。另外,处于进化中的基因一般涉及较多的生物学功能,表明在进化过程中这种新功能的分化很可能是随着基因的进化而产生的[17, 49]。气味受体基因家族的进化从嗅觉识别的角度反映了物种化学感受的生态适应性,对豌豆蚜OR6分支气味受体基因的克隆与表达模式分析是解析气味受体家族特异性扩张现象的第一步,也是探究豌豆蚜生物进化和群体遗传学的关键,为研究半翅目物种的化学感受进化机制奠定了分子基础。
参考文献
[1] BRUCE T J, WADHAMS L J, WOODCOCK C M. Insect host location: A volatile situation [J]. Trends in Plant Science, 2005, 10(6): 269274.
[2] CAREY A F, CARLSON J R. Insect olfaction from model systems to disease control [J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(32): 1298712995.
[3] HALLEM E A, HO M G, CARLSON J R. The molecular basis of odor coding in the Drosophila antenna [J]. Cell, 2004, 117(7): 965979.
[4] BENTON R. On the origin of smell: Odorant receptors in insects [J]. Cellular and Molecular Life Sciences, 2006, 63(14): 15791585.
[5] CARRAHER C, DALZIEL J, JORDAN M D, et al. Towards an understanding of the structural basis for insect olfaction by odorant receptors [J]. Insect Biochemistry and Molecular Biology, 2015(66): 3141.
[6] 杜立嘯, 刘杨, 王桂荣. 昆虫外周嗅觉系统信号转导机制研究进展[J]. 中国科学:生命科学, 2016, 46(5): 573583.
[7] WICHER D, MIAZZI F. Functional properties of insect olfactory receptors: Ionotropic receptors and odorant receptors [J]. Cell and Tissue Research, 2021, 383(1): 719.
[8] 申思凡, 张真, 孔祥波, 等. 昆虫气味受体研究技术及其在林业昆虫中的应用研究进展[J]. 林业科学, 2020, 56(5): 150159.
[9] CLYNE P J, WARR C G, FREEMAN M R, et al. A novel family of divergent seventransmembrane proteins: Candidate odorant receptors in Drosophila [J]. Neuron, 1999, 22(2): 327338.
[10]HILL C A, FOX A N, PITTS R J, et al. G proteincoupled receptors in Anopheles gambiae [J]. Science, 2002, 298(5591): 176178.
[11]ROBERTSON H M, WANNER K W. The chemoreceptor superfamily in the honey bee, Apis mellifera: Expansion of the odorant, but not gustatory, receptor family [J]. Genome Research, 2006, 16(11): 13951403.
[12]BOHBOT J, PITTS R J, KWON H W, et al. Molecular characterization of the Aedes aegypti odorant receptor gene family [J]. Insect Molecular Biology, 2007, 16(5): 525537.
[13]ENGSONTIA P, SANDERSON A P, COBB M, et al. The red flour beetles large nose: An expanded odorant receptor gene family in Tribolium castaneum [J]. Insect Biochemistry and Molecular Biology, 2008, 38(4): 387397.
[14]SMADJA C, SHI P, BUTLIN R K, et al. Large gene family expansions and adaptive evolution for odorant and gustatory receptors in the pea aphid, Acyrthosiphon pisum [J]. Molecular Biology and Evolution, 2009, 26(9): 20732086.
[15]ROBERTSON H M, WARR C G, CARLSON J R. Molecular evolution of the insect chemoreceptor gene superfamily in Drosophila melanogaster [J]. Proceedings of the National Academy of Sciences of the United States of America, 2003(100): 1453714542.
[16]NOZAWA M, NEI M. Evolutionary dynamics of olfactory receptor genes in Drosophila species [J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(17): 71227127.
[17]MCBRIDE C S. Rapid evolution of smell and taste receptor genes during host specialization in Drosophila sechellia [J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(12): 49965001.
[18]EIRNLPEZ J M, REBORDINOS L, ROONEY A P, et al. The birthanddeath evolution of multigene families revisited [J]. Genome Dynamics, 2012(7): 170196.
[19]BENTON R. Multigene family evolution: Perspectives from insect chemoreceptors [J]. Trends in Ecology & Evolution, 2015, 30(10): 590600.
[20]MCKENZIE S K, KRONAUER D J C. The genomic architecture and molecular evolution of ant odorant receptors [J]. Genome Research, 2018, 28(11): 17571765.
[21]GUO Mengbo, DU Lixiao, CHEN Qiuyan, et al. Odorant receptors for detecting flowering plant cues are functionally conserved across moths and butterflies [J]. Molecular Biology and Evolution, 2021, 38(4): 14131427.
[22]HOU Xiaoqing, YUVARAJ J K, ROBERTS R E, et al. Functional evolution of a bark beetle odorant receptor clade detecting monoterpenoids of different ecological origins [J]. Molecular Biology and Evolution, 2021, 38(11): 49344947.
[23]LEGAN A W, JERNIGAN C M, MILLER S E, et al. Expansion and accelerated evolution of 9exon odorant receptors in Polistes paper wasps [J]. Molecular Biology and Evolution, 2021, 38(9): 38323846.
[24]International Aphid Genomics Consortium. Genome sequence of the pea aphid Acyrthosiphon pisum[J/OL]. PLoS Biology, 2010, 8(2): e1000313. DOI: 10.1371/journal.pbio.1000313.
[25]LI Yiyuan, PARK H, SMITH T E, et al. Gene family evolution in the pea aphid based on chromosomelevel genome assembly [J]. Molecular Biology and Evolution, 2019, 36(10): 21432156.
[26]ROBERTSON H M, ROBERTSON E C N, WALDEN K K O, et al. The chemoreceptors and odorant binding proteins of the soybean and pea aphids [J]. Insect Biochemistry and Molecular Biology, 2019(105):6978.
[27]ENDO T, IKEO K, GOJOBORI T. Largescale search for genes on which positive selection may operate [J]. Molecular Biology and Evolution, 1996, 13(5): 685690.
[28]YANG Ziheng, BIELAWSKI J P. Statistical methods for detecting molecular adaptation [J]. Trends in Ecology & Evolution, 2000, 15(12): 496503.
[29]王大海. 麥长管蚜转录组测序、RNAi靶标基因筛选及其和豌豆蚜的比较转录组学分析[D]. 北京: 中国农业科学院, 2013.
[30]蒙裕欢. 物种全基因进化分析揭示季节性物种受到更强的正选择[D]. 广州: 华南理工大学, 2012.
[31]付博华. 野桑蚕转录组测序与正向选择基因分析[D]. 重庆: 西南大学, 2015.
[32]智艳芳. 揭示适应性进化机制的新方法[D]. 西安: 陕西师范大学, 2017.
[33]陈艳红. 系统发育框架下鸟类食性差异的分子进化研究[D]. 武汉: 武汉大学, 2018.
[34]蒋梦婉. 基因组测序分析揭示青海田鼠和棕色田鼠低氧适应性进化[D]. 郑州: 郑州大学, 2020.
[35]张琳. 菲律宾菊头蝠组蝙蝠的系统发育与适应性进化研究[D]. 长春: 东北师范大学, 2020.
[36]LEARY G P, ALLEN J E, BUNGER P L, et al. Single mutation to a sex pheromone receptor provides adaptive specificity between closely related moth species [J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(35): 1408114086.
[37]ENGSONTIA P, SANGKET U, CHOTIGEAT W, et al. Molecular evolution of the odorant and gustatory receptor genes in lepidopteran insects: Implications for their adaptation and speciation [J]. Journal of Molecular Evolution, 2014, 79(1/2): 2139.
[38]THOMPSON J D, GIBSON T J, PLEWNIAK F, et al. The clustalx windows interface: Flexible strategies for multiple sequence alignment aided by quality analysis tools [J]. Nucleic Acids Research, 1997, 25(24): 48764882.
[39]HUANG Tianyu, ZHANG Ruibin, YANG Lulu, et al. Identification and functional characterization of ApisOr23 in pea aphid Acyrthosiphon pisum[J]. Journal of Integrative Agriculture, 2022, 21(5): 14141423.
[40]YANG Chunxiao, PAN Huipeng, LIU Yong, et al. Selection of reference genes for expression analysis using quantitative realtime PCR in the pea aphid, Acyrthosiphon pisum (Harris) (Hemiptera, Aphidiae)[J/OL]. PLoS ONE, 2014, 9(11): e110454. DOI: 10.1371/journal.pone.0110454.
[41]CAO Song, LIU Yang, WANG Bing, et al. A single point mutation causes oneway alteration of pheromone receptor function in two Heliothis species [J/OL]. iScience, 2021, 24(9): 102981. DOI: 10.1016/j.isci.2021.102981.
[42]LIU Yipeng, CUI Zhongyi, SI Pinfa, et al. Characterization of a specific odorant receptor for linalool in the Chinese citrus fly Bactrocera minax (Diptera: Tephritidae)[J/OL]. Insect Biochemistry and Molecular Biology, 2020, 122: 103389. DOI: 10.1016/j.ibmb.2020.103389.
[43]ZHANG Ruibin, LIU Yang, YAN Sanchun, et al. Identification and functional characterization of an odorant receptor in pea aphid, Acyrthosiphon pisum[J]. Insect Science, 2019, 26(1): 5867.
[44]LI Ruiting, HUANG Lingqiao, DONG Junfeng, et al. A moth odorant receptor highly expressed in the ovipositor is involved in detecting hostplant volatiles [J/OL]. eLife, 2020, 9:e53706. DOI: 10.7554/eLife.53706.
[45]LEGEAI F, MALPEL S, MONTAGNE N, et al. An expressed sequence tag collection from the male antennae of the noctuid moth Spodoptera littoralis: A resource for olfactory and pheromone detection research [J/OL]. BMC Genomics, 2011, 12: 86. DOI: 10.1186/147121641286.
[46]李彬,张赛,王晨蕊,等. 绿盲蝽八个普通气味受体基因的克隆及功能鉴定[J]. 昆虫学报, 2020, 63(9): 10481058.
[47]NOUHAUD P, GAUTIER M, GOUIN A, et al. Identifying genomic hotspots of differentiation and candidate genes involved in the adaptive divergence of pea aphid host races [J]. Molecular Ecology, 2018, 27(16): 32873300.
[48]PURANDARE S R, BRISSON J A. Divergent chemosensory gene expression accompanies ecological specialisation of pea aphid morphs [J]. Ecological Entomology, 2020, 45(2): 364368.
[49]FERNANDEZ R, MARCETHOUBEN M, LEGEAI F, et al. Selection following gene duplication shapes recent genome evolution in the pea aphid Acyrthosiphon pisum[J]. Molecular Biology and Evolution, 2020, 37(9): 26012615.
(責任编辑:田 喆)

