莱茵衣藻碳酸酐酶基因家族的生物信息学分析
马彦波,邓必成,侯余勇,路思晗,郭之乐,王巍杰
(1. 华北理工大学,生命科学学院,河北 唐山 063210;2. 中国科学院天津工业生物技术研究所,天津 300308)
碳酸酐酶(carbonic anhydrase,CA)是一种催化CO2和HCO3-之间转化的酶,广泛存在于自然界中的各种生物体内[1]。在藻类中,CA起着重要的作用,因为它们需要通过光合作用来吸收CO2并将其转化为有机物质[2,3]。α-CA和β-CA是藻类中的主要CA分类成员[4]。α-CA是一种单体酶,具有高度保守性,在不同种藻类中表现出相似的结构和功能特征[5]。β-CA是一种大多数情况下以二聚体形式存在的酶,其基因组分布较为广泛,具有更多样化的结构和功能[6]。藻类中CA的活性受到光照、温度、营养素等环境因素的调控,并且与藻类的生长、代谢以及碳循环等方面密切相关[7,8]。因此,对于藻类中CA的研究不仅有助于加深对这些生物体代谢和生态系统功能的理解,还可能为解决海洋碳循环和气候变暖等问题提供科学依据。
莱茵衣藻(Chlamydomonas reinhardtii)是研究绿藻代谢的模式微藻,具有较高的生长速度,但生物量的积累有限[9]。CO2在水中的溶解度很低,而CA可以将大气中的CO2转化为HCO3-,是微藻CCM(Carbon Concentrating Mechanism)机制中的重要组成部分,促进无机碳的吸收利用[10,11]。在藻类系统中,CA的功能是将细胞外HCO3-转化为1,5-二磷酸核酮糖羧化酶/加氧酶(RuBisCo)的底物[12,13],这也是CCM机制的共同特征,以增强微藻的碳捕获能力[14]。因此,对莱茵衣藻CA的生物信息学分析有助于为“双碳”政策下微藻固碳的应用提供理论基础。
1材料与方法
1.1 莱茵衣藻CA基因家族成员鉴定
在Ensemble Plant(https://plants.ensembl.org/index.html)数据库中下载物种莱茵衣藻的全基因组数据,包括CDS、Protain sequence和GFF3文件。从TAIR(https://www.arabidopsis.org/)数据库下载拟南芥已鉴定的α-CA和β-CA蛋白全长序列,通过本地BLAST搜索莱茵衣藻的全基因组序列(E-value值为1e-5)。将获得的蛋白序列利用NCBI-BLAST去除重复序列后,提交到NCBI-Batch-CDD、Pfam和SMART进一步验证结构域,获得莱茵衣藻的α-CA和β-CA基因家族成员[15]。
1.2 理化性质分析
利用ExPASyprotparam tool(https://web.expasy.org/protparam/)网站分析莱茵衣藻碳酸酐酶蛋白的理化性质[16],利用WOLF PSORT(https://wolfpsort.hgc.jp/)网站对莱茵衣藻碳酸酐酶蛋白的亚细胞定位进行预测。
1.3 同源性分析
在NCBI(https://www.ncbi.nlm.nih.gov/)数据库中下载杜氏盐藻、雨生红球藻、微拟球藻、小球藻的全基因组序列,按照1.1所述方法筛选出各物种的碳酸酐酶基因家族成员。利用MEGA11软件将莱茵衣藻的碳酸酐酶蛋白与杜氏盐藻、雨生红球藻、微拟球藻和小球藻的α-CA和β-CA亚家族成员的蛋白进行系统进化分析,构建系统发育树,选择Neighbor-Joining(邻接法)构建系统发育树,Bootstrap值设为1 000次,其余选择默认参数。利用iTOL(https://itol.embl.de/)在线软件注译发育树。
1.4 三级结构预测和蛋白质互作
利用Swiss-model (https://swissmodel.expasy.org/)网站预测莱茵衣藻CA蛋白的三级结构,String(https://cn.string-db.org/)构建莱茵衣藻CA家族蛋白之间的相互作用网络关系。
1.5 蛋白基因结构和保守基序分析
在Ensemble Plant(https://plants.ensembl.org/index.html)数据库中下载物种莱茵衣藻的全基因组数据,利用TBtools软件提取α-CA和β-CA的基因结构信息并进行数据分析。利用在线工具MEME Suite 5.4.1(https://meme-suite.org/meme/meme_5.4.1/)对莱茵衣藻碳酸酐酶蛋白家族的保守基序进行预测,设置Motif数量为10,最大宽度50,最小宽度6,进一步使用TBtools软件实现Motif保守基序的可视化。
1.6 染色体定位和顺式作用元件分析
利用TBtools软件预测莱茵衣藻CA蛋白家族各基因的染色体位置,从莱茵衣藻基因组中导出CA基因上游2 000 bp启动子区域序列,然后利用Plant CARE(https://bioinformatics.psb.ugent.be/webtools/plantcare/html/)网站预测莱茵衣藻CA蛋白启动子区域顺式作用元件。
1.7 添加碳酸氢盐促进莱茵衣藻CA的表达
实验以TAP(Tris Acetate-Phosphate)培养基培养为对照组,在对照组中添加5 mmol/L的HCO3-为实验组,固定时间间隔进行取样,测定680 nm处的吸光度值,并对CA基因进行表征。
2结果与分析
2.1 莱茵衣藻CA家族成员鉴定及理化性质分析
按照1.1所述方法检索莱茵衣藻CA蛋白序列,进一步通过CDD、Pfam(α-CA:PF00484; β-CA:PF00194)和SMART蛋白质预测工具验证结构域,获得3个α-CA和5个β-CA蛋白,将其分别重新命名为Cr1~3及Cr4~5。利用ExPASyprotparam tool在线软件分析蛋白理化性质,WOLF PSORT网站对亚细胞定位进行预测,结果见表1。
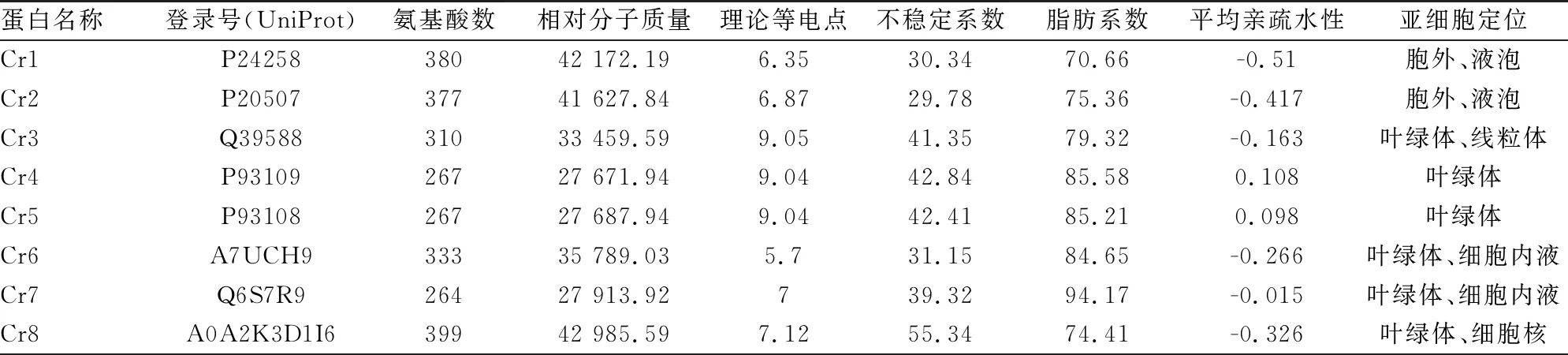
表1 莱茵衣藻CA蛋白家族的理化性质
由表1可看出,α-CA亚家族的Cr1~3编码的氨基酸残基数量为310~380 个,分子量在33.46~42.17 kDa,理论pI值2个家族成员小于7.0,另外1个家族成员Cr3大于7.0,均呈亲水性。β-CA亚家族的Cr4~8所编码的氨基酸数量为267~399个,分子量在27.67~42.99 kDa,其理论pI值3个成员大于7.0,另外2个家族成员均在5~7之间,平均亲疏水性2个成员疏水,另外3个成员亲水。
2.2 系统进化分析
为了分析莱茵衣藻8个CA家族成员与杜氏盐藻、雨生红球藻、微拟球藻和小球藻CA家族成员的系统进化关系,将杜氏盐藻11个CA(Ds1~11)的氨基酸序列,雨生红球藻10个CA(Hl1~10)的氨基酸序列,微拟球藻14个CA(Ng~14)的氨基酸序列,及小球藻5个CA(Cv~5)的氨基酸序列,连同这8个碳酸酐酶的CA成员一起比对,采用NJ方法构建系统发育树(见图1)。结果表明,不同藻来源的α-CA成员,均很好地聚为一类,而β-CA亚家族成员被分为2类,说明不同CA亚家族成员在进化上具有相对独立性。从整体上看,莱茵衣藻与雨生红球藻2个CA亚家族成员之间的亲缘关系更近一些。
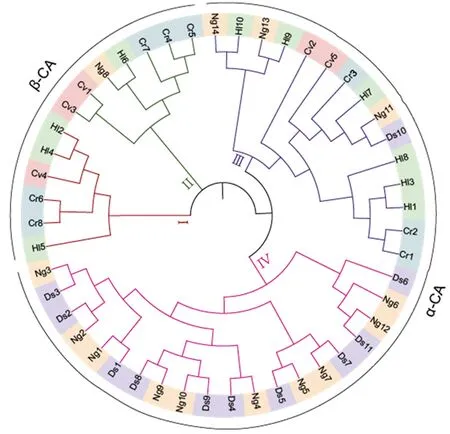
图1 莱茵衣藻与杜氏盐藻、雨生红球藻、微拟球藻、小球藻CA家族成员的进化关系
2.3 CA蛋白三级结构预测和互作网络分析
三级结构预测结果(见图2)显示,Cr4、Cr5与Cr7为等位于同一进化树分支的成员,三级结构高度相似,Cr6与Cr8在结构上完全相同且位于同一亚族的相同分支上。但Cr1、Cr2与Cr3比较特殊,虽然位于同一分支上,但三级结构有着明显差异,推测某些非同源的部分基因决定了其蛋白结构,后续蛋白特征结构与基因结构分析也说明了这一点。
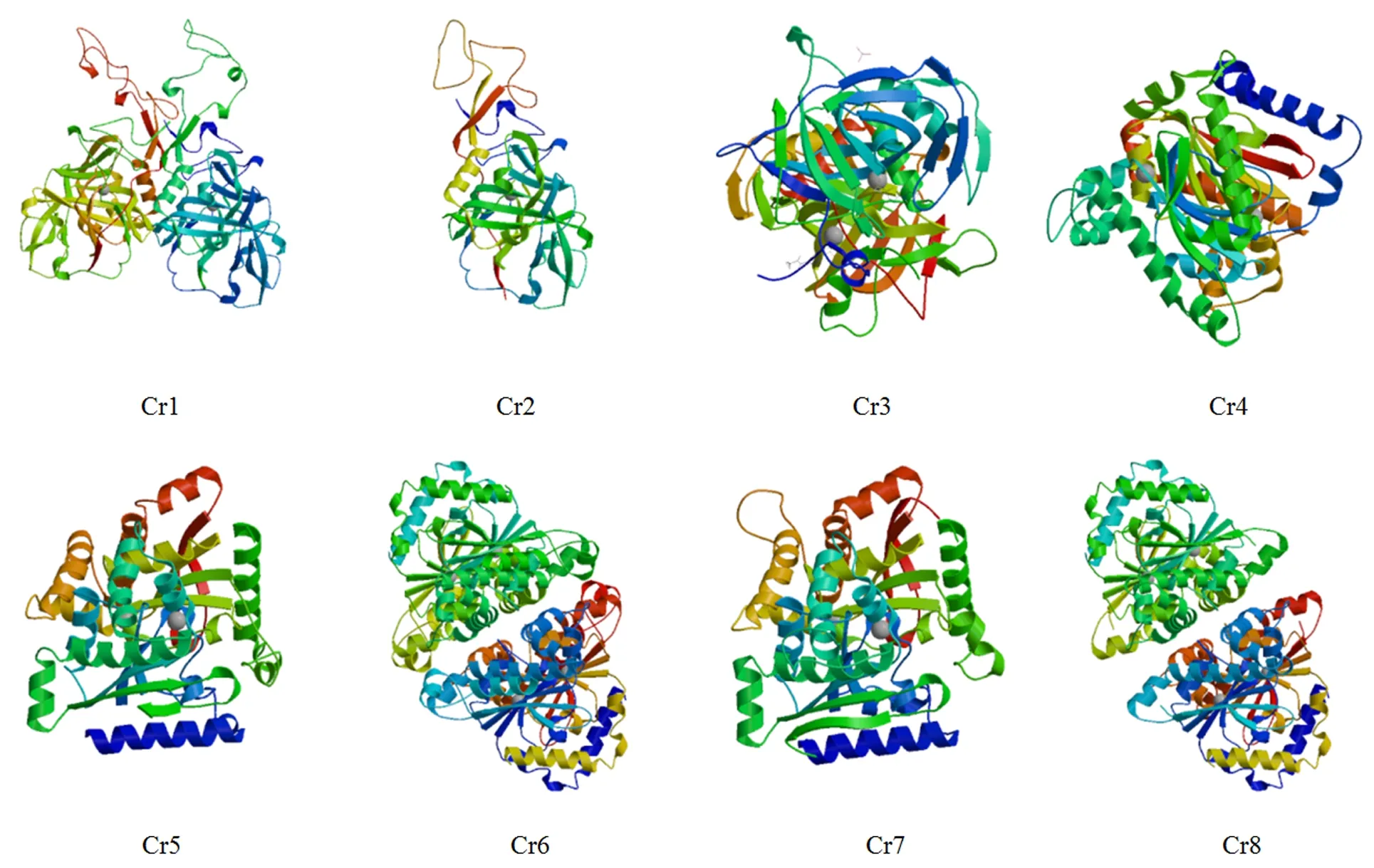
图2 莱茵衣藻CA家族蛋白三级结构预测图
为了更好地了解莱茵衣藻CA基因家族功能,将8个莱茵衣藻CA蛋白上传至STRING在线数据库中,以拟南芥蛋白质数据库为参考进行比对,构建出莱茵衣藻CA蛋白的互作网络(见图3所示),对莱茵衣藻CA蛋白互作信息进行评估和预测。结果显示,Cr4没有与其他蛋白存在互作关系,Cr2分别与Cr1和Cr3之间存在直接的蛋白质相互作用关系。通过分析蛋白质互作网络,发现Cr2的互作蛋白最多,有6个;其他蛋白之间的互作均在3~4个之间。

图3 莱茵衣藻CA家族蛋白互作网络预测
2.4 蛋白基因结构和保守基序分析
为了进一步了解CA蛋白特征区域,通过MEME在线网站选用10个Motif对CA家族蛋白进行分析(见图4)。每个Motif中氨基酸残基字符越大,其氨基酸位点保守性越好;基序的氨基酸残基大字符越多,该基序越保守稳定。
根据保守基序分析结果(如图5所示)可知,莱茵衣藻所有α-CA家族成员均含有Motif2和Motif10,而在β-CA亚家族中Motif1、Motif5和Motif6稳定存在,说明Motif1、Motif5和Motif6是β-CA主要功能结构域,但Motif5在Cr7中没有显现出来,其基序空缺可能导致其蛋白结构和功能的变化,推测其具有功能差异性。

图4 Motif1-10的多级共有序列
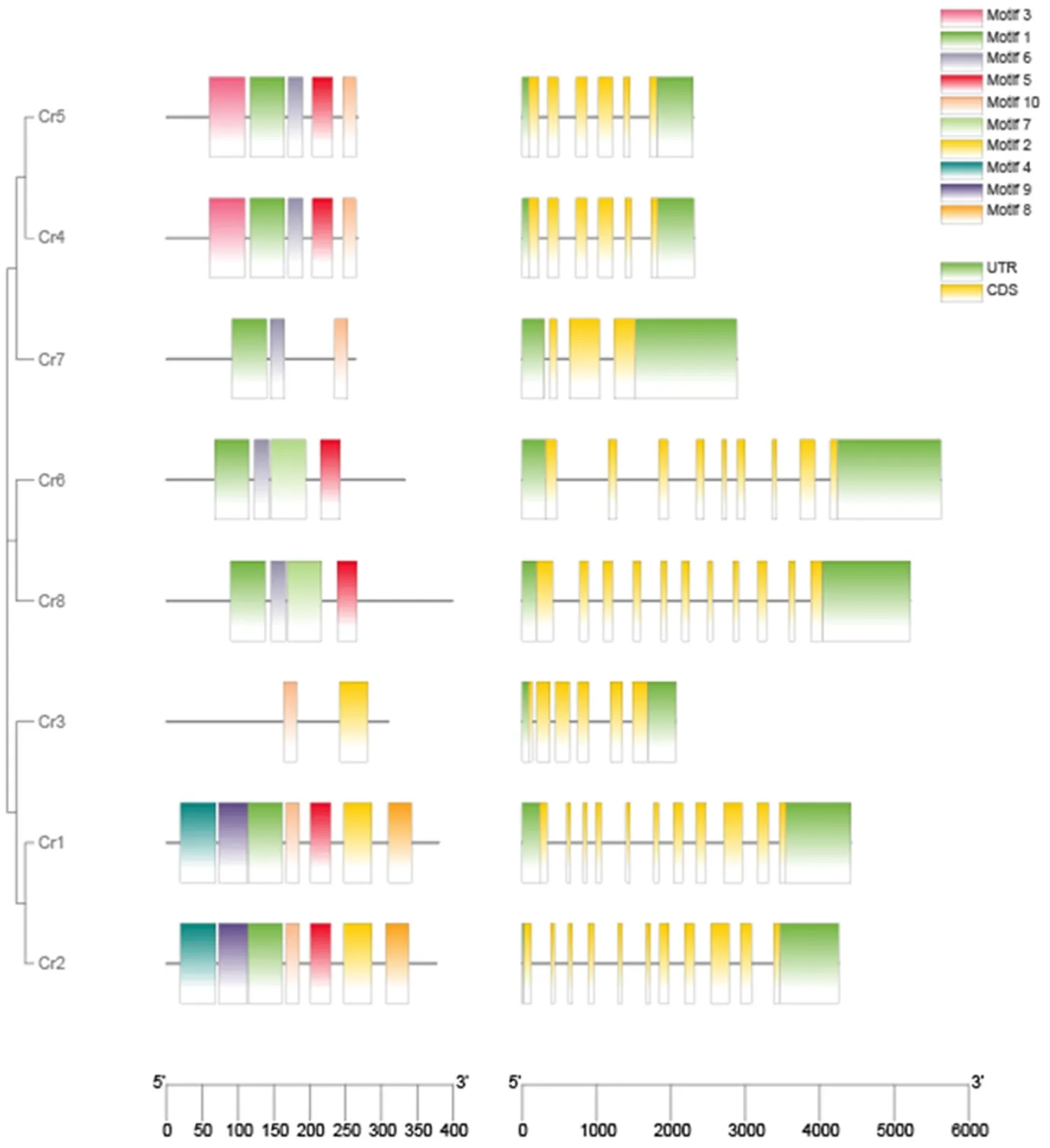
图5 莱茵衣藻CA家族保守结构域与基因结构图
图5基因结构分析显示,莱茵衣藻α-CA亚家族各成员外显子个数在6~11之间,而β-CA成员则在6~9之间,α-CA亚家族成员的外显子数目要多于β-CA亚家族,推测α-CA亚家族成员的基因结构较β-CA亚家族复杂。
2.5 染色体定位和顺式作用元件分析
在染色体定位分布图(如图6所示)中,每个CA基因的位置可以用坐标度来确定,8个CA基因定位于莱茵衣藻的5条染色体上。其中4号、5号和9号染色体上分布最多,各有2个CA基因;12、13号染色体上各有1个CA基因。莱茵衣藻CA各成员在染色体的位置都相距较远,推测没有发生串联互换,说明CA家族基因的保守性。
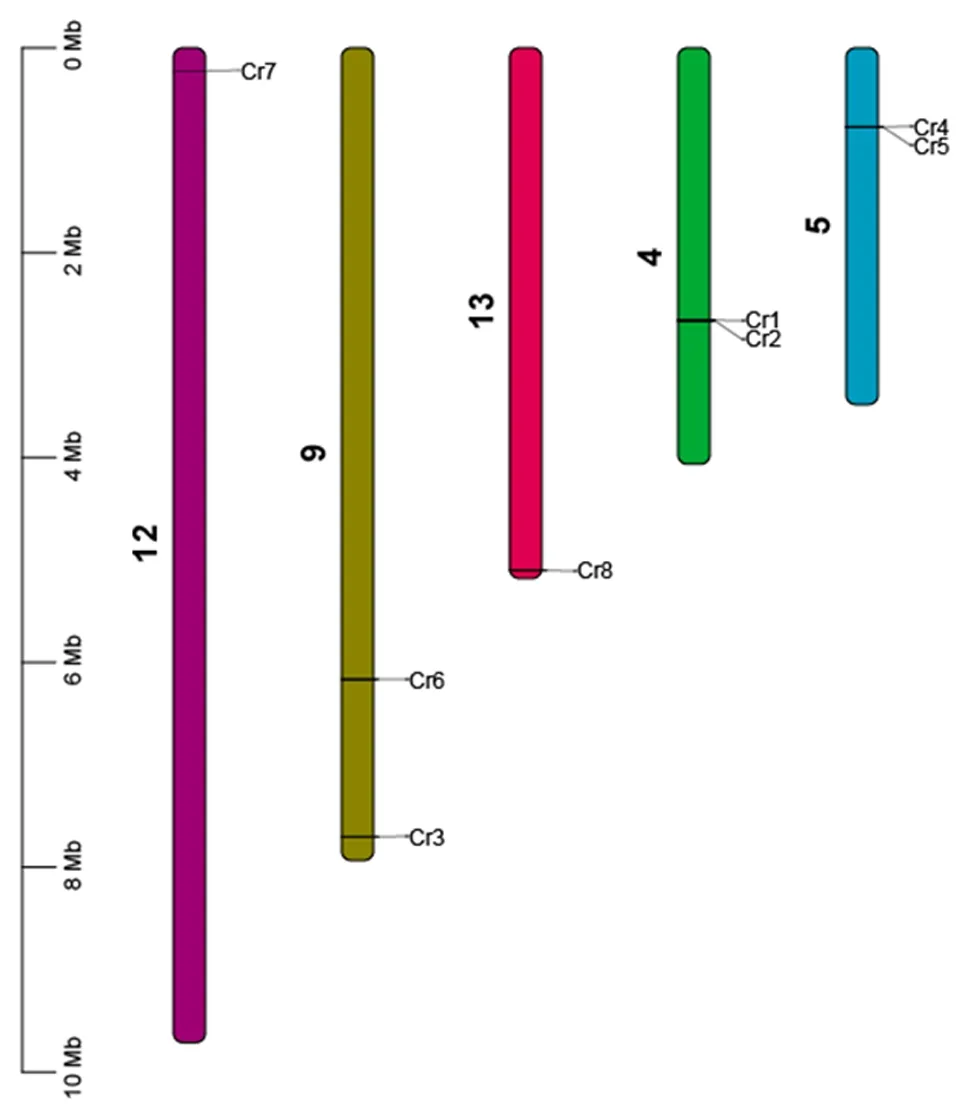
图6 莱茵衣藻CA家族染色体定位图
通过PlantCARE工具对莱茵衣藻8个CA基因的启动子顺式作用元件进行分析(见图7所示)。
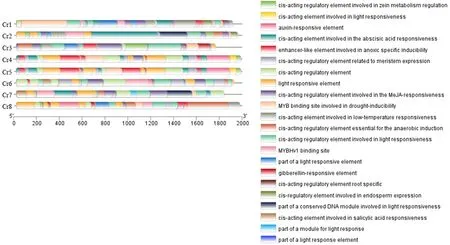
图7 莱茵衣藻CA家族顺式作用元件分布图
结果表明,在这些基因的启动子中主要含有激素应答顺式作用元件,例如,参与Me JA响应的CGTCA-motif和TGACG-motif;参与ABA响应的ABRE元件;参与水杨酸反应的TCA-element;参与生长素响应的TGA-element;参与胚乳表达的调节元件GCN4-motif;参与赤霉素响应的P-box元件;与缺氧调控相关的GC-motif元件;与厌氧诱导调控相关的ARE元件;与分生组织表达相关的CAT-box元件。除此外,还含有多个参与光响应、非生物胁迫等相关的顺式作用元件及多种转录因子结合位点。如控制光反应的相关元件TCT-motif、ACE、TCCC-motif、CAG-motif、AE-box;参与低温响应的LTR元件,以及MYC转录因子结合元件G-box和C-box。结果表明,莱茵衣藻的碳酸酐酶基因有可能响应了逆境及激素信号从而参与调控植物生长发育的过程。
2.6 添加碳酸氢盐生理实验和相对表分析
为了进一步对莱茵衣藻CA家族基因功能进行验证,实验结果表明,添加碳酸氢盐促使莱茵衣藻生物量的积累,在莱茵衣藻生长的前期,以无机碳源(HCO3-)为主要碳源进行代谢生长,与对照组相比,实验组中莱茵衣藻的生物量的积累显著高于对照组,在第24 h,实验组生物量是对照组生物量的2倍(见图8所示)。笔者曾经发表的文章结果表明[17],CA家族基因在莱茵衣藻的代谢生长方面有着重要意义,尤其是在CO2和HCO3-的相互转化和利用;基因表征的实验结果也表明,CAH2(Cr1)和CAH3(Cr3)在莱茵衣藻吸收利用无机碳有着重要的作用,莱茵衣藻生长的初期CAH2和CAH3就有着不同程度上的上调表达,其他无机碳转运子(CCP1/CCP2/LCIA/LCIB/LCIC等)的协同作用下,促进了莱茵衣藻对无机碳源的利用和对生物量的积累。总之,对莱茵衣藻CA蛋白家族的生物信息学研究,可以为莱茵衣藻生物固碳和提升生物量的研究提供更多的理论基础。
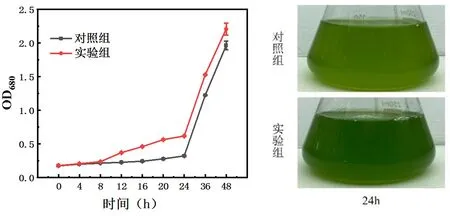
图8 莱茵衣藻生长曲线
3结论
(1)采用生物信息学分析方法分析莱茵衣藻CA基因家族的8个CA家族成员,结果表明莱茵衣藻CA基因家族的蛋白序列长度最小为264 aa,最大为399 aa,分子量大小和理论pI分别介于33.46~42.17 kDa和5.7~9.05之间,其中只有Cr4和Cr5具有疏水性,其余的均为亲水性。
(2)系统进化分析表明杜氏盐藻、雨生红球藻、微拟球藻和小球藻CA家族的α-CA成员聚为一类,β-CA分成2类;蛋白质三级结构和互作网络预测表明,CA蛋白家族成员中,Cr4、Cr5和Cr7三级结构高度相似,Cr6和Cr8三级结构高度相似,而在蛋白质互作中,Cr4没有参与之间的互作关系,Cr6与Cr8在结构上完全相同且位于同一亚族的系统进化树相同分支上,在互作关系中,Cr2的互作蛋白最多,这也与笔者曾经发表论文中基因表征结果一致[17]。
(3)莱茵衣藻的8个CA基因均含有特定保守基序;染色体定位发现基因Cr1和Cr2在4号染色体,Cr4和Cr5在5号染色体,Cr3和Cr6在9号染色体,Cr7和Cr8分别在12和13号染色体上;实验结果表明,CA在莱茵衣藻吸收、转化和利用HCO3-方面发挥着重要作用[17]。

