麦鱼线粒体基因组序列测定与结构特征分析
鲍鸣 奚业文 海思娆 王晨龙 陈闯将 王希春 李西雷



摘 要:通过PCR扩增和一代测序技术获得麦鱼 (Rhinogobius sp.) 线粒体基因组全序列,并对其序列结构进行了分析。结果表明:1) 麦鱼线粒体基因组全长16 511 bp,其碱基G+C的含量为47.7%,A+T的含量为52.3%,呈现AT偏好性;2) 共含有蛋白质编码基因13个,rRNA基因2个,tRNA基因22个和D-loop区,除ND6、tRNA-Gln、tRNA-Tyr、tRNA-Ala、tRNA-Asn、tRNA-Cys、tRNA-Ser、tRNA-Glu、tRNA-Pro基因在L链上编码之外,其他基因均在H链上进行编码;3) 基于线粒体基因组全序列和Cytb基因,分别构建了鰕虎鱼类中部分属的进化树,其表明麦鱼与波氏吻鰕虎鱼、褐吻鰕虎鱼、子陵吻鰕虎鱼聚在一个分支上,均属于吻鰕虎鱼属,并且其与波氏吻鰕虎鱼的亲缘关系最近。
关键词:麦鱼(Rhinogobius sp.);线粒体基因组;序列分析;系统发育进化;物种鉴定
麦鱼(Rhinogobius sp.) 属鲈形目(Perciformes),鰕虎鱼亚目(Gobioidei),鰕虎鱼科(Gobiidae),吻鰕虎鱼属(Rhinogobius),为安徽省池州市东至县所特有的小型鱼类之一,因盛产于刈麦季节而定名,与著名的庐山石鱼属于同一种类[1]。迄今对于麦鱼的研究甚少,麦鱼准确的物种鉴定、分类地位及遗传多样性尚未确定,现仅有根据形态学特征进行的分类研究,其养殖潜力尚待评估与开发[1]。
线粒体基因组数据特点显著,如结构紧密、能够自主复制、编码效率高、无组织特异性、无重组现象、进化速率快以及遵循严格的母系遗传等,在系统与进化生物学和群体遗传学等许多生物学研究中已广泛应用[2-9]。目前对鰕虎鱼科多属种的分类较为混乱,当前最被接受的分类是NELSON(2006)提出的9科5亚科分类方法,中国鰕虎鱼分类与此一致[10-11]。根据分布范围已经证实过去记载于我国的栉鰕虎鱼属种类实际上应归为吻鰕虎鱼属[12]。在已有的研究中,研究者根据形态特点结合生态习性将麦鱼鉴定为吻鰕虎鱼和洞庭吻鰕虎鱼[1],伍汉霖等[13]、刘蝉馨[14]、罗智等[15]根据生物及生态学特性研究鰕虎鱼类的分类地位。曲丽艳[10]通过核基因及线粒体COI基因对28种鰕虎鱼进行了系统发育分析,将传统分类中归为鰕虎鱼亚科的睛尾蝌蚪鰕虎鱼聚到小鰕虎亚科,其他大部分结果与形态研究一致。ZHONG等[16]通过线粒体基因对波氏吻鰕虎鱼(Rhinogobius Cliffordpopei)进行的系统发育分析表明,波氏吻鰕虎鱼与其他吻鰕虎鱼属鱼类聚在一个分支,但吻鰕虎鱼属与缟鰕虎鱼属的进化关系尚不清楚。
本试验使用PCR扩增和一代测序技术,测定了麦鱼的线粒体基因组全序列,分析了基因的排序及其组成,通过与26种鰕虎鱼类线粒体基因组进行比对,构建鰕虎鱼类的系统进化树,进一步鉴定了麦鱼的分类地位,补充了鰕虎鱼科的线粒体基因组数据,也为吻鰕虎鱼属的分子鉴定和系统发育研究提供了参考基础。
1 材料与方法
1.1 试验材料
本试验中所用的麦鱼样品均由2022年7月采自安徽省池州市东至县。采集样本的肌肉组织以及肝脏组织,去离子水清洗,-80 ℃冰箱中保存备用。
1.2 线粒体DNA提取
使用灭菌的剪刀切割30 g左右的肌肉组织于液氮中,-80 ℃保存待用,线粒体DNA严格按照上海诺伦生物医药技术有限公司细胞线粒体DNA抽提试剂盒说明书进行,1%琼脂糖凝胶电泳检测。
1.3 线粒体基因组测序
引物设计根据麦鱼种在NCBI上的近缘物种的线粒体基因序列,PCR扩增程序为:94 ℃预变性30 s,94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸1 min/kb,30个循环后,72 ℃延伸10 min。取10 μL PCR产物于1%琼脂糖凝胶中电泳检测,DNA纯化样品直接测序。
1.4 序列的拼接与分析
使用BioEdit进行序列分析,删去可信度低的测序片段,并统计其长度,再利用NCBI在線功能BLAST与数据库里面已经报道过的鰕虎鱼亚科进行比对分析,并使用Contig Express进行序列的拼接,最终补全序列,将麦鱼全线粒体基因组序列提交至GenBank数据库,获得序列号为QDA22533.1—QDA22545.1。
使用NCBI在线分析Open Reading Frame Finder预测确定编码位置。用NCBI中已有的鰕虎鱼类线粒体基因组进行比对,注释蛋白质编码基因(Protein coding genes,PCGs)及D-Loop区。软件MEGA7.0分析线粒体基因序列的碱基组成、密码子使用情况以及碱基偏移程度。
利用在线tRNAscan-SE软件进行麦鱼tRNA基因定位,并利用The Mfold Web Server 在线软件有效预测麦鱼线粒体tRNA的二级结构。
1.5 系统进化树的构建
使用 MEGA 7.0基于K-2-P模型采用自展法(bootstrap)1 000次检验分支可信度,构建邻接法(NJ法)分子系统进化树,将在GeneBank中下载的同一属26种鰕虎鱼类的线粒体基因组全序列以及Cytb基因序列与本试验得到的麦鱼线粒体基因组全序列(16 511 bp)进行多重序列比对和分析,以了解鰕虎鱼类之间的发育关系。使用的基因基本信息见表1。
2 结果与分析
2.1 麦鱼线粒体基因组结构与特征
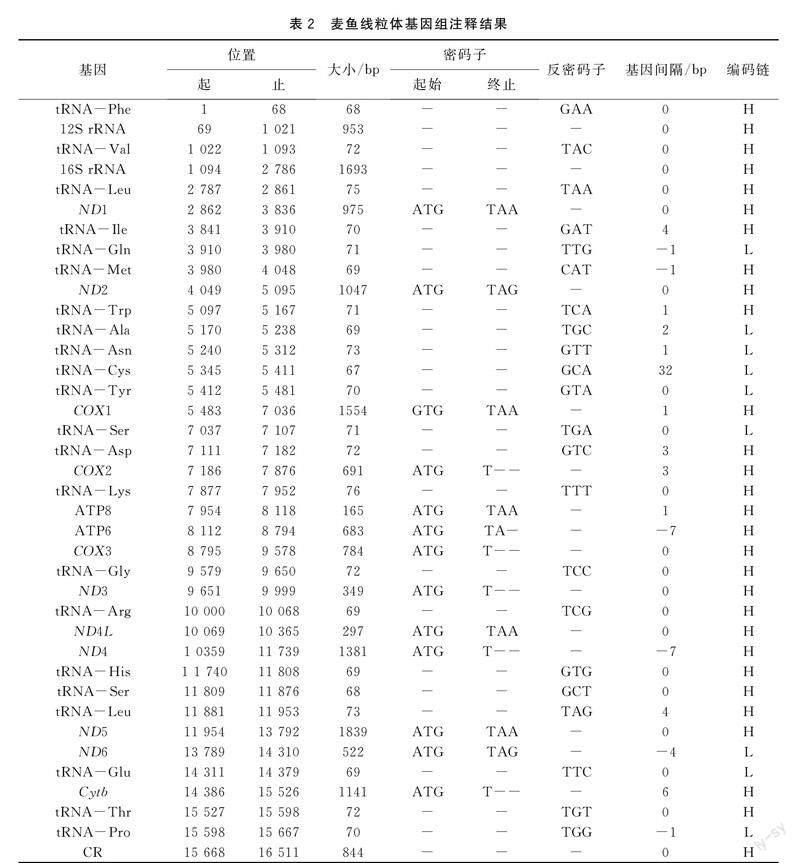
麦鱼线粒体基因组全长16 511 bp,碱基含量分别为A (26.7%) 、T (25.6%) 、G (16.9%) 、C (30.8%) ;G+C含量为47.7%,A+T含量为52.3%,说明本试验中麦鱼线粒体基因组呈现AT偏好性。麦鱼线粒体全基因组序列中共含37个基因,分别为PCGs 13个、tRNA基因22个、rRNA基因2个和d-loop区 1个(见封三图1)。ND6基因以及tRNA基因中的tRNA-Gln、tRNA-Tyr、tRNA-Ala、tRNA-Asn、tRNA-Cys、tRNA-Ser、tRNA-Glu和tRNA-Pro均在L-链上进行编码,余下的28个蛋白质基因及tRNA基因则均在H-链上进行编码(表2) 。
麦鱼线粒体基因组结构与众多鱼类基因组结构相同,碱基排列较紧密,但仍存在部分重叠与间隔[3-5]。其基因间隔共有11处,间隔长度共计58 bp,其中间隔最大处出现于tRNA-Cys基因与tRNA-Asn基因之间,间隔为32 bp。而基因重叠共计出现6次,共计21 bp,长度不一,其中重叠长度最多的一处出现在ATP8与ATP6之间以及ND4L基因与ND4基因之间,重叠7 bp。既没有出现重叠也没有出现间隔处总计22处 (表2) 。
2.2 线粒体基因组蛋白质编码基因
本次试验获得的麦鱼线粒体基因组的13个蛋白质编码基因序列总长度为11 428 bp,含3 810个密码子 (终止密码子除外) ,在H链上进行编码的有12个基因(ND1,ND2、COX1、COX2、ATP8、ATP6、COX3、ND3、ND4L、ND4、ND5、Cytb) ,仅ND6基因在L链上进行编码。蛋白质编码基因拥有两种起始密码子(ATG和GTG),除了COX1蛋白质编码基因的起始密码子为GTG以外,其余12个蛋白质编码基因的起始密码子均为ATG。终止密码子有四种(TAG、TAA、TA-、T--),使用情况相较起始密码子更为复杂。其中ND1、COX1、ATP8、ND4L、ND5基因的终止密码子为TAA;ND2、ND6基因的终止密码子为TAG;而COX2、COX3、ND3、ND4、Cytb基因则是以不完全的T作为终止密码子;ATP6基因的终止密码子为TA-。终止密码子使用情况与太平洋鳕[3]、黄条鰤[4]相同。
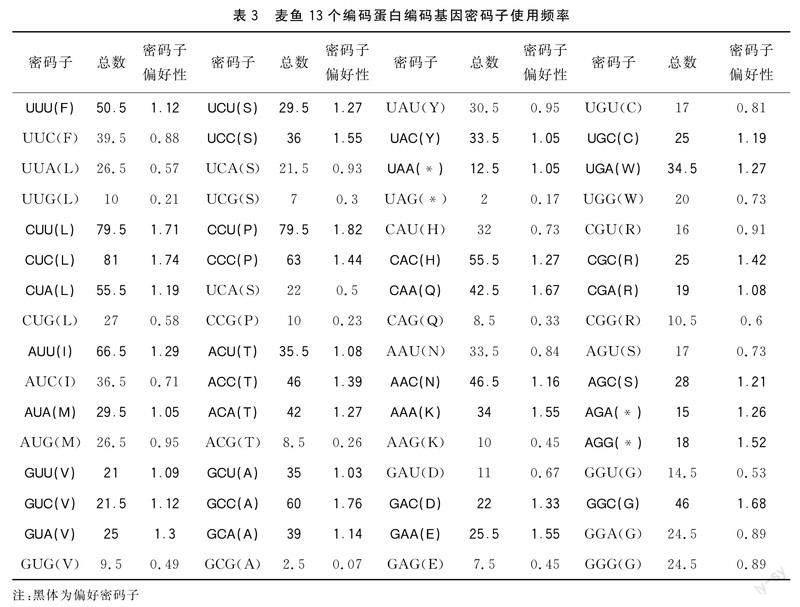
软件分析蛋白质编码基因密码子的使用频率(表3)以及同义密码子使用频率的结果表明:13个蛋白质编码基因中有35个偏好密码子(RSCU密码子),第三位为C或T的密码子具有很高的使用频率,以G为结尾的密码子使用频率最小。第三位碱基以A或者T结尾的密码子中,UUA(L)、UCA(S)、UAU(Y)、CAU(H)、AAU(N)、GAU(D)、UGU(C)、CGU(R)、AGU(S)、GGU(G)、GGA(G)密码子的RSCU小于1,其他密码子RSCU均大于1。
2.3 线粒体基因组tRNA基因及rRNA基因
麦鱼线粒体基因组共计22个tRNA基因,其长度分布于68 bp到76 bp之间,总计1 556 bp,不均匀地分布于13个蛋白质编码基因之间。分析结果显示,tRNA基因之间存在着相同程度的重叠及不同程度的间隔,tRNA-Ile与tRNA-Gln、tRNA-Thr与tRNA-Pro、tRNA-Gln与tRNA-Met分别都存在相同程度的核苷酸重叠,三组重叠的核苷酸都是1bp。其间隔的长度从1 bp至32 bp不等,如tRNA-Asn基因与tRNA-Cys基因之间的间隔为32 bp。tRNA-Gln、tRNA-Asn、tRNA-Ala、tRNA-Cys、tRNA-Tyr、tRNA-Glu、tRNA-Ser、tRNA-Pro基因均在L链进行编码,其余14个tRNA基因在H链编码。tRNA的二级结构预测结果显示(详见图2),tRNA-Val、tRNA-Phe等21个tRNA基因均可形成明显的三叶草结构,tRNA-Ser (GCT) 基因缺失二氢尿嘧啶臂 (DHU臂) 。
另外,麦鱼线粒体基因组包括rRNA基因2个,12S rRNA以及16S rRNA基因,均处于H链上,位于tRNA-Phe与tRNA-Leu之间,由tRNA-Val基因将两者分开。12S rRNA基因和16S rRNA基因的A+T含量分别为52.8%和52.7%,呈现AT碱基偏好性。
2.4 系统进化树分析
2.4.1 线粒体全基因组NJ进化树分析 系统进化树(见图3)分为两个大支,第一个分支由大弹涂鱼属、青弹涂鱼属、副平牙鰕虎鱼属、狼牙鰕虎鱼属 、鳗鰕虎鱼属、齿弹涂鱼属、弹涂鱼属、缟鰕虎魚属、竿鰕虎鱼属、鲻鰕虎鱼属等构成;第二个分支由尖塘鳢属、乌塘鳢属、吻鰕虎鱼属构成。
由图3可知本次试验获得的麦鱼线粒体基因组和子陵吻鰕虎鱼、褐吻鰕虎鱼、波氏吻鰕虎鱼聚在较近的分支上,与波氏吻鰕虎鱼亲缘关系最近。并进一步证实过去记载于我国的栉鰕虎鱼属种类实际上应归为吻鰕虎鱼属,结果与前人的分类结果相契合[17]。
2.4.2 Cytb基因NJ进化树分析 本分析采用当前在物种鉴别、遗传多样性和群体分化中常用的Cytb基因作为分子标记,研究麦鱼的系统发育地位[3,18-20]。该系统进化树(见封三图4) 主要分为三个分支,青弹涂鱼属、副平牙鰕虎鱼属、狼鰕虎鱼属、齿弹涂鱼属和弹涂鱼属聚为一个大支,缟鰕虎鱼属为一个分支,吻鰕虎鱼属、乌塘鳢属、竿鰕虎鱼属、尖塘鳢属和鲻鰕虎鱼属聚为一个分支,乌塘鳢属与吻鰕虎鱼属和本次研究的麦鱼距离最近,由图4可知,根据Cytb基因构建的进化树与线粒体全基因组构建的进化树结果几乎完全一致。
3 讨论
通过研究获得麦鱼线粒体基因组全长16 511 bp,包括22个tRNA 基因、13个蛋白编码基因,2个rRNA 和1段 D-loop 控制区,在玻璃缺鳍鲶(Kryptopterus vitreolus)[5]中也有此类结构。其线粒体基因组中,A+T碱基 (52.3%) 大于G+C碱基 (47.7%) 含量,呈现AT偏好性,这与太平洋鳕 (Gadus macrocephalus)[3]、黄条鰤(Seriola aureovittata)[4]、玻璃缺鳍鲶[5]、吻鰕虎鱼属(Rhinogobius)[21]等鱼类相似。
在麦鱼线粒体基因组中,蛋白质编码基因既存在于H链也存在于L链上。Brown等[6]报道,H链上的蛋白质编码基因不容易形成有保护的双链,因该H链大多是水解的单链。与黄条鰤[4]相似,麦鱼线粒体基因组中绝大多数蛋白质编码基因都位于H链上,易于发生相应的水解和氧化,而位于L链上的ND6基因会稳定很多,这也体现了ND6基因的差异性和重要性。
在麦鱼mtDNA的tRNA基因和rRNA基因中,含有22个tRNA,它们的长度分布于68 bp到76 bp之间,总计1 556 bp,其中绝大部分tRNA的二级结构都满足三叶草的形状,穿插于不同的蛋白质编码基因与rRNA中间。仅tRNA-Ser (GCT) 基因缺失二氢尿嘧啶臂 (DHU臂),这与毛明光等[3]研究的太平洋鳕十分相似。麦鱼线粒体基因组中含有2个rRNA,16S rRNA和12S rRNA,其与相邻基因并无间隔和重叠。线粒体基因组中含有的重复序列、部分插入序列和序列等都属于物种进化的特征[7]。
本研究的麦鱼与波氏吻鰕虎鱼、褐吻鰕虎鱼、子陵吻鰕虎鱼聚在一个分支上,均属于吻鰕虎鱼属,并且其与波氏吻鰕虎鱼的亲缘关系最近。此外,吻鰕虎鱼属与中华乌塘鳢、尖塘鳢、斑驳尖塘鳢所聚成的塘鳢属比较接近,这与曲丽艳[10]、孙希福[18]、金逍逍等[22]、ZHONG等[16]等的分析结果基本相似。本次试验的麦鱼不管是线粒体基因组全序列进化树,还是使用Cytb基因构建的进化树分析,都出现了乌塘鳢属、吻鰕虎鱼属与麦鱼的距离最近的结果,但是对于其他分析结果有所不同,缟鰕虎鱼属在线粒体基因组分析中和其他属归为一支,在使用Cytb基因进行进化树分析时,单独地划为一个分支。产生这样的结果是因为脊椎动物线粒体基因的不同区域的进化速率不同,所以线粒体各个基因的系统发育信息的表现也会有所不同。
目前,线粒体基因组中的相关基因被大量应用于系统发育、物种分类等相关研究领域[2]。KIM等[23]、黄小林等[8]基于mtDNA中的12S rRNA、ND4L基因构建系统进化树研究矛尾刺鰕虎鱼(Acanthogobius hasta)、篮子鱼(Siganidae Siganus)的进化地位。随着分子生物学手段的普遍使用,国内外学者对鰕虎鱼科鱼类系统发育研究也越来越多,AGORRETA等[24]分析了222种欧洲的鰕虎鱼目鱼类在系统进化上的关系,THACKER等证实了丝鳍鱚属是鰕虎鱼科的姐妹属[25],研究了鰕虎鱼科在棘鳍鱼目中的系统分布[26]。普遍来说线粒体基因的进化速度大于物种的进化速度,不同的地理环境、生活习惯也都有机会导致物种DNA的变异,要解释清楚还需要进一步加深对于线粒体基因的研究。
参考文献:
[1] 安徽省池州地区升金湖水产资源调查小组.池州地区的麦鱼[J].动物学杂志,1976,4(19):36-37.
[2] 李雪娟,杨婧,王俊红,等.线粒体基因组数据等分析方法和软件[J].应用昆虫学报,2013,50(1):298-304.
[3] 毛明光,顾杰,刘瑞婷,等.太平洋鳕线粒体全基因组测序及结构特征分析[J].水生生物学报,2019,43(1):17-26.
[4] 史宝,柳学周,刘永山,等.黄条鰤线粒体全基因組测序及结构特征分析[J].中国水产科学,2019,26(3):405-415.
[5] 孙志鹏,吕伟华,匡友谊,等.玻璃缺鳍鲶线粒体全基因组序列测定与分析[J].水产学杂志,2018,31(2):12-18.
[6] BROWN W M,PRAGER E M,WANG A,et al.Mitochondrial DNA sequences of primates:tempo and mode of evolution[J].Journal of Molecular Evolution,1982,18(4):225-239.
[7] 高祥刚,鹿志创,田甲申,等.斑海豹线粒体基因组序列分析及比较研究[J].水产科学,2017,36(4):5.
[8] 黄小林,杨育凯,林黑着,等.篮子鱼属线粒体基因组序列系统发育信息分析[J].生物学杂志,2018,35(5):33-36.
[9] 杨杨,宋小晶,唐文乔,等.克氏光唇鱼线粒体基因组测定及光唇鱼属的系统发育分析[J].动物学杂志,2018,53(2):207-219.
[10] 曲丽艳.基于形态、线粒体及核基因对东海28种鰕虎鱼类的系统进化分析[D].上海:上海海洋大学,2018:42-47.
[11] NELSON J S.Fish of the world[M].4th ed.New York:John Wiley and Sons, 2006:601.
[12] 郭昶畅.中国沿海石首鱼科鱼类的鉴定、分类和分子系统发育研究[D].厦门:厦门大学,2017.
[13] 伍汉霖,钟俊生.中国动物志-硬骨鱼纲、鲈形目(五)、鰕虎鱼亚目[M].北京:科学出版社,2008.
[14] 刘蝉馨.纹缟鰕虎鱼的生物学和生态学[J].水产科学,1985(4):13-17.
[15] 罗智,李晓东,白海娟,等.野生和养殖矛尾复鰕虎鱼营养组成和形态学的比较研究[J].上海水产大学学报,2008,17(2):182-186.
[16] ZHONG L Q,WANG M H,LI D,et al.Complete mitochondrial genome of freshwater goby Rhinogobius cliffordpopei(Perciformes,Gobiidae):genome characterization and phylogenetic analysis[J].Genes & Genomics,2018,40:1137-1148.
[17] 李帆,钟俊生.中国浙江省吻鰕虎鱼属一新种 (鲈形目:鰕虎鱼科) [J].动物学研究,2007,28(5):539-544.
[18] 孙希福.基于形态学和分子生物学资料探讨中国沿海10种鰕虎鱼类的系统发育关系[D].青岛:中国海洋大学,2009.
[19] 陈雷,程远志,王日昕,等.2种鰕虎鱼细胞色素b基因全序列克隆与分析[J].南方水产,2010,6(5):1673-2227.
[20] AKIHITO,IWATA A,KOBAYASHI T,et al.Evolutionary aspects of gobioid fishes based upon a phylogenetic analysis of mitochondrial cytochrome b genes[J].GENE.2000,259(1-2):5-15.
[21] WANG D,DAI C X,LI Q,et al.Complete mitochondrial genome and phylogenic analysis of Rhinogobius cliffordpopei(Perciformes,Gobiidae)[J].Taylor & Francis,2019,4(2):2473-2474.
[22] 金逍逍,孙悦娜,王日昕,等.鰕虎鱼类线粒体全基因组序列结构特征分析及系统发育关系探讨[J].遗传.2013,(12):1391-1402.
[23] KIM I C,KWEON H S,Kim Y J,et al.The complete mitochondrial genome of the javeline goby Acanthogobius hasta (Perciformes,Gobiidae) and phylogenetic considerations[J].GENE.2004,336(2):147-153.
[24] AGORRETA A,MAURO D S,SCHLIEWEN U,et al.Molecular phylogenetics of Gobioidei and phylogenetic placement of European gobies[J].Molecular Phylogenetics and Evolution,2013,69:619-633.
[25] THACKER C E,SATOH T P,KATAYAMA E,et al.Molecular phylogeny of Percomorpha resolves Trichonotus as the sister lineage to Gobioidei (Teleostei:Gobiiformes) and confirms the polyphyly of Trachinoidei[J].Molecular Phylogenet Evolution,2015:172-179.
[26] THACKER C E.Phylogeny of Gobioidei and placement within Acanthomorpha,with a new classification and investigation of diversification and character evolution[J].Copeia,2009(1):93-104.
The analysis of complete mitochondrial genome sequences and gene organization of Rhinogobius sp.(Perciformes,Gobiidae)
BAO Ming1,XI Yewen1,HAI Sirao2,WANG Chenlong2,CHEN Chuangjiang2,WANG Xichun2,LI Xilei2
(1.Aquatic Technology Promotion Station of Anhui,Hefei 230601,China;2.College of Animal Science and Technology,Anhui Agricultural University,Hefei 230036,China)
Abstract:The PCR and the first-generation sequencing technology were used to obtain the complete mitochondrial genome (mtDNA) sequences of Rhinogobius sp..The mtDNA was annotated and its structure was analyzed.In present study,the circular genome was 16 511 bp in length.The G+C content was 47.7% and the A+T content was 52.3%,which showed a tendency of high AT.The mitochondrial genome encodes 13 proteins and 2 rRNAs,22tRNAs and a D-Loop region.Except for ND6,tRNA-Gln,tRNA-Tyr,tRNA-Ala,tRNA-Asn,tRNA-Cys, tRNA-Ser,tRNA-Glu,tRNA-Pro,all the other genes were encoded on the heavy strand (H strand) .Based on the whole mitochondrial genome sequence and the Cytb gene,the pthlogenetic trees were constructed separately with several species in Gobiidae.It showed that Rhinogobius sp.clustered with R. cliffordpopei,R.brunneus and R.giurinus on a branch,belonging to Rhinogobius and having the closest relationship with R. cliffordpopei.
Key words:Rhinogobius sp.; mitochondrial genome; sequence analysis; phylogenetic evolution; species identification
(收稿日期:2023-05-17)
作者簡介:鲍鸣(1979.7-),女,研究生,工程师,研究方向:水产品质量安全、渔业环境监测等。E-mail: baominglj@126.com。

