桃叶珊瑚属植物的叶绿体基因组结构特征及系统发育分析
李 娟, 童家赟, 范智超, 童 毅
( 广州中医药大学 中药学院, 广州 510006 )
桃叶珊瑚属(AucubaThunb.),隶属于丝缨花科(Garryaceae)(Angiosperm Phylogeny Group et al., 2016),为常绿小乔木或灌木。该属植物四季常青,冬季红果,为庭园绿化的优良观叶观果树种(Xiang &Boufford, 2005)。本属部分种类民间药用,主要以根、叶和果实入药,具有清热解毒、祛风除湿、活血化瘀等功效(国家中医药管理局中华本草编委会,1997;江纪武,2005;南京中医药大学,2006;艾铁民,2013)。最先从桃叶珊瑚属植物中发现的桃叶珊瑚苷(aucubin, AU)具有广泛药理活性,包括抗氧化、抗衰老、抗炎、保肝等(Zeng et al., 2020)。
然而,桃叶珊瑚属植物基础的分类修订和物种划分远未完成,这直接影响今后桃叶珊瑚属植物的化学和药理研究,也给药用桃叶珊瑚新资源的开发、推广、生产指导等带来困难。虽然桃叶珊瑚属的花、果实或全株体态使其很容易与其他类群区别,但由于可用作桃叶珊瑚属属下物种鉴定的形态特征较为复杂(如叶形、叶缘、毛被等),种间缺乏有效区分的形态分类性状且可能存在广泛的杂交和多倍化事件,导致属内物种划分存在很大困难,该属系统而全面的种间系统发育关系亟待建立(Xiang &Boufford, 2005)。
在植物进化过程中,叶绿体基因组的结构和序列相对保守,编码基因的数量、结构、组成和排列顺序基本一致,极少发生重组等变异,被广泛应用于植物的物种鉴定、系统发育和物种起源研究中(刘昶和黄林芳,2020)。目前,在美国国家生物信息中心(NCBI)上共收录了7条桃叶珊瑚属植物叶绿体基因组序列,黄媛等(2022)基于这7条叶绿体基因组的68个蛋白质编码基因对桃叶珊瑚属进行系统发育分析,结果支持了丝缨花目、丝缨花科和桃叶珊瑚属的单系性。
本研究通过浅层基因组测序技术,以已发表的青木(A.japonica)的叶绿体基因组(序列号NC_058874.1)作为参考,组装注释了花叶青木(A.japonicavar.variegata)、峨眉桃叶珊瑚(A.omeiensis)、桃叶珊瑚(A.chinensis)、密花桃叶珊瑚(A.confertiflora)、纤尾桃叶珊瑚(A.filicauda)、窄斑叶珊瑚(A.albopunctifoliavar.angustula)以及黄杨叶丝缨花(Garryabuxifolia)的叶绿体基因组。通过对桃叶珊瑚属植物的叶绿体基因组进行序列比较及系统发育分析,拟探讨以下问题:(1)6种桃叶珊瑚属植物叶绿体基因组序列有何结构特征;(2)这些序列之间有何差异;(3)桃叶珊瑚属的系统发育地位如何,6种桃叶珊瑚属植物之间有何亲缘关系。以期为今后深入研究桃叶珊瑚属植物的分类修订、系统发育、生物地理、分子鉴定和物种进化等研究提供叶绿体基因组信息的参考资料。
1 材料
桃叶珊瑚属植物的分子材料主要来源于西南和华南分布地野外采集的新鲜叶片,每个个体采集2~3片新鲜健康叶片,分别用湿纸巾和吸水纸擦去表面的污渍和水,剪碎后放入茶包袋,用硅胶进行快速干燥。所有凭证标本均保存在广州中医药大学标本室(GUCM),经该文作者分类鉴定,确定这6种桃叶珊瑚属植物分别为峨眉桃叶珊瑚、桃叶珊瑚、密花桃叶珊瑚、花叶青木、纤尾桃叶珊瑚和窄斑叶珊瑚(图1)。黄杨叶丝缨花的干燥叶片取自加州大学伯克利分校植物园(UC Botanical Garden)栽培植物(分子标本采集号SZ5417)。青木的叶绿体全基因组序列(序列号NC_058874.1)从NCBI数据库(https://www.ncbi.nlm.nih.gov/)下载,用作叶绿体基因组组装及注释的参考,所有新测序的叶绿体基因组序列以及注释信息均已上传至NCBI数据库,并获得登录号。采集信息及GenBank登录号见表1。
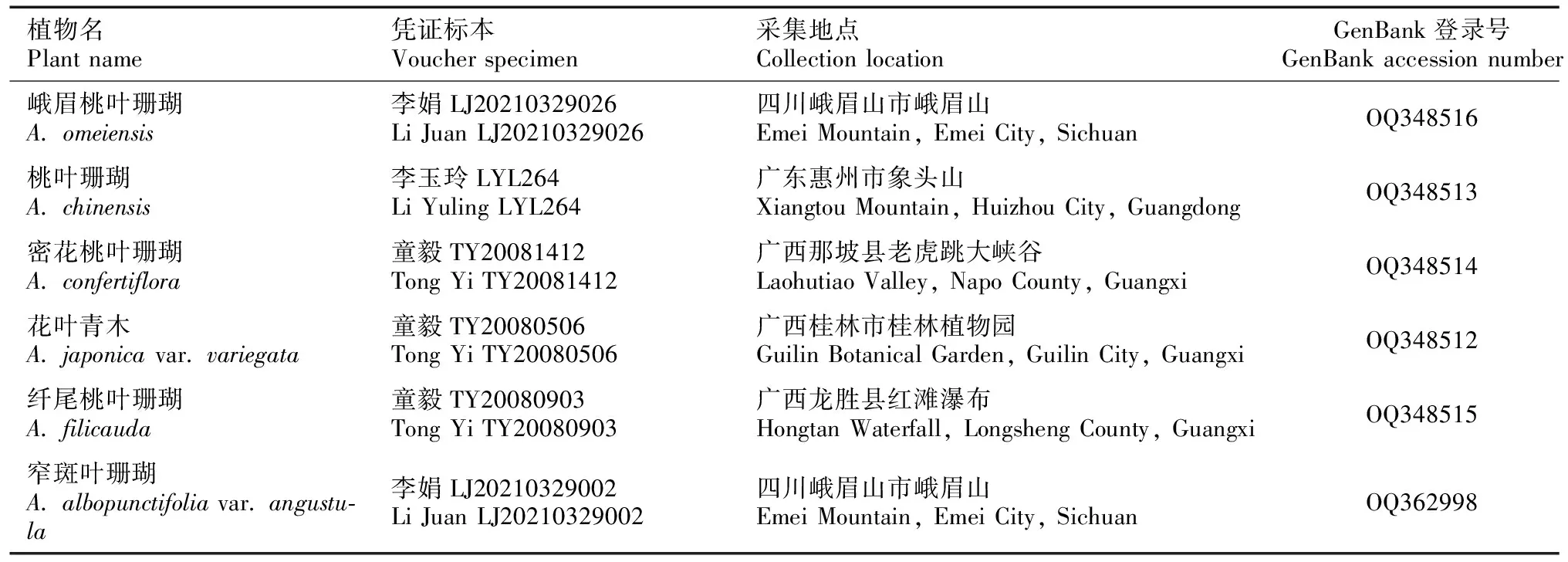
表 1 桃叶珊瑚属6种植物采集信息及GenBank登录号Table 1 Collected informations and GenBank accession numbers of six Aucuba species
2 方法
2.1 总基因组DNA提取与测序
采用改良的CTAB方法(Doyle &Doyle, 1987),从硅胶干燥的叶片中提取桃叶珊瑚属植物的总DNA;提取总DNA后,使用B-500超微量分光光度计(上海元析仪器有限公司)以及琼脂糖凝胶电泳检测所提DNA的质量和浓度,检测合格的DNA测序工作委托深圳华大基因科技有限公司使用DNBSEQ测序平台进行二代测序;最终每个样品获得3 Gb的clean data。
2.2 叶绿体全基因组的组装、注释以及物理图谱的绘制
首先,使用GetOrganelle-1.7.3.5.(Jin et al., 2020)软件进行叶绿体基因组拼接组装,k-mer值设置65、105、127,线程t设置24,其他命令使用默认参数,最终的组装命令为get_organelle_from_reads.py -1 sample_1.fastq.gz -2 sample_2.fastq.gz -F embplant pt -o output-plastome -R 10 -t 24 -k 65,105,127;然后,以青木的叶绿体全基因组序列作为参考,利用PGA-master(Qu et al., 2019)软件对组装出的叶绿体基因组进行注释,使用Geneious R 9.0.2(Basic, 2012)软件对PGA-master的注释结果进行手动校正;最后,使用在线软件OGDraw(Lohse et al., 2013)绘制叶绿体基因组物理图谱。
2.3 叶绿体基因组特征分析
使用Geneious R 9.0.2软件分别统计6种桃叶珊瑚属叶绿体全基因组序列、两个单拷贝区以及一对反向重复区的长度、各区GC含量以及基因注释结果等信息。
2.4 密码子偏好性及最优密码子分析
对6种桃叶珊瑚植物叶绿体基因组中的蛋白编码基因序列(CDS)进行筛选,剔除重复基因以及长度小于300 bp的基因,序列中碱基类型仅包含A、T、C、G,每条序列均含有起始密码子(ATG)和终止密码子(TAG、TGA和TAA),序列中间没有终止密码子。最终每个叶绿体基因组序列均获得52条符合条件的CDS,使用CodonW(Peden,2005)软件计算这些序列的同义密码子相对使用度(RSCU)以及有效密码子数(ENC),所得数据在Excel进行整理,并用TBtools(Chen et al., 2020)软件绘制热图。密码子中RSCU值大于1的被确定为高频密码子(Wang et al., 2018)。同时满足高频及高表达条件的密码子被认定为最优密码子(Sharp &Li, 1987)。高表达基因具有较低的ENC值,低表达基因具有较高的ENC值,按照ENC值的大小对每个物种的基因进行排序,选取高低两端各10%的基因分别作为低表达基因组和高表达基因组,计算高表达基因组和低表达基因组每个密码子的差值ΔRSCU,ΔRSCU大于0.08则定为高表达密码子,筛选出高达表密码子中RSCU值大于1的高频密码子,即得最优密码子(Sharp &Li, 1987)。

A. 峨眉桃叶珊瑚; B. 桃叶珊瑚; C. 密花桃叶珊瑚; D. 花叶青木; E. 纤尾桃叶珊瑚; F. 窄斑叶珊瑚。A. A. omeiensis; B. A. chinensis; C. A. confertiflora; D. A. japonica var. variegata; E. A. filicauda; F. A. albopunctifolia var. angustula.图 1 6种桃叶珊瑚属植物图Fig. 1 Pictures of six Aucuba species
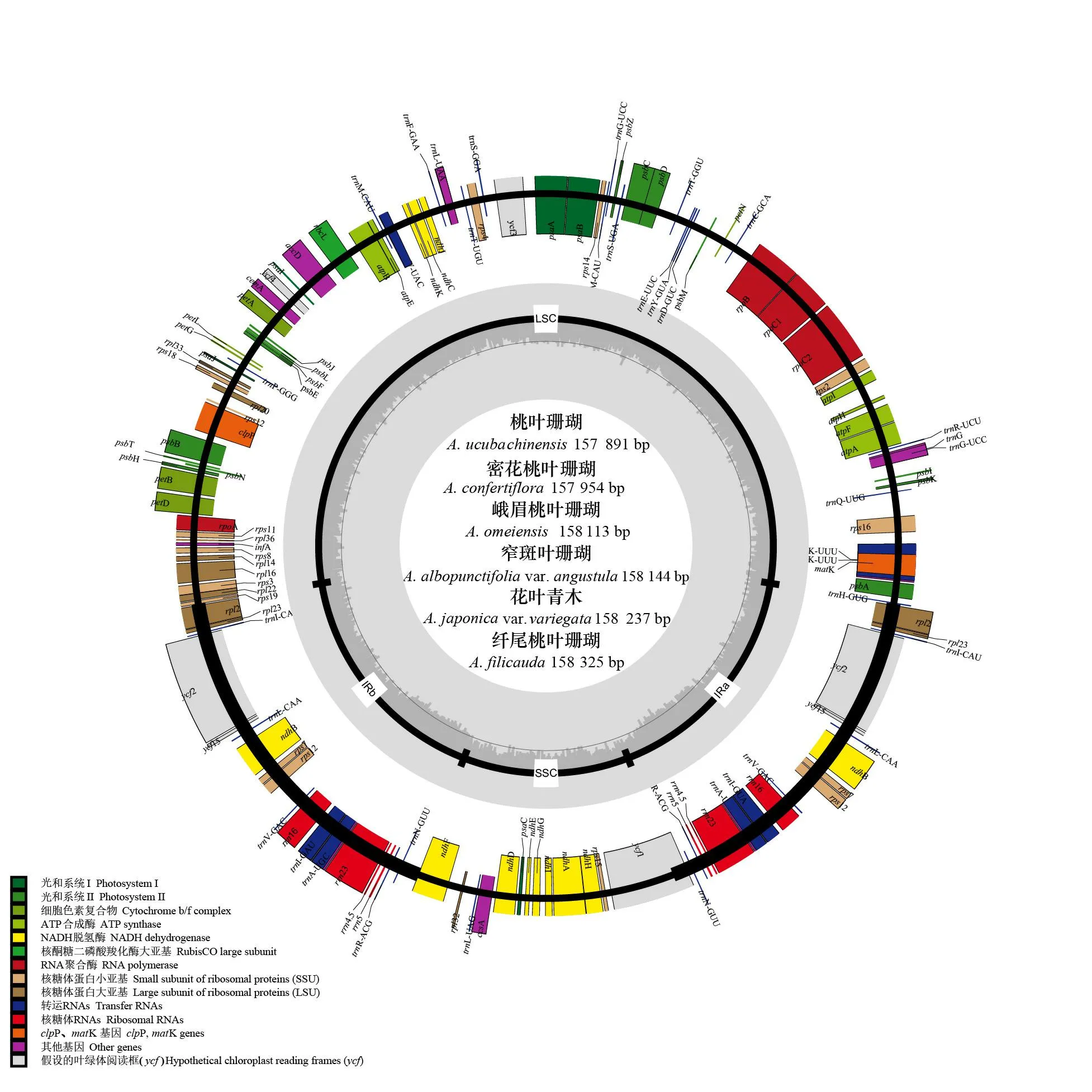
图 2 桃叶珊瑚属6种植物叶绿体基因组的物理图谱Fig. 2 Chloroplast genome map of six Aucuba species
2.5 重复序列分析
通过REPuter(Stefan et al., 2001)在线软件查找叶绿体基因组的重复序列,包括正向重复序列(forward repeats)、反向重复序列(reverse repeats)、互补重复序列(complement repeats)以及回文序列(palindromic repeats)4种重复类型,参数设定为重复碱基单元n≥30 bp,汉明距离(Hamming distance)为3。此外,使用Tandem Repeats Finder(TRF)(Benson, 1999)串联重复序列查找工具的默认参数检测串联重复序列(tandem repeats)。使用MISA(Beier et al., 2017)软件的Perl脚本检测叶绿体基因组的SSR,从单核苷酸到六核苷酸的重复次数阈值设定依次为 10、5、4、3、3,即1个碱基重复≥10次,2个碱基重复≥5次,3个碱基重复≥4次,4个、5个、6个碱基重复≥3次。
2.6 IR边界的收缩和扩张分析
使用在线软件IRscope(Ali et al., 2018)对6条序列进行IR 边界收缩和扩张的可视化分析,得到叶绿体基因组的简化示意图。
2.7 序列比较分析
以峨眉桃叶珊瑚作为参考序列,利用在线软件mVISTA(Frazer et al., 2004)对6个叶绿体基因组进行序列一致性比较分析,采用Shuffle-LAGAN运行模式,生成6个叶绿体基因组差异的可视化结果。
2.8 叶绿体基因组序列共线性分析
基因数量及基因顺序的保守性称为共线性,使用Geneious R 9.0.2软件里的Mauve插件(Darling et al., 2004),对6条序列进行共线性分析,用于可视化该属各物种基因排列顺序的一致性和基因重排与倒位现象。
2.9 核苷酸多态性分析
使用DnaSP v6.0(Rozas et al., 2017)软件对6条序列进行滑动窗口分析,检测叶绿体基因组的高变热点区及其大小、变异位点数等信息,并计算核苷酸多样性(nucleotide diversity,Pi)。将比对好的序列导入DnaSP v6.0,运行参数设定步长(step size)为200 bp,窗口长度(window length)为600 bp,筛选出Pi值大于0.01且片段长度不小于150 bp的片段作为桃叶珊瑚属植物叶绿体基因组高变区域,并根据基因注释的结果确定它们在叶绿体基因组上的位置。
2.10 系统发育分析
为了揭示桃叶珊瑚属的系统发育关系,从NCBI下载了丝缨花目下的杜仲科杜仲(序列号NC_037948),加上本次组装所得的黄杨叶丝缨花(序列号OQ348517),作为外类群,共计9条叶绿体基因组序列构建桃叶珊瑚属的系统发育树。使用Geneious R 9.0.2里的MAFFT(Rozewicki et al., 2019)插件进行多序列比较,切掉序列头尾不一样长的碱基后用于系统发育树的构建。通过最大似然法(ML)和贝叶斯推断法(BI)重建桃叶珊瑚属系统发育关系。使用IQtree(Minh et al., 2020)构建ML树;使用JModeltest(Posada, 2020)软件进行比对后核酸序列替代模型的选择,以贝叶斯信息准则(Bayesian information criterion, BIC)为标准选出最佳建树模型,使用MrBayes(Ronquist &Huelsenbeck, 2003)构建BI树。
3 结果与分析
3.1 桃叶珊瑚属6种植物叶绿体基因组结构和基本特征
6条桃叶珊瑚属叶绿体全基因组的物理图谱见图2。由图2可知,6条序列均为典型的双链环状DNA,分子序列全长为157 891~158 325 bp,其总GC含量为37.7%~37.8%。LSC区的序列长度为87 210~87 563 bp,GC含量为35.8%~35.9%,SSC区的序列长度为18 531~18 580 bp, GC含量为31.5%~31.6%,IR区的序列长度为26 067~26 143 bp,GC含量为43.0%~43.1%(表2)。6条序列均编码114个基因(不含IR区重复的基因),包括80个蛋白编码基因(CDS)、30个转运RNA(tRNA)、4个核糖体RNA(rRNA)(表3)。
3.2 密码子使用偏好
6条桃叶珊瑚属叶绿体基因组CDS的相对同义密码子使用度(RSCU)见图3。 RSCU>1,表明该密码子是偏好密码子,使用较频繁;RSCU=1表明该密码子无使用偏好性;RSCU<1表明该密码子使用频率较低。在分析时剔除终止密码子UAA、UGA和UAG,以及色氨酸的唯一密码子UGG和蛋氨酸的唯一密码子AUG, 因为这些密码子不存在偏好性。由图3可知,6条序列均有59个同义密码子,RSCU>1的高频密码子数均为29个,其中,有28个以A或U结尾,有1个以G结尾,说明桃叶珊瑚属植物叶绿体基因组的密码子偏好以A或U结尾。
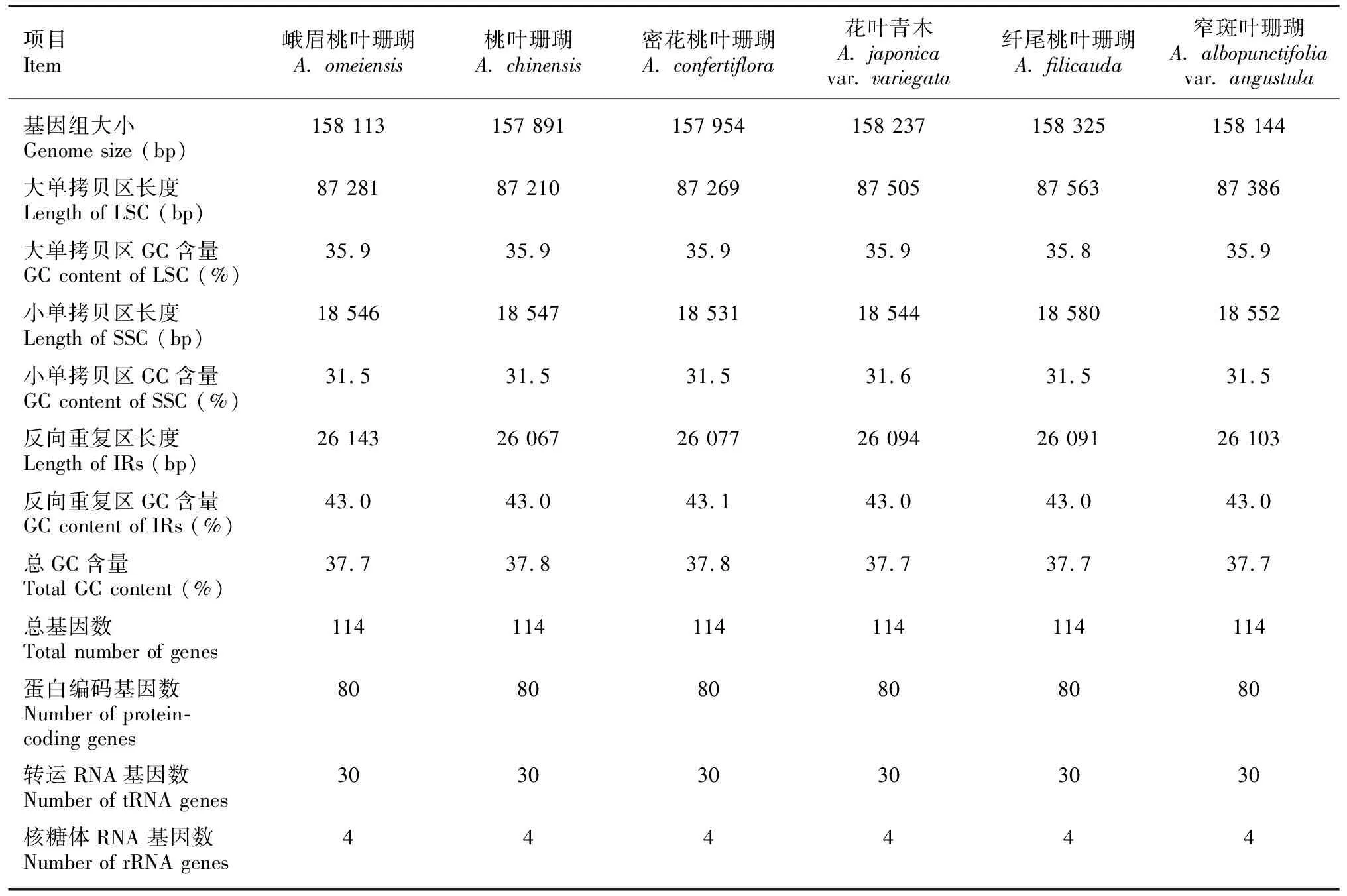
表 2 桃叶珊瑚属6种植物叶绿体基因组基本特征Table 2 Basic characteristics of chloroplast genomes in six Aucuba species
6条序列共筛选出100个最优密码子(表4),其中,除了窄斑叶珊瑚、花叶青木、纤尾桃叶珊瑚和密花桃叶珊瑚共同拥有的UUG以G结尾之外,其余最优密码子皆为A/U结尾,6条序列共有的12个最优密码子,分别为AAA、ACU、AGU、CAA、CCU、CGU、GAA、GCU、GGU、GUU、UCU、UGU。其中,峨眉桃叶珊瑚、花叶青木和密花桃叶珊瑚分别特有的最优密码子为CAU、CUU和GAU。
3.3 重复序列和SSR分析
利用软件REPuter共鉴定出6条序列共270个散在重复序列,包括133个正向重复序列、8个反向重复序列、2个互补重复序列、127个回文重复序列(表5)。6条序列中,正向重复序列最多,回文重复序列次之,反向重复序列和互补重复序列较少,仅在桃叶珊瑚和窄斑叶珊瑚中分别检测到了一条互补重复序列,其他4种植物均未检测到。
通过TRF对串联重复序列进行检测,统计情况见表6。由表6可知,6条序列中串联重复序列共133个,其中检测到串联重复序列最少的是密花桃叶珊瑚,为20个,而花叶青木最多,达25个。桃叶珊瑚属植物叶绿体基因组的串联重复序列在LSC区和IR 区的分布明显多于SSC区。
MISA软件检测结果(表7)显示,6条序列共检测到412个SSR位点,共识别出4种SSR,包括单核苷酸 SSR(367个)、二核苷酸 SSR(26个)、三核苷酸 SSR(9个)、四核苷酸 SSR(10个)。SSR的类型以单核苷酸A/T重复为主,占比所有SSR位点数的86.88%~99.41%,其中,纤尾桃叶珊瑚和花叶青木被识别出最多的SSR个数,均为73个,桃叶珊瑚被识别出最少的SSR个数,为61个。
3.4 IR/SC边界的扩张与收缩分析
桃叶珊瑚属植物叶绿体全基因组的2个IR区在LSC及SSC之间存在LSC/IRb、SSC/IRb、SSC/IRa以及LSC/IRa 4这4个边界(图4)。4个边界相对保守,但各种之间仍具较小差异。LSC/IRb的边界均位于rps19基因内部,6条序列的rps19基因均向IRb区扩张了33 bp;SSC/IRb边界均位于ndhF基因内部,6条序列的ndhF基因均向IRb区扩张了42 bp;SSC/IRa边界均位于ycf1基因内部,密花桃叶珊瑚和花叶青木的ycf1基因向IRa区扩张了1 082 bp,纤尾桃叶珊瑚、窄斑叶珊瑚和峨眉桃叶珊瑚的ycf1基因向IRa区扩张了1 079 bp,桃叶珊瑚的ycf1基因向IRa区扩张了1 061 bp;LSC/IRa边界位于trnH附近,峨眉桃叶珊瑚与密花桃叶珊瑚的LSC/IRa边界与trnH的距离为14 bp,其余4种的LSC/IRa边界与trnH的距离均为7 bp。

颜色越深表示该密码子的RSCU值越高;高频密码子(RSCU>1)已用红色字体标出。The darker color in the figure indicates the higher RSCU value of the codon; high-frequency codons (RSCU>1) marked in red.图 3 桃叶珊瑚属6种植物叶绿体基因组RSCU热图Fig. 3 RSCU heat map of chloroplast genomes of six Aucuba species

表 3 桃叶珊瑚属6种植物叶绿体基因组的基因组成Table 3 Chloroplast genome compositions of six Aucuba species
3.5 序列变异分析
为了比较桃叶珊瑚属内物种间叶绿体基因组序列的差异,以峨眉桃叶珊瑚的叶绿体基因组序列为参考,将该6条序列进行全局对比,mVISTA图的结果(图5)显示了桃叶珊瑚属6条序列的高度相似性,单拷贝区比反向重复区更保守,编码区比非编码区(non-coding sequences, NCS)相对更保守。位于LSC区的trnC-GCA-petN以及位于IR区的rps7-trnV-GAC均为基因间隔区,在这些区域相似度较低,存在不同程度的变异。此外,rRNA基因和tRNA基因区域的一致度最高,序列最为保守。LSC和SSC区序列相比于IR区有更多的变异,表明IR区在进化过程中更为保守。
3.6 叶绿体基因组共线性分析
叶绿体基因组的多重基因组比对法检测出6条序列之间只有一个局部共线区(locally collinear blocks, LCBs)(图6),所有基因的种类、数量和排列顺序在属内均高度一致,叶绿体基因组完全共线,没有重排重组现象发生,进一步说明该属叶绿体基因组具有较高的保守性。
3.7 序列变异热点分析
滑窗分析结果(图7)显示,6条序列的核苷酸多样性(Pi)在0~0.018 89之间,平均Pi值为0.003 51,表明6种植物的叶绿体基因组高度相似,序列非常保守;其中IR区的核苷酸多样性(Pi=0.000 85)明显低于LSC区(Pi=0.004 47)和SSC(Pi=0.006 14)区,SSC显示出最高的核苷酸多样性,而IR区在整个叶绿体基因组中更为保守。共筛选出10个高变片段(表8),包括位于LSC区的rps16、rps16-trnQ-UUG、rpoB-trnC-GCA、petN-psbM、trnC-GCA-petN、psbM-trnD-GUC和accD-psaI 7个片段,以及位于SSC区的ndhE、ndhE-ndhG和ycf1 3个片段,其中,ycf1的分化程度最高。在这些高变区域中,仅有rps16、ndhE和ycf1 3个片段是编码序列,其他高变片段均位于基因间隔区(intergenic spacer,IGS),这些高变片段可作为桃叶珊瑚属植物物种鉴定的条形码候选片段。
目前,叶绿体基因组中被广泛应用于植物鉴定的3条通用的条形码为matK、rbcL和trnH-GUG-psbA,但是在本研究中,这3个片段在桃叶珊瑚属叶绿体基因组序列中均显示了较低的Pi值,变异程度不理想,从Pi值的角度分析不适合做分子标记。
3.8 系统发育分析
根据BIC方法用JModeltest筛选出核苷酸的最佳替代模型均为GTR+G。分别使用软件MrBayes和IQtree构建BI树和ML树。系统发育分析结果(图8)表明,基于叶绿体全基因组构建的BI树和ML树展现出了完全一致的拓扑结构,桃叶珊瑚属所有物种形成一个支持率高的单系(BS=100%,PP=1),与丝缨花属构成姊妹类群,共同组成丝缨花科。桃叶珊瑚属属内分化为两个进化支,峨眉桃叶珊瑚、密花桃叶珊瑚以及桃叶珊瑚共同组成进化支Ⅰ(Clade Ⅰ, BS=100%, PP=1),花叶青木、青木、纤尾桃叶珊瑚以及窄斑叶珊瑚共同组成进化支Ⅱ(Clade Ⅱ, BS=100%, PP=1)。
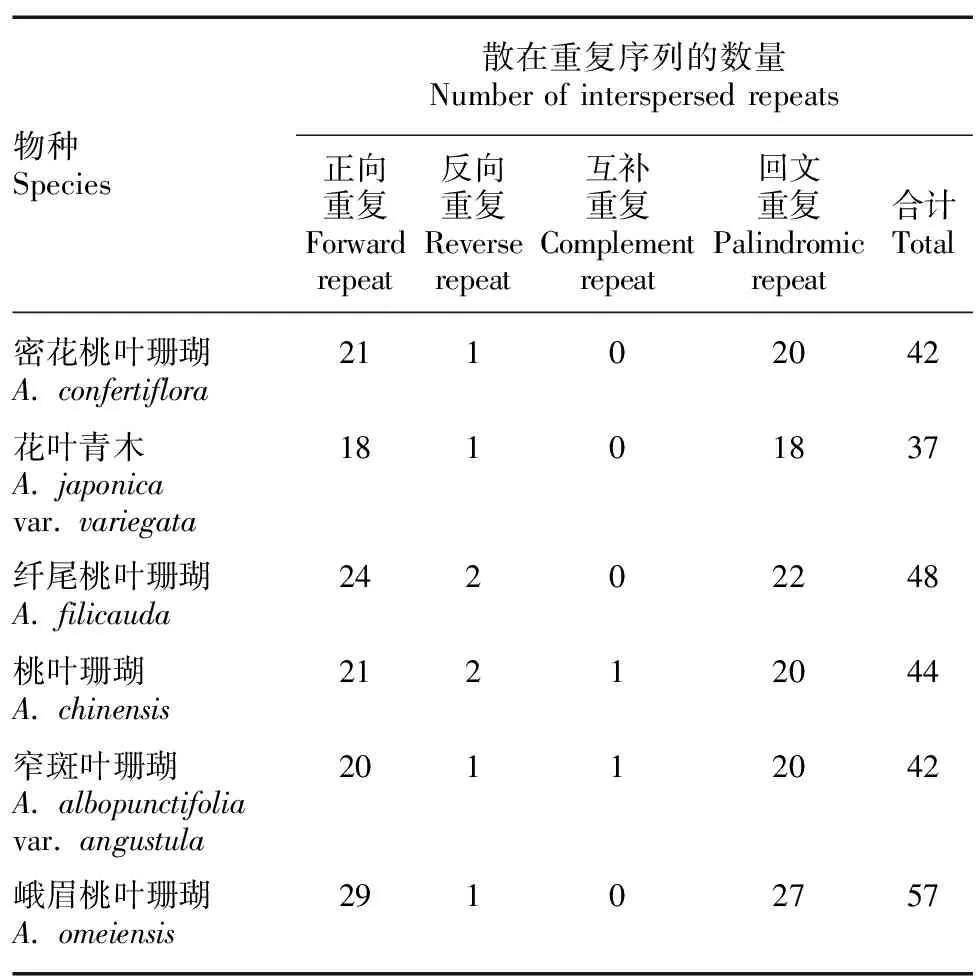
表 5 桃叶珊瑚属6种植物叶绿体基因组中散在重复序列的类型和数量Table 5 Types and numbers of interspersed repeats in chloroplast genomes of six Aucuba species

表 6 桃叶珊瑚属6种植物叶绿体基因组中串联重复序列统计Table 6 Total counts of tandem repeats in chloroplast genomes of six Aucuba species
4 讨论与结论
4.1 叶绿体基因组的结构特征
本研究组装注释了桃叶珊瑚属6种植物的叶绿体基因组,并进行了序列比较分析,结果显示6条序列高度相似,无论是基因组结构及大小,还是基因的含量及组成,皆是相对保守。桃叶珊瑚属叶绿体基因组拥有典型的环状四分体结构,6条序列全长157 891~158 325 bp,6条序列均编码114个基因,包括80个蛋白质编码基因、30个tRNA基因以及4个rRNA基因,总GC量为37.7%~37.8%。LSC、SSC、IR区的GC含量分别为35.8%~35.9%、31.5%~31.6%、43.0%~43.1%,与大多数被子植物一样,桃叶珊瑚属叶绿体基因组的IR区相较于LSC区和SSC区都更为稳定,有着最高的GC含量。
密码子对于遗传信息的正确表达至关重要,密码子的使用偏好在不同物种甚至某一物种内的不同基因都具有特异性,这是由于基因与物种长期进化过程中选择、突变以及漂变等因素的综合作用,亲缘关系越近或者生存环境越相似的物种越有可能采用相似的密码子选择策略(Romero, et al., 2000; Xu, et al., 2011),通过比较密码子偏好性差异可以推断基因是否受到不同程度的翻译选择,对于探讨桃叶珊瑚属物种的进化模式有着重要的作用。6条序列均有59个同义密码子,RSCU>1的高频密码子数均为29个,其中,除了UUG以G结尾之外,其余28个高频密码子均以A/U结尾,这表明桃叶珊瑚属叶绿体基因组的密码子偏好以A/U结尾;在6条序列中共筛选出100个最优密码子,包含12个共有最优密码子,分别为AAA、ACU、AGU、CAA、CCU、CGU、GAA、GCU、GGU、GUU、UCU、UGU,均是以A/U结尾,尤其是以U结尾较多,这与双子叶植物偏好使用A/U结尾密码子的研究结果一致(Kawabe &Miyashita, 2003)。
叶绿体重复序列的存在和丰富程度可增加生物种群遗传多样性,并且与多种系统发育信号有关(Adeyemo et al., 2021),SSR和重复序列在叶绿体基因组中广泛存在,具有丰富的多态性位点,易复制、遗传信息含量高等优点,在研究物种遗传多样性、亲缘关系、品种鉴定和标记辅助育种中具有较高的可靠性(Adeyemo et al., 2021;贾守宁等,2023)。6条叶绿体基因组序列共检测到412个SSR位点,SSR的类型以单核苷酸A/T重复为主且主要位于LSC区; 6条序列共检测到270条散在重复序列以及133条串联重复序列。该属叶绿体基因组上的重复序列的功能还需进一步深入研究。

图 4 桃叶珊瑚属6种植物叶绿体基因组LSC、IR和SSC边界比较示意图Fig. 4 Comparison of the LSC, IR and SSC boundaries in chloroplast genomes of six Aucuba species

表 7 桃叶珊瑚属6种植物叶绿体基因组SSR在不同区域的数量分布Table 7 Number and distribution of SSR in chloroplast genomes of six Aucuba species
4.2 叶绿体基因组的结构比较分析
比较基因组学分析结果表明,桃叶珊瑚属叶绿体基因组序列具有高度的相似性,单拷贝区比反向重复区更为保守,编码区比非编码区更为保守;叶绿体基因组IR/SC边界分布的基因类型完全相同;基因组基因结构和排列顺序整体上相似,所有基因的种类、数量和排列顺序均高度一致,6条序列完全共线,没有重排重组现象发生;根据课题组前期研究(未发表)发现,目前通用的DNA条形码片段(psbA-trnH,rbcL,matK,ITS,ITS2)及其片段组合的变异程度不理想,对桃叶珊瑚属植物的物种分辨率不高,不适合作为该属植物的通用条形码,本文从桃叶珊瑚属叶绿体基因组LSC区和SSC区共筛选出10个变异率较高,长度适中的高变片段,这些高变区可作为桃叶珊瑚属物种鉴定的潜在条形码,可以进一步结合双亲遗传的核基因片段联合分析,为桃叶珊瑚属植物的物种鉴定,杂交起源,多倍体物种的形成和系统进化分析提供可靠的分子标记。
4.3 系统发育分析
系统发育分析结果表明,桃叶珊瑚属为一个支持率高的单系(BS=100%, PP=1),与丝缨花属亲缘关系较近,共同构成丝缨花科;丝缨花科与杜仲科关系较近,这与分类系统APG Ⅳ和Huang等(2022)的研究结果一致。
桃叶珊瑚属属内分化为两个支持率高的进化支,峨眉桃叶珊瑚、密花桃叶珊瑚以及桃叶珊瑚共同组成进化支Ⅰ,共同的特征为乔木、花绿色;花叶青木、青木、纤尾桃叶珊瑚以及窄斑叶珊瑚共同组成进化支Ⅱ,共同的特征为灌木、花红色。这两组特征是否分别稳定地成为两个进化支的共衍特征,还需该属更多种类和居群的支持。在进化支Ⅱ内部,花叶青木和青木聚为一支,亲缘关系较近。通过回溯花叶青木与青木的叶绿体全基因组序列,发现这两条叶绿体基因组序列,无论是基因数量还是基因位置,都保持高度一致,仅在基因间隔区存在4个碱基位点的变异。这也在一定程度上印证了Ranney的观点——青木中表达的斑点叶性状很可能是一个具有多重等位基因和数量遗传的核基因,而不是严格的母系遗传,可能有多个具有不同遗传模式的基因可以产生桃叶珊瑚属植物带斑叶的性状(Ranney et al.,2018)。因此,“花叶”表现型,在桃叶珊瑚属类群中并不是一个稳定可靠的性状,基于“花叶”特征建立的变种或新种划分需格外谨慎。
向秋云在FOC中将峨眉桃叶珊瑚归并入原变种桃叶珊瑚(Xiang &Boufford, 2005),本研究系统发育分析结果表明峨眉桃叶珊瑚与密花桃叶珊瑚关系更近,二者再与桃叶珊瑚构成姐妹群。Ranney使用流式细胞仪对桃叶珊瑚属植物的相对基因组大小进行了研究,发现峨眉桃叶珊瑚的1Cx值 (10.6 pg)显著高于包括桃叶珊瑚 (7.37 pg)在内的其他种类(6.8~7.5 pg),表明峨眉桃叶珊瑚经历了比其他类群更显著的倍性不变的基因组扩张(即基因组扩张非染色体加倍引起)(Ranney et al., 2018);结合标本与野外宏观形态性状分析,三者构成了物种关系复杂的复合种群。因此,关于峨眉桃叶珊瑚的物种划分问题,还需要综合更多形态特征和细胞学证据,以及核基因数据的分析,特别是共线性单拷贝核基因,方能得到较为清楚的解析。此外,一方面,桃叶珊瑚属植物很可能存在广泛的杂交和多倍化事件;另一方面,传统用以种间鉴别的叶形态特征在本属中变异极大,导致该属植物种间区别特征存在过渡变异和交叉类型,缺乏稳定可靠的区别特征,诸多种类难以准确辨别。并且,目前我们对于该属植物形态特征仍未有足够的把握,因此,对于物种鉴定不确切和物种划分明显不合理的种类,本研究未予囊括。对桃叶珊瑚属植物系统全面的属下分类系统的构建和属下种间关系的解决有赖于更广泛的采样,更多的性状证据(尤其是染色体数据)和更丰富的具足够变异位点的单拷贝核基因和叶绿体基因组的联合分析建树,来共同厘清桃叶珊瑚属的属下分类问题,为以后深入研究桃叶珊瑚属植物的药用和园艺用途等奠定分类学基础。
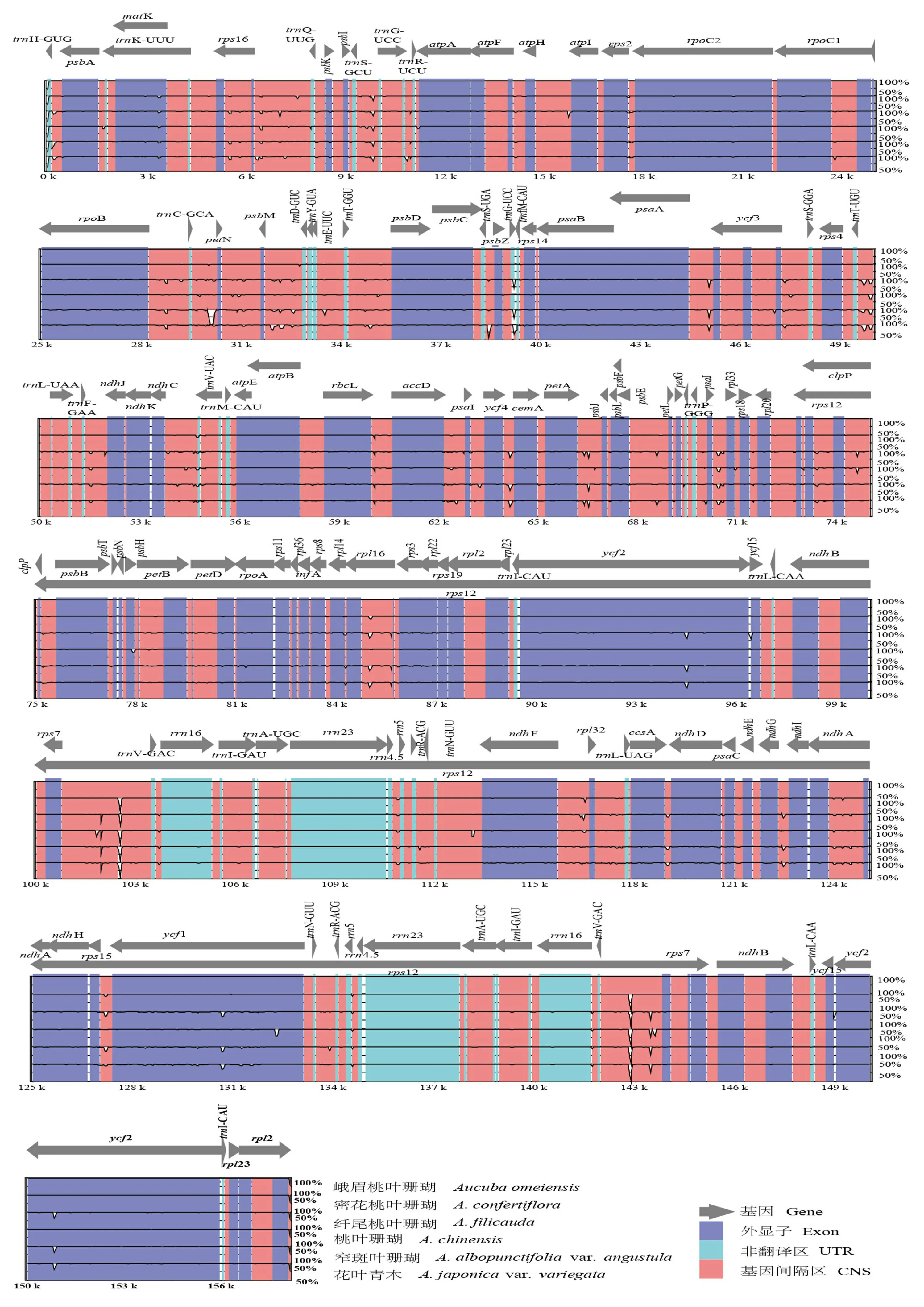
灰色箭头表示基因的方向; 深蓝色表示编码区; 红色区域表示非编码序列; 浅蓝色区域表示tRNA或rRNA; Y轴表示从50%到100%序列之间的相似性。Gray arrows indicate gene orientation; dark blue indicates coding region; red regions indicate non-coding sequences; light-blue regions indicate tRNA or rRNA; Y axis indicates the similarity among sequences, ranging from 50% to 100%.图 5 桃叶珊瑚属6种植物叶绿体基因组序列可视化比对分析Fig. 5 Plastome comparison of six Aucuba species

图 6 桃叶珊瑚属6种植物叶绿体全基因组Mauve共线性分析Fig. 6 Mauve genome alignments of the whole chloroplast genomes of six Aucuba species
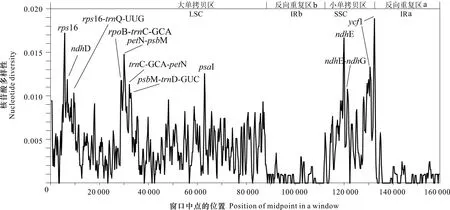
窗口长度600 bp, 步长200 bp。10个高变区(高Pi 值)均被标记。Window lengths is 600 bp, step size is 200 bp. Ten regions with the highest Pi values are marked out.图 7 基于滑窗分析检测桃叶珊瑚属植物叶绿体全基因组的高变区Fig. 7 Hypervariable regions within the chloroplast genomes of six Aucuba species using a sliding window analysis
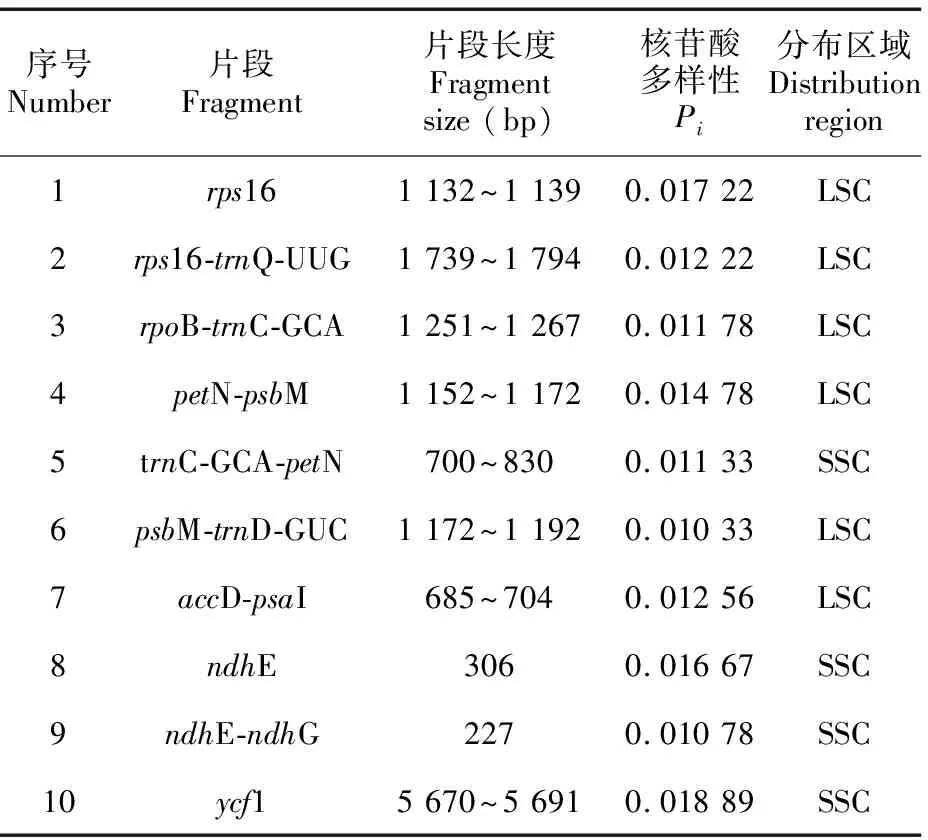
表 8 桃叶珊瑚属6种植物叶绿体基因组10个高变区信息Table 8 Ten hypervariable regions information of chloroplast genomes of six Aucuba species
致谢特别感谢河北师范大学石硕老师在黄杨叶丝缨花分子样品收集工作中给予的大力帮助。

