基于多尺度特征和反向注意力的肝脏肿瘤自动分割方法
张 瑞,唐乔湛,李斯卉,宋江玲
(西北大学 医学大数据研究中心,陕西 西安 710127)
肝脏是腹腔内最大的实质性器官,具有代谢、解毒、免疫防御等生理功能。肝脏肿瘤是发生在肝脏的异常组织增生,可分为肝脏良性肿瘤和肝脏恶性肿瘤(也称为肝癌)。据统计,我国肝癌发病率占所有癌症的9%,居癌症发病谱第4位;肝癌死亡率占所有癌症的14%,居癌症死亡谱第2位[1]。文献[2]通过对10 966例肝癌患者的临床资料进行回顾性分析,发现早期肝癌患者经治疗,其5年生存率高达62.5%~77.2%,而中、晚期肝癌患者的5年生存率仅23.8%。因此,尽早对肝脏肿瘤进行良恶性诊断对于及时诊断患者肝脏情况、维护肝脏健康、提高肝癌生存率具有重要意义[3]。而肝脏肿瘤的准确分割是实现这一目标的重要前提。
肝脏肿瘤分割是指在相应医学影像中对肝脏肿瘤区域进行精确定位。计算机断层扫描(computed tomography, CT)是一种能够提供高分辨率图像的影像学检查,具有扫描速度快、对人体伤害小等优点,是临床中主要用于肝脏肿瘤分割的检查手段[4]。传统的肝脏肿瘤分割主要是通过专业的放射科医生在CT上进行手动勾勒。然而,随着患者数量的增加,且每位患者的CT包含几十张甚至几百张切片图像,逐个切片进行人工分割与标记极其耗时费力,会大幅增加医生的工作压力。同时,人工标记容易掺杂医生的主观意识,不同水平的医生对肝脏病变的认识和判断往往不同,容易出现误诊和漏诊现象。随着科学技术的发展,利用计算机视觉和人工智能进行医学图像分割的方法大量涌现,这在很大程度上减轻了医生的工作量,降低了误诊和漏诊现象。同时也一定程度缓解了偏远地区医疗资源不平衡的问题,实现医疗资源下沉。
目前,肝脏肿瘤分割算法主要分为3类:基于传统图像分割的算法、基于机器学习的分割算法和基于深度学习的分割算法。其中,传统图像分割算法主要包括阈值分割法[5]、水平集分割法[6]、区域生长分割法[7]等。基于机器学习的分割算法主要是将特征工程和分类器相结合,具体地,通过对医学图像进行特征提取(如图像的轮廓信息、图像的强度分布、图像的纹理特征、图像的频域特征等),进而结合分类器(如K-均值算法[8]、SVM[9]、AdaBoost[10]等)来完成前景像素和背景像素的分类,以实现肝脏肿瘤分割。上述第1类方法在分割阶段需要进行人工交互来完成算法的初始化设置,且算法的分割效果受初始化设置的影响较大;第2类方法依赖于人工设计的特征,其特征表示能力有限,导致分割方法泛化性能差,分割结果不够精准。
随着深度学习技术的发展,其强大的特征自主挖掘能力,使其在医学影像分割领域得到了广泛应用[11]。其中,2015年Ronneberger等人提出的U-Net网络[12],由于其结构简洁,尤其针对少量数据仍具有较好的分割性能,近年来在医学影像分割任务中受到极大关注。很多基于U-Net及其变体的模型与方法被提出用于实现肝脏肿瘤分割,包括H-DenseUNet[13]、U-Net++[14]、RIU-Net[15]、MS-FANet[16]等。
尽管基于深度学习的肝脏肿瘤自动分割方法得到了一定的发展,但仍存在局限性,具体体现在两方面:一是不同患者的肝脏肿瘤在大小、位置、形态等方面存在显著差异,易导致误检、漏检现象,已有方法通过金字塔结构、空洞卷积等提取不同尺度的特征,并将上述多尺度特征直接进行拼接或相加操作,忽略了大目标的细节信息和小目标的全局信息[17-20];二是肝脏肿瘤的边缘模糊导致边缘分割精度低,已有方法通过引入额外的边缘分支、设计边缘损失函数以提高网络的边缘分割能力,但上述方法需要引入边缘先验知识,且边缘分割效果受边缘先验知识的影响较大[21-25]。图1 (a)~(c)是不同患者的CT切片中肝脏及肝脏肿瘤分布示意图,其中蓝色线是肝脏区域的边缘,红色线是肝脏肿瘤区域的边缘。从图1可以发现,(a)中患者的肝脏肿瘤大小各异、位置分散,少数肝脏肿瘤的边缘模糊;(b)中患者的肝脏肿瘤边缘与肝脏边缘重合;(c)中患者的肝脏肿瘤极小。

图1 肝脏及肝脏肿瘤分布示意图Fig.1 Liver and liver tumor distribution diagram
基于上述分析,本文在U-Net网络框架下,提出了一种融合多尺度特征提取模块和基于深度监督的反向注意力模块的肝脏肿瘤自动分割模型(multi-scale reverse attention u-net, MRA U-Net),其主要架构为编码器-解码器结构。具体地,在编码器上的多尺度特征提取模块使网络能够根据分割目标大小自适应地选择合适的尺度特征,从而缓解肝脏肿瘤大小、位置、形态存在显著性差异的问题;在编码器和解码器之间的基于深度监督的反向注意力模块能够引导网络关注编码器特征中有关分割目标的边缘特征,进一步为解码器特征补充细节特征,实现对肝脏肿瘤边缘的精细分割;此外,针对医学图像分割中的类别不平衡问题,本文设计了一种混合加权损失,使网络在训练过程中关注难分的像素点,以减少误诊、漏诊现象。
1 方法
1.1 MRA U-Net整体结构
本文提出的肝脏肿瘤分割网络MRA U-Net是一个对称的编码器-解码器结构,其框架结构如图2所示。在编码器部分,本文设计了多尺度特征提取模块,以对CT图像的不同尺度特征进行提取,得到包含大目标细节信息和小目标全局信息的多尺度特征。具体地,首先,通过多尺度卷积层将输入特征沿通道维度划分成特征子集,并采用卷积操作和类残差连接提取不同特征子集上的多尺度特征。随后,采用通道注意力模块对上述特征进行融合,以适应不同大小的分割目标。解码器部分采用U-Net网络的解码器。而在编码器和解码器之间的跳跃连接上,本文设计了基于深度监督的反向注意力模块,通过从解码器上产生反向注意力特征图,以引导网络关注编码器特征中有关分割目标的边缘特征,进而为解码器特征补充分割目标的细节特征。最后,通过对基于深度监督的反向注意力模块输出的融合边缘信息的分割结果进行深度监督,使网络更好地学习分割目标的边缘,实现网络对肝脏肿瘤的精准定位。接下来,本文将详细介绍多尺度特征提取模块和基于深度监督的反向注意力模块的构造方式。
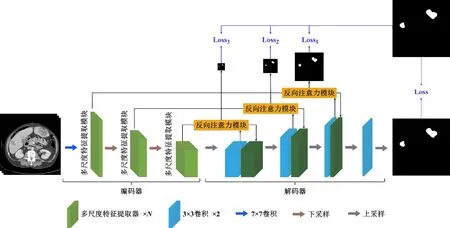
图2 MRA U-Net示意图Fig.2 MRA U-Net schematic
1.2 多尺度特征提取模块
肝脏肿瘤的大小、位置、形态等存在显著差异,大部分多尺度特征提取方法在扩大感受野的同时忽略了大目标的细节信息和小目标的全局信息。为了提高网络对不同大小目标的分割能力,本文在编码器上设计了多尺度特征提取模块,如图3所示,该模块共包括N个多尺度特征提取器,每个特征提取器是由6个卷积层和1个注意力层构成的残差结构。
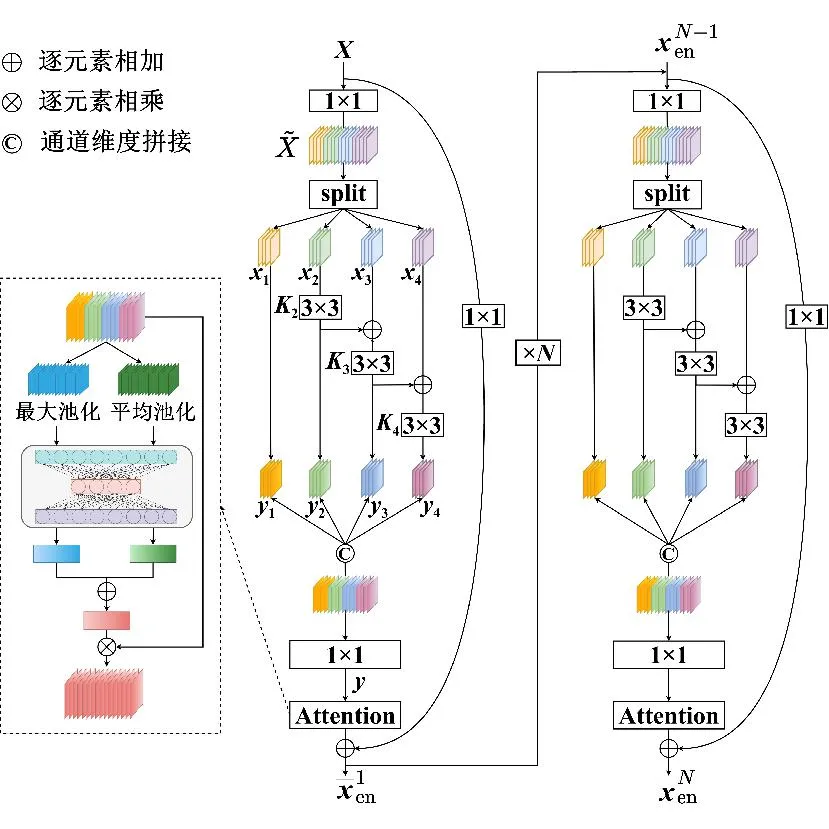
图3 多尺度特征提取模块示意图Fig.3 Multi-scale convolution diagram
每个特征提取器进行特征提取的过程主要分为以下4个步骤。
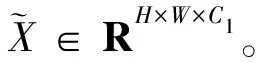
xh=split(Conv1×1(X))(h=1,2,…,s)
(1)
式中:split表示划分特征子集的操作;Conv1×1表示1×1卷积操作。
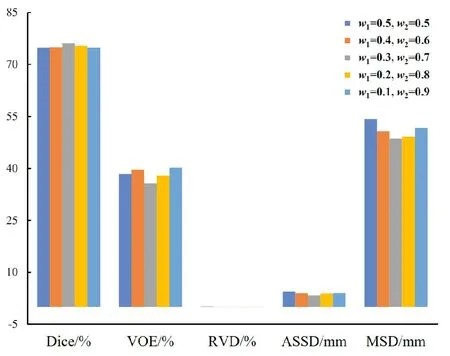
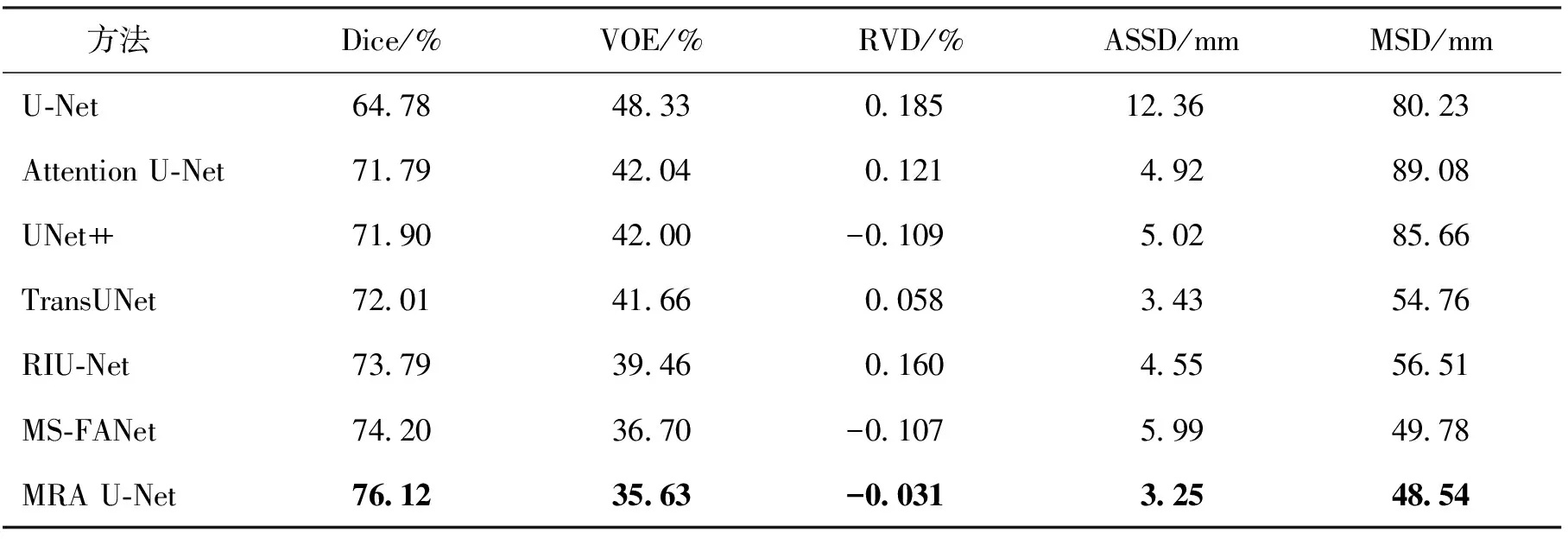
2)特征子集x1直接作为输出y1,特征子集x2经过3×3卷积操作K2后得到输出特征y2,其余每个特征子集xh(2 (2) 由于特征经过3×3卷积操作后会得到一个更大的感受野,因此yh具有比yh-1更大的感受野,即每个yh代表一种尺度下的图像特征。 3)将所有yh∈RH×W×C1/s沿通道维度进行拼接后经过1×1卷积操作得到多尺度特征y∈RH×W×C2, y=Conv1×1(concat(y1,y2,…,ys)) (3) 进而采用通道注意力模块(如图3中的子图所示)学习特征y每个通道的注意力分数,并据此得到加权多尺度特征Att(y), Att(y)=y×σ(MLP(MaxPool(y))+ MLP(AvgPool(y))) (4) 式中:concat表示特征拼接操作;MaxPool和AvgPool分别表示最大池化和平均池化;MLP表示共享的全连接层;σ表示Sigmoid激活函数。 (5) 式中:ReLU表示ReLU激活函数。 在传统的U-Net编码器-解码器结构中,解码器特征中包含高级的语义信息,使网络能够在图像中分辨出分割目标,而编码器特征中包含丰富的边缘、纹理等信息,这些特征通过跳跃连接的方式输入到解码器中,从而为解码器特征补充分割目标的位置信息,实现对分割目标的定位。但这种简单的跳跃连接方式使编码器无法反向利用解码器中的高级语义信息,从而导致编码器难以利用分割目标的整体信息对其局部细节进行微调,使得定位不够精确。因此,本文设计了一个基于深度监督的反向注意力模块(见图4),利用解码器特征产生反向注意力特征图,进而引导网络关注编码器特征中有关分割目标的边缘特征,以完成对于病灶区域局部细节的定位。 图4 基于深度监督的反向注意力模块示意图Fig.4 Schematic diagram of reverse attention module based on deep supervision (6) 式中:Deconv表示反卷积操作;σ表示Sigmoid激活函数。 (7) 式中:Conv3×3表示3×3卷积操作;ReLU表示ReLU激活函数。 (8) 式中:Conv1×1表示1×1卷积操作。 (9) 式中:aj(j=1,2,…,M)表示深度监督不同阶段损失的权重。 针对肝脏肿瘤分割中常常出现的类别不平衡问题,本文设计了一种混合加权损失函数,使网络在训练过程中关注难分的像素点,以减少误诊、漏诊现象。 Loss=w1lossce+w2losstversky (10) 式中:wi(i=1,2)表示各损失函数的权重;lossce表示交叉熵损失(cross-entropy loss, CE)[26];losstversky表示Tversky损失[27]。具体表示为 (11) losstversky(p,g)= (12) 结合深度监督部分的损失,本文在网络训练过程中使用的总损失为 Losstotal=Loss+Lossdeep (13) 本文的实验验证采用MICCAI2017 LiTS挑战赛数据集[28],该数据集包含从7个临床站点收集的201名患者的CT扫描图,其中训练集包含131名患者的CT扫描图(标签可见),在线测试集包含70名患者的CT扫描图(标签不可见)。训练集中CT的标注是由具有3年肿瘤成像经验的放射科医生使用ITK-SNAP软件完成,包含肝脏肿瘤、肝脏、背景3类标签。由于不同临床站点使用的扫描设备跟扫描协议不同,所有CT的层内分辨率范围为0.56~1.0 mm,层厚范围为0.45~6.0 mm,轴向切片的大小为512×512,切片数范围为42~1 026。考虑到该挑战赛不再接收在线测试集上分割结果的提交,本文仅使用标签可见的131名患者的CT扫描图作为全部数据集。 本文对于数据集的预处理包含重采样、调窗、归一化等步骤。具体地,首先统计所有CT的空间采样率,得到空间采样率的中位数为[0.76, 0.76, 1.0],并将所有CT重采样至[0.76, 0.76, 1.0];其次,通过调窗操作将所有CT的强度值截断到[-200,200]HU范围内,以增强CT的对比度,减少无关组织器官对肝脏肿瘤分割结果的影响;最后,对所有CT进行归一化,并按照轴位进行切片,将相邻的3张CT切片和中间CT切片对应的标签分别作为输入图像和真实标签,使网络学习相邻切片间的信息。数据集中包含肝脏的切片有19 156张,包含肿瘤的切片有7 190张,数据预处理流程如图5所示。 图5 CT预处理流程图Fig.5 CT preprocessing flow chart 本文实验使用Adam进行优化, 初始学习率为0.000 1, 学习率的更新公式为 式中:i表示当前迭代次数;ti表示全部迭代次数。数据集的批量大小设置为8,训练轮数为 150 轮,本文采用翻转、旋转、对比度增强等方式对数据集进行数据增强, 并将图像大小调整至256×256,混合损失函数中w1为0.3,w2为0.7,Tversky损失函数中α为0.1,β为0.9。本文所有表格中显示的实验结果均为5折交叉验证结果的平均值, 所有实验均在Linux Ubuntu 18.04.4 LTS平台上借助显存为12 GB的NVIDIA GeForce GTX 2080Ti、Python 3.7以及Pytorch 1.7.1 框架完成的。 2.3.1 评价指标 为了验证所提分割网络的性能,本文采用的模型性能评价指标有Dice系数(dice coefficient, Dice)[29]、体积重叠误差(volumetric overlap error, VOE)[30]、相对体积差(relative volume difference, RVD)[31]、平均对称表面距离(average symmetric surface distance, ASSD)[31]、最大对称表面距离(maximum symmetric surface distance, MSD)[32]。若网络的分割结果和真实标签分别为A和B,则各评价指标的计算公式如下。 1)Dice用于计算网络的分割结果和真实标签之间的相似性,其值越大,分割效果越好,其数学表达式为 (14) 2)VOE用于计算网络的分割结果和真实标签的体素之间的体积重叠误差,值越接近0,分割效果越好,其公式为 (15) 3)RVD计算网络的分割结果和真实标签的体素之间的相对体积重叠误差,值越接近0,分割效果越好,定义为 (16) 4)ASSD计算网络的分割结果边界S(A)上的体素到真实标签边界S(B)以及真实标签边界S(B)上的体素到网络的分割结果边界S(A)的所有距离的平均值,值越接近0,分割效果越好,表示为 ASSD= (17) 式中:d表示欧几里得距离。 5)MSD将ASSD中计算距离平均值替换为计算最大距离,值越接近0,分割效果越好,具体公式为 S(A))) (18) 2.3.2 消融实验 本文通过设计消融实验,以验证多尺度特征提取模块、基于深度监督的反向注意力模块以及混合损失函数的有效性。表1展示了消融实验结果,其中最优结果使用黑色加粗字体进行标记。 表1 不同模块和不同损失在LiTS数据集上的消融实验Tab.1 Ablation experiments with different modules and different losses on LiTS dataset 多尺度特征提取模块的有效性。 通过比较表1中“基线模型(U-Net)”和“基线模型+多尺度特征提取模块”的分割性能,发现多尺度特征提取模块的增加将Dice提高了7.9%,将VOE、ASSD、MSD分别降低了7.28%、7.67 mm、29.77 mm,同时使RVD更接近0,提升了网络的分割性能。一方面,多尺度特征提取模块从不同的特征子集上提取了丰富的多尺度特征,能够同时包含大目标的细节信息和小目标的全局信息;另一方面,采用通道注意力模块对多尺度特征进行通道加权,使网络能够根据分割目标的大小自适应地选择合适的多尺度特征。上述结果说明本文所设计的多尺度特征提取模块是有效的。 基于深度监督的反向注意力模块的有效性。在肝脏肿瘤大小、位置、形态存在显著差异的情况下,仅通过Dice反映的网络分割结果和真实标签的相似程度无法很好地度量分割效果,需要通过距离度量来定量分析网络对于分割目标边缘的分割能力。通过比较表1中“基线模型(U-Net)”和“基线模型+基于深度监督的反向注意力模块”的分割性能,发现基于深度监督的反向注意力模块的加入使ASSD、MSD降低了7.38 mm、9.88 mm,同时使Dice增加了6.71%,从而说明基于深度监督的反向注意力模块的嵌入对于肝脏肿瘤边缘分割精度的提升是有意义的。 损失函数的有效性。为了验证本文所设计的混合加权损失函数的有效性,采用不同的损失函数设置对MRA U-Net进行优化并评估网络的分割性能。具体地,本文设置的情况有“CE+Tversky”“CE+Dice”“CE”“Tversky”,通过对比可以发现,采用“CE+Tversky”对网络进行优化时,Dice高达76.12%,验证了混合加权损失函数的有效性。其中,Tversky损失通过对假阳性样本和假阴性样本加权,降低网络的误诊率和漏诊率,进而提升网络的分割性能。 2.3.3 超参数分析 为了进一步探讨本文使用的混合损失函数中交叉熵损失和Tversky损失的权重设置对模型效果的影响,本文采用不同的损失权重设置进行实验以对比网络的分割性能,实验结果如图6中的直方图所示,可以发现,当w1=0.3,w2=0.7时,网络的分割性能最佳。此时Tversky损失作为混合损失函数的主要部分,使网络在前期训练时重点关注难分的像素,随着Tversky损失的下降,交叉熵损失引导网络学习分割结果中全体像素的分布与真实标签中全体像素分布的差异,提升网络的整体分割性能。 图6 MRA U-Net在不同混合损失函数权重下的分割性能Fig.6 Segmentation performance of MRA U-Net under different hybrid loss function weights 2.3.4 对比实验 为了验证本文所提方法的优越性,将本文所提方法和U-Net、Attention U-Net、UNet++、TransUNet、RIU-Net、MS-FANet的分割性能进行了比较。如表2所示,本文所提方法相较次优方法在Dice上提高了1.92%,同时将VOE、ASSD、MSD降低了1.07%、2.74 mm、1.24 mm,并且本文所提方法的RVD更接近0。根据上述量化结果可知,本文所提方法在分割结果和真实标签的相似度、分割目标的边缘分割效果等方面表现良好,同时反映出多尺度特征提取模块和基于深度监督的反向注意力模块相结合的有效性。 表2 不同方法在LiTS数据集上的分割性能Tab.2 Segmentation performance of different methods on LiTS dataset 2.3.5 分割结果可视化 2.3.4小节从定量的角度分析了本文所提方法的有效性,本小节通过对不同方法的分割结果进行可视化,以从定性的角度分析本文所提方法与其他方法的性能差异。图7展示了采用不同方法对不同患者CT切片进行分割的可视化结果。根据图7中第1行的可视化结果能够发现,U-Net着重关注与对比度相关的特征,其将与肝脏肿瘤灰度值相似的组织误分割为肝脏肿瘤,同时Attention U-Net也存在上述问题,但其误分割现象比U-Net少。从图7中第3行的可视化结果可以看出,本文所提的MRA U-Net对于小目标肝脏肿瘤的分割能力较强,能够有效避免漏诊现象的出现。通过图7中第2、4、5行的可视化结果能够发现,本文所提的MRA U-Net分割的肝脏肿瘤边缘比其他方法的分割结果更精确,验证了基于深度监督的反向注意力模块对网络边缘分割能力的提升。 图7 不同方法的分割结果可视化Fig.7 Visualization of segmentation results by different methods 本文提出了一种融合多尺度特征提取模块和基于深度监督的反向注意力模块的肝脏肿瘤自动分割模型。首先,设计了一种新的多尺度特征提取模块,并据此构造编码器用以提取CT图像的多尺度特征,通过通道注意力机制使网络能够根据分割目标的大小自适应地选择合适的多尺度特征;其次,在编码器-解码器的跳跃连接上嵌入基于深度监督的反向注意力模块,通过从解码器特征中产生反向注意力,以引导网络关注编码器特征中有关分割目标的边缘特征,为解码器特征补充分割目标的细节信息并采用深度监督缓解梯度消失、促进网络的训练。此外,本文还设计了一种混合损失函数,以解决图像分割中存在的类别不平衡问题。经数值实验证明,本文所提方法在肝脏肿瘤的自动分割中性能表现良好。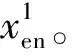
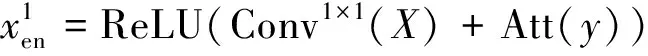

1.3 基于深度监督的反向注意力模块

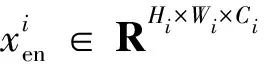



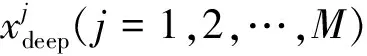

1.4 损失函数

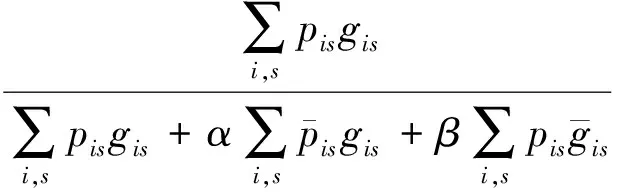
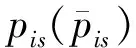
2 数值实验
2.1 数据集及预处理

2.2 实验环境及参数设置
2.3 实验结果与分析
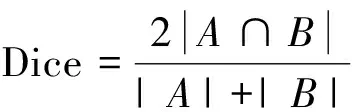





3 结语

