基于ISSR分子标记技术的土沉香遗传多样性分析
于 彤,申文辉,覃日亮,韦文龙,李俊晓,谭长强,滕维超,曹艳云,郝海坤
(1.广西壮族自治区林业科学研究院,广西南宁 530002;2.广西大学,广西南宁 530004;3.广西优良用材林资源培育重点实验室,广西南宁 530002;4.柳江区冲马岭林场,广西柳州 545100;5.浦北县绿化工作站,广西浦北 535300;6.齐齐哈尔大学,黑龙江齐齐哈尔 161000)
土沉香(Aquilariasinensis)是瑞香科(Thymelaeaceae)沉香属多年生常绿乔木,又名牙香树、沉香和白木香,是我国特有的珍贵树种,分布于我国广东、海南、广西和福建等地[1]。土沉香以含树脂的心材入药,具有较好的医疗价值和经济效益[2]。由于无节制地砍伐,野生土沉香资源受到严重破坏,现已被列为国家珍稀濒危三级保护植物及国家二级重点保护野生植物[3]。
近年来,分子标记技术的迅速发展为物种的遗传变异、亲缘关系及种质资源多样性等研究提供支持。简单重复间序列(Inter-Simple Sequence Repeat,ISSR)是一种基于简单序列重复(Simple Sequence Repeat,SSR)特征开发的分子标记技术,其特点在于能高效地扩增简单序列重复区间,产生高度多态性,具有很好的稳定性和操作简便性。目前该技术在植物的品种鉴定[4]、遗传作图[5]和遗传多样性[6]等研究中得到了广泛应用。
土沉香的研究主要集中在组织培养[7-8]和植物生理[9]等方面,对土沉香种质资源遗传多样性的研究较少。本研究采用ISSR分子标记技术,分析广西3个居群、云南1个居群和海南9个居群共13个居群147 个土沉香家系的遗传分化,探讨居群间的亲缘关系及家系间的遗传差异,为我国野生土沉香资源的保护和合理利用提供理论依据。
1 材料与方法
1.1 试验材料
2017年6月,在广西、海南和云南选取树龄10~20年、生长健壮且具有代表性的土沉香单株;2017年7月,在广西壮族自治区林业科学研究院苗圃(108°35′E,22°92′N)内嫁接。试验材料包括来自广西马山(MS)、浦北(PB)和北流(BL)3 个居群的40个家系,海南儋州(DZ)、琼中(QZ)、琼海(QH)、万宁(WN)、屯昌(TC)、澄迈(CM)、海口(HK)、乐东(LD)和陵水(LS)9个居群的92个家系及云南(YN)1个居群的15个家系,共147个家系。
1.2 试验方法
1.2.1 DNA提取及检测
采用改良的十六烷基三甲基溴化铵(CTAB)法[10]提取土沉香的基因组DNA;采用1.5%琼脂糖凝胶电泳(电压120 V,电泳45 min,在GelDoc XR+ 凝胶成像仪上成像),检测DNA 样品质量;采用紫外分光光度计(UV1800PC,上海精若科学仪器有限公司)检测DNA 质量浓度;取A260/A280值大于1.8 的DNA样品稀释至40 mg/L,于-20 ℃冰箱保存备用。
1.2.2 PCR引物筛选、ISSR-PCR扩增及检测
参考加拿大哥伦比亚大学公布的100 条ISSR引物序列设计[11],该100 条引物均由上海生工生物工程有限公司合成。每个居群随机选取3个家系的DNA 样品进行引物筛选,最终筛选出条带清晰、反应稳定的引物进行PCR 扩增。PCR 反应总体系为20 μL,含有40 mg/L 的DNA 样品1.5 μL、20 μmol/L的引物1.5 μL、10×PCR Buffer 5 μL、2.5 mmol/L 的Mg2+2 μL、2.5 mmol/L 的dNTPs 1.25 μL、5 U/μL 的Taq 酶0.25 μL 和8.5 μL ddH2O。PCR 扩增反应在Biometra TProfessional PCR 扩增仪上进行。反应程序为:94 ℃预变性5 min;94 ℃变性30 s,55~60 ℃退火30 s,72 ℃延伸1 min,38 个循环;72 ℃延伸8 min,4 ℃结束反应保存。PCR 产物在2%琼脂糖凝胶电泳(电压130 V,电泳50 min)。电泳后,在Gel-Doc XR + 凝胶成像仪上拍照保存结果。每条引物均重复扩增和电泳3 次,选取清晰并稳定的条带作为最终结果。
1.3 数据处理
采用Gel-pro 32 软件与人工相结合的方法读取条带,根据条带有无的方法进行统计,“有”条带赋值为1,“无”条带赋值为0,得到并导出0、1 矩阵[12]。采用POPGEN 32 软件对遗传数据进行多样性统计,计算遗传特征值,即有效等位基因数(Ne)、等位基因数(Na)、多态性位点百分率(Percentage of Polymorphic Loci,PPL)、居群总基因多样性(Ht)、居群内基因多样性(Hs)、Nei's 遗传距离(D)、Nei's 基因多样性指数(H)、香农信息指数(I)、遗传分化系数(Gst)和基因流(Nm)等。采用NTSYS 2.10E 软件构建13 个土沉香居群的UPGMA 系统树状图,采用SPSS 软件中的Mantel检验分析地理距离与遗传距离的相关性。
2 结果与分析
2.1 ISSR-PCR扩增结果
从100 条ISSR 引物序列筛选出条带重复性好、稳定且清晰的34 条引物用于土沉香147 个家系的ISSR-PCR 扩增。34条ISSR引物共获得扩增位点数291 个,其中多态性位点数230 个,平均PPL 为78.97%(表1)。平均每条引物扩增位点数为8.56个,平均每条引物多态性位点数为6.76 个。PPL 达100%的引物有7条,分别为引物807、823、835、844、848、880和884。引物886扩增位点数最多(19个)。
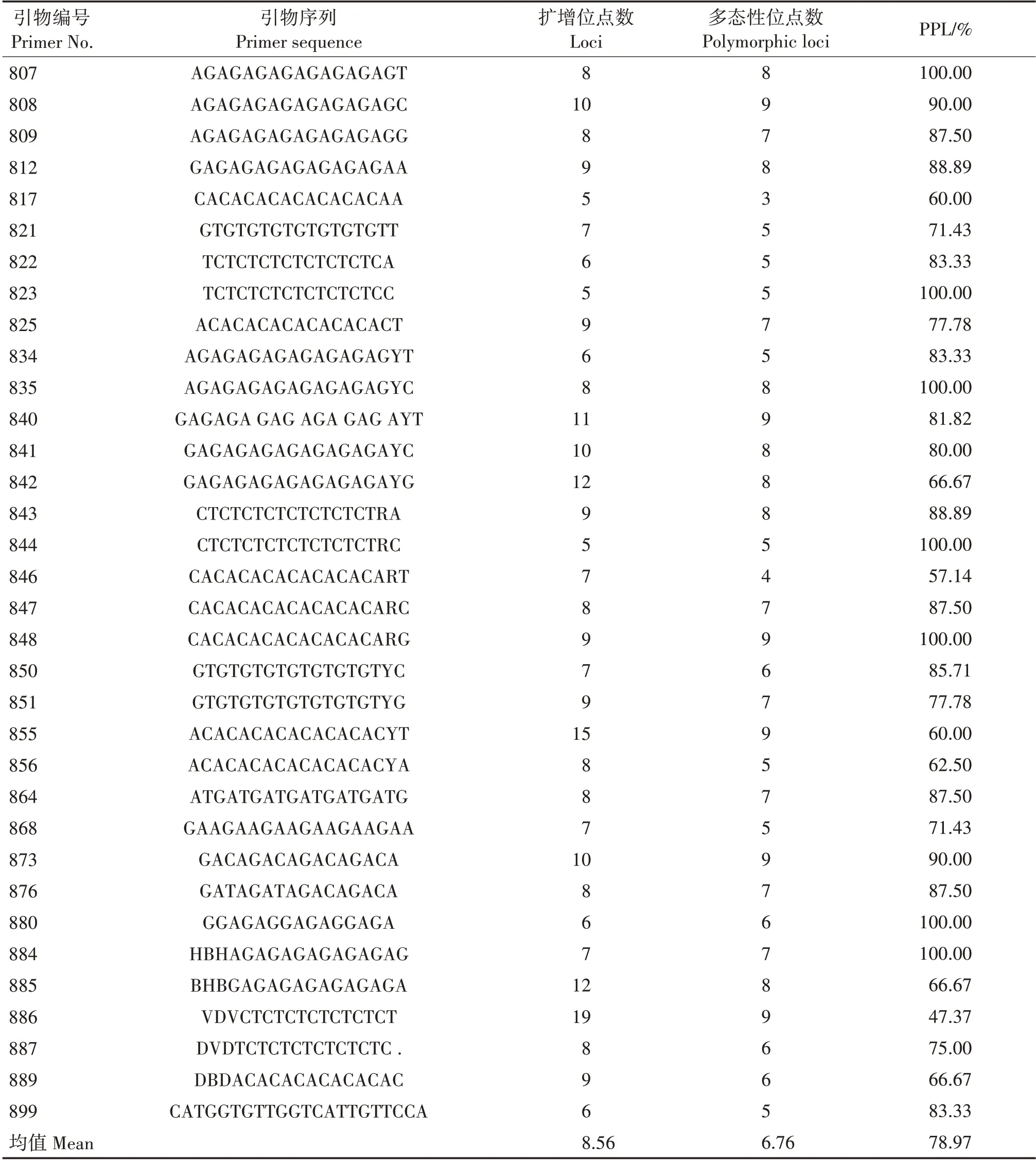
表1 34条ISSR引物在土沉香147个家系中的多态性检测Tab.1 Polymorphism test of 34 ISSR primers in 147 families of A.sinensis
2.2 遗传多样性分析
13个土沉香居群的Na为1.430 4~1.734 8,居群水平均值为1.591 6,家系水平均值为1.982 6;Ne为1.309 2~1.470 2,居群水平均值为1.399 1,家系水平均值为1.518 3;H为0.171 9~0.270 6,居群水平均值为0.226 1,家系水平均值为0.310 4;I为0.250 5~0.400 4,居群水平均值为0.332 0,家系水平均值为0.472 8(表2)。各指数最高值均出现在广西马山居群,最低值均出现在海南陵水居群。
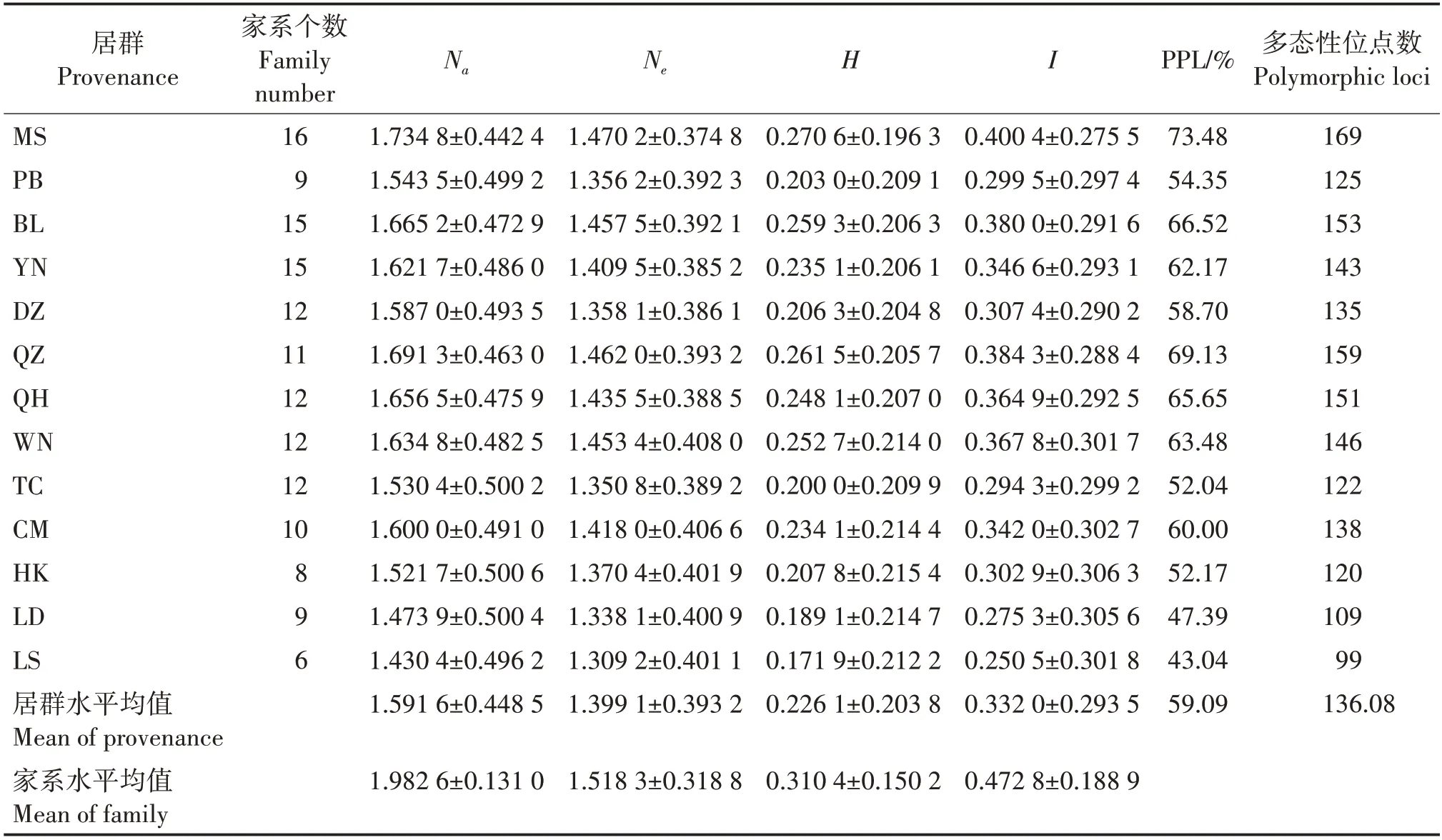
表2 13个土沉香居群遗传多样性指数Tab.2 Genetic diversity indices of 13 populations of A.sinensis
13个土沉香居群的PPL为43.04%~73.48%,居群水平均值为59.09%;多态性位点数为99~169,居群水平均值为136.08;PPL 表现为广西马山>海南琼中>广西北流>海南琼海>海南万宁>云南>海南澄迈>海南儋州>广西浦北>海南海口>海南屯昌>海南乐东>海南陵水。
2.3 遗传分化分析
13 个土沉香群居的Ht、Hs、Gst和Nm均值分别为0.307 6、0.226 1、0.265 0和1.387 1(表3)。

表3 13 个土沉香居群遗传分化分析Tab.3 Genetic differentiation analysis on 13 populations of A.sinensis
2.4 13个土沉香居群的遗传距离和聚类分析
13个土沉香居群的遗传距离为0.050 2~0.172 8,其中海南屯昌与海南澄迈居群间的遗传距离最小,海南乐东与广西浦北居群间的遗传距离最大;13个土沉香居群的遗传一致度为0.841 3~0.951 0,其中海南乐东与广西浦北居群间的遗传一致度最小,海南屯昌与海南澄迈居群间的遗传一致度最大(表4)。

表4 13 个土沉香居群的遗传距离(D)与遗传一致度(I)Tab.4 Genetic distances(D)and genetic consistencies(I)of 13 populations of A.sinensis
以遗传距离0.9 为阈值,13 个土沉香居群可被划分为3类,第一类为广西马山、广西浦北和云南居群;第二类为广西北流、海南儋州、海南琼中、海南琼海和海南万宁居群;第三类为海南屯昌、海南澄迈、海南海口、海南乐东和海南陵水居群(图1)。
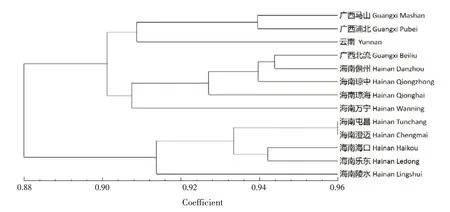
图1 基于Nei's遗传距离的13个土沉香居群UPGMA聚类图Fig.1 UPGMA clustering diagram of 13 populations of A.sinensis based on Nei's genetic distance
2.5 13 个土沉香居群遗传距离与地理距离相关性分析
13 个土沉香居群间的遗传距离与地理距离不相关(图2)。Pearson 为0.031,表示遗传距离与地理距离的解释率为3.1%,说明土沉香的遗传多样性不可以用地理距离解释。
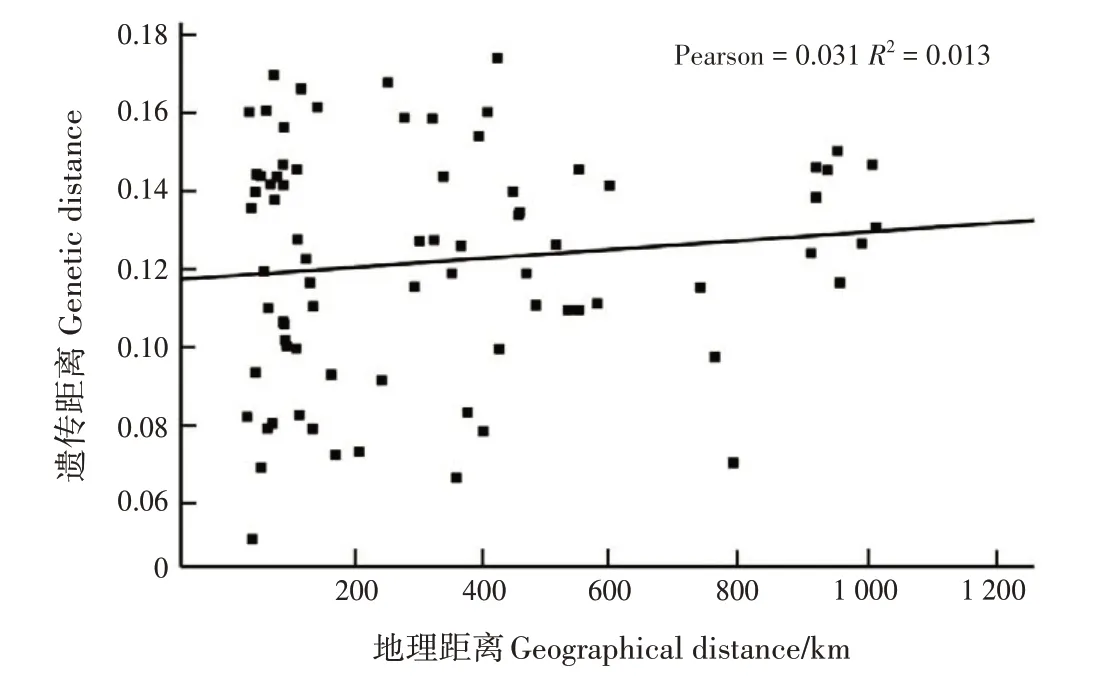
图2 13个土沉香居群遗传距离与地理距离相关性散点图Fig.2 Scatter plots of correlations among genetic distances and geographical distances of 13 populations of A.sinensis
3 讨论与结论
本研究首次采用ISSR分子标记技术对13 个土沉香居群147个家系的遗传多样性及亲缘关系进行分析,筛选得到34 条ISSR 引物,共获得扩增位点数291 个,其中多态性位点数230 个,PPL 均值为78.97%,表现出丰富的多态性。H居群水平均值为0.226 1,家系水平均值为0.310 4;I居群水平均值为0.332 0,家系水平均值为0.472 8。说明土沉香居群具有较高的遗传多样性,与黄久香等[13]研究土沉香遗传多样性的结论较一致。广西马山的土沉香居群遗传多样性丰富,各指数值均最高,海南陵水的土沉香居群遗传多样性最低。
遗传分化系数是用于衡量植物群体间分化程度的重要指标,为遗传分化的研究提供有力的解释和说明[14]。0 ≤Gst≤0.05时,群体间遗传分化水平较低;0.05<Gst≤0.15 时,群体间遗传分化水平为中度;0.15<Gst≤0.25 时,群体间遗传分化水平较高;0.25 基因流是指基因在同种植物群体内或群体之间的运动,有助于提高植物群体的遗传多样性水平,防止种群分化[16]。当Nm>1 时,基因流动性较强[17]。本研究中,Nm均值为1.387 1,说明13 个土沉香居群间存在较强程度的基因流动。 遗传距离可用来衡量物种之间或同一物种群体之间遗传差异(基因组差异)的程度。本研究中,海南屯昌与海南澄迈居群间的遗传距离最小、遗传一致度最大,海南乐东与广西浦北居群间的遗传距离最大、遗传一致度最小。13 个土沉香居群可被划分为3 类,第一类为广西马山、广西浦北和云南居群;第二类为广西北流、海南儋州、海南琼中、海南琼海和海南万宁居群;第三类为海南屯昌、海南澄迈、海南海口、海南乐东和海南陵水居群。土沉香居群间的遗传距离与地理距离不相关,土沉香的遗传多样性不可以用地理距离解释。这可能是因为3个地区种源的土沉香是由1 个原生种源扩张,由于扩张到采样点的时间较短,繁殖代数较少,还未形成地理距离差异。 植物种源的亲缘关系对于种群的遗传多样性保护具有重要意义。本研究中,基于34 条ISSR 引物对13 个土沉香居群147 个家系进行分子层面分析,发现13 个土沉香居群147 个家系具有较高的遗传多样性和极高的遗传分化水平,可为土沉香种质资源保护、开发与利用和优种栽培等领域提供理论依据。 利益冲突:所有作者声明无利益冲突。 作者贡献声明:于彤负责研究计划制定、数据收集与分析和论文撰写与修改;申文辉负责研究计划制定;覃日亮、韦文龙负责试验调查;李俊晓负责数据收集与分析;谭长强负责研究计划制定、数据收集与分析和论文修改;滕维超负责论文撰写;曹艳云、郝海坤负责文献检索。

