CRISPR/Cas9基因编辑技术在植物抗病性改良中的应用综述
朱宗财 王志军 高能 武冬梅
摘要:植物病害是影响作物生长的重要因素之一,对世界粮食安全构成很大的威胁,培育优良的抗病品种成为最优策略。CRISPR/Cas9基因编辑技术自问世以来备受关注,因该系统简单、高效、稳定的特点,逐渐成为分子育种领域重要的技术手段。本综述简单回顾了CRISPR/Cas9基因编辑技术的技术原理和在植物中的應用情况,系统总结了该技术在植物抗真菌、细菌和病毒方面的应用。还列举了可用于提高植物抗病性的基因位点以及真菌、细菌和病毒等病原物的致病相关基因位点编辑应用情况,探讨了该技术主要的优势和不足,以及未来应用的前景和挑战,以期为今后研究提供参考和借鉴。
关键词:CRISPR/Cas9;基因编辑技术;植物抗病性;抗病性改良;抗病育种
中图分类号:S432.1 文献标志码:A
文章编号:1002-1302(2024)03-0001-11
植物病害是影响作物生长的重要因素之一,因植物病害引起的作物减产高达20%左右,对世界粮食安全构成很大的威胁[1]。虽然化学药剂的使用一定程度上可以降低植物病害带来的危害,但由于化学药剂的特性,对人类健康和自然环境带来潜在的风险[2],因而,严重依赖化学药剂的农业生产方式是不可持续的,迫切需要绿色环保、有效可靠的方式为农业生产保驾护航[3]。近年来,随着一些生物农药的成功研制,为植物病害的防治带来了新的手段,但存在防治效果缓慢、易受环境因素的制约与干扰等问题,所以目前植物病害的防治仍然以化学防治为主,生物农药仅占全球作物保护市场的5%[4]。因此,培育优良的抗病品种成为最优策略[5]。
不管是传统的作物育种还是近年来迅速发展的分子育种,本质上两者都是促进基因间的交流,但分子育种的优势在于能够定向的设计和操作,实现从传统的经验育种到定向高效的精准育种的跃升[6]。CRISPR/Cas9基因编辑技术因能够精准地对生物体基因组特定目标基因进行修饰,而逐渐成为分子育种领域的重要技术手段,广泛应用于农业、医疗、工业等领域[7-8]。
1 CRISPR/Cas9系统
CRISPR/Cas系统广泛存在于原核生物基因中,是细菌和古细菌为应对病毒和质粒的不断攻击而进化出的RNA介导的适应性防御系统[9]。CRISPR/Cas系统分为3类,分别是Ⅰ型、Ⅱ型、Ⅲ型,其中Ⅰ型和Ⅲ型CRISPR系统利用Cas蛋白与crRNAs的复合物介导对靶标DNA识别与剪切,Ⅱ型CRISPR系统则是通过RNA引导的内切酶Cas9以及2种非编码crRNA和tracrRNA识别并剪切靶标DNA[10]。2012年,Jinek等发现一种双链RNA,即tracrRNA-crRNA,它能够指导Cas9蛋白在特定位点剪切双链DNA,并且将tracrRNA-crRNA二元复合体改造为单链RNA嵌合体后,仍能够指导Cas9蛋白[11]。2013年Cong等首次利用CRISPR/Cas9系统实现了基因敲除[12]。随着研究的不断深入,CRISPR/Cas9系统不仅仅用于基因敲除,而且在内源基因表达的调控、染色体位点活细胞标记、单链RNA编辑、高通量基因筛选等方面也得到了应用,并且因该系统简单、高效、稳定的特点,迅速吸引了众多科学家在动植物育种、药物筛选等相关领域展开研究[13]。
1.1 CRISPR/Cas9系统的技术原理
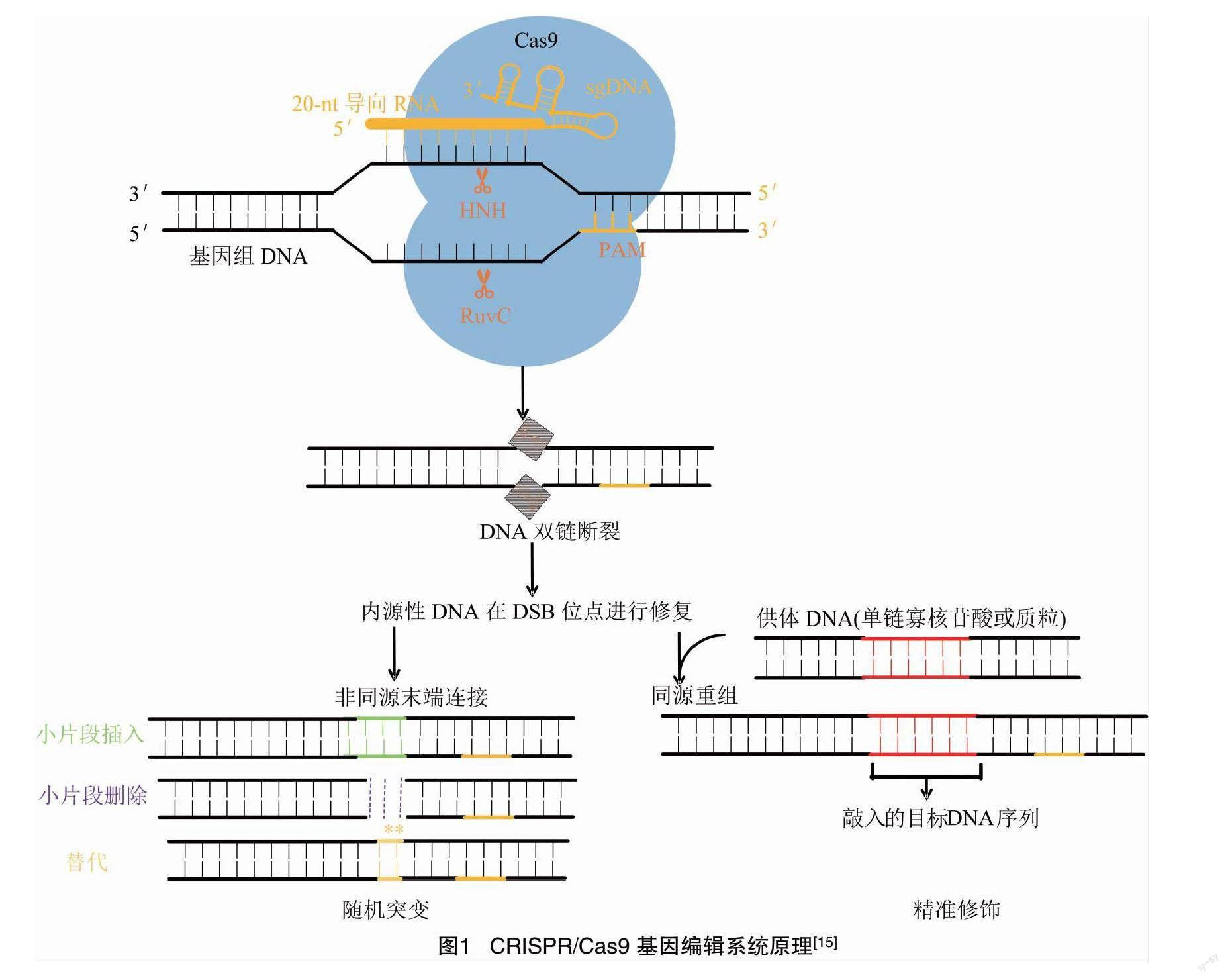
CRISPR/Cas9系统由单向导RNA(single guide RNA,简称sgRNA)和核酸内切酶Cas9组成[11],其中crRNA和tracrRNA融合形成sgRNA,crRNA则是RNA酶Ⅲ加工CRISPR转录成的一条长的RNA分子(Precursor crRNAs,pre-crRNAs)后形成的,tracrRNA 则是与crRNA互补的一段序列[14]。Cas9包含HNH和RuvC核酸酶结构域,其中HNH域是单个域,而RuvC域由3个子域组成,分别是RuvC-Ⅰ、RuvC-Ⅱ和RuvC-Ⅲ,RuvC-Ⅰ在Cas9氨基端,RuvC-Ⅱ和RuvC-Ⅲ位于HNH两侧[15]。Cas9在sgRNA的引导下到达靶标基因位点解开DNA双链,使crRNA与互补链杂交,另一条链保持游离的单链状态,然后由Cas9蛋白的HNH活性位点剪切crRNA的互补DNA 链(目标链),RuvC活性位点剪切非互补链(非目标链),最终产生DNA双链断裂(double-strand break,简称DSB)[9,15]。当产生DSB后,会触发细胞内DNA修复机制,在无修复模板时,非同源末端连接(nonhomologous end joining,简称NHEJ)修复机制被激活,在DSB位置出现随机插入、缺失或替换;当存在同源性供体DNA修复模板时,通常会触发同源定向修复(homology directed repair,简称HDR)机制,形成手术刀式的精确修饰[15]。在实际应用中,crRNA序列是与靶标基因同源的20个核酸序列,tracrRNA为通用序列,并且靶标基因位点紧邻PAM序列(通常是PAM序列上游 3 bp 处),当PAM序列为NGG(N为任意碱基)序列时切割效率最高[16](图1)。
1.2 CRISPR/Cas9系统在植物中的应用
CRISPR/Cas9基因编辑技术因操作方便、成本低廉、编辑效率高等特点,已经成为植物基因编辑的有力工具之一[17]。随着研究的不断深入,经过改造的CRISPR/Cas9系统已广泛应用于拟南芥、烟草、高粱、水稻、小麦、玉米等不同植物基因组的定向编辑,作用方式包括基因敲除、基因组缺失、顺式调控元件的破坏、基因敲入和病毒感染的抑制等,并且获得较高的诱导突变率和可稳定遗传的基因组编辑植株[18-19]。该技术不仅可以对单个靶标进行编辑,而且能够实现多靶标编辑,Ma等开发了一种能够对多个基因位点(最多8个)编辑的CRISPR/Cas9系统,并成功应用于水稻和拟南芥的水稻类成花素家族(FT-like)功能的研究[20]。CRISPR/Cas9基因编辑技术的基因敲入或替换主要依赖于细胞的HDR机制,由于HDR仅在S期和G2期晚期活跃,而NHEJ在除有丝分裂外的整个细胞周期中均比较活跃。因此,基因敲入或替换的编辑效率较低[21],但仍在许多植物中实现了基因片段的精确敲入或替换。例如,在玉米、水稻等植物中已有相关报道[22-24]。近年来,CRISPR/Cas9基因编辑技术也开始应用于植物与病原物互作的研究中,对于提高植物抗病性具有重大意义[25]。
2 CRISPR/Cas9系统在植物抗真菌中的应用
2.1 编辑寄主基因产生抗病性
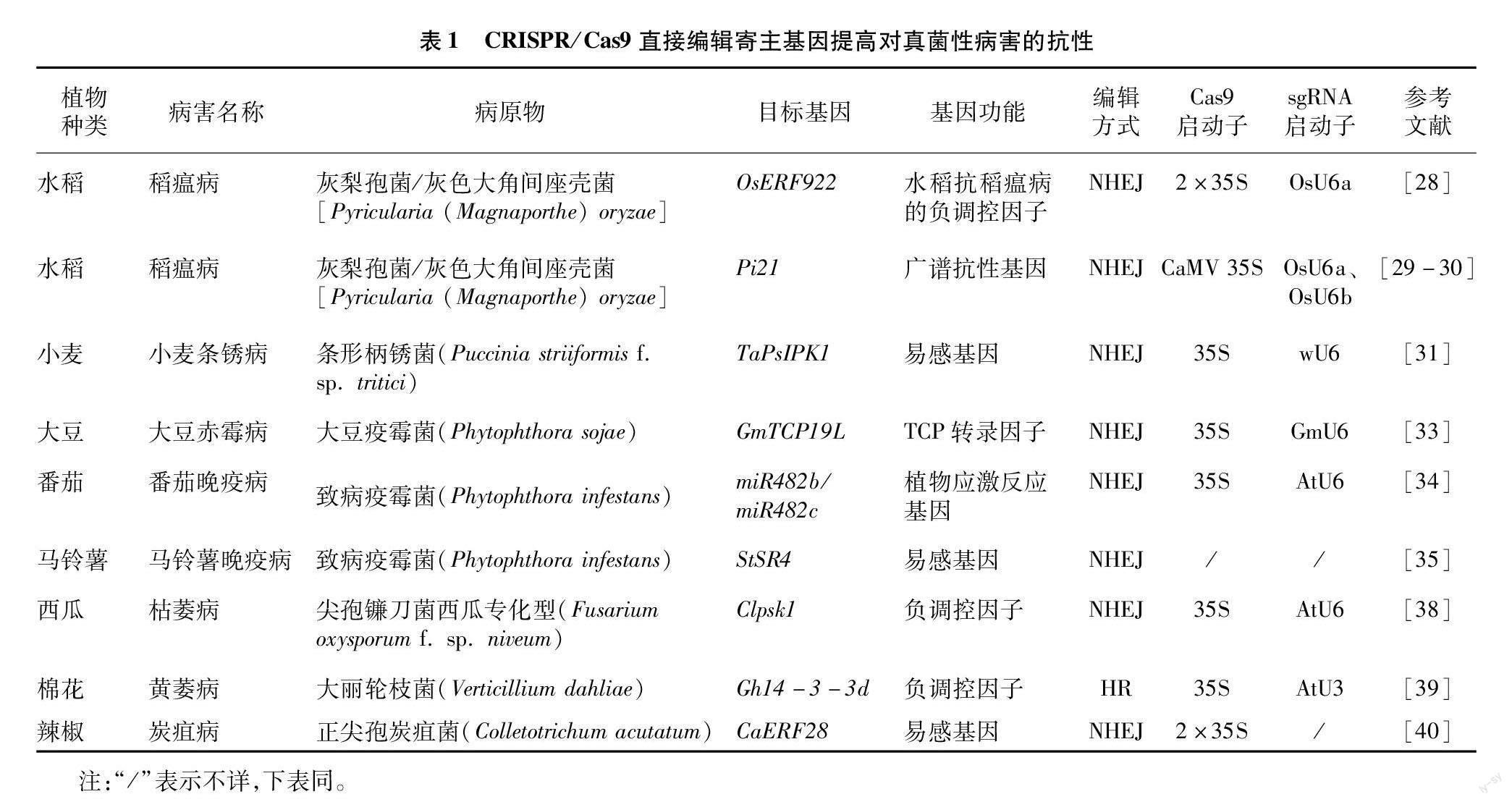
CRISPR/Cas9基因编辑技术的出现为真菌研究领域带来了巨大的变革。2015年,Ndvig等通过CRISPR/Cas9基因编辑技术有效地将定向突变引入到构巢曲霉菌(Aspergillus nidulans)中[26]。同年Matsu-Ura等发现,该技术同样可以有效编辑粗糙链孢霉菌(Neurospora crassa),并猜想可能还适用于链孢霉属自然分离物和丝状真菌的基因编辑[27]。利用CRISPR/Cas9基因编辑技术已成功构建OsERF922和Pi21等基因缺失的突变株系,与野生型相比,突变株系对稻瘟病的抗性均有所提高,而不影响主要农艺性状[28-30]。小麦受体类细胞质激酶基因TaPsIPK1是一个已知的被锈病效应蛋白靶向的易感基因,通过CRISPR/Cas9基因编辑技术敲除TaPsIPK1基因的突变株系对小麦条锈病病原菌(Puccinia striiformis f. sp. tritici)能够产生广谱抗性,农艺性状同样未受到影响[31]。TCP转录因子在植物生长发育、激素合成、信号转导以及对生物和非生物胁迫的响应中发挥重要作用,而大豆在感染大豆赤霉病后,TCP基因会被显著诱导[32]。因此,Fan等利用CRISPR/Cas9基因编辑技术在大豆品种Williams 82中特异性诱导GmTCP19L靶向突變,发现GmTCP19L直接或间接调节大豆对赤霉病的抗性[33]。CRISPR/Cas9基因编辑技术不仅用于粮食作物病害的研究,还应用于蔬菜病害的研究。研究显示,通过CRISPR/Cas9基因编辑技术同时敲除番茄中的miR482b和miR482c基因,能够提高番茄对晚疫病的抗性,并且同时敲除miR482b和miR482c比单独敲除miR482b的抗性更强[34]。晚疫病同样也是危害马铃薯的主要病害之一,在我国发生较为普遍。Moon等将StSR4基因通过CRISPR/Cas9基因编辑技术引入马铃薯原生质体中,产生StSR4基因缺失的突变株系,尽管对晚疫病的抗性有所提高,但马铃薯的生长受到抑制[35]。
尖孢镰刀菌(Fusarium oxysporum)和大丽轮枝菌(Verticillium dahliae)是土传真菌病原中最具代表性的病原之一,危害棉花、番茄、香蕉和甜瓜在内的许多重要作物,造成巨大的经济损失。植物磺肽素(phytosulfokine,简称PSK)是一种二硫酸五肽植物激素,在植物生长发育以及植物免疫调节方面发挥重要作用[36-37]。通过CRISPR/Cas9基因编辑技术敲除编码PSK前体的Clpsk1基因,发现Clpsk1功能丧失后的西瓜幼苗,增加了对尖孢镰刀菌的抗性[38]。陆地棉为异源四倍体,导致传统基因编辑技术的应用存在一定困难,因此Zhang等利用CRISPR/Cas9基因编辑技术成功将Gh14-3-3d基因插入陆地棉中,突变株系显著增加了对大丽轮枝菌的抗性[39]。通常CRISPR/Cas9系统包含2个启动子,分别用来启动Cas9蛋白和sgRNA。2021年,Mishra等首次使用单个启动子的CRISPR/Cas9系统,对辣椒的易感基因CaERF28进行缺失突变,与野生型相比,抗炭疽病能力明显增强[40]。CRISPR/Cas9系统直接编辑寄主基因提高对真菌病害的抗性详见表1。
2.2 编辑真菌基因产生抗病性
小麦颖枯病是由颖枯壳针孢(Parastagonospora nodorum)引起的小麦死体营养型真菌病害,研究显示CRISPR/Cas9基因编辑技术可用于P. nodorum反向遗传的研究,并且在NHEJ途径和HR途径均进行了验证[41]。子囊菌亚门稻绿核菌属稻曲病菌(Ustilaginoidea virens)是稻曲病的致病因子,研究人员以U. virens的Gln-tRNA为启动子构建了Cas9-gRNA载体,并转化至U. virens菌株的原生质体中,有效地靶向缺失U. virens中的USTA和UvSLT2基因[42]。由于疫霉(Phytophthora)同源重组率低,所以无法有效对其进行基因编辑。2017年Fang等利用CRISPR/Cas9基因编辑技术在大豆疫霉(P. sojae)中建立基因编辑体系,而且在其他疫霉的基因编辑研究中提出优化方案,为更深层次的研究提供了新思路[43]。环磷酸腺(cyclic adenosine monophosphate,简称cAMP)信号通路在控制植物病原体的形态变化和致病性方面发挥重要作用[44]。PcPdeH是一种编码高亲和力磷酸二酯酶(phosphodiesterase,PDE)的基因,是cAMP信号通路的关键调节因子[45]。在辣椒疫霉(P. capsici)中利用CRISPR/Cas9基因编辑技术获得PcPdeH缺失的突变菌株,突变菌株表现出营养生长缺陷的现象,并且与野生型菌株相比毒力显著降低[46]。尖孢镰刀菌(F. oxysporum)作为典型的土传病害,能够引起植物产生多种病害,最常见的则为枯萎病,植物感染后表现出褪绿、坏死、发育迟缓和萎蔫等症状[46]。Wang等基于同源性靶向整合(homology-independent targeted integration,简称HITI)和同源性重组整合(homology-dependent recombination Integration,简称HDRI),利用CRISPR/Cas9基因编辑技术成功建立尖孢镰刀菌(F. oxysporum)等真菌的内源性基因标记(endogenous gene tagging,EGT)系统,为阐明尖孢镰刀菌(F. oxysporum)毒力因子及发病机制提供了强有力的技术手段[47]。
3 CRISPR/Cas9系统在植物抗细菌中的应用
3.1 编辑寄主基因产生抗病性
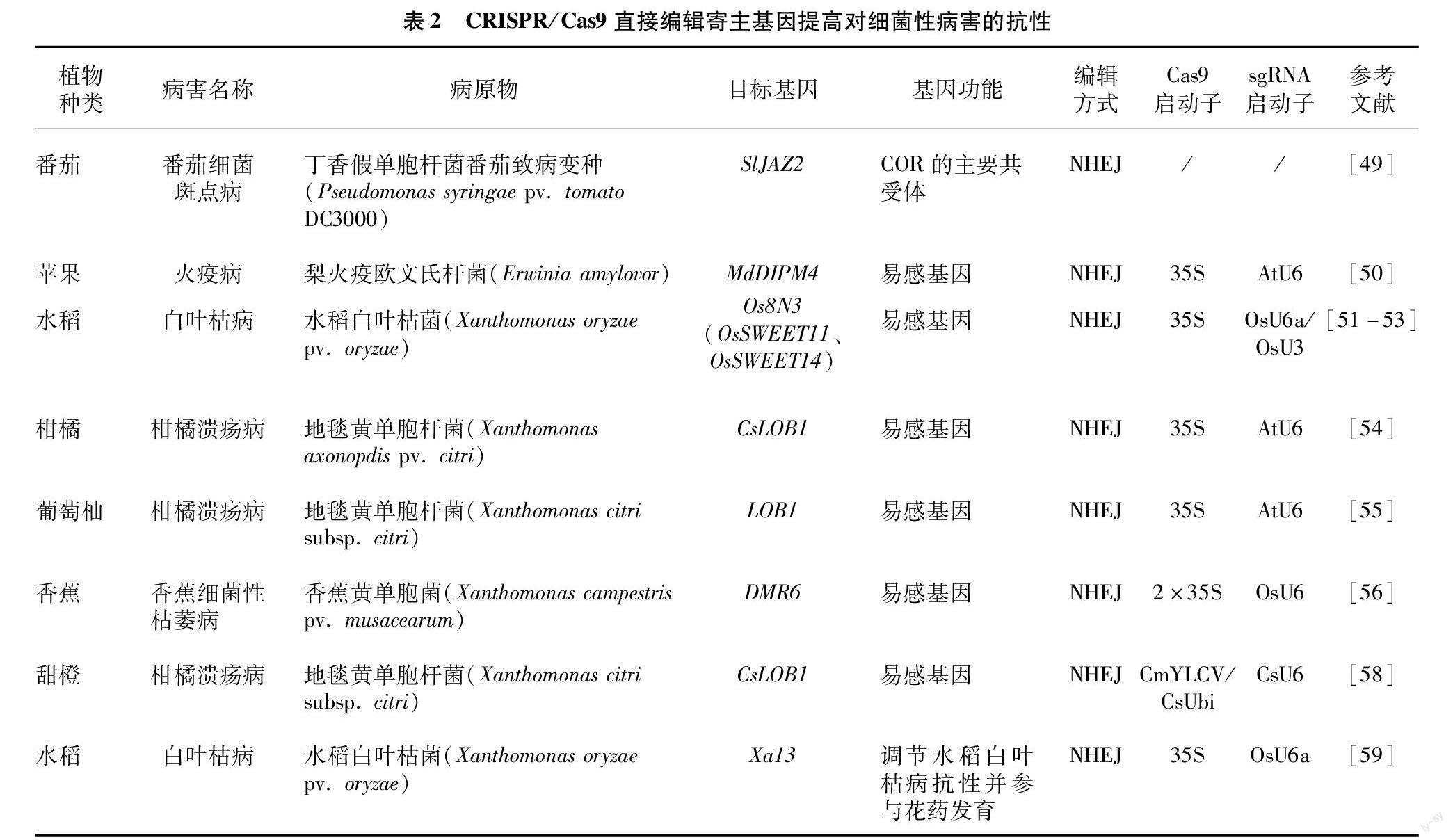
植物细菌病害防控水平取决于植物病原细菌学的基础研究水平和创新能力[48],假单胞杆菌属(Pseudomonas)作为植物病原细菌主要类群之一,造成的危害主要是叶斑、叶枯、萎蔫。丁香假单胞菌番茄致病变种(P. syringae pv. tomato DC3000,Pto DC3000)是番茄细菌性斑点病的致病因子,主要通过气孔和伤口入侵番茄,而Pto DC3000入侵番茄后,则产生刺激气孔打开和促进细菌定殖的冠菌素(coronatine,COR),SlJAZ2蛋白是气孔保卫细胞中COR的主要供受体,Ortigosa等通过CRISPR/Cas9基因编辑技术编辑SlJAZ2基因,产生C端结构域缺失的突变体,进而通过COR阻止气孔重新开放,增加了对Pto DC3000的抗性[49]。苹果火疫病是由梨火疫欧文氏杆菌(Erwinia amylovora)侵染所引起的一类毁灭性病害,E. amylovora的致病基因DspA/E编码的致病效应子与苹果易感蛋白MdDIPM4相互作用触发植物感染,因此,有研究利用CRISPR/Cas9基因编辑技术敲除MdDIPM4,以降低对E. amylovor的敏感性[50]。水稻白叶枯病是由稻黄单胞菌白叶枯致病变种(Xanthomonas oryzae pv. oryzae,Xoo)引起的一种细菌性病害,提高水稻自身抗性是一种经济有效的办法。水稻Os8N3(也称为OsSWET11)基因是转录激活类因子(transcription activator-like effector,TAL effector)誘导的易感基因。利用CRISPR/Cas9基因编辑技术敲除水稻Os8N3(OsSWET11)基因的突变株系,显著增强了对Xoo的抗性,且农艺性状未发生显著变化[51-53]。柑橘溃疡病由该菌的亚种地毯黄单胞杆菌(X. axonopdis pv. citri)引起的典型病害之一,会造成柑橘叶子、茎和果实产生坏死、隆起的病变,导致落叶落果,严重影响作物的产量和品质。X. axonopdis pv. citri通过PthA4(一种转录激活效应器)诱导溃疡易感性基因LOB1的表达,因此,Peng等利用CRISPR/Cas9基因编辑技术对晚锦橙易感基因CsLOB1进行基因编辑,由于晚锦橙含有3个拷贝的CsLOB1G等位基因和1个拷贝的CsLOB1-等位基因,因此,设计了5个sgRNA进行编辑,仅对CsLOB1G敲除后明显增强了对柑橘溃疡病的抗性[54]。Jia等同样利用CRISPR/Cas9基因编辑技术对葡萄柚的双等位基因TI-LOBP和TI-LLOBP进行敲除,双突变株系的抗性显著增强[55]。香蕉作为一种重要的经济作物,同样受到黄单胞杆菌属(Xanthomonas)的危害,其致病菌为香蕉黄单胞菌(X. campestris pv. musacearum,Xcm),目前,只有野蕉(Musa balbisiana)对Xcm具有抗性,而其余栽培种均不具备对Xcm的抗性[56]。有研究显示,通过CRISPR/Cas9基因编辑技术对易感基因DMR6进行基因编辑,获得DMR6缺失的突变株系,显著增强了香蕉对病原细菌的抗性[57]。
由于传统的CRISPR/Cas9基因编辑技术在编辑双等位基因存在编辑效率不高的问题,所以甜橙仍然难以产生双等位/纯合突变体。因此,Huang等对CRISPR/Cas9系统进行改进,将35S启动子替换为CsUbi(citrus sinensis ubiquitin,简称CsUbi)启动子或CmYLCV(cestrum yellow leaf curling virus,简称CmYLCV)启动子,该系统能够显著增强甜橙的编辑效率[58]。目前,大多数报道主要针对一个特定基因的sgRNA突变,且大部分突变位点被选择在基因编码区,但由于一些基因含有特殊功能,因此不能采取此方法来进行编辑。例如,Xa13基因为多功能性基因,不仅参与调节水稻对白叶枯病的抗性,而且还参与花药的发育。因此,Li等通过CRISPR/Cas9基因编辑技术仅对Xa13基因启动子的部分序列进行编辑,获得了抗水稻白叶枯病但不影响正常花药发育和结实的突变株系[59]。CRISPR/Cas9直接编辑寄主基因提高对细菌性病害的抗性详见表2。
3.2 编辑细菌基因产生抗病性
明确植物病原物致病机制通常需要挖掘产生致病因子基因的功能,但大多数分子生物学技术仅适用于某种类型致病变种的基因编辑。因此,CRISPR/Cas9基因编辑技术的优势可以得到充分体现。假单胞菌属(Pseudomonas)是自然界种类最多、生态意义最大的革兰氏阴性菌群,丁香假单胞菌(P. syringae)是与植物相关性最大的一类假单胞菌,可引起植物多种病害的发生。例如,丁香假单胞菌番茄致病变种(P. syringae pv. tomato)和丁香假单胞杆菌菜豆晕疫病致病变种(P. syringae pv. phaseolicola)等。Martí等将Cas9核酸酶和sgRNA融合表达至pSEVA质粒后,转化至P. syringae pv. tomato和P. syringae pv. phaseolicola,从而达到基因编辑的目的[60]。黄褐假单胞菌(P. fulva)能够侵染辣椒、番茄、茄子、马铃薯等多种植物,Zhang等基于HDR和噬菌体λ-Red重组系统,利用CRISPR/Cas9系统开发了一种高效便捷的P. fulva基因编辑方法,并且编辑效率可达100%[61]。黄单胞杆菌属(Xanthomonas)是一类重要的植物病原细菌,产生了多种致病种,主要危害水稻、番茄和柑橘等多种作物。Xoo菌株通常使用高度保守的Ⅲ型分泌系统(T3SS)将毒力效应物输送到水稻细胞中,并进一步抑制宿主的免疫,而对Xoo菌株基因组的编辑将有助于增加对其致病因子的研究。Jiang等利用内源性I-C CRISPR/Cas系统实现了Xoo菌株各种精确、高效的基因编辑,包括缺失、插入、碱基替换等,此系统将有助于植物病原细菌致病机制的研究,为宿主-病原物互作研究提供理论基础[62]。目前,尚未见利用CRISPR/Cas9基因编辑技术对植物病原细菌欧氏杆菌属(Erwinia)、土壤杆菌属(Agrobacterium)和棒杆菌属(Corynebacterium)进行相关研究的报道。
4 CRISPR/Cas9系统在植物抗病毒中的应用
4.1 编辑寄主基因产生抗病性
以CRISPR/Cas9基因编辑技术为代表的抗病毒工程成为预防和控制植物病毒的有效策略,马铃薯Y病毒属是植物病毒中最大的一个属,可危害多种作物,造成重大的经济损失[63]。真核翻译起始因子4E(eukaryotic translation initiation factor 4E,eIF4E)编码一个与甲基化的鸟嘌呤结合的帽子结合蛋白,能够触发蛋白质翻译起始复合物的组装,是双子叶植物应对马铃薯Y病毒属(potato virus Y,PVY)的一个S基因。因此,eIF4E已成为CRISPR/Cas9基因组编辑技术设计植物抗病毒的主要靶标位点。例如,在番茄中,利用CRISPR/Cas9基因编辑技术对eIF4E1进行敲除编辑,显著增强了番茄植株对辣椒斑驳病毒(pepper mottle virus,简称PepMoV)的抗性[64]。在小麦中,为增加对小麦黄花叶病毒(wheat yellow mosaic virus,WYMV)的抗性,同样利用CRISPR/Cas9基因组编辑技术对eIF4E进行编辑,获得了抗性株系[65]。烟草不仅是重要的非粮食经济作物,而且还是一种模式植物,感染马铃薯Y病毒后,可造成烟草大量减产,严重影响烟草产业的发展。通过双sgRNA表达CRISPR/Cas9系统来敲除烟草中eIF4E基因家族的多个拷贝,同时缺失eIF4E1-S、eIF4E1-T、eIF4E2-S和eIF4E2-T能够增加烟草对PVY的持久抗性[66]。AGO蛋白是重要的抗病毒蛋白之一,其中AGO2的抗病毒机制尚未明确,因此,Ludman等通过CRISPR/Cas9基因编辑技术获得了AGO2缺失的本氏烟株系,证明AGO2是植物应对PVX、TuMV和TCV免疫反应的重要组成部分[67]。2014年,中东地区出现了一种危害番茄的病毒,番茄褐色皱果病毒(tomato brown rugose fruit virus,ToBRFV),并开始在世界范围内迅速传播,严重威胁了番茄产业的发展。烟草病毒增殖蛋白1(TOBAMOVIRUS MULTIPLICATION1,TOM1)基因对烟草花叶病毒的高效复制起着至关重要的作用,并且在许多植物中发现TOM1的同源基因。因此,Ishikawa等利用CRISPR/Cas9基因编辑技术对TOM1同源基因进行敲除,获得了抗ToBRFV的突变株系[68]。植物bZIP类转录因子参与植物的生长发育、激素信号、抗病性及抗逆性等多种生物胁迫过程,为进一步明确bZIP类转录因子抗病毒机制,研究人员通过CRISPR/Cas9基因编辑技术获得了NbbZIP28基因突变株系,突变株系与野生型植株相比,农艺性状未发生显著变化,但NbbZIP28敲除后导致植株对病毒的敏感性上升[69]。番茄黄化曲叶病毒(tomato yellow leaf curl virus,TYLCV)病是番茄的主要病害之一,一些番茄品种携带了TYLCV的易感基因SlPelo,因此,Pramanik等通过CRISPR/Cas9基因编辑技术在优质番茄品种BN-86中,获得了SlPelo基因的敲除株系,以提高番茄的抗病性[70]。CRISPR/Cas9直接编辑寄主基因提高对病毒性病害的抗性详见表3。
4.2 编辑病毒基因产生抗病性
CRISPR/Cas9不仅能够编辑寄主基因来提高抗病毒能力,而且可以直接编辑病毒基因来增强植物对病毒的抗性。例如,CRISPR/Cas9基因编辑技术编辑衣壳蛋白(CP)、复制蛋白(Rep)、基因间隔区域(IR)和一些开放阅读框(ORF)等病毒相关结构来增强植物病毒抗性[71]。木尔坦棉花曲叶病毒(cotton leaf curl multan virus,CLCuMuV)属于双生病毒科(Geminiviridae)菜豆金色花葉病毒属(Begomovirus) 能够引起棉花曲叶病的发生。通过
构建IR和转录激活蛋白(TrAP)双sgRNA的CRISPR/Cas9系统,突变株系对CLCuMuV的抗性显著增强,表明该系统可应用于抗DNA病毒植物株系的选育,为抗植物双生病毒科病毒提供新策略[72]。为增加CRISPR/Cas9系统的稳定性,Ghorbani等构建了由rgsCaM启动子驱动Cas9的CRISPR/Cas9系统,以番茄黄化曲叶病毒(tomato yellow leaf curl virus,TYLCV)的CP和IR为靶点设计sgRNA,与野生型相比,对TYLCV的抗性显著增强,并且以rgsCaM启动子驱动的Cas9效率显著高于传统的35S启动子[73]。Ali等则基于TYLCV的CP、Rep和IR设计了3种sgRNA,结果显示这3种sgRNA均减少了烟草中病毒的积累,但基于IR设计的sgRNA效果更加明显[74]。属于双生病毒科(Geminiviridae)曲顶病毒属(Curtovirus)的甜菜曲顶病毒 (beet severe curly top virus,简称BSCTV)也危害多种作物。Ji等以CP和Rep为靶标设计sgRNA,在拟南芥和本氏烟中进行瞬时表达,BSCTV侵染后,过表达植株表现出强烈的抗病毒能力[75]。有研究报道,长基因间隔区(long intergenic region,LIR)也被当作提高植物抗双生病毒设计sgRNA的靶标位点,在烟草中瞬时表达后,接种菜豆黄矮病毒(bean yellow dwarf virus,BeYDV),病毒的拷贝数和症状明显减弱[76]。为提高植物抗小麦矮缩病毒(wheat dwarf virus,WDV)的能力,研究人员针对WDV基因组的不同片段,设计了4个sgRNA,分别是MP 3′端和CP 5′端(重叠ORF)、Rep/RepA 5′端、LIR和Rep 3′端,突变株系的农艺性状未发生变化,但抗WDV的能力显著增强[77]。Malik等基于CRISPR/Cas9基因编辑技术,将靶向保守基因组区域的各种gRNA构建体单独或串联(3个构建体一起)用于触发对鹰嘴豆褪绿矮缩病毒(chickpea chlorotic dwarf virus,CpCDV)的抗性,结果显示,基于CpCDV基因组的LIR、Rep和RepA设计的sgRNA串联构成的CRISPR/Cas9系统对CpCDV的抗性最强[78]。
相对于DNA病毒,RNA病毒在农业生产造成的危害更大。例如,烟草花叶病毒(tobacco mosaic virus,简称TMV)和黄瓜花叶病毒(cucumber mosaic virus,简称CMV),可对多种作物产生危害。然而利用CRISPR/Cas9基因编辑技术编辑RNA病毒的报道较少。Zhang等以CMV基因组的开放阅读框1a(open reading frames 1a,ORF 1a)、ORF CP和3′端非编码区 (3′ untranslated regions,3′ UTRs)为靶标设计sgRNA,在本氏烟和拟南芥中转化后,表现出抗TMV和CMV的能力,并且后代植物同样表现出抗性,表明这种抗性是可遗传的[79]。葡萄卷叶相关病毒(grapevine leafroll-associated virus 3,GLRaV-3)是葡萄卷叶病的致病因子之一,由于缺乏天然抗性资源,因此,关于葡萄抗GLRaV-3的研究相对有限。Jiao等则利用FnCas9以GLRaV-3的5 ku 蛋白(5 ku protein,p5)、热休克蛋白70同系物(Heat shock protein 70 homolog,Hsp70h)、热休克蛋白90同系物(Hsp90h)、CP以及次外壳蛋白(CPm)为靶标设计sgRNA,结果表明,Hsp70h和CP表现出最强抗性,能够有效赋予葡萄抗GLRaV-3特性[80]。CRISPR/Cas9编辑病毒基因使植物产生抗病性详见表4。
5 讨论与展望
CRISPR/Cas9基因编辑技术自问世以来,就备受关注。CRISPR/Cas9基因编辑技术不仅可以针对植物自身进行定向编辑,而且能够直接编辑真菌、细菌和病毒等病原物来提高植物抗病性。CRISPR/Cas9系统在启动子的选择上,Cas9通常以35S作为启动子,但部分研究为提高启动效率以及特异性也选择CmYLCV/CsUbi、Ubi4-2、rgsCaM以及2×35S为启动子,sgRNA通常为U6启动子,仅有少部分为U3启动子。CRISPR/Cas9基因编辑技术由于可以实现多个sgRNA的同时编辑,因此,较其他基因编辑技术编辑效率高,利用此特点,可以对某些家族基因的多个基因位点或含有多个等位基因进行编辑,更有利于基因功能研究[66]。CRISPR/Cas9基因编辑技术还存在一个明显的优势,就是可以针对基因的某段序列进行突变,所以针对某些多功能基因,则可以避免干扰植物正常的发育。例如,Xa13为多功能性基因,不仅具有隐性抗细菌性枯萎病的功能,而且还参与花药的发育[59]。当然,CRISPR/Cas9基因编辑技术也存在一些不足。例如,对靶标基因位点和PAM序列有一定的要求。只有靶标位点紧邻PAM序列(通常是PAM序列上游3 bp处),PAM序列为NGG(N为任意碱基)序列时切割效率最高,对于一些特殊靶标基因位点,则会出现编辑效率不高的现象[16]。无论是对植物本身,还是针对病原物的基因编辑,主要是通过NHEJ途径敲除靶标基因,而通过HDR途径进行基因编辑的则较少,这主要是源于NHEJ在除有丝分裂外的整个细胞周期中均比较活跃,HDR仅在S期和G2晚期活跃[21],因此,在HDR途径仍要加大研究力度,充分发挥CRISPR/Cas9基因编辑技术的潜力。
在植物抗真菌方面,主要以编辑寄主基因产生抗病性为主,包括一些易感基因、转录因子以及一些植物激素调节因子,而且以基因敲除为主[81]。多数研究显示,转基因植物农艺性状未发生变化,但并未经过多代验证,仍存在一些风险,部分研究表明,对某些基因编辑后,造成农艺性状发生变化。例如,利用CRISPR/Cas9基因編辑技术将StSR4基因敲除后,突变株系尽管对晚疫病的抗性有所提高,但同时也导致马铃薯出现矮化的现象[35]。在植物抗细菌方面,针对植物基因的编辑主要是以假单胞杆菌属、欧文氏杆菌属和黄单胞杆菌属为主,对于细菌基因组的编辑主要以假单胞杆菌属和黄单胞杆菌属为主,目前,尚未见利用CRISPR-Cas9基因编辑技术对植物病原细菌欧氏杆菌属、土壤杆菌属和棒杆菌属进行相关研究的报道。与植物真菌和细菌不同的是,关于植物抗病毒的研究,对于病毒本身的编辑要多于寄主植物。在编辑寄主植物时,围绕eIF4E进行的相关研究较多。例如,关于PepMoV、WYMV以及PVY等多个病毒的研究中均以eIF4E为靶标设计sgRNA[64-66]。虽然相对于DNA病毒,RNA病毒在农业生产中造成的危害更大,但是利用CRISPR/Cas9基因编辑技术对于病毒的研究则是以DNA病毒为主,针对RNA病毒基因组的研究较少。
作为一种强大而通用的基因编辑和调控工具,CRISPR/Cas9基因编辑技术已经在加速各个科学领域的发展,并成为农业可持续发展的源泉[82]。目前,基于CRISPR/Cas9基因编辑技术的植物抗病性研究取得了一定的成果,但多数研究以室内研究为主,在田间条件下的抗病性并未经过验证,在未来应该要加强田间试验,选育稳定遗传抗病性,且不影响农艺性状的优良品系。许多科学家认为,CRISPR/Cas9基因编辑技术只涉及到植物本身DNA的变化,而不设计外源基因的插入,应该是属于诱变,但欧盟法院作出判决,认为使用基因组编辑技术创造的生物将被监管为转基因生物,而此决定势必会影响相关研究的积极性[83]。虽然CRISPR/Cas9基因编辑技术存在一些不足和挑战,但在未来仍然是作物抗病育种研究的优势技术,特别是在野生抗性资源稀缺时,CRISPR/Cas9基因编辑技术诱导的突变将为植物抗病育种提供多种选择,促进农业可持续发展。
参考文献:
[1]Serge S,Laetitia W. Modeling the impact of crop diseases on global food security[J]. Annual Review of Phytopathology,2020,58(1):313-341.
[2]Liu X R,Ao K,Yao J,et al. Engineering plant disease resistance against biotrophic pathogens[J]. Current Opinion in Plant Biology,2021,60:101987.
[3]Kang S,Lumactud R,Li N X,et al. Harnessing chemical ecology for environment-friendly crop protection[J]. Phytopathology,2021,111(10):1697-1710.
[4]周 蒙. 中国生物农药发展的现实挑战与对策分析[J]. 中国生物防治学报,2021,37(1):184-192.
[5]Dangl J L,Horvath D M,Staskawicz B J. Pivoting the plant immune system from dissection to deployment[J]. Science,2013,341(6147):746-751.
[6]朱宗财. 烟草NtRBP45在烟草花叶病毒侵染寄主过程中的功能研究[D]. 阿拉尔:塔里木大学,2020:7.
[7]赖昕彤,王柯岚,由雨欣,等. 基于CRISPR/Cas系统的DNA碱基编辑研究进展[J]. 生物技术通报,2022,38(6):1-12.
[8]Chen K L,Wang Y P,Zhang R,et al. CRISPR/cas genome editing and precision plant breeding in agriculture[J]. Annual Review of Plant Biology,2019,70:667-697.
[9]陈修贵. CRISPR/Cas9系统介导的棉花GhCLA1和GhVP基因编辑的研究[D]. 武汉:华中农业大学,2017:2.
[10]Nishimasu H,Ran F A,Hsu P D,et al. Crystal structure of Cas9 in complex with guide RNA and target DNA[J]. Cell,2014,156(5):935-949.
[11]Jinek M,Chylinski K,Fonfara I,et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science,2012,337(6096):816-821.
[12]Cong L,Ran F A,Cox D,et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science,2013,339(6121):819-823.
[13]Gupta D,Bhattacharjee O,Mandal D,et al. CRISPR-Cas9 system:a new-fangled dawn in gene editing[J]. Life Sciences,2019,232:116636.
[14]李 江,耿立召,許建平. CRISPR/Cas9系统中引导RNA的研究进展[J]. 生物技术通报,2019,35(4):108-115.
[15]Jiang F G,Doudna J A. CRISPR-Cas9 structures and mechanisms[J]. Annual Review of Biophysics,2017,46:505-529.
[16]Ma Y W,Zhang L F,Huang X X. Genome modification by CRISPR/Cas9[J]. The FEBS Journal,2014,281(23):5186-5193.
[17]Zhang Y L,Ma X L,Xie X R,et al. CRISPR/Cas9-based genome editing in plants[J]. Progress in Molecular Biology and Translational Science,2017,149:133-150.
[18]Ma X L,Zhu Q L,Chen Y L,et al. CRISPR/Cas9 platforms for genome editing in plants:developments and applications[J]. Molecular Plant,2016,9(7):961-974.
[19]景润春,卢 洪. CRISPR/Cas9基因组定向编辑技术的发展与在作物遗传育种中的应用[J]. 中国农业科学,2016,49(7):1219-1229.
[20]Ma X L,Zhang Q Y,Zhu Q L,et al. A robust CRISPR/Cas9 system for convenient,high-efficiency multiplex genome editing in monocot and dicot plants[J]. Molecular Plant,2015,8(8):1274-1284.
[21]Yang Y H,Xu C D,Shen Z Y,et al. Crop quality improvement through genome editing strategy[J]. Frontiers in Genome Editing,2022,3:819687.
[22]Svitashev S,Young J K,Schwartz C,et al. Targeted mutagenesis,precise gene editing,and site-specific gene insertion in maize using Cas9 and guide RNA[J]. Plant Physiology,2015,169(2):931-945.
[23]Begemann M B,Gray B N,January E,et al. Precise insertion and guided editing of higher plant genomes using Cpf1 CRISPR nucleases[J]. Scientific Reports,2017,7:11606.
[24]Wang M G,Lu Y M,Botella J R,et al. Gene targeting by homology-directed repair in rice using a geminivirus-based CRISPR/Cas9 system[J]. Molecular Plant,2017,10(7):1007-1010.
[25]Zaynab M,Sharif Y,Fatima M,et al. CRISPR/Cas9 to generate plant immunity against pathogen[J]. Microbial Pathogenesis,2020,141:103996.
[26]Ndvig C S,Nielsen J B,Kogle M E,et al. A CRISPR-Cas9 system for genetic engineering of filamentous fungi[J]. PLoS One,2015,10(7):e0133085.
[27]Matsu-Ura T,Baek M,Kwon J,et al. Efficient gene editing in Neurospora crassa with CRISPR technology[J]. Fungal Biology and Biotechnology,2015,2(1):1-7.
[28]Wang F J,Wang C L,Liu P Q,et al. Enhanced rice blast resistance by CRISPR/Cas9-targeted mutagenesis of the ERF transcription factor gene OsERF922[J]. PLoS One,2016,11(4):e0154027.
[29]Nawaz G,Usman B,Peng H W,et al. Knockout of Pi21 by CRISPR/Cas9 and iTRAQ-based proteomic analysis of mutants revealed new insights into M. oryzae resistance in elite rice line[J]. Genes,2020,11(7):735.
[30]Zhou Y B,Xu S C,Jiang N,et al. Engineering of rice varieties with enhanced resistances to both blast and bacterial blight diseases via CRISPR/Cas9[J]. Plant Biotechnology Journal,2022,20(5):876-885.
[31]Wang N,Tang C L,Fan X,et al. Inactivation of a wheat protein kinase gene confers broad-spectrum resistance to rust fungi[J]. Cell,2022,185(16):2961-2974.
[32]Lin F,Zhao M X,Baumann D D,et al. Molecular response to the pathogen Phytophthora sojae among ten soybean near isogenic lines revealed by comparative transcriptomics[J]. BMC Genomics,2014,15(1):1-13.
[33]Fan S J,Zhang Z,Song Y,et al. CRISPR/Cas9-mediated targeted mutagenesis of GmTCP19L increasing susceptibility to Phytophthora sojae in soybean[J]. PLoS One,2022,17(6):e0267502.
[34]Hong Y H,Meng J,He X L,et al. Editing miR482b and miR482c simultaneously by CRISPR/Cas9 enhanced tomato resistance to Phytophthora infestans[J]. Phytopathology,2021,111(6):1008-1016.
[35]Moon K B,Park S J,Park J S,et al. Editing of StSR4 by Cas9-RNPs confers resistance to Phytophthora infestans in potato[J]. Frontiers in Plant Science,2022,13:997888.
[36]Sauter M. Phytosulfokine peptide signalling[J]. Journal of Experimental Botany,2015,66(17):5161-5169.
[37]Hammes U Z. Novel roles for phytosulfokine signalling in plant–pathogen interactions[J]. Plant,Cell & Environment,2016,39(7):1393-1395.
[38]Zhang M,Liu Q L,Yang X P,et al. CRISPR/Cas9-mediated mutagenesis of Clpsk1 in watermelon to confer resistance to Fusarium oxysporum f. sp. niveum[J]. Plant Cell Reports,2020,39(5):589-595.
[39]Zhang Z N,Ge X Y,Luo X L,et al. Simultaneous editing of two copies of Gh14-3-3d confers enhanced transgene-clean plant defense against Verticillium dahliae in allotetraploid upland cotton[J]. Frontiers in Plant Science,2018,9:842.
[40]Mishra R,Mohanty J N,Mahanty B,et al. A single transcript CRISPR/Cas9 mediated mutagenesis of CaERF28 confers anthracnose resistance in chilli pepper (Capsicum annuum L.)[J]. Planta,2021,254(1):5.
[41]Haseena K,McDonald Megan C,Williams Simon J,et al. Assessing the efficacy of CRISPR/Cas9 genome editing in the wheat pathogen Parastagonspora nodorum[J]. Fungal Biology and Biotechnology,2020,7(1):4.
[42]Liang Y F,Han Y,Wang C F,et al. Targeted deletion of the USTA and UvSLT2 genes efficiently in Ustilaginoidea virens with the CRISPR-Cas9 system[J]. Frontiers in Plant Science,2018,9:699.
[43]Fang Y F,Cui L K,Gu B,et al. Efficient genome editing in the oomycete Phytophthora sojae using CRISPR/Cas9[J]. Current Protocols in Microbiology,2017,44(1):21A.1.1-21A.1.26.
[44]Borges-Walmsley M I,Walmsley A R. cAMP signalling in pathogenic fungi:control of dimorphic switching and pathogenicity[J]. Trends in Microbiology,2000,8(3):133-141.
[45]Li X,Liu Y,Tan X Q,et al. The high-affinity phosphodiesterase PcPdeH is involved in the polarized growth and pathogenicity of Phytophthora capsici[J]. Fungal Biology,2020,124(3/4):164-173.
[46]Gordon T R. Fusarium oxysporum and the Fusarium wilt syndrome[J]. Annual Review of Phytopathology,2017,55:23-39.
[47]Wang Q,Coleman J J. CRISPR/Cas9-mediated endogenous gene tagging in Fusarium oxysporum[J]. Fungal Genetics and Biology,2019,126:17-24.
[48]郭澤建,侯明生. 中国植物病理学会2011年学术年会论文集[C]. 北京:中国农业科学技术出版社,2011:265.
[49]Ortigosa A,Gimenez-Ibanez S,Leonhardt N,et al. Design of a bacterial speck resistant tomato by CRISPR/Cas9-mediated editing of SlJAZ2[J]. Plant Biotechnology Journal,2019,17(3):665-673.
[50]Pompili V,Dalla Costa L,Piazza S,et al. Reduced fire blight susceptibility in apple cultivars using a high-efficiency CRISPR/Cas9-FLP/FRT-based gene editing system[J]. Plant Biotechnology Journal,2020,18(3):845-858.
[51]Kim Y A,Moon H,Park C J. CRISPR/Cas9-targeted mutagenesis of Os8N3 in rice to confer resistance to Xanthomonas oryzae pv. oryzae[J]. Rice,2019,12(1):1-13.
[52]Zafar K,Khan M Z,Amin I,et al. Precise CRISPR-Cas9 mediated genome editing in super basmati rice for resistance against bacterial blight by targeting the major susceptibility gene[J]. Frontiers in Plant Science,2020,11:575.
[53]Zeng X,Luo Y F,Vu N T Q,et al. CRISPR/Cas9-mediated mutation of OsSWEET14 in rice cv. Zhonghua11 confers resistance to Xanthomonas oryzae pv. oryzae without yield penalty[J]. BMC Plant Biology,2020,20(1):1-11.
[54]Peng A H,Chen S C,Lei T G,et al. Engineering canker-resistant plants through CRISPR/Cas9-targeted editing of the susceptibility gene CsLOB1 promoter in citrus[J]. Plant Biotechnology Journal,2017,15(12):1509-1519.
[55]Jia H G,Omar A A,Orbovic' V,et al. Biallelic editing of the LOB1 promoter via CRISPR/Cas9 creates canker-resistant ‘Duncan grapefruit[J]. Phytopathology,2022,112(2):308-314.
[56]Tripathi L,Tripathi J N,Shah T,et al. Molecular basis of disease resistance in banana progenitor Musa balbisiana against Xanthomonas campestris pv. musacearum[J]. Scientific Reports,2019,9(1):7007.
[57]Tripathi J N,Ntui V O,Shah T,et al. CRISPR/Cas9-mediated editing of DMR6 orthologue in banana (Musa spp.) confers enhanced resistance to bacterial disease[J]. Plant Biotechnology Journal,2021,19(7):1291-1293.
[58]Huang X E,Wang Y C,Wang N.Highly efficient generation of canker-resistant sweet orange enabled by an improved CRISPR/Cas9 system[J]. Frontiers in Plant Science,2022,12:769907.
[59]Li C Y,Li W,Zhou Z H,et al. A new rice breeding method:CRISPR/Cas9 system editing of the Xa13 promoter to cultivate transgene-free bacterial blight-resistant rice[J]. Plant Biotechnology Journal,2020,18(2):313-315.
[60]Martí E S. CRISPR/Cas9 system optimization in Pseudomona syringae using a heterologous repair system[J]. Biosaia,2020,9:1.
[61]Zhang N,He J T,Muhammad A,et al. CRISPR/Cas9-mediated genome editing for Pseudomonas fulva,a novel Pseudomonas species with clinical,animal,and plant-associated isolates[J]. International Journal of Molecular Sciences,2022,23(10):5443.
[62]Jiang D D,Zhang D D,Li S N,et al. Highly efficient genome editing in Xanthomonas oryzae pv. oryzae through repurposing the endogenous type I-C CRISPR-Cas system[J]. Molecular Plant Pathology,2022,23(4):583-594.
[63]沈 川,李 夏,吳云锋. CRISPR/Cas基因编辑系统在植物抗病毒防御中的应用[J]. 植物病理学报,2023,53(3):343-357.
[64]Yoon Y J,Venkatesh J,Lee J H,et al. Genome editing of eIF4E1 in tomato confers resistance to pepper mottle virus[J]. Frontiers in Plant Science,2020,11:1098.
[65]Kan J H,Cai Y,Cheng C Y,et al. CRISPR/Cas9-guided knockout of eIF4E improves wheat yellow mosaic virus resistance without yield penalty[J]. Plant Biotechnology Journal,2023,21(5):893-895.
[66]Le N T,Tran H T,Bui T P,et al. Simultaneously induced mutations in eIF4E genes by CRISPR/Cas9 enhance PVY resistance in tobacco[J]. Scientific Reports,2022,12(1):14627.
[67]Ludman M,Burgyán J,Fátyol K. Crispr/Cas9 mediated inactivation of argonaute 2 reveals its differential involvement in antiviral responses[J]. Scientific Reports,2017,7(1):1-12.
[68]Ishikawa M,Yoshida T,Matsuyama M,et al. Tomato brown rugose fruit virus resistance generated by quadruple knockout of homologs of TOBAMOVIRUS MULTIPLICATION1 in tomato[J]. Plant Physiology,2022,189(2):679-686.
[69]何青云,劉笑玮,焦裕冰,等. 烟草NbbZIP28突变体的创建及其对病毒侵染胁迫的响应[J]. 中国农业科学,2018,51(14):2689-2699.
[70]Pramanik D,Shelake R M,Park J,et al. CRISPR/Cas9-mediated generation of pathogen-resistant tomato against tomato yellow leaf curl virus and powdery mildew[J]. International Journal of Molecular Sciences,2021,22(4):1878.
[71]Hinge V R,Chavhan R L,Kale S P,et al. Engineering resistance against viruses in field crops using CRISPR-Cas9[J]. Current Genomics,2021,22(3):214-231.
[72]Yin K Q,Han T,Xie K,et al. Engineer complete resistance to cotton leaf curl multan virus by the CRISPR/Cas9 system in Nicotiana benthamiana[J]. Phytopathology Research,2019,1(1):1-9.
[73]Ghorbani Faal P,Farsi M,Seifi A,et al. Virus-induced CRISPR-Cas9 system improved resistance against tomato yellow leaf curl virus[J]. Molecular Biology Reports,2020,47(5):3369-3376.
[74]Ali Z,Abulfaraj A,Idris A,et al. CRISPR/Cas9-mediated viral interference in plants[J]. Genome Biology,2015,16(1):238.
[75]Ji X,Zhang H W,Zhang Y,et al. Establishing a CRISPR-Cas-like immune system conferring DNA virus resistance in plants[J]. Nature Plants,2015,1:15144.
[76]Baltes N J,Hummel A W,Konecna E,et al. Conferring resistance to geminiviruses with the CRISPR–Cas prokaryotic immune system[J]. Nature Plants,2015,1:15145.
[77]Kis A,Hamar é,Tholt G,et al. Creating highly efficient resistance against wheat dwarf virus in barley by employing CRISPR/Cas9 system[J]. Plant Biotechnology Journal,2019,17(6):1004-1006.
[78]Malik M A M,Haider M S,Zhai Y,et al. Towards developing resistance to chickpea chlorotic dwarf virus through CRISPR/Cas9-mediated gene editing using multiplexed gRNAs[J]. Journal of Plant Diseases and Protection,2023,130(1):23-33.
[79]Zhang T,Zheng Q F,Yi X,et al. Establishing RNA virus resistance in plants by harnessing CRISPR immune system[J]. Plant Biotechnology Journal,2018,16(8):1415-1423.
[80]Jiao B,Hao X,Liu Z,et al. Engineering CRISPR immune systems conferring GLRaV-3 resistance in grapevine[J]. Horticulture Research,2022,9:uhab023.
[81]夏雄飞,潘俊良,韩长志. CRISPR/Cas9基因编辑技术在植物病原真菌中的应用研究进展[J]. 江苏农业科学,2022,50(12):22-27.
[82]Swati T,Robin K,Vivak K,et al. Engineering disease resistant plants through CRISPR-Cas9 technology[J]. GM Crops & Food,2021,12(1):125-144.
[83]Borrelli V M G,Brambilla V,Rogowsky P,et al. The enhancement of plant disease resistance using CRISPR/Cas9 technology[J]. Frontiers in Plant Science,2018,9:1245.

