16S rRNA甲基化酶在氨基糖苷类抗生素耐药革兰阴性菌中的分布
周颖杰, 余 慧, 郭庆兰, 徐晓刚, 叶信予, 吴 湜, 郭 燕, 王明贵
氨基糖苷类抗生素在临床广泛应用,它通过作用于细菌核糖体,抑制其蛋白质合成,并破坏细胞膜完整性而杀灭细菌[1]。由于这类药物具杀菌作用,抗菌谱广、有较长的抗生素后效应,与β内酰胺类等抗菌药物有协同作用,目前是治疗革兰阳性及阴性菌感染,尤其是耐多药革兰阴性菌感染的常用联合治疗药物之一[2]。
2003年,一种由质粒介导的新耐药机制—16S rRNA甲基化酶(16S rRNA methylase)被发现,该酶使革兰阴性杆菌对卡那霉素组和庆大霉素组的多种临床常用氨基糖苷类抗生素高度耐药。目前已发现的16S rRNA甲基化酶基因有rmtA[3]、rmtB[4]、rmtC[5]、rmtD[6]、npmA[7]和armA[8]。这些基因位于质粒或转座子上,易于传播,是造成细菌对氨基糖苷类抗生素高度耐药的重要原因。然而,16S rRNA甲基化酶系列基因在不同国家和地区的不同细菌中分布可有很大差异,因此有必要进行调查,明确本地区16S rRNA甲基化酶在氨基糖苷类抗生素耐药菌中的分布,并通过同源性分析,分析该基因的传播方式,将有助于临床抗感染治疗和新药开发[9]。
本研究对我院临床分离的氨基糖苷类抗生素耐药革兰阴性菌进行16S rRNA甲基化酶的检测。
材料与方法
一、材料
(一)菌株来源 收集我院(三级甲等医院,床位数1 300张)2007年10月1日—12月31日临床分离的所有连续非重复的革兰阴性杆菌。药敏试验质控菌大肠埃希菌ATCC25922和铜绿假单胞菌ATCC27853为本研究所保存菌株。16S rRNA甲基化酶基因阳性对照株由 Yoshichika教授赠与[3]。
(二)培养基及主要试剂 Mueller-Hinton(MH)琼脂培养基、阿米卡星和庆大霉素纸片为英国OXOID公司产品,阿米卡星、卡那霉素、妥布霉素、庆大霉素、奈替米星、新霉素、大观霉素、环丙沙星、头孢噻肟、头孢他啶、头孢吡肟和美罗培南标准品购自中国药品生物制品检定所,PCR引物由上海生工生物工程技术服务有限公司合成。
二、方法
(一)药敏试验 纸片扩散法药敏试验的操作和结果判读根据CLSI 2007年版[10]。采用琼脂对倍稀释法对纸片扩散法提示耐药的菌株进行阿米卡星等12种抗菌药的最低抑菌浓度(MIC)测定,操作和结果判读根据CLSI 2009年版[11]。
(二)16S rRNA甲基化酶基因检测 琼脂稀释法测定对阿米卡星和庆大霉素耐药的菌株(阿米卡星MIC≥64 mg/L,庆大霉素MIC≥16 mg/L)进行armA,rmtA,rmtB,rmtC,rmtD和npmA基因检测。制备反应模板采用煮沸法。armA,rmtA,rmtB,rmtC,rmtD基因检测所用引物见参考文献[6,12],npmA基因检测所用引物为 5′-GGAGGGCTATCTAATGTGGT-3′和 5′-GCCCAAAGAGAAT TAAACTG-3′。PCR产物用1%琼脂凝胶进行电泳,紫外光检测仪检测结果。
(三)ERIC(Enterobacterial repetitive intergenic consensus sequence)-PCR分析 ERIC-PCR的引 物 为 5′-AAGTAAGTGACTGGGGTGAGCG-3′[13]。反应条件为94 ℃,5 min;4个循环:94℃1 min,26℃2 min,72℃1 min;40个循环:94℃1 min,40℃1 min,72℃1 min;最后,72℃10 min延伸。反应产物用2%的琼脂凝胶进行电泳,紫外光检测仪检测结果。
(四)统计学 采用SPSS 17.0 for windows进行统计分析。
结 果
一、临床分离菌株的耐药情况
共收集741株连续非重复临床分离的革兰阴性菌,MIC结果提示其中211株细菌同时对阿米卡星(MIC≥64 mg/L)和庆大霉素(MIC≥16 mg/L)耐药。
二、甲基化酶基因的检出
对以上211株耐药菌进行了PCR检测。193株(193/211,91.5%)检出16S rRNA甲基化酶基因,其中armA检出63.0%(133/211),rmtB检出28.4%(60/211)。未检测到rmt A,rmtC,rmtD和npmA基因。
将阿米卡星每个MIC水平的甲基化酶阳性菌株数两两做 Fisher′s Exact检验,发现256 mg/L与512 mg/L两组 P<0.01,512 mg/L与 1 024 mg/L两组,P=0.008,甲基化酶检出的差异有统计学意义;1 024 mg/L与>1 024 mg/L两组,P=1,差异无统计学意义(表1)。将庆大霉素每个MIC水平甲基化酶阳性菌株数分别做Fisher′s Exact检验,发现32 mg/L与128 mg/L+256 mg/L两组P=0.028,甲基化酶检出率的差异有统计学意义,128 mg/L+256 mg/L+512 mg/L与>512 mg/L两组,P=1,差异无统计学意义(表1)。
104株阿米卡星MIC≥512 mg/L及庆大霉素MIC≥128 mg/L的肠杆菌科细菌中,甲基化酶基因检出率为100%,armA和rmtB各约占一半(分别为49与55株)(表2);94株阿米卡星MIC≥512 mg/L及庆大霉素MIC≥128 mg/L不发酵糖菌中,甲基化酶基因的检出率为94.7%,主要为armA(84株)(表2)。
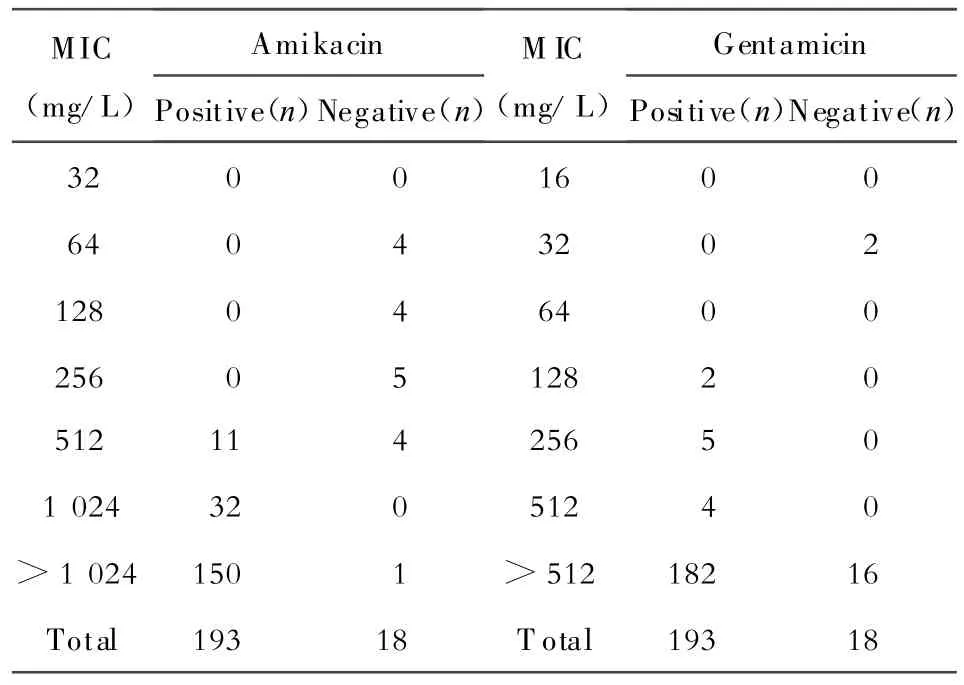
表1 不同阿米卡星和庆大霉素MIC菌株中16S rRNA甲基化酶基因的分布Table 1.The distribution of 16S rRNA methylase genes in strains with different MICs of amikacin and gentamicin
三、药敏试验结果
所有含甲基化酶基因的菌株对阿米卡星、卡那霉素、妥布霉素、庆大霉素和奈替米星均呈高度耐药,但有些菌株对新霉素和大观霉素敏感(表3)。甲基化酶基因阳性的菌株,阿米卡星的MIC均≥512 mg/L,庆大霉素MIC均≥128 mg/L;甲基化酶阴性的菌株,除5株阿米卡星MIC≥512 mg/L,庆大霉素MIC>512 mg/L,包括1株鲍曼不动杆菌,4株铜绿假单胞菌外,其余13株阿米卡星MIC为64~256 mg/L,庆大霉素MIC 2株为32 mg/L,11株MIC>512 mg/L(表1)。
甲基化酶基因阳性菌株与阴性菌株相比,对头孢菌素类、氟喹诺酮类及碳青霉烯类抗菌药物耐药水平更高(表3)。
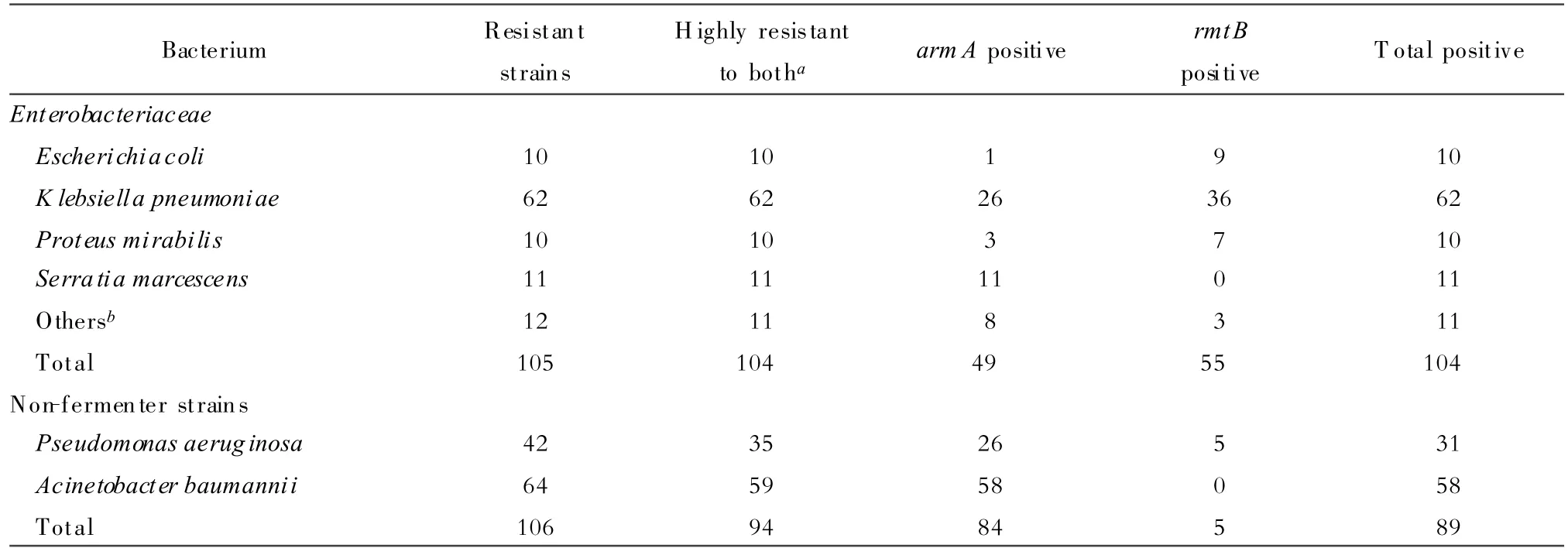
表2 对阿米卡星及庆大霉素耐药革兰阴性菌中16S rRNA甲基化酶基因的分布(n)Table 2.The prevalence of 16S rRNA methylase genes in Gram-negative strains resistant to amikacin and gentamicin(n)
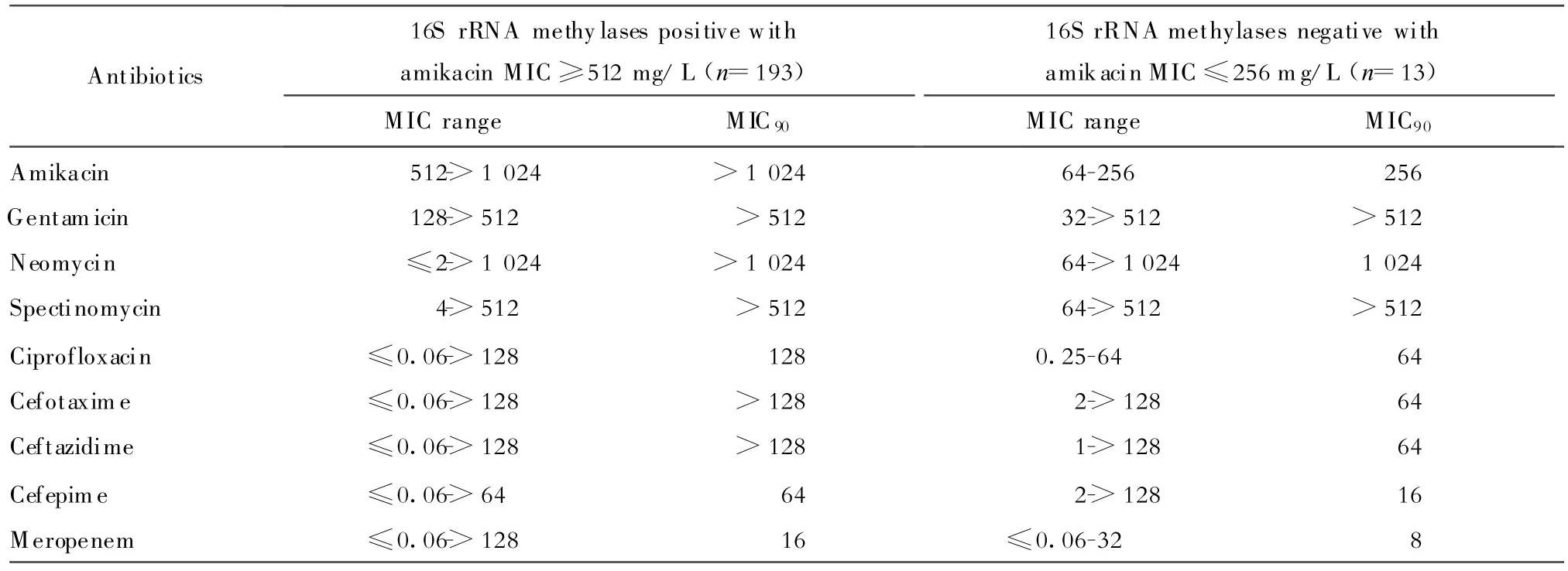
表3 16S rRNA甲基化酶阳性和阴性菌株对不同抗菌药物的MIC(mg/L)比较Table 3.MICs(mg/L)of various antimicrobials against 16S rRNA methylase gene positive or negative isolates
四、ERIC-PCR分析结果
198株16S rRNA甲基化酶基因阳性的菌株中,166株成功地进行了 ERIC-PCR分析,其中62株肺炎克雷伯菌分8个克隆,57株鲍曼不动杆菌分7个克隆,19株铜绿假单胞菌分为6个克隆,10株大肠埃希菌分6个克隆,10株奇异变形杆菌分3个克隆,2株摩根摩根菌分2个克隆,4株弗劳地柠檬酸杆菌和2株产酸克雷伯菌分别为1个克隆。同一个ERIC-PCR克隆的菌株可以有不同的甲基化酶基因。
讨 论
氨基糖苷类抗生素作用于原核细胞核糖体30S亚单位中16S rRNA的A位(此为mRNA的译码区),使细菌在合成蛋白的过程中发生错误翻译,抑制肽链延长,造成错误的蛋白质插入细胞膜,导致细胞膜断裂,使细胞内重要物质外漏,细胞迅速死亡[2]。根据在核糖体的作用部位,氨基糖苷类抗生素可以分为:①与A位点结合的4,6-二脱氧链霉胺类,其中包括卡那霉素组(阿米卡星)和庆大霉素组;4,5-二脱氧链霉胺类,包括新霉素、巴龙霉素等。②与非A位点结合的链霉素和大观霉素[9]。
随着氨基糖苷类抗生素在临床广泛应用,细菌对其耐药率也持续上升。细菌对氨基糖苷类抗生素的耐药机制过去认为主要有3种:①细菌外膜蛋白通透性改变或细胞内膜转运异常,使药物在菌体内的蓄积减少。②产生氨基糖苷类钝化酶包括氨基糖苷类乙酰转移酶、磷酸转移酶及核苷转移酶。③核糖体蛋白或16S rRNA突变,使药物作用靶位改变,药物进入细菌后不能有效地与核糖体结合而产生耐药[14-16]。2003年,日本[3]和法国[8]学者发现16S rRNA甲基化酶,该酶使细菌30S核糖体亚单位中16S rRNA A位点的1个或几个碱基甲基化,使氨基糖苷类抗生素不能与其作用靶点结合,从而介导革兰阴性杆菌对多种临床常用氨基糖苷类抗生素耐药[9]。
自2003年16S rRNA甲基化酶发现以来,该酶在肠杆菌科细菌、铜绿假单胞菌及鲍曼不动杆菌中均有发现[12,17-23]。很多国家及地区对该酶的流行病学进行了研究[3-8],其检出率在各地区不同菌种及不同耐药水平的细菌中,从0.03%[12]到95%[17]不等。其分布也有地区及菌种差异:RmtA至今仅在日本的铜绿假单胞菌及韩国的1株铜绿假单胞菌中发现[12,18];RmtD仅在拉丁美洲发现[19-20];鲍曼不动杆菌中仅有ArmA[21-22]。
以上研究中,氨基糖苷类抗生素耐药菌株数大多少于100株。本研究对211株连续非重复临床分离的氨基糖苷类抗生素耐药菌株进行了检测,其中包括肠杆菌科细菌及不发酵糖菌,有助于了解该酶在氨基糖苷类抗生素耐药革兰阴性菌中的流行情况。
研究发现,在阿米卡星MIC≥512 mg/L及庆大霉素MIC≥128 mg/L的菌株中,104株肠杆菌科细菌100%检出甲基化酶,94株不发酵糖菌95%检出甲基化酶。这些甲基化酶阳性的菌株对卡那霉素、妥布霉素、庆大霉素和奈替米星亦均呈高度耐药,但有些菌株对新霉素和大观霉素敏感,这是由于新霉素和大观霉素的作用机制是与非A位点核糖体结合,因此不受甲基化酶对A位点甲基化的影响。而对在阿米卡星MIC为64~256 mg/L的13株细菌中,未检出甲基化酶,这些菌株中2株庆大霉素MIC为32 mg/L,11株MIC>512 mg/L。
我国台湾Yan等[23]发现,35株armA或rmtB阳性的大肠埃希菌和肺炎克雷伯菌,其阿米卡星MIC≥512 mg/L,而46株未检出该类基因的细菌MIC为16~64 mg/L,与本研究的发现相似。另外,本研究发现甲基化酶阴性菌株中存在庆大霉素的MIC>512 mg/L的菌株,可能因氨基糖苷类钝化酶引起[2],需要进一步研究证实。所以,采用阿米卡星的MIC值预测是否存在16S rRNA甲基化酶较合适。
本研究有4株铜绿假单胞菌和1株鲍曼不动杆菌对阿米卡星高度耐药,但未检出现有的6种甲基化酶基因,需要对该5株细菌可能存在的其他耐药机制进一步检测和研究,以明确其高度耐药的原因。
至今在中国大陆仅检测到armA和rmtB[22,24-25],本研究也未发现其他4种甲基化酶基因。与其他文献报道类似,本研究在鲍曼不动杆菌中只检出armA[21-22]。目前,rmtA仅在日本和韩国的铜绿假单胞菌中发现[12,18],本研究的42株铜绿假单胞菌中也未发现有rmt A。
不少文献报道甲基化酶基因通常与其他耐药基因同时存在,如 blaTEM、blaCTX-M、blaSHV、blaOX A[4,7,21,23,26]、和qnr[24],所以,含甲基化酶基因的菌株可能同时对β内酰胺类和氟喹诺酮类抗菌药物耐药。本研究的结果也发现,甲基化酶基因阳性的菌株比阴性菌株对头孢菌素类、碳青霉烯类和喹诺酮类抗菌药物的耐药水平更高。
ERIC-PCR的结果表明,耐药菌株并非单克隆传播,因此,推测该基因的传播同时有水平和垂直2种方式。Chen等[24]的研究已经证实这一点。
综上所述,几乎所有阿米卡星MIC>512 mg/L的革兰阴性菌临床株中均检出16S rRNA armA或rmtB甲基化酶基因。
[1] Magnet S,Blanchard JS.Molecular insights into aminoglycoside action and resistance[J].Chem Rev,2005,105(2):477-498.
[2] 汪复,张婴元.实用抗感染治疗学[M].上海:人民卫生出版社,2004:231-232.
[3] Yokoyama K,Doi Y,Yamane K,et al.Acquisition of 16S rRNA methylase gene in P.aeruginosa[J].Lancet,2003,362(9399):1888-1893.
[4] Doi Y,Yokoyama K,Yamane K,et al.Plasmid-mediated 16S rRNA methylase in S.marcescens conferring high-level resistance to aminoglycosides[J].Antimicrob Agents Chemother,2004,48(2):491-496.
[5] Wachino J,Yamane K,Shibayama K,et al.Novel plasmidmediated 16S rRNA methylase,rmtC,found in a P.mirabilisisolate demonstrating extraordinary high-level resistance against various aminoglycosides[J].Antimicrob Agents Chemother,2006,50(1):178-184.
[6] Doi Y,Oliveria Garcia D,Adams J,et al.Coproduction of novel 16S rRNA methylase RmtD and metallo-β-lactamase SPM-1 in a panresistant P.aeruginosa isolate from Brazil[J].Antimicrob Agents Chemother,2007,51(3):852-856.
[7] Galimand M,Courvalin P,Lambert T.Plasmid-mediated highlevel resistance to aminoglycosides in Enterobacteriaceae due to 16S rRNA methylation[J].Antimicrob Agents Chemother,2003,47(8):2565-2571.
[8] Wachino J,Shibayama K,Kurokawa H,et al.Novel plasmid-mediated 16S rRNA m1A1408 methyltransferase,NpmA,found in a clinically isolated E.coli strain resistant to structurally diverse aminoglycosides[J].Antimicrob Agents Chemother,2007,51(12):4401-4409.
[9] 周颖杰,王明贵.质粒介导氨基糖苷类抗生素新耐药机制:16S rRNA甲基化酶[J].中国感染与化疗杂志,2010,10(2):155-159.
[10] Clinical and Laboratory Standards Institute.Performance Standards for Antimicrobial Susceptibility Testing[S].Seventeenth Info rmational Supplement,2007,M100-S17.
[11] Clinical and Laboratory Standards Institute.Performance Standards for Antimicrobial Susceptibility T esting[S].Nineteenth Info rmational Supplement,2009,M100-S19.
[12] Yamane K,Wachino J,Suzuki S,et al.16S rRNA methylase-producing,gram-negative pathogens,Japan[J].Emerg Infect Dis,2007,13(4):642-646.
[13] Versalovic J,Koeuth T,Lupski JR.Distribution of repetitive DNA sequences in eubacteria and application to fingerprinting of bacterial genomes[J].Nucleic Acids Res,1991,19(24):6823-6831.
[14] Hancock RE.Aminoglycoside uptake and mode of actionwith special reference to streptomycin and gentamicin.I.Antag onists and mutants[J].J Antimicrob Chemother,1981,8(4):249-276.

