脊髓性肌萎缩症SMN 1基因携带者筛查技术研究进展
江雨 周裕林
(厦门市妇幼保健院中心实验室,福建 厦门 361003)
1 背景介绍
脊髓性肌萎缩症(spinal muscular atrophy,SMA)是全球最常见的致死性常染色体隐性遗传疾病之一,活婴发生率为1/5000~1/10 000,人群携带率为1/35~1/50[1]。
临床上根据SMA发病年龄和病程的不同,将患者分为Ⅰ型、Ⅱ型、Ⅲ型及Ⅳ型,其中占患者比例约70%的Ⅰ型临床表型最为严重,常于6个月内起病,多数于2岁内夭折[2]。1995年,SMA相关基因被定位于5号染色体长臂1区(5ql1.2~13.3),并发现了反向重复的运动神经元生存基因(survival motor neuron,SMN)[3]。该基因有 2个序列高度相似的拷贝:靠近端粒的决定性基因SMN1及靠近着丝粒的修饰SMN2(图1),两者之间仅存在5个碱基的差异,而在编码区内只存在1个碱基的差异,即第7号外显子上的840C>T。由于SMN所在的染色体区域存在众多重复和假基因簇等易导致结构不稳定的序列,导致SMN基因发生缺失或转换的频率较高,相应的表现为人群SMN基因拷贝数组合复杂多变。

图1 SMN基因在染色体5q13中的位置图谱
脊髓性肌萎缩症危害严重且无有效治疗方法,尤其是占多数比例的Ⅰ型患儿一旦发病进展迅速、存活期短,生育SMA患儿无疑将给患者家庭带来经济和精神的沉重负担,因此开展人群携带者筛查,对高危胎儿进行产前诊断是降低该病发生率的惟一途径。2008年,全美医学遗传学会(American College of Medical Genetics,ACMG)建议不分地域种族,对所有孕龄人群无差别地实施SMN1携带者基因筛查,对高风险胎儿实施产前诊断或植入前诊断,从而减少SMA患儿的出生[4]。随即在2009年,全美妇产科医师协会(American College of Obstetricians and Gynecologists,ACOG)提议各国或地区有必要掌握本地区SMA流行病学数据,并在具备结果准确、成本低廉的检测方法前提下将SMA携带者检查纳入孕前优生范畴[5]。目前已报道的SMA携带者检测方法众多,不同方法的检出率、可靠性及经济性等指标也不尽相同,本文就SMN基因拷贝数分析中3种常见的技术及2种新方法作综述如下。
2 SMN1基因携带者筛查常见技术
据统计约95%的SMA患者表现为SMN1基因第7外显子位点纯合缺失[6],另外5%患者为SMN1杂合缺失,即属于含有微小突变的单拷贝SMN1基因。因此临床上SMA携带者检测的主要目标为SMN1基因单拷贝人群的检出。目前文献报道中最常见的SMN基因拷贝数分析技术为变性高效液相色谱(denaturing high performance liquid chromatography,DHPLC)、多 重 连 接 探 针 扩 增(multiplex ligation-dependent probe amplification,MLPA)及实时荧光定量PCR(real-time PCR)技术。不同技术在SMN1基因单拷贝携带者分析中互有优缺(表1),现分述如下。

表1 DHPLC、MLPA、实时定量PCR在SMN基因拷贝数变异分析中的比较
DHPLC是一类在单核苷酸多态性(Single Nucleotide Polymorphism,SNP)研究中常见的分析技术,具有成本低、检测快速、通量高的优点。其原理是在部分变性的条件下,通过杂合与纯合二倍体在柱中保留时间的差异判断是否存在DNA突变。在脊髓性肌萎缩症检测中,DHPLC分型的原理是通过将扩增产物洗脱峰形及峰高(面积)比例与参照样本比对,进而推测样本SMN基因的实际拷贝数[7,8]。实践应用中,DHPLC的分辨力常受到实验设计、检测环境等诸多因素的影响,存在检测可靠性和稳定性欠佳的问题。因而该技术在SMA携带者中一般仅用于初级筛查,诊断结果常需辅以其他方法进行验证。
MLPA是2002年由Schouten等[9]开发的一种基因拷贝数分析技术。其基本原理为特异性探针与靶序列DNA进行杂交,之后通过连接、PCR扩增,产物毛细管电泳分离及数据收集、软件计算等分析靶序列拷贝数变异。每个MLPA探针包括2个寡核苷酸片段,每个探针都包括一段引物序列和一段特异性序列。在MLPA反应中,2个寡核苷酸片段都与相邻的靶序列进行杂交,之后使用连接酶进行连接。此连接反应高度特异,只有当靶序列与探针特异性序列完全互补时,连接酶才能将2段探针连接成一条完整的核酸单链;只有当连接反应完成,才能进行随后的PCR扩增并收集到相应探针的扩增信号,如果检测的靶序列发生点突变或缺失,那么相应探针的扩增峰便会降低或缺失。因此,根据扩增峰的改变就可判断靶序列是否存在拷贝数的异常。目前荷兰MRC-Holland公司生产的SMA检测试剂盒(SALSA MLPA KIT P021)是可供临床实施脊髓性肌萎缩症诊断及携带者检测的惟一商品化产品。其包含的探针组合可同时检测SMN1及SMN2基因的第7、8外显子,针对纯合缺失型患者的检出率可达100%。在携带者检测中,由于具有可同步扩增的稳定拷贝数内参基因及校正品质控,其检测准确性及可靠性较DHPLC更佳,目前MLPA已成为SMA基因诊断的主流技术[10-12]。
近年来,随着相关仪器设备价格的迅速降低,实时定量PCR几乎已成为所有医疗机构的常规配置。由于具有操作步骤少、成本低、灵敏度高、无需开盖检测等诸多优势,实时定量PCR已成为当前分子诊断领域的 重要技术[13]。2002 年,Feldkotter M等[14]率先尝试用定量PCR方法分析SMN基因拷贝数,发展至今该平台已衍生出多种SMA携带者的检测方法,具体主要包括以下3类(表2):①SYBR GreenⅠ染料法:Feldkotter M 等[14]将一个SMN1:SMN2=2:0的参照样本梯度稀释,模拟不同SMN1拷贝数的标准品,并通过未知样本与标准品荧光crossing point(Cp)值的拟合判断样本的实际拷贝数,报道灵敏度为96.2%,特异性为100%。Soloviov OO等[15]则通过引入ALB内参基因,并依照相对定量算法2-△△Ct(Livak method)计算样本的SMN1基因拷贝数。相较在此之前采用的放射性或银离子标记的SMN基因分析方法,荧光染料实时定量PCR法的应用大幅度地提升了检测的易操作性,为大规模携带者筛查提供了可能。但是由于SYBR GreenⅠ等荧光染料本身并无序列特异性,无法区分扩增产物与引物二聚体及非特异产物产生的荧光信号差异。此外,目标基因与内参基因的扩增效率差异是否稳定符合2-△△Ct相对定量算法的数学基础也有待验证,因而该方法在携带者检测中的可靠性尚值得商榷。②通用引物结合特异性探针方法[16]:此类方法采用一对可同时扩增SMN1和SMN2基因第7外显子C840T位点的通用引物,并通过针对差异位点设计的特异性Taqman 探针区分SMN1和SMN2。本课题组通过对采用此类方法的文献内容进行重复,发现探针区分SMN基因的特异性存在局限,无SMN1拷贝的患者同样会产生一定强度的SMN1探针杂交信号,从而干扰了携带者筛查检测中的SMN1基因拷贝数分析。③特异性引物结合通用探针方法[17]:此法是通过针对SMN基因第7外显子C840T位点设计竞争性引物,分别扩增SMN1及SMN2基因,再通过两者共用的Taqman 探针检测荧光信号。相较由探针区分SMN1及SMN2基因,竞争性引物区分避免了SMN2基因的干扰,提高了分析的准确性。此外,由于体系中加入了内参基因、校正品以及外部标准曲线等多层次质控,使得检测的稳定性得以保证。课题组通过此类方法完成了对56例确定携带者的检测(数据未发表),检出率达到96.4%(54/56,2个携带者为同一染色体上存在2个SMN1拷贝)。另一方面,我们看到,虽然平台普及程度较高,但相较于DHPLC及MLPA等使用专用技术平台的SMN基因分型方案,实时定量PCR由于设备型号规格众多,不同研究采用的具体技术差异往往较大,未形成可供临床指导的标准化实施方案。因此至今未见应用此技术开展大规模人群携带者筛查的报道。
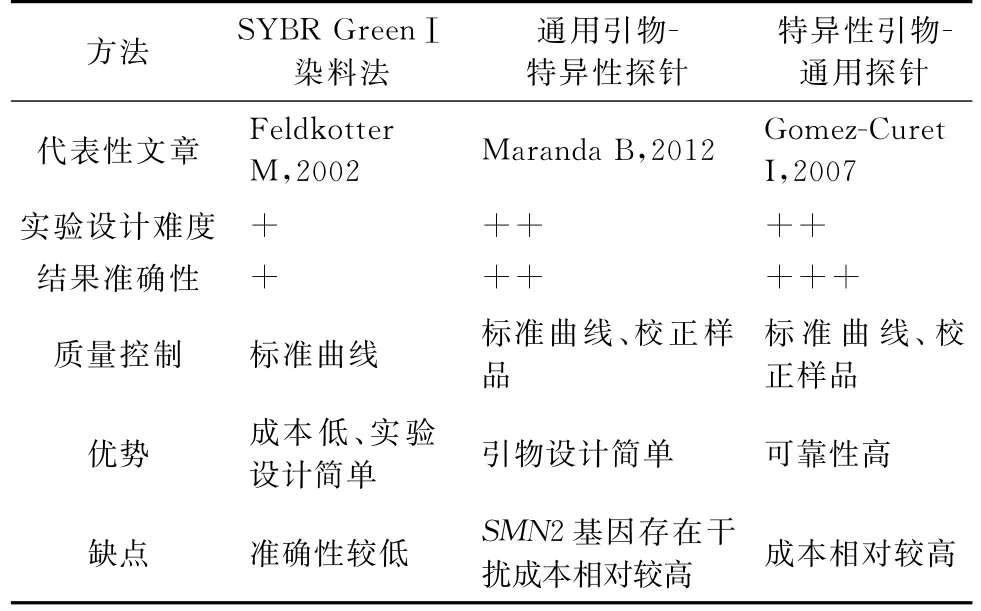
表2 常见定量PCR检测SMN基因拷贝数方法比较
3 SMN1基因携带者筛查新技术
由于现有SMN1基因携带者筛查技术在检测成本、稳定性和准确性等存在不同程度的缺陷,因此近年来研究界陆续发展了一些新的技术。这其中高分辨溶解曲线分析(high resolution melting analysis,HRMA)和数字PCR(Digital-PCR)技术在未来的SMA携带者筛查应用中最具潜力。
高分辨熔解曲线分析是一类近年来蓬勃发展的新型定量PCR衍生技术,因其在SNP研究中具有操作简便、检测快速、成本低廉等诸多优势而倍受关注[18,19]。该技术的原理是通过实时监测升温过程中饱和荧光染料与PCR扩增产物的结合情况,SNP位点异源双链由于不完全匹配会在升温过程中率先解开,荧光染料从局部解链的DNA分子上释放,从荧光强度与时间曲线上就可以判断是否存在SNP。而且不同SNP位点、杂合子与纯合子等都会影响熔解曲线的峰形,因此HRMA能够有效区分不同SNP位点与不同基因型。从检测原理来看,由于SMN1和SMN2第7外显子仅存在一个碱基的差异,因此两者不同的拷贝数组合可使产物熔解曲线峰形发生变化,从而可通过HRMA实现区分。2009年,Chen WJ等[20]率先应用HRMA结合非标记探针完成了对患者及正常人的区分,但未能实现对正常人SMN1基因拷贝数的定量分析。此后,Morikawa S[21]和 Er TK 等[22]分别尝试了 HRMA方法的可行性,但均未解决SMN1单拷贝分型技术。2012年,Dobrowolski SF等[23]通过对扩增引物和检测体系的改进,提出HRMA技术可对包括患者及个别类型携带者实现区分。从现有的报道来看,HRMA无需特异性探针及外部标准曲线,降低了检测成本、简化了操作步骤并易实现自动化,在SMA大规模人群筛查中具备潜力。但与此同时,由于人群中SMN基因拷贝数组合多态性高,已知类型就多达 19 种(表 3)[23],因此 HRMA 对不同SMN拷贝数比例间细微差异的分辨能力尚需验证。此外,由于SMN1:SMN2=1(2):0与SMN1:SMN2=1(2):1(2)携带者与正常人具有相同比例的SMN拷贝数组合,两者之间的准确区分亦是HRMA技术亟待解决的问题。

表3 人群中SMN 1、SMN 2拷贝数组合类型及SMN 1/SMN 2拷贝数比值
数字PCR最早在上世纪末是由Vogelstein B等[24]提出来的一种DNA绝对定量概念,但直到近两年才有成熟的检测平台面市。其原理是基于单分子PCR方法并结合统计学计算方法对DNA进行定量:将大量的稀释后的DNA溶液分散至微反应器或微滴中,使每个反应器(微滴)的DNA模板数少于或者等于1个拷贝。这样经过PCR循环之后,有DNA模板的反应器(微滴)可以检测到荧光信号,没有DNA模板的反应器(微滴)则检出不到信号(图2)。最后根据统计学中的柏松分布原理,推算出原始DNA的浓度或是基因拷贝数。从检测原理来看,数字PCR技术可准确定量样本基因的拷贝数,而无需使用相对定量方法中的内参基因、标准品及校准品等复杂质控,在进一步提升检测准确性的同时简化了操作流程。Zhong Q等[25]应用最新的数字平台PCR对包括患者、携带者及正常人的20例样本进行了准确的SMN基因分型,取得了预想中的结果。从近年来层出不穷的报道中可以看到数字PCR技术方兴未艾,各类研究及应用必将日趋深入和广泛,但是由于目前相关平台的可靠性仍处验证初期,同时也面临着检测成本较高及通量较低的问题,因此短时间内难以作为SMA携带者大规模人群筛查技术投入临床应用。
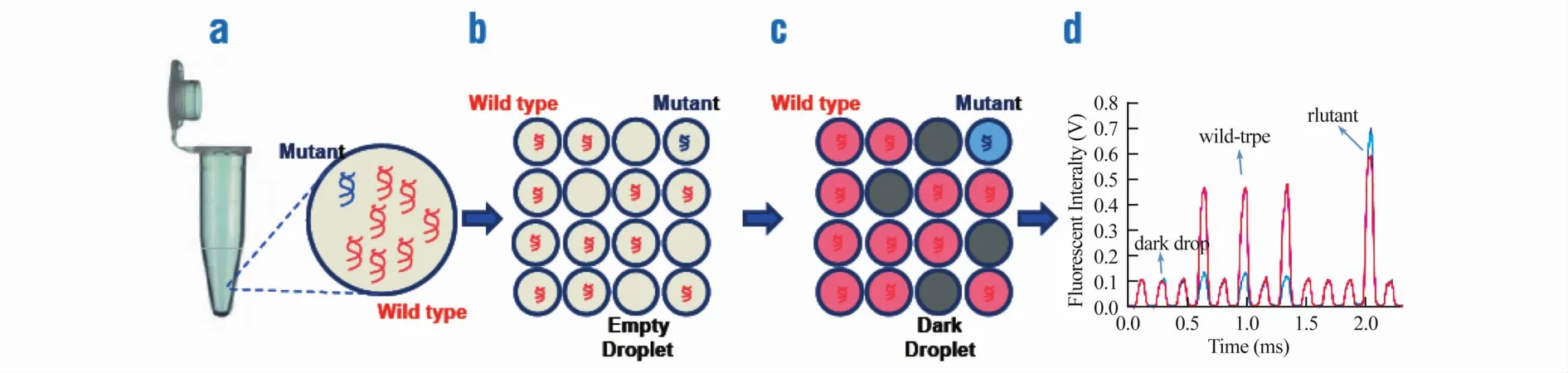
图2 数字PCR技术原理示意图
4 国内外SMA基因携带者检测临床应用现状及展望
由于脊髓性肌萎缩症携带者无临床表现,因此基因检测是检出携带人群的惟一可行方案。目前一些发达国家和地区在脊髓性肌萎缩症携带者产前优生检查中已积累了丰富的实践经验,并建立起了从遗传咨询到产前诊断的完整实施方案,SMA患儿的出生数量呈逐年下降趋势,取得了较好的社会效益[11,26-28]。从应用层面上看,由于具有稳定的商品化解决方案,MLPA是目前国外携带者检测的主流技术。国内虽然也可见相关研究报道,但长期以来却难以作为常规筛查技术投入应用。分析原因其一可能由于MLPA分析所需的毛细管测序仪价格昂贵,配套的进口试剂耗材价格高,导致国内医疗机构该检测平台的普及度不高;其次,MLPA操作步骤多、检测时间长、对技术人员的操作分析能力要求较高,在医疗资源紧缺的国内未受青睐。相对而言,定量PCR平台在国内的普及更为广泛,目前大多数二级以上医疗机构检验科或实验室已实现配置。但是由于型号规格不尽相同,缺乏可供借鉴的标准化实施方案,文献报道的方法往往不容易准确重复,使得目前有能力应用此技术开展临床SMA携带率检测的医疗机构数量十分有限。技术实施上的障碍再加上疾病相关的宣传教育滞后,以至于SMA遗传优生检查项目在国内长期处于空白状态,进而导致每年有大量的SMA患儿出生,给社会及患者家庭造成了沉重的负担。基于国情现状,笔者认为基于定量PCR平台的SMA携带者检测技术更适合于作为筛查技术在我国普及应用,建立并推广经充分验证的标准化实施方案是开展SMA优生筛查的必要前提。
[1]Wilson RB,Ogino S.Carrier frequency of spinal muscular atrophy[J].Lancet,2008,372:1542.
[2]Munsat TL,Davies KE.International SMA consortium meeting.(26-28 June 1992, Bonn, Germany)[J].Neuromuscul Disord,1992,2:423-428.
[3]Roy N,Mc Lean MD,Besner-Johnston A,et al.Refined physical map of the spinal muscular atrophy gene (SMA)region at 5q13 based on YAC and cosmid contiguous arrays[J].Genomics,1995,26:451-460.
[4]Prior TW.Carrier screening for spinal muscular atrophy[J].Genet Med,2008,10:840-842.
[5]ACOG committee opinion No.432:spinal muscular atrophy[J].Obstet Gynecol,2009,113:1194-1196.
[6]Lunn MR,Wang CH.Spinal muscular atrophy[J].Lancet,2008,371:2120-2133.
[7]肖雪,蔡兰云,王蕊艳,等.Dhplc技术在儿童型脊髓性肌萎缩症的基因诊断及携带者基因筛查中的应用[J].江西医学院学报,2009:99-102.
[8]Chen WJ,Wu ZY,Lin MT,et al.Molecular analysis and prenatal prediction of spinal muscular atrophy in Chinese patients by the combination of restriction fragment length polymorphism analysis,denaturing high-performance liquid chromatography,and linkage analysis[J].Arch Neurol,2007,64:225-231.
[9]Schouten JP,McElgunn CJ,Waaijer R,et al.Relative quantification of 40 nucleic acid sequences by multiplex ligation-dependent probe amplification[J].Nucleic Acids Res,2002,30:e57.
[10]He J,Zhang QJ,Lin QF,et al.Molecular analysis of SMN1,SMN2,NAIP,GTF2H2,and H4F5 genes in 157 Chinese patients with spinal muscular atrophy[J].Gene,2013,518:325-329.
[11]Su YN,Hung CC,Lin SY,et al.Carrier screening for spinal muscular atrophy(SMA)in 107,611 pregnant women during the period 2005-2009:a prospective population-based cohort study[J].PLoS One,2011,6:e17067.
[12]Goncalves-Rocha M,Oliveira J,Rodrigues L,et al.New approaches in molecular diagnosis and population carrier screening for spinal muscular atrophy[J].Genet Test Mol Biomarkers,2011,15:319-326.
[13]Weaver S,Dube S,Mir A,et al.Taking qPCR to a higher level:Analysis of CNV reveals the power of high throughput qPCR to enhance quantitative resolution[J].Methods,2010,50:271-276.
[14]Feldkotter M,Schwarzer V,Wirth R,et al.Quantitative analyses of SMN1 and SMN2 based on real-time lightCycler PCR:fast and highly reliable carrier testing and prediction of severity of spinal muscular atrophy[J].Am J Hum Genet,2002,70:358-368.
[15]OO Soloviov GL, SS Podlesnaya,LA Livshits.Implementation of the quantitative Real-Time PCR for the molecular-genetic diagnostics of spinal muscular atrophy[J].Biopolymers and Cell,2010,26:51-55.
[16]Maranda B,Fan L,Soucy JF,et al.Spinal muscular atrophy:clinical validation of a single-tube multiplex real time PCR assay for determination of SMN1 and SMN2 copy numbers[J].Clin Biochem,2012,45:88-91.
[17]Gomez-Curet I,Robinson KG,Funanage VL,et al.Robust quantification of the SMN gene copy number by real-time Taq Man PCR[J].Neurogenetics,2007,8:271-278.
[18]Li SW,Lin K,Ma P,et al.FADS Gene Polymorphisms Confer the Risk of Coronary Artery Disease in a Chinese Han Population through the Altered Desaturase Activities:Based on High-Resolution Melting Analysis[J].PLoS One,2013,8:e55869.
[19]Cui G,Zhang L,Xu Y,et al.Development of a high resolution melting method for genotyping of risk HLA-DQA1 and PLA2R1 alleles and ethnic distribution of these risk alleles[J].Gene,2013,514:125-130.
[20]Chen WJ,Dong WJ,Lin XZ,et al.Rapid diagnosis of spinal muscular atrophy using high-resolution melting analysis[J].BMC Med Genet,2009,10:45.
[21]Morikawa S,Harahap IS,Kaszynski RH,et al.Diagnosis of spinal muscular atrophy via high-resolution melting analysis symmetric polymerase chain reaction without probe:a screening evaluation for SMN1 deletions and intragenic mutations[J].Genet Test Mol Biomarkers,2011,15:677-684.
[22]Er TK,Kan TM,Su YF,et al.High-resolution melting(HRM)analysis as a feasible method for detecting spinal muscular atrophy via dried blood spots[J].Clin Chim Acta,2012,413:1781-1785.
[23]Dobrowolski SF,Pham HT,Downes FP,et al.Newborn screening for spinal muscular atrophy by calibrated shortamplicon melt profiling[J].Clin Chem,2012,58:1033-1039.
[24]Vogelstein B,Kinzler KW.Digital PCR[J].Proc Natl Acad Sci U S A,1999,96:9236-9241.
[25]Zhong Q,Bhattacharya S,Kotsopoulos S,et al.Multiplex digital PCR:breaking the one target per color barrier of quantitative PCR[J].Lab Chip,2011,11:2167-2174.
[26]Sugarman EA,Nagan N,Zhu H,et al.Pan-ethnic carrier screening and prenatal diagnosis for spinal muscular atrophy:clinical laboratory analysis of>72,400 specimens[J].Eur J Hum Genet,2012,20:27-32.
[27]Muralidharan K,Wilson RB,Ogino S,et al.Population carrier screening for spinal muscular atrophy a position statement of the association for molecular pathology[J].J Mol Diagn,2011,13:3-6.
[28]Sukenik-Halevy R,Pesso R,Garbian N,et al.Large-scale population carrier screening for spinal muscular atrophy in Israel-effect of ethnicity on the false-negative rate[J].Genet Test Mol Biomarkers,2010,14:319-324.

