油菜开花时间遗传调控的研究进展
谭勇俊,鲁军雄,刘忠松
(湖南农业大学农学院,长沙410128)
开花是显花植物生长过程中一个重要转折点,代表植物从营养生长转向生殖生长。选择合适的开花时机对植物避开不利的自然环境条件和获得尽可能多的后代具有重要意义[1]。
油菜与模式植物拟南芥同属十字花科,具有较近的亲缘关系[2]。研究表明,拟南芥开花受到温度、光周期和生长年限、营养条件等因素影响,调控网络复杂,参与调控的基因有170多个[3]。根据信号因子的来源以及参与基因的不同,将拟南芥开花调控途径分为 5 条[3,4],即:春化途径(vernalization pathway)、自主途径(autonomous pathway)、光周期途径(photoperiod pathway)、赤霉素途径(gibberellin pathway)和年龄途径(aging pathway)。
春化是指某些植物需经过一段时间的低温处理才能正常开花的现象。春化途径中的2个关键基因是 FLC[5]和 FRI[6],FLC 编码具有 MADS - box 结构的转录因子,抑制开花调控途径下游的 FT、SOC1、LFY等基因的表达从而抑制开花。
光周期途径是光周期控制开花的一条途径。其中处于中心位置的基因是CO(CONSTANS)。CO基因在叶片中表达,其表达量直接或间接地受多种感知不同波长的色素的控制,具有明显的昼夜周期性[7]。CO基因的蛋白产物为一种转录调控因子,能促进叶片中FT基因转录。叶片中FT基因编码的蛋白合成后被转运到茎尖分生组织,促进开花[8]。
1 油菜及其近缘植物开花时间的QTL定位
对油菜开花时间的研究主要集中于白菜型油菜和甘蓝型油菜。由于甘蓝型油菜是白菜与甘蓝种间杂交后加倍形成的异源四倍体,含有来源于甘蓝的基因组(C基因组),因此,有关甘蓝的研究成果对甘蓝型油菜的相关研究具有参考意义。早期主要通过分离群体的表型分析来研究油菜开花时间的控制基因数目、遗传力以及各基因之间的效应。Zaman等[9]的研究认为,开花时间为多基因控制,且存在主效基因。在分子标记以及QTL定位方法提出后,研究者们纷纷将其应用于油菜及相关植物开花时间的研究:Teutonico等[10]在白菜中定位出2个主效QTL;Ferreira等[11]在甘蓝型油菜LG9(该连锁群对应A2染色体)上定位了1个主效QTL;Carmago等[12]在甘蓝中也检测到3个开花时间QTL。所进行的QTL定位使用的分子标记主要是RFLP和一些形态学标记,分离群体主要为临时群体F2群体,同时,因标记类型的限制,用于定位的标记数也较少(不到 200个)。随着研究的深入,SSR、AFLP、SCAR、SRAP、CAPS、SNP、DArT 等多种类型标记被应用于开花时间QTL定位,定位所使用的标记数量也迅速增加(几百至上千个),且主要使用DH、RIL群体等永久群体并在多个环境条件下考察表型数据,从而能区分环境方差以及实验误差的影响。
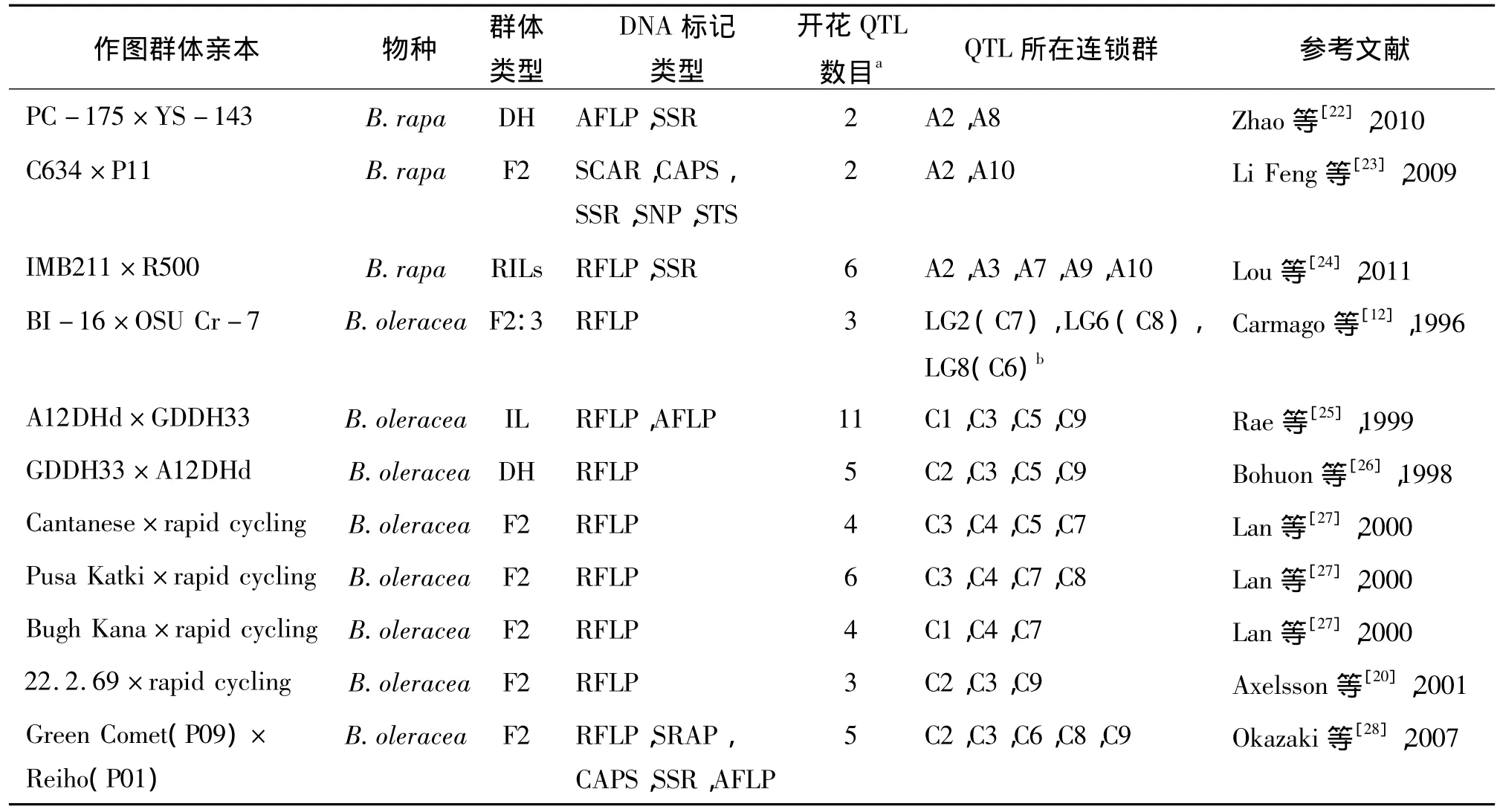
在甘蓝型油菜大部分定位结果中,A2、A3、C2、C3、C9连锁群存在开花时间主效QTL,并且QTL的置信区间重合或非常接近;其次,A1、A7、A10、C6 等染色体也在多个试验中检测到含有开花时间QTL。
已有的油菜开花时间QTL定位结果中存在QTL认定标准不统一的问题。因使用的计算软件、算法以及对数据的处理不一致,不同研究者得到的QTL数量存在较大差异,其中表现最突出的是在对微效QTL的认定上。
甘蓝型油菜及其亲本部分已发表的开花期QTL定位结果统计如表1。

表1 甘蓝型油菜及其亲本种开花时间QTL定位结果
(续表1)
2 油菜开花关键基因的克隆
油菜及其近缘植物中开花相关基因的研究主要集中于FLC、CO等少数几个关键基因。
2.1 FLC基因
FLC即FLOWERING LOCUS C,属于 MADS-box转录因子家族,主要参与春化途径,对开花起负调控作用。Kole等[29]在白菜的回交分离群体中发现,FLC基因序列与春化主效 QTL共分离,推断FLC基因在白菜中与在拟南芥中功能相同。Tadege等[30]将拟南芥 FLC基因转入甘蓝型油菜中,发现拟南芥FLC基因的表达对甘蓝型油菜的开花时间具有推迟作用;而将甘蓝型油菜cDNA文库中分离得到的5个拟南芥FLC基因的同源序列进行表达分析发现,其表达量与春化及开花时间相关,转入这些基因序列的拟南芥植株的开花时间也出现推迟,说明FLC基因在拟南芥与甘蓝型油菜中功能比较保守且作用机理相近。Kim等利用白菜BAC测序获得的基因全长进行的表达分析以及转化拟南芥实验也验证了这一结论[31]。但 Razi等[32]对甘蓝 QTL区间与FLC拷贝的位置的研究认为,FLC参与春化作用,但并不是造成甘蓝开花时间差异的主要基因。在甘蓝型油菜中,至今已找到FLC基因的9个拷贝,这些拷贝已分别定位于 A2、A3、A10、C2、C3 和C9等6条染色体上,其中A3、C3和C9染色体各有2个拷贝[33],A10染色体的FLC基因拷贝是A10染色体上开花时间QTL的候选基因[34]。
2.2 FRI基因
FRI正向调控FLC基因的表达,对开花起负调控作用。早期通过对油菜和拟南芥的比较作图发现,开花时间QTL所在位置与拟南芥FRI基因所在的4号染色体顶端存在共线性关系[35]。目前,已经在油菜及其相关物种中克隆得到了多个FRI基因拷贝。Wang等[36]在甘蓝型油菜BAC文库中克隆得到了4个FRI基因的拷贝,发现其中定位于A3染色体的拷贝位于一个开花时间QTL的置信区间内;进一步的研究发现,多个材料中该拷贝的序列多态性与对应材料的开花时间相关。Irwin等[37]在甘蓝中也鉴定出了2个FRI拷贝。
2.3 CO 基因
CO即CONSTANS,是含有锌指结构的转录因子,主要参与光周期途径,是开花的正向调控因子。Robert等[38]以拟南芥CO基因序列为探针,从甘蓝型油菜基因组文库中分离了4个CO基因拷贝,均具有特异性的锌指结构;定位发现N10(A10)和N19(C9)连锁群各有2个拷贝;转化拟南芥对应突变体发现,其中位于A10染色体的一个拷贝可使植株提前开花。Martynov等[39]通过同源克隆得到白菜CO基因的2个同源序列;Lagercrantz等[40]在黑芥中定位了CO基因的2个拷贝并使用RT-PCR的方法检测到两者都表达。进一步研究发现,仅其中拷贝BniCOa对开花时间有影响,而拷贝BniCOb虽表达,但不影响开花时间[41]。
2.4 FT 基因
FT基因即FLOWERING LOCUS T基因,它在叶片中表达的产物被运输到生长点,促进开花,是一直寻找的“成花素”。目前,FT基因在甘蓝型油菜中已克隆出6个拷贝,其中A2、C2染色体各1个拷贝,A7、C6染色体各有 2个拷贝[42]。A2染色体上FT基因拷贝在白菜和甘蓝型油菜中都转录,C2染色体拷贝在甘蓝和甘蓝型油菜中因插入以及甲基化修饰而不转录,A7与C6染色体上FT基因拷贝在冬性油菜中发生沉默[43],可能与油菜感光性的强弱有关。
除以上几个与开花相关的主要基因外,Lee等[44]在多个白菜品系中克隆并分析SVP基因,发现SVP基因影响开花时间,但其表达量不受春化的影响。An等[45]通过使用酵母双杂交技术发现甘蓝的AP1基因与SVP之间存在互作,这与拟南芥中情况一致。Baum等[46]分析了LEAFY基因在十字花科植物中的差异以及进化情况。
3 展望
油菜开花时间是一个复杂的数量性状,受光周期、温度、激素水平、营养条件等多因素的影响。前期研究因相关信息的缺乏,主要以拟南芥等模式植物研究结果为参考,具有一定价值。但油菜是多倍体,经历了多倍化,与拟南芥存在显著差异,依赖拟南芥的研究结果具有局限性。白菜、甘蓝基因组测序的完成以及甘蓝型油菜全基因组测序的开展,为进一步的研究提供了更加有利的条件。
油菜中已经定位了大量开花时间QTL,但其中只有少数进行了解析[48,49],这与物种相对复杂的基因组背景、标记定位精度以及现有技术的不足等有关。相关物种全基因组测序结果、DArT和SNP等高通量标记、转录组测序以及遗传转化体系的成熟将大大加速QTL的解析。
培育早熟品种是当前油菜育种的主要目标之一。而油菜开花时间调控网络非常复杂,某一基因可能参与多条调控途径,同一基因的不同拷贝可能具有不同的功能,因此,在透彻研究开花调控机理的基础上,对开花控制途径中的多个基因进行选择或者修饰,应能培育出理想的品种。
[1] Amasino R.Seasonal and developmental timing of flowering[J].Plant J,2010,61(6):1001 -1013.
[2] Yang YW,Lai KN,Tai PY,et al.Rates of nucleotide substitution in angiosperm mitochondrial DNA sequences and dates of divergence between Brassica and other angiosperm lineages[J].J Mol Evol,1999,48(5):597 -604.
[3] Fornara F,A De Montaigu,Coupland G.SnapShot:Control of flowering in Arabidopsis[J].Cell,2010,141(3):550,550 e1-2.
[4] Srikanth A,Schmid M.Regulation of flowering time:all roads lead to Rome[J].Cell Mol Life Sci,2011,68(12):2013-2037.
[5] Michaels SD,Amasino RM.FLOWERING LOCUS C encodes a novel MADS domain protein that acts as a repressor of flowering[J].Plant Cell,1999,11(5):949-956.
[6] Johanson U,West J,Lister C,et al.Molecular analysis of FRIGIDA,a major determinant of natural variation in Arabidopsis flowering time[J].Science,2000,290:344-347.
[7] Sawa M,Nusinow DA,Kay SA,et al.FKF1 and GIGANTEA complex formation is required for day-length measurement in Arabidopsis[J].Science Signalling,2007,318:261.
[8] Corbesier L,Vincent C,Jang S,et al.FT protein movement contributes to long-distance signaling in floral induction of Arabidopsis[J].Science,2007,316:1030 -1033.
[9] Zaman MW.Introgression in Brassica napus for adaptation to the growing conditions in Bangladesh[J].Theor Appl Genet,1989,77(5):721 -728.
[10] Teutonico RA,Osborn TC.Mapping loci controlling vernalization requirement in Brassica rapa[J].Theor Appl Genet,1995,91(8):1279 -1283.
[11] Ferreira ME,Satagopan J,Yandell BS,et al.Mapping loci controlling vernalization requirement and flowering time in Brassica napus[J].Theor Appl Genet,1995,90(5):727-732.
[12] Camargo LEA,Osborn TC.Mapping loci controlling flowering time in Brassica oleracea [J].Theor Appl Genet,1996,92(5):610-616.
[13] Butruille DV,Guries RP,Osborn TC.Linkage analysis of molecular markers and quantitative trait loci in populations of inbred backcross lines of Brassica napus L.[J].Genetics,1999,153(2):949-964.
[14] Zhao J,Becker HC,Zhang D,et al.Oil content in a European× Chinese rapeseed population[J].Crop Science,2005,45(1):51 -59.
[15] Udall JA,Quijada PA,Lambert B,et al.Quantitative trait analysis of seed yield and other complex traits in hybrid spring rapeseed(Brassica napus L.)2.Identification of alleles from unadapted germplasm[J].Theor Appl Genet,2006,113(4):597 -609.
[16] Delourme R,Falentin C,Huteau V,et al.Genetic control of oil content in oilseed rape(Brassica napus L.)[J].Theor Appl Genet,2006,113(7):1331 -1345.
[17] Long Y,Shi J,Qiu D,et al.Flowering time quantitative trait loci analysis of oilseed brassica in multiple environments and genomewide alignment with Arabidopsis[J].Genetics,2007,177(4):2433-2444.
[18] Cai CC,Tu JX,Fu TD,et al.The genetic basis of flowering time and photoperiod sensitivity in rapeseed(Brassica napus L.)[J].Genetika,2008,44(3):381 -388.
[19] Raman H,Raman R,Eckermann P,et al.Genetic and physical mapping of flowering time loci in canola(Brassica napus L.)[J].Theor Appl Genet,2013,126(1):119-132.
[20] Axelsson T,Shavorskaya O,Lagercrantz U.Multiple flowering time QTLs within several Brassica species could be the result of duplicated copies of one ancestral gene[J].Genome,2001,44(5):856 -864.
[21] Lou P,Zhao J,Kim J S,et al.Quantitative trait loci for flowering time and morphological traits in multiple populations of Brassica rapa[J].J Exp Bot,2007,58(14):4005-4016.
[22] Zhao J,Kulkarni V,Liu N,et al.BrFLC2(FLOWERING LOCUS C)as a candidate gene for a vernalization response QTL in Brassica rapa[J].J Exp Bot,2010,61(6):1817-1825.
[23] Li F,Kitashiba H,Inaba K,et al.A Brassica rapa linkage map of EST-based SNP markers for identification of candidate genes controlling flowering time and leaf morphological traits[J].DNA Res,2009,16(6):311 -323.
[24] Lou P,Xie Q,Xu X,et al.Genetic architecture of the circadian clock and flowering time in Brassica rapa[J].Theor Appl Genet,2011,123(3):397 -409.
[25] Rae AM,Howell EC,Kearsey MJ.More QTL for flowering time revealed by substitution lines in Brassica oleracea[J].Heredity(Edinb),1999,83(5):586 -596.
[26] Bohuon Ejr,Ramsay LD,Craft JA,et al.The association of flowering time quantitative trait loci with duplicated regions and candidate loci in Brassica oleracea[J].Genetics,1998,150(1):393 -401.
[27] Lan TH,Paterson AH.Comparative mapping of quantitative trait loci sculpting the curd of Brassica oleracea[J].Genetics,2000,155(4):1927 -1954.
[28] Okazaki K,Sakamoto K,Kikuchi R,et al.Mapping and characterization of FLC homologs and QTL analysis of flowering time in Brassica oleracea[J].Theor Appl Genet,2007,114(4):595 -608.
[29] Kole C,Quijada P,Michaels SD,et al.Evidence for homology of flowering-time genes VFR2 from Brassica rapa and FLC from Arabidopsis thaliana[J].Theor Appl Genet,2001,102(2):425 -430.
[30] Tadege M,Sheldon CC,Helliwell CA,et al.Control of flowering time by FLC orthologues in Brassica napus[J].Plant J,2001,28(5):545-553.
[31] Kim SY,Park BS,Kwon SJ,et al.Delayed flowering time in Arabidopsis and Brassica rapa by the overexpression of FLOWERING LOCUS C(FLC)homologs isolated from Chinese cabbage(Brassica rapa L.:ssp.pekinensis)[J].Plant Cell Rep,2007,26(3):327 -336.
[32] Razi H,Howell EC,Newbury HJ,et al.Does sequence polymorphism of FLC paralogues underlie flowering time QTL in Brassica oleracea[J].Theor Appl Genet,2008,116(2):179-192.
[33] Udall JA,Quijada PA,Osborn TC.Detection of chromosomal rearrangements derived from homologous recombination in four mapping populations of Brassica napus L.[J].Genetics,2005,169(2):967 -979.
[34] Hou J,Long Y,Raman H,et al.A Tourist-like MITE insertion in the upstream region of the BnFLC.A10 Gene is associated with vernalization requirement in rapeseed(Brassica napus L.)[J].BMC Plant Biology,2012,12(1):238.
[35] Osborn TC,Kole C,Parkin IA,et al.Comparison of flowering time genes in Brassica rapa,B.napus and Arabidopsis thaliana[J].Genetics,1997,146(3):1123 -1129.
[36] Wang N,Qian W,Suppanz I,et al.Flowering time var-iation in oilseed rape(Brassica napus L.)is associated with allelic variation in the FRIGIDA homologue BnaA.FRI.a[J].J Exp Bot,2011,62(15):5641 -5658.
[37] Irwin JA,Lister C,Soumpourou E,et al.Functional alleles of the flowering time regulator FRIGIDA in the Brassica oleracea genome[J].BMC Plant Biol,2012,12:21.
[38] Robert LS,Robson F,Sharpe A,et al.Conserved structure and function of the Arabidopsis flowering time gene CONSTANS in Brassica napus [J].Plant Mol Biol,1998,37(5):763-772.
[39] Martynov VV,Khavkin EE.Polymorphism of the CONSTANS gene in Brassica plants[J].Russian Journal of Plant Physiology,2005,52(2):242-248.
[40] Lagercrantz U,Axelsson T.Rapid evolution of the family of CONSTANS LIKE genes in plants[J].Mol Biol Evol,2000,17(10):1499-1507.
[41] Sjodin P,Hedman H,Shavorskaya O,et al.Recent degeneration of an old duplicated flowering time gene in Brassica nigra[J].Heredity(Edinb),2007,98(6):375-384.
[42] Wang J,Long Y,Wu B,et al.The evolution of Brassica napus FLOWERING LOCUS T paralogues in the context of inverted chromosomal duplication blocks[J].BMC Evol Biol,2009,9:271.
[43] Wang J,Hopkins CJ,Hou J,et al.Promoter variation and transcript divergence in Brassicaceae lineages of FLOWERING LOCUS T[J].PLoS One,2012,7(10):e47127.
[44] Lee JH,Park SH,Lee JS,et al.A conserved role of SHORT VEGETATIVE PHASE(SVP)in controlling flowering time of Brassica plants[J].Biochim Biophys Acta,2007,(7-8):455-461.
[45] An YH,Li XF,Zhu JX,et al.The screening of interaction factors with BoCAL and BoAP1 related to curd formation[J].Journal of Molecular Cell Biology,2007,40(2):130-136.
[46] Baum DA,Yoon HS,Oldham RL.Molecular evolution of the transcription factor LEAFY in Brassicaceae[J].Mol Phylogenet Evol,2005,37(1):1 -14.
[47] Wang X,Wang H,Wang J,et al.The genome of the mesopolyploid crop species Brassica rapa[J].Nat Genet,2011,43(10):1035 -1039.
[48] 王 晶.甘蓝型油菜中控制开花的主效 QTL簇的解析和“成花素”基因家族的克隆及分析[D].武汉:华中农业大学农学院,2009.
[49] 邹潇潇.甘蓝型油菜开花关键调控因子BnFLC基因家族的克隆和分析[D].武汉:华中农业大学农学院,2012.

