小麦R2R3-MYB转录因子TaMYB3-4D的克隆及功能分析
王延谦,张 波,陈文杰,刘登才,刘宝龙,张怀刚*
(1 青海省作物分子育种重点实验室,西宁 810001;2 中国科学院大学,北京 100049;3 中国科学院高原生物适应与进化重点实验室,西宁 810001)
小麦R2R3-MYB转录因子TaMYB3-4D的克隆及功能分析
王延谦1,2,3,张 波1,3,陈文杰1,3,刘登才1,3,刘宝龙1,3,张怀刚1,3*
(1 青海省作物分子育种重点实验室,西宁 810001;2 中国科学院大学,北京 100049;3 中国科学院高原生物适应与进化重点实验室,西宁 810001)
MYB转录因子是植物最大的转录因子家族之一,广泛参与植物各种生理生化过程。该研究通过对小麦基因组测序数据库进行同源搜索,利用电子克隆技术从紫色籽粒小麦品种‘高原115’中分离得到了一个新的MYB基因TaMYB3-4D。结果表明,TaMYB3-4D仅含有一个内含子,其编码蛋白含有2个连续的MYB结构域,为典型的R2R3-MYB蛋白。TaMYB3-4D系统发生关系上与调控花青素合成的MYB基因亲缘关系较近。TaMYB3-4D与bHLH基因ZmR瞬时表达能够诱导白色胚芽鞘中花青素的合成。此外,TaMYB3-4D基因仅在‘高原115’含花青素的种皮和胚芽鞘中表达,在根、茎、叶中均未表达。研究表明,TaMYB3-4D基因是一个具有调控花青素合成代谢功能的R2R3-MYB基因,很有可能参与小麦花青素的生物合成。
小麦;MYB转录因子;花青素
MYB转录因子是植物最大的转录因子家族之一。这些MYB转录因子均含有一个或多个较为保守的DNA结合域——MYB结构域,每个MYB结构域(50~53氨基酸残基)含有3个规则分布的色氨酸残基[1-3]。根据所含MYB结构域的数目与位置,植物中的转录因子可分为4个亚类:4R-MYB(含4个相连的MYB结构域);3R-MYB(R1R2R3-MYB);R2R3-MYB和MYB-related protein(含1个或2个间隔的MYB结构域)[4-6]。最早的MYB基因(υ-myb)是1982年从鸟类的原癌病毒中发现的[7]。在植物中发现的第一个MYB转录因子是ZmC1[8]。在拟南芥、玉米、水稻、矮牵牛、葡萄、苹果等植物中已经发现了大量的MYB转录因子,其中R2R3-MYB基因的比例较大[9-12]。拟南芥中已发现125个R2R3-MYB基因,占拟南芥基因组编码基因的0.2%~0.6%[13]。R2R3-MYB转录因子广泛参与植物的生理生化过程,如调控初级或次生代谢途径,控制细胞分化和细胞周期,抵御植物生长发育中的各种生物或非生物胁迫[14]等。植物中分离到并进行功能验证的R2R3-MYB转录因子大多调控初级和次生代谢途径,特别是调控类黄酮类次生代谢途径。如拟南芥中AtMYB11调控所有组织中黄酮醇的生物合成,AtMYB90调控营养组织中花青素的生物合成,AtMYB123调控种皮的原花青素合成[15-17]。
花青素属于重要的次生代谢产物—类黄酮化合物,是一种水溶性色素,赋予植物器官和组织五颜六色,具有吸引昆虫帮其传粉的作用。植物中的花青素还具有保护植物免受紫外线的伤害的功能。同时其对人类也具有重要的作用,如其抗氧化、抗病毒、抗癌、抗衰老、增强人体免疫力等功效[18-19]。在植物中发现的第一个MYB转录因子ZmC1就是调控玉米籽粒的颜色[8]。普通小麦中与花青素合成代谢相关的性状有紫色花药、红色叶耳、紫色胚芽鞘、紫色茎干、红粒、紫粒和蓝粒等。这些性状的QTL都进行了初步定位[20-21]。小麦的红粒性状已经定位到小麦的3AL、3BL、3DL染色体组上,并且克隆到了MYB类转录因子TaMYB10-3A、TaMYB10-3B和TaMYB10-3D基因,利用自然群体证明了目标基因是红粒性状的主效基因[22]。与红粒性状类似,控制小麦胚芽鞘紫色基因定位于7A、7B、7D的短臂上,距着丝粒15~20 cM,在4BL上还有一个加性位点,推测也是由MYB类转录因子控制[20]。李振声先生的蓝粒小麦的主效基因定位到长穗偃麦草的4E染色体上,并对结构基因查尔酮合成酶(CHS)和类黄酮-3′5′-羟化酶(F3′5′H)在蓝粒小麦花青素合成代谢中的作用进行了分析,但这些基因都不是蓝粒性状的主效基因[23]。
本研究以富含花青素的紫色籽粒小麦品种——‘高原115’为研究材料,从中分离克隆出一个R2R3-MYB转录因子TaMYB3-4D,并进行功能分析,为小麦中花青素合成代谢的深入研究奠定基础。
1 材料和方法
1.1 材 料
供试小麦品种‘高原115’是中国科学院西北高原生物研究所培育的普通小麦品种,2001年通过了青海省农作物品种审定委员会审定。在其籽粒和胚芽鞘中都有明显的花青素积累,表现为紫色。在温室条件下培养3 d后剪取胚芽鞘,10 d后剪取根和叶片。大田种植情况下,开花后21 d分别剪取茎秆、叶片及未成熟的种子。所有样品液氮冻存后保存于-80 ℃中用于提取DNA和RNA。瞬时表达所用小麦品种‘中国春’由青海省作物分子育种重点实验室保存,‘中国春’的胚芽鞘根据Himi等[24]的流程准备。
1.2 植物基因组DNA、总RNA及cDNA准备
参照Yan等[25]改良的CTAB法提取‘高原115’的基因组DNA。采用植物总RNA快速提取试剂盒RNAprep Pure Plant Kit(Tiangen)提取‘高原115’的根、茎、叶、未成熟种子以及胚芽鞘的总RNA。用微量紫外检测仪NANODROP2000C(Thermo)测量DNA及RNA样品浓度。用反转录试剂盒RevertAid First Strand cDNA Synthesis Kit(Thermo)将1 μg植物总RNA反转录为cDNA。
1.3 TaMYB3-4D基因的克隆
玉米中ZmC1是植物中克隆到的第一个与花青素合成代谢相关的MYB类转录因子,用ZmC1基因核苷酸序列在普通小麦基因组数据库(https://urgi.versailles.inra.fr/blast/blast.php)中进行同源搜索,发现在4DL上存在一个高度同源的重叠群:IWGSC_chr4DL_V2_ab_k71_contigs_longerthan_200_14366997。通过对核苷酸序列的生物学信息分析,将目标基因命名为TaMYB3-4D,设计了1对扩增TaMYB3-4D基因编码区的特异引物Tamyb3-4D-F和Tamyb3-4D-R(表1)。
TaMYB3-4D编码区的扩增使用高保真DNA聚合酶(Thermo-Fisher Scientific)在GeneAmp PCR System 9700(Applied Biosystems)PCR扩增仪进行。PCR程序为:98 ℃预变性2 min;98 ℃变性15 s,65 ℃退火30 s,72 ℃延伸30 s,35个循环;72 ℃延伸10 min。PCR反应产物在1.0%琼脂糖凝胶上进行电泳分离检测,将目的带切胶并用琼脂糖凝胶回收试剂盒Tiangen TIANgel Midi Purification Kit(天根,Tiangen)回收目的基因。将回收的PCR产物连接至pGEM-T Easy(Promega Corporation)载体上,转化大肠杆菌DH5α,阳性克隆送至华大公司测序。
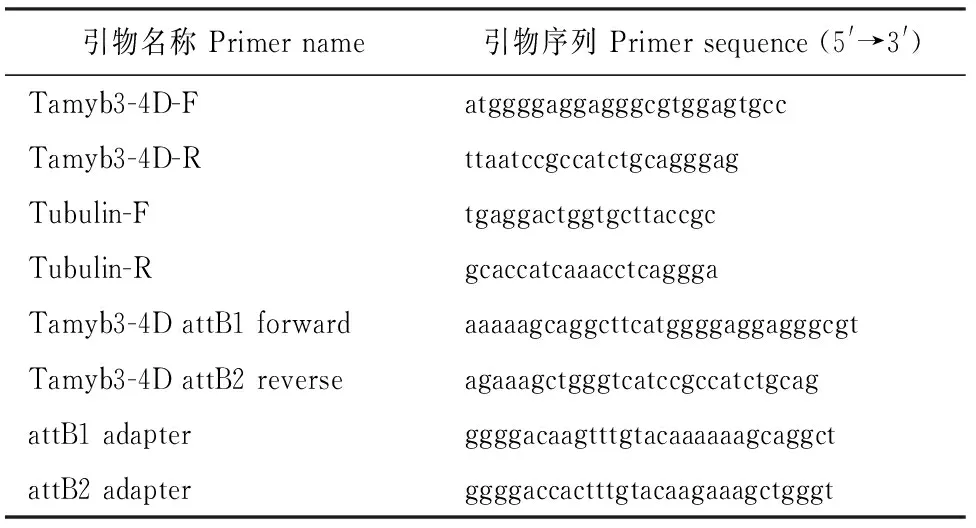
表1 引物名称及序列
1.4 生物信息学分析
使用Vector NTI 10软件(Invitrogen)进行序列拼接及比对。利用Primer Primer 5.0软件进行引物设计。使用MEGA 4.0构建MYB蛋白的系统进化树[26]。利用Gene Structure Display Server(http://gsds.cbi.pku.edu.cn/) 绘制基因结构图。
1.5 瞬时表达分析
利用Gateway克隆技术两步法将TaMYB3-4D克隆到中间载体pDONR207中,使用引物分别为Tamyb3-4D attB1 forward和Tamyb3-4D attB2 reverse;attB1 adapter 和 attB2 adapter。然后使用LR反应将TaMYB3-4D构建到含有玉米ubiquitin启动子的瞬时表达载体pBract214,获得瞬时表达载体pBract214-TaMYB3-4D(Gateway Cloning Kit,Invitrogen)。具体操作步骤参照Gateway试剂盒说明书。pBract214-ZmR(玉米bHLH转录因子)和pBract214-ZmC1(玉米MYB转录因子)为青海省作物分子育种重点实验室保存,构建方法与pBract214-TaMYB3-4D相同。瞬时表达质粒通过基因枪轰击转入白色的中国春胚芽鞘中[27]。胚芽鞘在光照培养箱中培养16 h后,在实体显微镜下观察并照相。
1.6 RT-PCR分析
以微管蛋白基因Tubulin为内标基因,引物为Tubulin-F和Tubulin-R(表1)。使用特异性引物Tamyb3-4D-F和Tamyb3-4D-R进行RT-PCR,检测TaMYB3-4D基因在‘高原115’不同组织(根、茎、叶、未成熟种子及胚芽鞘)中的表达情况。
2 结果与分析
2.1 TaMYB3-4D基因全长序列的克隆
利用基因全长特异引物TaMYB3-4D-F和TaMYB3-4D-R(表1),在‘高原115’基因组DNA和种皮cDNA中扩增得到了TaMYB3-4D基因组序列及CDS序列(图1,A、B)。测序结果表明,其基因组序列为791 bp;CDS序列为675 bp,编码224个氨基酸,与PCR产物的电泳结果是一致的。编码一个典型的R2R3-MYB蛋白,在R2和R3结构域中均含有保守的色氨酸残基W,这表明TaMYB3-4D很有可能是R2R3-MYB转录因子(图1,C)。
2.2 生物信息学分析
在模式植物拟南芥中,R2R3-MYB基因的功能已研究得较为全面。在本研究中,为了推测TaMYB3-4D基因的潜在功能,构建了拟南芥和小麦R2R3-MYB基因的系统进化树(图2)。TaMYB3-4D与拟南芥的AtMYB123/TT2以及小麦第3同源群的TaMYB10-A1、TaMYB10-B1、TaMYB10-D1基因关系最近,而这4个基因均调控类黄酮的生物合成。其中AtMYB123/TT2与拟南芥种皮的原花青素合成有关,TaMYB10-A1、TaMYB10-B1和TaMYB10-D1则与小麦红色种皮相关[28]。该结果暗示TaMYB3-4D基因可能与小麦类黄酮生物合成途径有关。与其它调控类黄酮生物合成的MYB转录因子的同源性分析显示,TaMYB3-4D推导的氨基酸序列与玉米ZmC1及小麦TaMYB3-4B的一致性最高,分别为57.51%和77.55%;与葡萄VvMYBA1一致性为30.86%,与拟南芥AtMYB123/TT2为38.46%,而与小麦TaMYB10达到41.10%。ZmC1是在玉米中发现的第一个与花青素合成有关的MYB转录因子,它的结构与功能已研究得较为清楚[29]。氨基酸序列的比对发现,TaMYB3-4D比ZmC1少了49个氨基酸残基;在保守区的一致性很高,仅有14个氨基酸残基的替换,同时在R3结构域中均存在一个与bHLH蛋白相互作用的结构原件;在C-末端有一个与ZmC1相似的转录激活区(图1,C和图3,A)。MYB基因结构图显示,TaMYB3-4B和TaMYB3-4D仅有1个内含子,而其它的MYB基因则有2个内含子(图3,B)。TaMYB3-4D一个内含子的丢失可能伴随着部分编码区的删除,导致其编码氨基酸序列的变短。
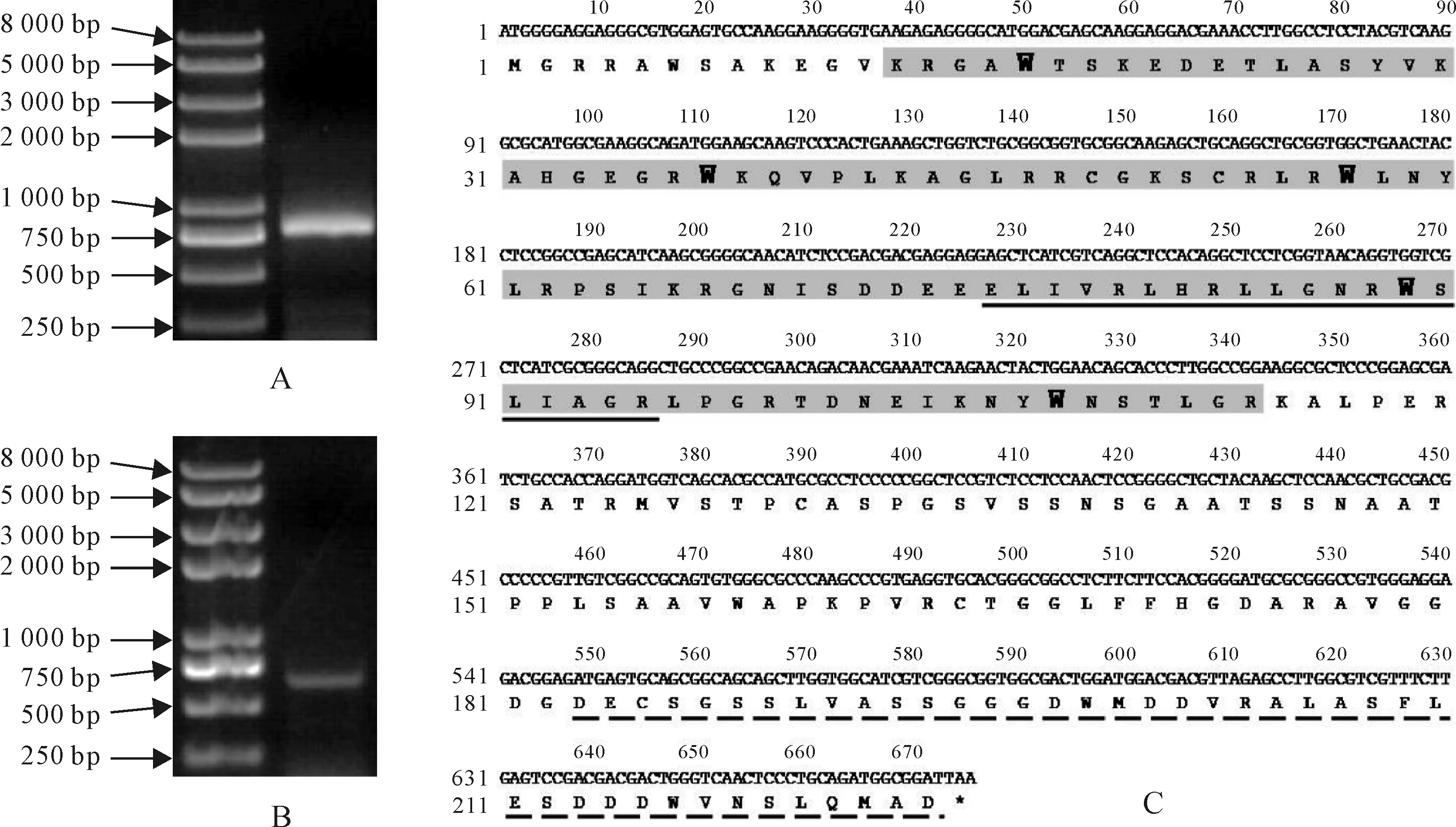
图1 TaMYB3-4D基因的PCR克隆、编码区序列及推测的氨基酸序列A.‘高原115’基因组DNA;B.‘高原115’种皮cDNA;C.TaMYB3-4D基因编码区及氨基酸序列:阴影部分为保守的MYB结构域,黑体字为MYB结构域保守的色氨酸(W)残基,横线部分表示与bHLH蛋白作用的D/ELx2R/Kx3Lx6Lx3R结构元件,虚线部分为可能的转录激活区

图2 拟南芥和小麦R2R3-MYB蛋白的系统进化关系AtMYB为拟南芥MYB基因,TaMYB为小麦MYB基因;圆点为本研究中TaMYB3-4D转录因子;方点为小麦第3同源群的MYB基因;箭头为拟南芥R2R3-MYB分类中的第5亚群
2.3 TaMYB3-4D基因的功能验证
在前人的研究中,玉米MYB转录因子ZmC1和bHLH转录因子ZmR的共表达能够诱导花青素的生物合成[27]。因此,在本试验中用ZmC1做对照,利用基因枪介导的瞬时表达技术来验证TaMYB3-4D的功能。从图4中可以看出,当TaMYB3-4D和ZmC1与ZmR共表达时,在‘中国春’胚芽鞘中均能诱导花青素的合成,这说明TaMYB3-4D是一个调控花青素生物合成的MYB转录因子。但是单独的TaMYB3-4D或者ZmC1在胚芽鞘中表达时,并没有观察到花青素斑点的存在(图4),这表明它们均需要与bHLH转录因子共同作用才能够激活结构基因的表达,从而诱导花青素的生物合成。
2.4 TaMYB3-4D在不同组织中的表达分析
以微管蛋白基因Tubulin为内标基因,通过RT-PCR分析了TaMYB3-4D基因在‘高原115’不同组织中的表达,结果只在未成熟的种子和胚芽鞘中检测到了TaMYB3-4D基因的表达,在其它组织(根、茎、叶)中并未检测到(图5)。而在田间和实验室的观察发现,‘高原115’的种皮及胚芽鞘均呈现紫色,这是否暗示着TaMYB3-4D基因可能与种皮及胚芽鞘中的花青素生物合成有关。

图3 植物中调控类黄酮生物合成的部分MYB基因的结构及氨基酸序列比对A为TaMYB3-4D与ZmC1和TaMYB3-4B氨基酸序列比对,箭头所示区域为MYB结构域,实线方框内表示与bHLH蛋白相互作用的结构元件,虚线方框内表示转录激活区;B为植物中调控类黄酮生物合成的部分MYB基因的结构,圆柱体代表外显子,线条代表内含子;NCBI登录号:VvMYBA1(AB111101)、AtMYB123(NC_003076)、ZmC1(AF320613)、TaMYB10-A1(AB599721)、TaMYB10-B1(AB599722)、TaMYB10-A1(AB191460)

图4 小麦胚芽鞘中基因枪介导的瞬时表达分析A.胚芽鞘部分放大图;B.瞬时表达后胚芽鞘全图;TaMYB3-4D、ZmR、ZmC1分别表示使用pBract214-TaMYB7D、pBract214-ZmR和pBract-ZmC1质粒轰击胚芽鞘;CK表示使用空载体pBract214轰击胚芽鞘
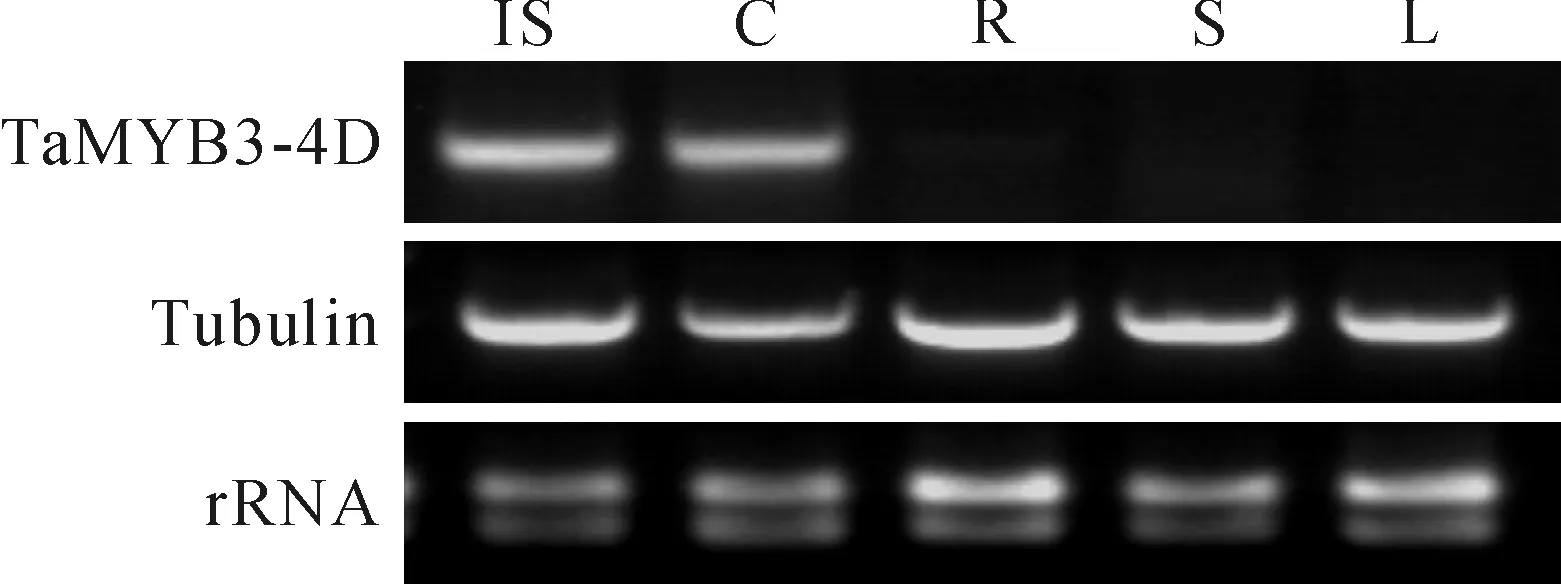
图5 TaMYB3-4D基因在‘高原115’不同组织中的表达情况IS.未成熟种子;C.胚芽鞘;R.根;S.茎;L.叶
3 讨 论
花青素能够保护植物免受紫外线的伤害,同时对人类也具有重要的作用,如其抗氧化、抗病毒、抗癌、抗衰老以及增强人体免疫力等。而作为调控花青素代谢的重要转录因子,小麦中与花青素合成代谢相关的一些MYB转录因子尚不能确定。因此,克隆小麦MYB基因并验证其功能,有助于解析小麦中花青素合成代谢的分子调控机理。
TaMYB3-4D与调控种皮中的花青素或原花青素代谢的拟南芥AtMYB123/TT2以及小麦TaMYB10进化关系最为接近,具有调控花青素合成代谢的保守结构域。在玉米bHLH转录因子ZmR的参与下TaMYB3-4D基因能够诱导胚芽鞘中花青素的合成,而单独TaMYB3-4D基因不能诱导花青素合成。Himi等[28]在TaMYB10以及其它参与调控类黄酮代谢的MYB转录因子的C-端中发现了一个保守的氨基酸模块IRTKAL/IRC,推测该模块在调控类黄酮的生物合成中起着重要的作用。在TaMYB3-4D推导的氨基酸序列中我们没有发现该保守模块,很可能这就是单独TaMYB3-4D基因不能调控花青素合成代谢的原因。当TaMYB3-4D
和玉米bHLH基因ZmR共表达时,大量的胚芽鞘细胞中积累了花青素,很有可能TaMYB3-4D与bHLH蛋白形成异源二聚体时能够弥补缺失的IRTKAL/IRC模块而调控花青素的代谢。这些都说明TaMYB3-4D是一个具有调控花青素合成代谢的MYB类转录因子。
TaMYB3-4D在普通小麦品种‘高原115’积累花青素合成的部位都有较高的表达量,而在无明显花青素合成代谢的部位或组织表达量较低。同时TaMYB3-4D具有调控花青素合成代谢的功能,因此该基因很有可能参与了调控‘高原115’中花青素合成代谢。在小麦中控制红粒性状的主效基因定位于3AL、3BL和3DL染色体,蓝粒性状定位于4E染色体,紫粒性状定位于2AL和7BL染色体,紫杆性状定位于7BS和7DS,紫色胚芽鞘定位于7AS、7BS和7DS染色体,紫色花药性状定位于7DS染色体上。这些已定位到的小麦中花青素合成代谢相关性状的主效基因还没有一个是在4D染色体上,因此TaMYB3-4D参与了小麦中花青素的合成代谢调控,但还不是小麦中花青素合成代谢相关性状的主效基因[30]。蓝粒小麦的主效基因定位于长穗偃麦草的4E染色体上[23],4E与普通小麦的第4同源群在进化关系上最为接近。4E染色体上有没有与TaMYB3-4D基因同源的MYB转录因子以及该转录因子对蓝粒性状形成中的作用还需要进一步的研究和验证。
本实验通过对小麦基因组测序数据库进行同源搜索,利用电子克隆技术从紫色籽粒小麦品种‘高原115’中分离得到了1个新的MYB基因TaMYB3-4D。TaMYB3-4D仅含有1个内含子,其编码蛋白含有两个连续的MYB结构域,为典型的R2R3-MYB蛋白。TaMYB3-4D系统发生关系上与调控花青素合成的MYB亲缘关系较近。TaMYB3-4D与bHLH基因ZmR瞬时表达能够诱导白色胚芽鞘中花青素的合成。此外,TaMYB3-4D基因仅在高含花青素的种皮和胚芽鞘中表达,在根、茎、叶中均未表达。这些结果表明,TaMYB3-4D是一个具有调控花青素合成代谢的MYB基因,很有可能参与小麦花青素的生物合成。
[1] GABRIELSEN O S,SENTENAC A,FROMAGEOT.Specific DNA binding domain by c-Myb:evidence for a double helix-turn-helix-related motif[J].Science,1991,253(5 024):1 140-1 143.
[2] JIA L,CLEGG M T,JIANG T.Evolutionary dynamics of the DNA-binding domains in putative R2R3-MYB genes identified from rice subspecies indica and japonica genomes[J].PlantPhysiology,2004,134(2):575-585.
[3] OGATA K S,MORIKAWA H,NAKAMURA A,etal.Solution structure of a specific DNA complex of the Myb DNA-binding domain with cooperative recognition helices[J].Cell,1994,79(4):639-648.
[4] DUBOS C,GROTEWOLD E,etal.MYB transcription factors inArabidopsis[J].TrendsinPlantScience,2010,15(10):573-581.
[5] JIN H,MARTIN C.Multifunctionality and diversity within the plant MYB-gene family[J].PlantMolecularBiology,1999,41(5):577-585.
[6] ROSINSKI J A,ATCHLEY W R.Molecular evolution of the Myb family of transcription factors:evidence for polyphyletic origin[J].JournalofMolecularEvolution,1998,46(1):74-83.
[7] KLEMPNAUER K H,GONDA T J,BISHOP J M.Nucleotide sequence of the retroviral leukemia genev-myband its cellular progenitorc-myb:the architecture of a transduced oncogene[J].Cell,1982,31(2):453-463.
[8] PAZ-ARES J,GHOSAL D,WIENAND U,etal.The regulatory c1 locus ofZeamaysencodes a protein with homology to myb proto-oncogene products and with structural similarities to transcriptional activators[J].TheEMBOJournal,1987,6(12):3 553-3 558.
[9] CHEN Y,YANG X,HE K,etal.The MYB transcription factor superfamily ofArabidopsis:expression analysis and phylogenetic comparison with the rice MYB family[J].PlantMolecularBiology,2006,60(1):107-124.
[10] DU H,ZHANG L,LIU L,etal.Biochemical and molecular characterization of plant MYB transcription factor family[J].Biochemistry(Mosccow),2009,74(1):1-11.
[11] VELASCO R,ZHARKIKH A,TROGGIO M,etal.A high quality draft consensus sequence of the genome of a heterozygous grapevine variety[J].PLoSOne,2007,2(12):e1326.
[12] RABINOWICZ P D,BRAUN E L,WOLFE A D,etal.Maize R2R3-MYB genes:sequence analysis reveals amplification in the higher plants[J].Genetics,1999,153(1):427-444.
[13] STRACKE R,WERBER M,WEISSHAAR B.The R2R3-MYB gene family inArabidopsisthaliana[J].CurrentOpinioninPlantBiology,2001,4(5):447-456.
[14] FELLER A,MACHEMER K,BRAUN E L,etal.Evolutionary and comparative analysis of MYB and bHLH plant transcription factors[J].ThePlantJournal,2011,66(1):94-116.
[15] STRACKE R,ISHIHARA H,HUEP G,etal.Differential regulation of closely related R2R3-MYB transcription factors controls flavonol accumulation in different parts of theArabidopsisthalianaseedling[J].ThePlantJournal,2007,50(4):660-677.
[16] GONZALEZ A,ZHAO M,LEAVITT J M,etal.Regulation of the anthocyanin biosynthetic pathway by the TTG1/bHLH/Myb transcriptional complex inArabidopsisseedlings[J].ThePlantJournal,2008,53(5):814-827.
[17] LEPINIEC L,DEBEAUJON I,ROUTABOUL J M,etal.Genetics and biochemistry of seed flavonoids[J].AnnualReviewofPlantBiology,2006,57:405-430.
[18] YANG C S,LANDAU J M,HUANG M T,etal.Inhibition of carcinogenesis by dietary polyphenolic compounds[J].AnnualReviewofNutrition,2001,21(1):381-406.
[19] ROSS J A,KASUM C M.Dietary flavonoids:bioavailability,metabolic effects,and safety[J].AnnualReviewofNutrition,2002,22(1):19-34.
[20] KHLESTKINA E K,PESTSOVA E G,RODER M S,etal.Molecular mapping,phenotypic expression and geographical distribution of genes determining anthocyanin pigmentation of coleoptiles in wheat (TriticumaestivumL.)[J].TheoreticalandAppliedGenetics,2002,104(4):632-637.
[21] NELSON J C,VANDEYNZE A E,AUTRIQUE E,SORRELLS M E,etal.Molecular mapping of wheat-homoeologous group-3[J].Genome,1995,38(3):525-533.
[22] HIMI E,NODA K.Isolation and location of three homoeologous dihydroflavonol-4-reductase (DFR) genes of wheat and their tissue-dependent expression[J].JournalofExperimentalBotany,2004,55(396):365-375.
[23] YANG G H,LI B,GAO J W,etal.Cloning and expression of two chalcone synthase and a flavonoid 3′5′-hydroxylase 3′-end cDNAs from developing seeds of blue-grained wheat involved in anthocyanin biosynthetic pathway[J].ActaBotanicaSinica,2004,46(5):588-594.
[24] HIMI E,NISAR A,NODA K.Colour genes (R and Rc) for grain and coleoptile upregulate flavonoid biosynthesis genes in wheat[J].Genome,2005,48(4):747-754.
[25] YAN Z,WAN Y,LIU K,etal.Identification of a novel HMW glutenin subunit and comparison of its amino acid sequence with those of homologous subunits[J].ChineseScienceBulletin,2002,47(3):220-225.
[26] TAMURA K,DUDLEY J,NEI M,etal.MEGA4:Molecular evolutionary genetics analysis (MEGA) software version 4.0[J].MolecularBiologyandEvolution,2007,24(8):1 596-1 599.
[27] AHMED N,MAEKAWA M,UTSUGI S,etal.Transient expression of anthocyanin in developing wheat coleoptile by maize C1 and B-peru regulatory genes for anthocyanin synthesis[J].BreedingScience,2003,53(1):29-34.
[28] HIMI E,MAEKAWA M,MIURA H,etal.Development of PCR markers forTamyb10 related toR-1,red grain color gene in wheat[J].TheoreticalandAppliedGenetics,2011,122(8):1 561-1 576.
[29] GROTEWOLD E,SAINZ M B,etal.Identification of the residues in the Myb domain of maize C1 that specify the interaction with the bHLH cofactor R[J].ProceedingsoftheNationalAcademyofSciencesoftheUnitedStatesofAmerica,2000,97(25):13 579-13 584.
[30] 刘宝龙.小麦品种高原115紫色籽粒中花青素合成调控机理研究[D].北京:中国科学院大学,2009.
(编辑:宋亚珍)
Cloning and Functional Verification of R2R3-MYBTranscription FactorTaMYB3-4Din Wheat
WANG Yanqian1,2,3,ZHANG Bo1,3,CHEN Wenjie1,3,LIU Dengcai1,3,LIU Baolong1,3,ZHANG Huaigang1,3*
(1 Qinghai Province Key Laboratory of Crop Molecular Breeding,Xining 810001,China;2 University of Chinese Academy of Sciences,Beijing 100049,China;3 Key Laboratory of Adaptation and Evolution of Plateau Biota (AEPB),Chinese Academy of Sciences,Xining 810001,China)
The MYB transcription factor is one of the largest gene families in plant,involving in various physiological and biochemical processes.In this study,one newMYBgeneTaMYB3-4Dwas isolated from the wheat cultivar ‘Gaoyuan 115’.TaMYB3-4Dwas close to the genes regulating anthocyanins biosynthesis in phylogenetic tree.TaMYB3-4Dpossessed only one intron,while the otherMYBgenes regulating anthocyanins biosynthesis had two introns.The deduced amino acid of TaMYB3-4D contained two consecutive MYB domains,the typical characteristic of the R2R3-MYB transcription factor.Transient expression experiments showed thatTaMYB3-4Dinduced the white coleoptile cells of wheat cultivar Chinese Spring (CS) to synthesize anthocyanins with the help ofbHLHtranscription factorZmRin light.Meanwhile,the RT-PCR analysis showed thatTaMYB3-4Dgene only expressed in purple seeds and red coleoptiles of ‘Gaoyuan 115’,but not in root,stems and leaves.All of these results suggested thatTaMYB3-4Dwas a R2R3-MYB transcription factor regulating anthocyanins biosynthesis in wheat.
Triticumaestivum;MYB transcription factor;anthocyanins biosynthesis
1000-4025(2015)04-0646-07
10.7606/j.issn.1000-4025.2015.04.0646
2014-12-27;修改稿收到日期:2015-01-30
中国科学院知识创新工程重要方向项目;国家自然科学基金(31260322);中国科学院西部之光项目
王延谦(1985-),男,在读博士研究生,主要从事小麦遗传育种研究。E-mail:wangyanqian2004@163.com
*通信作者:张怀刚,博士,研究员,博士生导师,主要从事小麦遗传育种研究。E-mail:hgzhang@nwipb.cas.cn
Q785;Q789
A

