磺胺类药物单链抗体的制备及其与磺胺噻唑相互作用研究
李永翰等

摘 要 抗体与对应的小分子待测物之间的相互作用模式决定了免疫分析的特性。本研究以磺胺类药物杂交瘤细胞株4C7为起点,应用基因工程技术制备出单链抗体scFv4C7,采用间接竞争ELISA法对比其与母本单克隆抗体的识别特性,同时采用同源建模构建scFv4C7的三维立体结构,并与磺胺噻唑(STZ)进行分子对接。间接竞争ELISA结果显示scFv4C7保留了亲本单克隆抗体的识别特性,分子对接结果显示STZ深陷入抗体的重链和轻链形成的“口袋”中,STZ分子更靠近重链,且主要与抗原互补决定区CDR H3相互作用。本研究为制备识别谱更广、亲和力更高的磺胺类药物抗体提供了必要的结构信息。
关键词 相互作用; 同源建模; 分子对接; 单链抗体; 磺胺噻唑
1 引 言
磺胺类药物(Sulfonamides, SAs)是目前应用范围最广、用量最大、种类最多的抗菌药物之一,其不合理使用在食品和环境中造成的残留给人类健康带来潜在危害。基于抗原抗体特异性反应的免疫分析具有灵敏度高、操作简便、成本低且适合现场快速检测而越来越受到重视。磺胺类药物多特异性抗体已有很多报道[1~2],这些抗体识别磺胺类药物的种类存在显著差异,难以应用于磺胺类药物的多残留检测。研究磺胺类药物与抗体之间的识别机制,探讨影响抗体识别的关键因素,对磺胺类药物半抗原设计的进行改进从而制备出性能更加优良的抗体具有重要的理论意义和应用价值。
单链抗体(Single chain variable fragment, scFv)是抗体分子中保留抗原结合部位的最小功能片段,以scFv为研究对象,通过同源建模、分子对接和构效关系等方法来研究抗原抗体之间的相互作用,可为了解抗体识别机制提供必要的结构信息 [3~6]。Wang等构建了河豚毒素的scFv,并通过同源建模和分子对接研究了scFv和河豚毒素的相互作用,预测相互作用的两个必需氨基酸为K183和I189,通过定点突变验证了该预测的可靠性[4]。He等利用基因工程技术将抗黄体酮scFv中的关键氨基酸W100定点突变成其它15种氨基酸,发现突变体与野生型scFv的特异性和亲和力存在明显差异,其中,将W100突变成R100后显著提高了抗体对孕酮衍生物(Progesterone3CMO)的亲和力[5],证明可通过基因工程技术改变结合位点的关键氨基酸来改变抗体的识别能力,并可能获得亲和力更高的抗体。
本研究通过制备磺胺类药物单克隆抗体4C7对应的scFv,利用同源建模和分子对接,通过实验研究和理论分析对磺胺噻唑与4C7的相互作用进行了初步研究,为全面了解4C7的识别机制、制备识别谱更广的磺胺类药物抗体奠定了基础。
2 实验部分
2.1 仪器与试剂
2720型PCR扩增仪(美国ABI公司); HQ60Ⅱ型漩涡混合器(北京北方同正生物技术发展有限公司); HDLAPPARATUS超净工作台(北京东联哈尔仪器制造有限公司); MiniPROTEAN4蛋白电泳设备(美国BioRad公司); GHP9160A隔水式恒温培养箱(北京陆希科技有限公司); H2C250恒温振荡培养箱(苏州市培英实验设备有限公司); Thermo Scientific Multiskan MK3酶标仪(美国Thermo Fisher Scientific 公司)。
磺胺类药物杂交瘤细胞株4C7和单克隆抗体Mab4C7由本实验室制备[2]; pJB33载体由本实验室保存; RV308菌种来自于美国标准生物品收藏中心; Qiagen RNeasy Mini kit、QIAshredder(Qiagen公司); SuperScriptTM FirstStrand Synthesis System for RTPCR(Invitrogen公司); 异丙基硫代半乳糖苷(IPTG)、Wizard DNA Cleanup System(Promega公司); 限制性内切酶SfiⅠ(美国NEB公司); T4 DNA连接酶、Ex Taq DNA聚合酶、DL2000 DNA Marker (TaKaRa公司); HisTrap HP组氨酸标记亲和层析柱(GE Healthcare); Amicon Ultra离心超滤管(Millipore公司); 羊抗鼠HRP标记抗体、抗cmyc抗体9E10(Jackson ImmunoResearch公司); 磺胺类药物标准品(SigmaAldrich公司); 96孔酶标板(Costar公司); 竞争ELISA中的缓冲液参照文献[2]介绍; 其它生化试剂均为国产分析纯。
2.2 引物合成
参照文献[7]设计鼠源抗体重链及轻链可变区的扩增引物,连接肽Linker采用Huston等[8]介绍的(Gly4Ser)3形式,并在引物序列上设计酶切位点以及保护碱基等基因附件(表1)。所有引物由上海英骏生物技术有限公司合成。
2.3 实验方法
2.3.1 杂交瘤细胞总RNA提取及cDNA的合成 培养杂交瘤细胞株4C7至对数生长期,细胞计数约107个时收集细胞,用试剂盒提取总RNA,取5 μL用1%琼脂糖凝胶电泳鉴定完整性,以提取的RNA为模版,利用SuperScriptTM FirstStrand Synthesis System for RTPCR合成cDNA。
2.3.2 VH、VL基因的克隆及scFv基因片段的拼接 以cDNA为模板,采用巢式PCR技术,分别以VHF1和VHR1为第一轮PCR引物,VHF2和VHR2为第二轮PCR引物,VHF2和VHR3为第三轮PCR引物,扩增出VH。分别以VLF1和VLR1为第一轮PCR引物,VLF2和VLR2为第二轮PCR引物扩增出VL。PCR反应体系按常规比例配成25 μL(反应程序: 94 ℃, 5 min; 94 ℃, 20 s; 55 ℃, 20 s; 72 ℃, 20 s; 72 ℃, 10 min; 共30次循环)。将PCR产物进行1%琼脂糖凝胶电泳鉴定, 回收目的片段, 获得VH和VL基因。endprint
取等摩尔纯化的VH、VL, 以VLF2和VHR3分别为上、下游引物, 通过重叠延伸PCR完成VLLinkerVH片段的拼接(拼接程序: 95 ℃, 5 min; 95 ℃, 1 min; 55 ℃, 2 min; 72 ℃, 1 min; 72 ℃, 10 min; 共30个循环), 1%琼脂糖凝胶电泳鉴定PCR产物, 切胶回收获得VLLinkerVH基因片段, 即为全长scFv。
2.3.3 scFvpJB33表达载体的构建
将scFv基因片段、pJB33载体分别用SfiⅠ双酶切,回收目的片段。将双酶切后的scFv(60 ng)和表达载体pJB33(100 ng)在T4 DNA Ligase作用下,25 ℃连接3 h。取20 μL的连接体系电击转化RV308感受态细胞,涂含34 mg/L氯霉素的2×YT平板,37 ℃过夜培养,挑取菌落,接种于含氯霉素的2×YT液体培养基中,37 ℃培养3 h后送金唯智生物科技(北京)有限公司测序。选择测序正确的菌落进行保种,备诱导表达用。
2.3.4 单链抗体的可溶性表达及纯化
在非变性条件下提取scFv, 并利用固定化金属亲和层析(IMAC)纯化:将构建好的表达载体接种于含34 mg/L氯霉素的2×YT液体培养基中,37 ℃,250 r/min培养至OD600=0.5~1.0时,加入IPTG至0.25 mmol/L诱导表达(16 ℃,150 r/min,16 h)。采用HisTrap HP组氨酸标记亲和层析柱纯化scFv4C7,用超滤管对获得的抗体溶液除盐和浓缩,SDSPAGE鉴定其大小。
2.3.5 单链抗体交叉反应率的测定
采用优化好的间接竞争ELISA 方法[2],测定scFv4C7对磺胺类药物的识别特性。并计算得到各竞争物的IC50和各竞争物对scFv4C7的交叉反应率。
2.3.6 同源建模和分子对接 将scFv基因序列提交至ExPASy数据库,得到相应的氨基酸序列。利用abYsis数据库在线工具分析scFv的VH和VL序列。采用SwissModel和Autodock 4.0进行同源建模和分子对接,由PyMOL获得三维结构图像,由LigPlus获得结合位点的作用力信息。
3 结果与讨论
3.1 磺胺类药物杂交瘤细胞总RNA提取
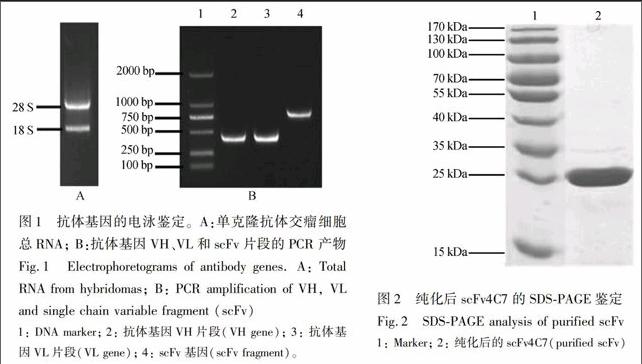
使用RNA提取试剂盒从杂交瘤细胞中提取总RNA,1%琼脂糖凝胶进行RNA电泳检测,如图1A所示,可清晰地观察到两条带:28 S和18 S,说明提取的总RNA完整度良好。
3.2 VH、VL和scFv基因片段的扩增鉴定
将提取的RNA反转录获得cDNA,以cDNA为模版,分别扩增出VH和VL基因,经1%琼脂糖凝胶电泳分析鉴定并回收,如图1B所示。将轻链与重链片段进行重叠延伸PCR,通过Linker连接得到scFv片段,如图1B所示。将测序正确的scFv目的片段和表达载体pJB33,分别用Sfi I双酶切后连接,电击转化RV308感受态细胞,构建完整的表达载体。
3.3 单链抗体的可溶性表达及纯化
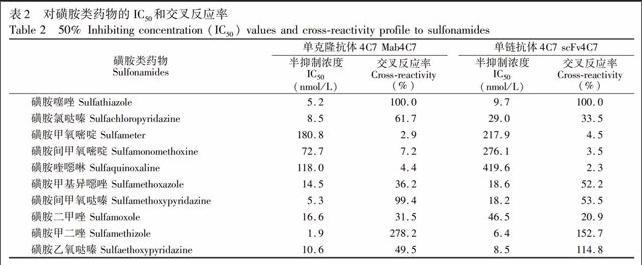
将可溶性表达的单链抗体scFv4C7纯化后用SDSPAGE鉴定,结果如图2所示,显示该其大小约25 kD,与预期结果一致。经浓缩后获得2.5 g/L的scFv4C7溶液。
3.4 单链抗体和单克隆抗体的生物学活性比较
间接竞争ELISA测定结果显示scFv4C7的IC50和交叉反应率与单克隆抗体Mab4C7基本一致,结果如表2所示,说明本研究制备的scFv具有与母本单克隆抗体一致的生物学活性。证明本研究克隆的Mab4C7可变区基因序列是正确的,可将其作为同源建模和分子对接的研究对象。
3.5.2 scFv4C7的同源建模 采用SwissModel构建scFv4C7的空间三维模型[9~11],如图3B所示,包含VL(紫色),Linker(黄色)和VH(绿色)三部分。利用PROCHECK获得Ramachandran plot,对模型中主链氨基酸残基的二面角φ和ψ进行合理性分析,初步评价模型的的可信度,如图3C所示,分析结果显示此同源模型中97%以上的氨基酸处于合理区域,证明此模型的可信度较高,具有较好的预测能力。
3.5.3 scFv4C7和STZ的分子对接 为了研究小分子与抗体的作用机制,本研究选择scFv识别较好的STZ,利用Autodock 4.0对scFv4C7和STZ进行分子模拟对接,如图3D所示,重链和轻链形成一个较深的“口袋”,该“口袋”为抗原结合区,几乎将小分子完全包围。二者的结合位点如图3E所示,轻链上的LAsn39,LTrp94,LArg51和重链上的HHis168, HTrp180, HAla229, HTyr231, HGly232和HGly233共同形成一个疏水型“口袋”,HARG230和HTRP235和STZ形成两个分子间氢键。在这11个氨基酸中8个位于重链,据此推断小分子与抗体结合时更靠近抗体的重链区域。在重链的抗原互补决定区(CDRs)中,CDR H3贡献的关键氨基酸(Tyr231,Gly232,Gly233)最多,说明CDR H3可能是抗原结合区中最关键的部分。
通过对此分子对接模型的分析,发现了抗体中与磺胺噻唑结合的关键部位(CDR H3)、主要作用力类型(疏水作用力和氢键)和关键氨基酸(HARG230和HTRP235)。本研究为制备识别谱更广、亲和力更高的磺胺类药物抗体提供了必要的结构信息。
References
1 Zhou Q, Peng D P, Wang Y L, Pan Y H, Wan D, Zhang X Y, Yuan Z H. Food Chem., 2014, 154: 52-62endprint
2 Wang Z H, Beier R C, Sheng Y J, Zhang S X, Jiang W X, Wang Z P, Shen J Z. Anal. Bioanal. Chem., 2013, 405(12): 4027-4037
3 QI YongHua, DONG YongJun, NING HongMei, ZHAO Kun. J. Northeast Agri. Univ., 2011, 42(9): 7-11
齐永华, 董永军, 宁红梅, 赵 坤. 东北农业大学学报, 2011, 42(9): 7-11
4 Wang R Z, Huang A L, Liu L C, Xiang S S, Li X F, Ling S M, Wang L, Lu T, Wang S H. Toxicon, 2014, 83: 22-34
5 He M, Hamon M, Liu H, Corper A L, Taussig M J. Protein Sci., 2006, 15(9): 2141-2148
6 WANG ZhanHui, DING ShuangYang, ZHANG SuXia, SHEN JianZhong. Acta Chimica Sinica, 2008, 66(23): 2613-2619
王战辉, 丁双阳, 张素霞, 沈建忠. 化学学报, 2008, 66(23): 2613-2619
7 Wen K, Nlke G, Schillberg S, Wang Z H, Zhang S X, Wu C M, Jiang H Y, Meng H, Shen J Z. Anal. Bioanal. Chem., 2012, 403(9): 2771-2783
8 Huston J S, Levinson D, MudgettHunter M, Tai M S, Novotny J, Margolies M N, RidgeR J, Bruccoleri R E, Haber E, Crea R. P. Natl. Acad. Sci. USA, 1988, 85(16): 5879-5883
9 Arnold K, Bordoli L, Kopp J, Schwede T. Bioinformatics, 2006, 22: 195-201
10 Schwede T, Kopp J, Guex N, Peitsch M C. Nucleic Acids Res., 2003, 31(13): 3381-3385
11 Guex N, Peitsch M C. Electrophoresis, 1997, 18(15): 2714-2723
12 Armstrong A A, Hildreth J E K, Amzel L M. J. Mol. Biol., 2013, 425(11): 2027-2038
Abstract The interaction between the antibody and the corresponding target molecule determines the characteristics of immunoassay. In this study, a single chain variable fragment antibody (scFv4C7) derived from the hybridoma strain 4C7 were prepared via genetic engineering technique. The recognition properties of scFv4C7 was determined and compared to those of the parent monoclonal antibody by indirect competitive enzymelinked immunosorbent assay(icELISA). Three dimensional structure of the scFv4C7 was presented by SwissModel, and sulfathiazole (STZ) was docked to the scFv4C7 model to obtain the structure of the binding complex. The results from the icELISA showed that the binding properties of scFv4C7 were comparable with the parent monoclonal antibody and STZ was almost completely buried in a deep binding pocket formed by the heavy chain and light chain of the antibody. The interaction between STZ and scFv4C7 was more closely related to the heavy chain and the complementaritydetermining region (CDR) H3 loop played more important role than other CDR loops. The study preliminary provided the necessary structural information for the preparation of antibody with broader specificity and higher affinity.
Keywords Proteinligand interaction; Homology modeling; Molecular docking; Single chain variable fragment antibody; Sulfathiazole
(Received 23 July 2014; accepted 14 October 2014)
This work was supported by the National Natural Science Foundation of China (No.31372475)endprint

